2. 河北省农业厅植物营养与海洋功能肥料重点实验室, 秦皇岛 066004;
3. 领先生物农业股份有限公司, 秦皇岛 066004;
4. 秦皇岛领先康地农业技术有限公司, 秦皇岛 066400
2. Key Laboratory of Plant Nutrition and Marine Functional Fertilizer of Hebei Provincial Agricultural Department, Qinhuangdao 066004;
3. Leading Bio-agricultural Co. Ltd, Qinhuangdao 066004;
4. Qinhuangdao Leading Comdee Agricultural Technology Co. Ltd, Qinhuangdao 066400
枯草芽孢杆菌(Bacillus subtilis)是芽孢杆菌属中的一种好氧革兰氏阳性菌[1]。枯草芽孢杆菌是一种广泛分布植物根际的有益微生物,能够产生很多抗菌谱广的具有抗菌活性的物质防治植物病害,具有良好的病害防治能力[2]。又因为枯草芽孢杆菌可以产生繁殖能力和抗逆性极强的芽孢休眠体,所以越来越多的枯草芽孢杆菌菌株被开发为微生物菌剂广泛应用于农业生产中。
枯草芽孢杆菌拥有着广泛的抑菌谱,具有较强的抑制病原菌生长的能力,其主要原因在于它可以分泌多种拮抗类物质,其中有通过非核糖体合成的低分子抗菌肽以及通过核糖体合成的大分子蛋白类细菌素[3],通过非核糖体途径合成的细菌素包括低分子量的伊枯草菌素(Iturin)、抗菌肽表面活性素(Surfactin)、芬枯草菌素(Fengycin)、溶杆菌素(Bacilysin)、Bacillaene、Rhizocticin和Mycobacillin等; 通过核糖体途径合成的细菌素包括Subtilin、Mersacidin、Ericin、Sublancin和Subtilosin A等[4-5]。
随着近现代测序技术的进步,尤其二代测序(Next generation sequencing,NGS)技术的出现以及测序成本的下降,为研究枯草芽孢杆菌提供了新的方法。利用全基因组序列信息对枯草芽孢杆菌近缘种群进行分类鉴定和抗性潜力快速挖掘具有快捷和准确性高等优点。枯草芽孢杆菌类群间的亲缘关系较近,16S rRNA序列相似度太高从而导致区分度较低[6],为了解决16S rRNA区分度低的问题,有研究在细菌的基因组中挑选了如gyrA[6]、rpoB[7]及gyrB[8]等在系统进化过程中保守但又具有相对较快的变异速率的基因作为补充。相比于单一基因分析,基于基因组水平的平均核苷酸一致性(Average nucleotide identity,ANI)[9-11]分析和DNA同源性(DNA-DNA hybridization,DDH)[12]分析对于枯草近缘种群具有更高的区分度。在挖掘次级抗菌代谢产物方面,antiSMASH在线工具自发布以来,在挖掘次级代谢产物基因簇领域得到了广泛的应用,成为了应用最广泛的挖掘次级代谢产物合成基因簇的应用工具[13]。
本试验对从土壤中分离到的一株具有良好生防效果的芽孢杆菌BS-6进行研究,通过拮抗试验发现BS-6对病原真菌的生长具有良好的抑制效果,通过全基因组测序得到BS-6的全基因组信息,结合16S rRNA序列、gyrA基因序列、平均核苷酸一致性(ANI)及DNA同源性(DDH)综合分析可以对芽孢杆菌BS-6的分类地位进行准确鉴定,利用antiSMASH在线工具对BS-6的抑菌相关功能基因进行分析探索其拮抗特性,为芽孢杆菌BS-6的农业生产应用提供理论依据。
1 材料与方法 1.1 材料供试菌株:芽孢杆菌菌株BS-6,该菌株为土壤中筛选到具有较好生防效果的根际微生物,由领先生物农业股份有限公司分离并保存。
植物病原真菌:马铃薯枯萎病菌(Fusarium oxysporum)、玉米纹枯病菌(Rhizoctonia solani)和苹果轮纹病菌(Botryospuaeria berengeriana de Not.),由领先生物农业股份有限公司分离并保存。
主要培养基:LB培养基,蛋白胨10.0 g/L、酵母粉5.0 g/L、氯化钠10.0 g/L,pH=7;PDA培养基,马铃薯200.0 g/L、葡萄糖20.0 g/L。
1.2 方法 1.2.1 植物病原真菌拮抗试验采用平板对峙法,首先在平皿中间分别放置马铃薯枯萎病菌(Fusarium oxysporum)、玉米纹枯病菌(Rhizoctonia solani)和苹果轮纹病菌(Botryospuaeria berengeriana de Not.)等3种植物病原菌的菌丝块。然后在菌丝块四周滴加10 μL BS-6的菌悬液,每种病原菌重复3次拮抗试验,设不接种BS-6的空白对照,于培养箱中20℃培养5 d。根据下列公式计算BS-6对植物病原菌的生长抑制率:
抑制率(%)=[(对照菌落直径-处理菌落直径)/(对照菌落直径-菌饼直径)]×100%
1.2.2 芽孢杆菌BS-6的全基因组测序将BS-6接于LB液体培养基中,37℃、180 r/min培养至对数期,在4℃下4 500 r/min离心10 min,舍去上清液收集菌体,利用液氮冷冻保存送至北京诺禾致源科技股份有限公司(Beijing Novogene Bioinformatics Technology Co.,Ltd)进行全基因组测序,获得其全基因组序列,将BS-6的全基因组信息上传至NCBI(https://www.ncbi.nlm.nih.gov),获得登录号为SSXZ00000000。
1.2.3 16S rRNA及gyrA基因序列分析根据全基因组注释信息,选择16S rRNA和gyrA基因序列分别提交至ezbiocloud.net数据库及NCBI数据库进行比对分析,利用MEGA 7.0.26软件Neighbor-Joining法构建系统发育树。
1.2.4 ANI和DDH分析使用Jspecies(http://jspecies.ribohost.com/jspeciesws//)中的Mummer[14]对研究中的菌株全基因组进行平均核苷酸一致性(Average nucleotide identity,ANI)值的计算。使用Genome-to-Genome Distance Calculation(http://ggdc.dsmz.de/distcalc2.php)[15]对研究中的菌株全基因组进行DDH值的计算
1.2.5 次级代谢产物合成基因簇分析本研究利用antiSMASH 3.0[16]bacterial version(https://antismash.secondarymetabolites.org/)对BS-6菌株中次生代谢物合成基因组簇进行预测。
2 结果 2.1 植物病原真菌拮抗试验拮抗试验结果如图 1所示,BS-6对马铃薯枯萎病菌(Fusarium oxysporum)、玉米纹枯病菌(Rhizo-ctonia solani)和苹果轮纹病菌(Botryospuaeria beren-geriana de Not.)的生长均具有良好抑制效果,抑菌率分别为80.00%、68.79%和76.19%。其中BS-6对马铃薯枯萎病的病原微生物尖孢镰刀菌(Fusarium oxysporum)具有显著的抑菌效果。
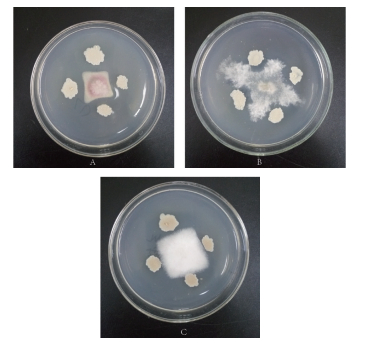
|
| 图 1 BS-6对马铃薯枯萎病菌(A)、苹果轮纹病菌(B)和玉米纹枯病菌(C)的抑制效果 |
经全基因组测序,获得芽孢杆菌BS-6基因组草图,上传至NCBI获得登录号为SSXZ00000000,基因组大小为3.99 Mb,GC含量为43.84%。将全基因组中注释到的16S rRNA序列提交至ezbicloud和NCBI的数据库中进行比对,选取部分同源性高于99%的菌株序列利用MEGA7软件的邻位连接法构建系统发育树(相似度重复计算1 000次)。芽孢杆菌BS-6基于16S rRNA序列的系统发育树如图 2所示。根据比对结果与菌株BS-6同源性高于99%的菌株均为芽孢杆菌属,系统发育树结果显示BS-6与模式菌株Bacillus halotolerans ATCC 25096聚为一类,但bootstrap支持值较低,可以确定BS-6为芽孢杆菌属,无法准确鉴定BS-6到种。
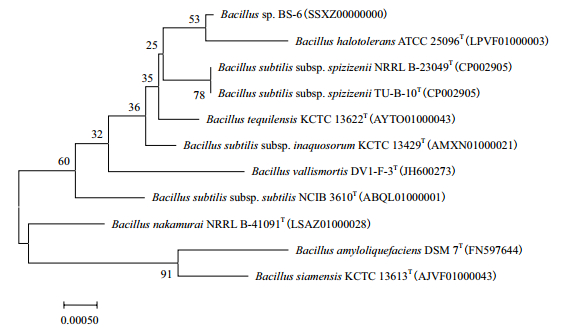
|
| 节间数字代表bootstrap支持值,上标T代表该菌株为模式菌株 图 2 基于16S rRNA序列比对结果构建的菌株BS-6的系统发育树 |
将测序得到的gyrA基因序列提交至NCBI进行BLAST分析,选取2株同源性较高的序列,同时根据16S rRNA比对结果选取其中具有gyrA基因序列信息的菌株构建系统发育树(图 3)。根据系统发育树,BS-6与Bacillus subtilis subsp. spizizenii W23聚为一类。
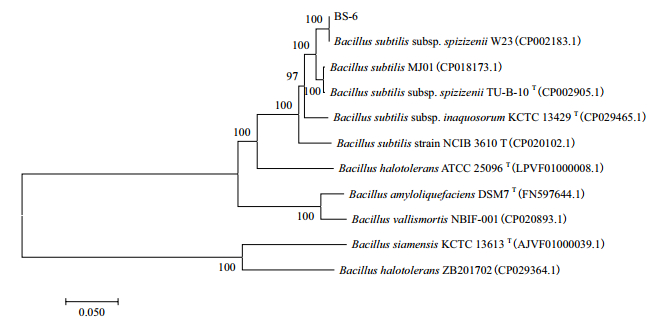
|
| 节间数字代表bootstrap支持值 图 3 基于gyrA序列比对结果构建的菌株BS-6的系统发育树 |
选择在16S rRNA序列和gyrA基因序列比对中的具有全基因组序列数据的模式菌株与BS-6进行ANI及DDH分析,该9株芽孢菌的ANI值及DDH值结果如表 1所示。
其中BS-6与模式菌株Bacillus subtilis subsp. spizizenii TU-B-10的ANI值为96.72%,高于网站给出的95%的种间鉴定标准。BS-6与模式菌株Bacillus subtilis subsp. spizizenii TU-B-10的DDH值为71.70%高于网站给出的70%的种间鉴定标准。
2.2.4 Bacillus subtilis BS-6次级代谢产物合成基因簇分析将Bacillus subtilis BS-6的全基因组序列上传至在线软件antiSMASH中进行次级代谢产物合成基因簇分析,共发现有11个与次级代谢产物合成相关的基因簇。同时对2.2.3中参与分类的芽孢菌株进行次级代谢产物合成基因簇分析,对基因组中已知功能的基因簇进行统计,统计结果如表 2所示。
Bacillus subtilis BS-6与亲缘关系较近的Bacillus subtilis subsp. spizizenii TU-B-10具有类似的次级代谢产物合成基因簇组成,Bacillus subtilis BS-6中具有7个次级代谢产物合成基因簇,且均具有抗菌作用,其中Mycosubtilin和Rhizocticin为Bacillus subtilis BS-6及其亲缘种特有的次级抗菌代谢产物合成基因簇。
3 讨论植物病害一直以来都是影响粮食产量的重要因素,传统的化学农药防治对病害控制具有速效性,但随着化学农药的大量使用,病原微生物的耐药性和农药残留产生的“副作用”也日益严重。近年来绿色高效的生物防治作为防治植物病害的重要措施逐渐受到人类的重视。其中芽孢杆菌特别是枯草芽孢杆菌作为生物防治的代表菌株具有良好的生防效果,被广泛的应用在农业生产中。
本团队从植物连作根际土壤中分离到了一株芽孢杆菌BS-6,前期溶血试验表明其溶血活性微弱,具有较高的安全性,经过应用试验和拮抗试验发现其具有良好的应用效果,本试验通过全基因组测序得到了BS-6的基因组序列,结合相关软件分析确定了BS-6的分类地位及其抗病防病能力的分析。
16S rRNA基因是细菌系统发育和种属鉴定的常用分类工具[17],但是枯草芽孢杆菌类群间的亲缘关系较近,导致16S rRNA序列区分度降低无法准确区分近缘种群[18],在本研究中利用BS-6的16S rRNA基因序列无法将其与其他芽孢杆菌进行准确区分,为了准确鉴定BS-6的分类地位,首先挑选了gyrA基因作为分类工具,根据系统发育分析发现BS-6与枯草芽孢杆菌聚为一类。又对BS-6及其亲缘种的平均核苷酸一致性(ANI)及DNA同源性(DDH)进行了分析,可以准确将BS-6鉴定为枯草芽孢杆菌。基于基因组水平的ANI和DDH分析验证了gyrA基因分析的结果,这表明gyrA基因作为枯草芽孢杆菌近缘种的分类工具具有良好的区分度。
antiSMASH是一种能够较完整的预测细菌和真菌次生代谢产物基因簇的在线生物信息学工具,特别是与生物抗生素合成有关的基因簇。本研究将与BS-6亲缘关系较近的几株芽孢杆菌的基因组提交至antiSMASH,对这几株亲缘关系较近的芽孢菌中的次生代谢产物基因簇进行分析,对比其在抑菌抗病方面的潜力大小。BS-6具有最多的7种生物抗生素合成有关的基因簇。枯草芽孢杆菌产生抗菌肽的途径主要包括核糖体途径和非核糖体2条途径。BS-6中的抗菌肽核糖体途径合成的抗菌肽主要有:常见的羊毛硫细菌素Subtilin; 稀有的羊毛硫细菌素类有Subtilosin A等; 非核糖体途径合成的抗菌物质主要有Surfactin、Mycosubtillin、Rhizocticin和Bacillibactin等[4-5]。
Subtilin是一类含有带正电荷的A类羊毛硫细菌素,对于革兰氏阳性菌具有很强的抑制活性[19-21]; Subtilosin A是一种抑菌谱非常广的抑菌肽,对单核细胞增生李斯特菌、肺炎克氏杆菌、金黄色葡萄球菌和大肠杆菌均有抑制作用[22-23]。
Surfactin是许多芽孢杆菌中都存在的广谱抗菌物质,同时也是一种很强的表面活性剂[24-25],具有双重身份的Surfactin具有乳化、发泡[26],抗病毒[27]、抗支原体、抗致病真菌、抗李斯特菌和杀幼虫的作用[28-30]。Bacillibactin是一种枯草芽孢杆菌分泌出来的对铁离子具有高亲和性的螯合剂,可以竞争性结合环境中对于病原菌生长和发挥活性必须的可溶性铁离子[31]。Bacillaene是由芽孢杆菌分泌的一种多聚烯类抗生素抑菌物质,主要抑制原核生物蛋白质的合成[32-33]。
相比于其它近缘种,抗菌物质Mycosubtillin和Rhizocticin的合成基因簇是BS-6基因组中特有的,据文献报道该两种物质均具有较强的抑制真菌活性[34-36]。其中Mycosubtillin对尖孢镰刀菌(Fusarium oxysporum)、白色念珠菌(Candida albicans)、酿酒酵母(Saccharomyces cerevisiae)和热带念珠菌(Candida tropicalis)具有抑菌活性[34, 37-38]。
拮抗试验结果发现,枯草芽孢杆菌BS-6对于真菌具有较强的抑制作用,尤其对尖孢镰刀菌(Fusarium oxysporum)的抑制效果最为显著,说明BS-6在防治由尖孢镰刀菌引发的植物病害时可能具有良好的效果,具有较大的应用潜力。
除以上7种芽孢杆菌属重要或特有的次生代谢产物基因簇外,BS-6中还存在4种功能未知的基因簇,其中CDPS(tRNA-dependent cyclodipeptide synthases)类1种,T3PKS(Type Ⅲ PKS)类1种,萜类(Terpene)2种,这表明BS-6中可能存在具有合成新的抑菌物质的基因簇,BS-6具有较大的农业应用潜力。
4 结论本研究通过全基因组测序得到了芽孢杆菌BS-6的基因组序列,通过对BS-6的16S rRNA基因、gyrA基因、平均核苷酸一致性及DNA同源性分析可以准确将BS-6鉴定为枯草芽孢杆菌。通过antiSMASH在线工具对BS-6中次生代谢产物基因簇进行分析,共发现11个相关基因簇,其中7种为芽孢杆菌属重要或特有的次生代谢产物基因簇,具有较强的抑制病原微生物生长的功能。本研究通过基因组分析发现菌株BS-6具有较强的应用潜力,为其在生物防治及大田作物抗病和增产方面的应用方面提供了理论依据。
| [1] |
Meinken C, Blencke HM, Ludwig H, et al. Expression of the glycolytic gapA operon in Bacillus subtilis:differential syntheses of proteins encoded by the operon[J]. Microbiology, 2003, 149(3): 751-761. |
| [2] |
Mukherjee S, Das P, Sen R. Towards commercial production of microbial surfactants[J]. Trends in Biotechnology, 2006, 24(11): 509-515. DOI:10.1016/j.tibtech.2006.09.005 |
| [3] |
赵朋超, 王建华, 权春善, 等. 枯草芽孢杆菌抗菌肽生物合成的研究进展[J]. 中国生物工程杂志, 2010, 30(10): 108-113. |
| [4] |
Leclère V, Béchet M, Adam A, et al. Mycosubtilin overproduction by Bacillus subtilis BBG100 enhances the organism's antagonistic and biocontrol activities[J]. Appl Environ Microbiol., 2005, 71(8): 4577-4584. DOI:10.1128/AEM.71.8.4577-4584.2005 |
| [5] |
Stein T. Bacillus subtilis antibiotics:structures, syntheses and specific functions[J]. Molecular Microbiology, 2005, 56(4): 845-857. DOI:10.1111/j.1365-2958.2005.04587.x |
| [6] |
Nguyen ATV, Nguyen DV, Tran MT, et al. Isolation and characterization of Bacillus subtilis CH16 strain from chicken gastrointestinal tracts for use as a feed supplement to promote weight gain in broilers[J]. Letters in Applied Microbiology, 2015, 60(6): 580-588. DOI:10.1111/lam.12411 |
| [7] |
Kim B, Lee S, Lyu M, et al. Identification of mycobacterial species by comparative sequence analysis of the RNA polymerase gene(rpoB)[J]. Journal of Clinical Microbiology, 1999, 37(6): 1714-1720. |
| [8] |
Wang LT, Lee FL, Tai CJ, et al. Comparison of gyrB gene sequences, 16S rRNA gene sequences and DNA-DNA hybridization in the Bacillus subtilis group[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(8): 1846-1850. DOI:10.1099/ijs.0.64685-0 |
| [9] |
Rosselló-Mora R. Updating prokaryotic taxonomy[J]. J Bacteriol, 2005, 187(18): 6255-6257. DOI:10.1128/JB.187.18.6255-6257.2005 |
| [10] |
Richter M, Rosselló-Móra R. Shifting the genomic gold standard for the prokaryotic species definition[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(45): 19126-19131. DOI:10.1073/pnas.0906412106 |
| [11] |
Konstantinidis KT, Tiedje J M. Genomic insights that advance the species definition for prokaryotes[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(7): 2567-2572. DOI:10.1073/pnas.0409727102 |
| [12] |
McCarthy BJ, Bolton ET. An approach to the measurement of genetic relatedness among organisms[J]. Proceedings of the National Academy of Sciences of the United States of America, 1963, 50(1): 156. DOI:10.1073/pnas.50.1.156 |
| [13] |
Adamek M, Spohn M, Stegmann E, et al. Antibiotics[M]. New York: Humana Press, 2017.
|
| [14] |
Richter M, Rosselló-Móra R, Oliver Glöckner F, et al. JSpeciesWS:a web server for prokaryotic species circumscription based on pairwise genome comparison[J]. Bioinformatics, 2016, 32(6): 929-931. DOI:10.1093/bioinformatics/btv681 |
| [15] |
Meier-Kolthoff JP, Auch AF, Klenk HP, et al. Genome sequence-based species delimitation with confidence intervals and improved distance functions[J]. BMC Bioinformatics, 2013, 14(1): 60-74. |
| [16] |
Weber T, Blin K, Duddela S, et al. antiSMASH 3. 0-a comprehen-sive resource for the genome mining of biosynthetic gene clusters[J]. Nucleic Acids Research, 2015, 43(W1): W237-W243. DOI:10.1093/nar/gkv437 |
| [17] |
冯兴, 潘康成, 张顺泉. 一株益生芽孢杆菌Pab02的16S rDNA测序鉴定[J]. 中国饲料, 2008(18): 4-7. DOI:10.3969/j.issn.1004-3314.2008.18.003 |
| [18] |
Fukushima M, Kakinuma K, Kawaguchi R. Phylogenetic analysis of Salmonella, Shigella, and Escherichia coli strains on the basis of the gyrB gene sequence[J]. Journal of Clinical Microbiology, 2002, 40(8): 2779-2785. DOI:10.1128/JCM.40.8.2779-2785.2002 |
| [19] |
Nagao JI. Properties and applications of lantibiotics, a class of bacteriocins produced by Gram-positive bacteria[J]. Journal of Oral Biosciences, 2009, 51(3): 158-164. DOI:10.1016/S1349-0079(09)80024-8 |
| [20] |
Spieβ T, Korn SM, Kötter P, et al. Autoinduction specificities of the lantibiotics subtilin and nisin[J]. Appl Environ Microbiol., 2015, 81(22): 7914-7923. DOI:10.1128/AEM.02392-15 |
| [21] |
Fuchs SW, Jaskolla TW, Bochmann S, et al. Entianin, a novel subtilin-like lantibiotic from Bacillus subtilis subsp. spizizenii DSM 15029T with high antimicrobial activity[J]. Appl Environ Microbiol, 2011, 77(5): 1698-1707. DOI:10.1128/AEM.01962-10 |
| [22] |
Babasaki K, Takao T, Shimonishi Y, et al. Subtilosin A, a new antibiotic peptide produced by Bacillus subtilis 168:isolation, structural analysis, and biogenesis[J]. The Journal of Biochemistry, 1985, 98(3): 585-603. DOI:10.1093/oxfordjournals.jbchem.a135315 |
| [23] |
Liu Q, Gao G, Xu H, et al. Identification of the bacteriocin subtilosin A and loss of purL results in its high-level production in Bacillus amyloliquefaciens[J]. Research in Microbiology, 2012, 163(6-7): 470-478. DOI:10.1016/j.resmic.2012.05.009 |
| [24] |
Jasim B, Sreelakshmi KS, Mathew J, et al. Surfactin, iturin, and fengycin biosynthesis by endophytic Bacillus sp. from Bacopa monnieri[J]. Microbial Ecology, 2016, 72(1): 106-119. DOI:10.1007/s00248-016-0753-5 |
| [25] |
Singh P, Cameotra SS. Potential applications of microbial surfactants in biomedical sciences[J]. Trends in Biotechnology, 2004, 22(3): 142-146. DOI:10.1016/j.tibtech.2004.01.010 |
| [26] |
Jain RM, Mody K, Joshi N, et al. Effect of unconventional carbon sources on biosurfactant production and its application in bioremediation[J]. International Journal of Biological Macromolecules, 2013, 62: 52-58. DOI:10.1016/j.ijbiomac.2013.08.030 |
| [27] |
Al-Ajlani MM, Sheikh MA, Ahmad Z, et al. Production of surfactin from Bacillus subtilis MZ-7 grown on pharmamedia commercial medium[J]. Microbial Cell Factories, 2007, 6(1): 17-25. DOI:10.1186/1475-2859-6-17 |
| [28] |
Chen WC, Juang RS, Wei YH. Applications of a lipopeptide biosurfactant, surfactin, produced by microorganisms[J]. Biochemical Engineering Journal, 2015, 103: 158-169. DOI:10.1016/j.bej.2015.07.009 |
| [29] |
Banat IM, De Rienzo MAD, Quinn GA. Microbial biofilms:biosurfactants as antibiofilm agents[J]. Applied Microbiology and Biotechnology, 2014, 98(24): 9915-9929. DOI:10.1007/s00253-014-6169-6 |
| [30] |
Mnif I, Hammami I, Triki MA, et al. Antifungal efficiency of a lipopeptide biosurfactant derived from Bacillus subtilis SPB1 versus the phytopathogenic fungus, Fusarium solani[J]. Environmental Science and Pollution Research, 2015, 22(22): 18137-18147. DOI:10.1007/s11356-015-5005-6 |
| [31] |
Hotta K, Kim CY, Fox DT, et al. Siderophore-mediated iron acquisition in Bacillus anthracis and related strains[J]. Microbiology, 2010, 156(7): 1918-1925. DOI:10.1099/mic.0.039404-0 |
| [32] |
Müller S, Strack SN, Hoefler BC, et al. Bacillaene and sporulation protect Bacillus subtilis from predation by Myxococcus xanthus[J]. Appl Environ Microbiol, 2014, 80(18): 5603-5610. DOI:10.1128/AEM.01621-14 |
| [33] |
Butcher RA, Schroeder FC, Fischbach MA, et al. The identification of bacillaene, the product of the PksX megacomplex in Bacillus subtilis[J]. Proceedings of the National Academy of Sciences, 2007, 104(5): 1506-1509. DOI:10.1073/pnas.0610503104 |
| [34] |
Besson F, Michel G. Action of mycosubtilin, an antifungal antibiotic of Bacillus subtilis, on the cell membrane of Saccharomyces cerevisiae[J]. Microbios, 1989, 59(239): 113-121. |
| [35] |
Thimon L, Peypoux F, Dana Maget R, et al. Interactions of bioactive lipopeptides, iturin A and surfactin from Bacillus subtilis[J]. Biotechnology and Applied Biochemistry, 1992, 16(2): 144-151. |
| [36] |
Borisova SA, Circello BT, Zhang JK, et al. Biosynthesis of rhizocticins, antifungal phosphonate oligopeptides produced by Bacillus subtilis ATCC6633[J]. Chemistry & Biology, 2010, 17(1): 28-37. |
| [37] |
Hourdou ML, Besson F. Surfactine et antibiotiques ituriniques:structure, propriétés et biosynthèse des lipopeptides de Bacillus subtilis[J]. Regard sur la biochimie, 1994(1): 35-42. |
| [38] |
Besson F, Peypoux F, Michel G, et al. Antifungal activity upon Saccharomyces cerevisiae of iturin A, mycosubtilin, bacillomycin L and of their derivatives; inhibition of this antifungal activity by lipid antagonists[J]. The Journal of Antibiotics, 1979, 32(8): 828-833. DOI:10.7164/antibiotics.32.828 |






