2. 中国农业科学院农业基因组研究所, 深圳 518120
2. Agricultural Genomics Institute at Shenzhen, Chinese Academy of Agricultural Sciences, Shenzhen 518120
小地老虎(Agrotis ipsilon)、棉铃虫(Helicoverpa armigera)、帝王蝶、黏虫(Mythimna separata)、稻飞虱(Nilaparvata lugens)、玉米螟(Ostrinia furnacalis、Ostrinia nubilalis)及东亚飞蝗(Locusta migratoria manilensis(Meyen))等都属于迁飞性昆虫[1-6]。迁飞是动物种群对栖息地资源发生季节性或偶然性变化时进化出的一种适应性对策, 昆虫的迁飞行为严重影响人类的生产和生活, 如危害农作物, 造成农作物大量减产、传播人和牲畜的疾病等。影响昆虫迁飞的因素众多, 包括生存环境、激素调控、遗传因素等, 而翅发育是昆虫迁飞的一个重要影响因素。昆虫翅膀的进化对昆虫觅食、求偶、避敌、抵御外界不良环境等起到重要作用[7-8]。随着果蝇等模式昆虫的深入研究, 一系列与翅发育有关的信号通路逐渐被揭示出来, 如Wnt、Hedgehog、Decapentaplegic(Dpp)、Hippo和c-Jun N-terminal Kinase(JNK)等信号通路, 其中Wnt通路是昆虫翅发育的关键信号通路。
Wnt信号通路包含3种Wnt信号传导通路:经典Wnt通路, 非经典Wnt /钙通路以及非经典planar cell polarity通路[9-11], 这3种途径都是通过Wnt蛋白配体与Frizzled(Fz)家族受体结合而激活的。经典Wnt通路引起基因转录的调节, 并且被认为部分地被SPATS1基因负调节[11]。非经典planar cell polarity通路调控细胞骨架, 骨架决定细胞的形状。非经典Wnt /钙通路, 调控细胞内的钙。Decapentaplegic(Dpp)信号通路中, Dpp是参与果蝇发育的关键形态发生素, 也是第一个经过验证的分泌形态发生素[12-13]。早期果蝇胚胎和15个成虫器的正确形成和发育需要Dpp调控, 这些组织将生长发育为成年果蝇中的四肢和其他器官和结构。DPP在调节组织的生长和器官组织大小方面起着重要的作用[12, 14]。Hedgehog(Hh)信号通路在控制细胞增殖、组织模式形成、干细胞维持和发育方面发挥着多种作用[15-18]。Hedgehog信号通路是一种向胚胎细胞传递信息的信号通路, 这些信息是胚胎细胞分化所必需的。胚胎的不同部位具有不同浓度的Hedgehog基因信号蛋白。该通路在成虫个体中也有作用。Hippo信号通路也被称为Salvador或者Warts通路, 通过调控细胞增殖和凋亡来控制动物的器官大小。该通路的名字来源于它的一个关键信号元件——蛋白激酶Hippo(Hpo)[19-20]。该基因的突变导致组织过度生长, 或类似“河马”表型。
小地老虎(Agrotis ipsilon)又名切根虫、土蚕等, 属于一种世界性的鳞翅目夜蛾科农业害虫[5, 21], 也是典型的迁飞性农业害虫。由于翅是在成虫阶段发育形成, 且属于昆虫胸部的组成部分, 本试验对小地老虎幼虫, 成虫头部、胸部、腹部和进行转录组测序, 探究可能决定小地老虎翅发育的关键通路及相关基因[22-23]。
1 材料与方法 1.1 材料小地老虎幼虫主要来源于中国农业科学院农业基因组研究所农业生态基因组学中心实验室, 小地老虎成虫主要来源于山东长岛野外采集。
1.2 方法 1.2.1 小地老虎总RNA的提取、文库构建和测序小地老虎幼虫6头作为一个重复, 共取18头做3组幼虫整虫转录组Ya、Yb、Yc的组织材料, 为避免肠道微生物污染, 幼虫整虫解剖去除围食膜, 再用液氮研磨。小地老虎成虫每6头作为一个重复, 分别取头、胸、腹, 做头(Ta、Tb、Tc)、胸(Xa、Xb、Xc)、腹(Fa、Fb、Fc)转录组材料。
取烘干研钵, 加入少量乙醇, 灼烧防止研钵内残留其他物质。待研钵烘干后, 加入液氮, 将组织样品加入研钵中研磨至粉状, 取适量放入装有1 mL TRIzol的RNAase free 1.5 mL离心管中。通常情况下, 1 mL TRIzol Reagent能够裂解50-100 mg组织样品。
向样品中加入0.2 mL氯仿(每1 mL TRIzol加0.2 mL氯仿)后, 用手轻轻摇动15 s左右, 使其混合并于常温孵育2-3 min, 然后将样品于12 000 r/min、4℃条件下进行离心15 min。将离心管倾斜为45°并用移液器将水相吸出, 然后移入新的离心管, 在此过程中应尽量避免吸入混合相或者有机层。取0.5 mL异丙醇(每1 mL TRIzol对应0.5 mL异丙醇)加入之前移出的水相样品中, 常温孵育10 min后在4℃、12 000 r/min条件下, 离心10 min, 倒掉上清液并留下RNA沉淀。加入1 mL 75%酒精(每1 mL的TRIzol至少加1 mL的75%乙醇), 轻微震荡混匀后在4℃、7 500 r/min条件下离心5 min, 倒掉上清液, 只留底部沉淀。风干RNA沉淀5-10 min, 禁止真空离心RNA沉淀。加入0.02-0.05 mL无核酸酶水, 使用移液器反复吸打, 然后在55-60℃条件下水浴10-15 min, 使RNA沉淀溶解, 将得到的RNA溶液于-80℃条件下长期保存。
利用Nanodrop2000对根据上述步骤提取出的total RNA, 检测其浓度和纯度, 利用琼脂糖凝胶电泳检测RNA完整性, 使用Agilent 2100测定total RNA RIN值。单次建库要求RNA总量5 μg, 浓度≥ 200 μg/mL, OD260/OD2280介于1.8-2.2之间。利用Illumina公司的TruSeqTM RNA sample preparation kit完成cDNA文库的构建, 并通过Illumina Hiseq测序平台的150 paired-end模式进行转录组测序。
1.2.2 转录组组装和ORF预测对于无参考基因组的转录组测序和相关研究, 获得RNA-seq高质量测序数据后, 需要将测序数据进行组装, 才能进行后续注释及其他生物学功能分析。Trinity(http://trinityrnaseq.sourceforge.net/, 版本号:v2.6.6)是目前从头组装Illumina短片段序列比较常用的软件, 本试验在对原始数据进行过滤接头和低质量数据后, 使用Trinity进行从头组装。使用TransDecoder(https://github.com/TransDecoder/TransDecoder, 版本号:5.5.0)软件识别转录本序列中候选的编码区域, 结合cd-hit(http://weizhongli-lab.org/cd-hit/, 版本号:v4.8.1)进行去冗余, 得到独立基因(Unigene)序列。
1.2.3 转录组功能注释使用Trinity软件组装得到转录本序列后, 要对生物学相关问题进行探究, 就要对这些转录本进行功能注释。将拼接所得所有(unigene)序列, 使用Interperscan(http://www.ebi.ac.uk/interpro/, 版本号:v5)进行结构域预测, 使用Blastp(https://www.ncbi.nlm.nih.gov/)分别与KEGG、eggNOG、NR、Trembl、Swissprot、数据库进行比对获得相应的功能注释信息, 然后进行KEGG通路富集分析。KEGG(Kyoto Encyclopedia of Genes and Genomes, 京都基因和基因组百科全书, http://www.genome.jp/kegg/)是基因组注释方面的公共数据库。KEGG数据库中丰富的通路信息将有助于我们从系统水平去了解基因的生物学功能, 例如代谢途径、遗传信息传递以及细胞过程等一些复杂的生物功能, 这大大提高了该数据库在实际生产和应用中的价值。根据与KEGG数据库比对结果, 获得转录本对应的KO编号, 根据KO编号可以获得某转录本可能参与的具体生物学通路。eggNOG(evolutionary genealogy of genes:Non-supervised Orthologous Groups)数据库包含了丰富的注释信息, 包含了KEGG等数据库信息。Swiss-Prot是一个手工注释的非冗余蛋白序列数据库。它结合了从科学文献中提取的信息和生物尿酸评估计算分析, TrEMBL(翻译的EMBL核苷酸序列数据库), 为那些不在Swiss-Prot中的蛋白质提供自动注释。NR(NCBI非冗余蛋白库)为综合数据库, 包括了SwissProt、PIR(Protein information resource)、PRF(Protein research foundation)、PDB(Protein data bank)蛋白质数据库中非冗余的数据以及从GenBank和RefSeq的CDS数据库中翻译所得的蛋白质数据。
1.2.4 差异表达基因分析使用组装软件将转录组组装好后, 利用RSEM(http://deweylab.biostat.wisc.edu/rsem/)进行表达量分析。RSEM利用bowtie的比对结果进行表达量统计, 首先得到每个样品比对到每个基因上的read count数目, 然后对其进行TPM数值转换, TPM数值可以反映基因的表达水平。使用R包(edgeR和DESeq)分别对小地老虎转录组进行基因表达差异分析, 取两者交集, 得到最终差异基因。
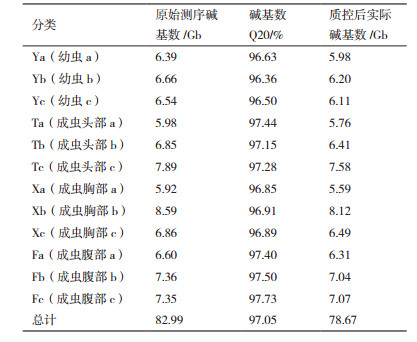
2 结果 2.1 原始测序数据质控测序小地老虎幼虫, 成虫头、胸和腹部, 各3个样本, 共计12个样本。小地老虎幼虫共测数据19.59 G, 小地老虎成虫头部20.73 G, 小地老虎成虫胸部21.36 G, 小地老虎成虫腹部21.32 G, 共计数据量82.99 G, 质控去掉Adapter和低于Q20的数据后最终得到78.67 G(表 1)。
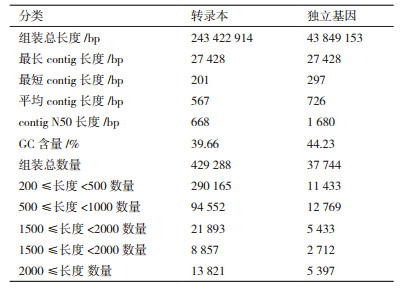
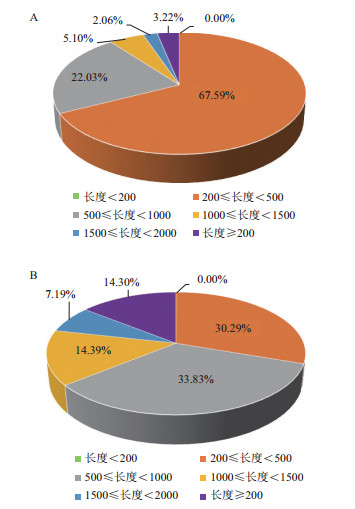
使用Trinity(http://trinityrnaseq.sourceforge.net/)软件, 对小地老虎转录组数据进行组装。最终组装得到转录组243.4 M, 最长序列27.4 K, 平均长度0.6 K, N50 0.7 K, denove转录组组装结果GC含量为39.66%(表 2)。使用TransDecoder对转录组进行ORF预测, 并去冗余后, 得到37 744个unigenes。转录组组装长度频数分布(图 1), 图 1-A为转录本(Transcripts)组装长度频数分布, 在200-500范围内的转录本比例最多, 为67.59%, 其次为在500-1 000范围内的转录本比例为22.03%。图 1-B为Unigenes组装长度频数分布, 在500-1 000范围内的转录本比例最多, 为33.83%, 其次为在200-500范围内的比例为30.29%。
|
| A:Transcripts(转录本)组装长度频数分布; B:unigenes(独立基因)组装长度频数分布 图 1 转录组组装长度频数分布 |
使用Interperscan进行结构域预测, 共有20 522个unigenes预测出结构域信息。将拼接所得独立基因, 使用Blastp分别与NR、Sport、Trembl、KEGG数据库进行比对, 比对后取besthit获得相应的注释信息。
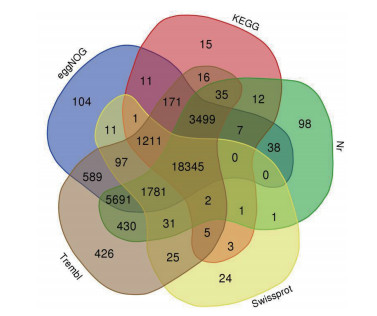
KEGG数据库注释功能基因23 334个, 注释通路373条, KO 6 161个; eggNOG数据路注释功能基因31 556个, Orthologous Groups(OG, 同源群)8 211个; Nr数据库共注释功能基因29 971个; Swissprot数据库注释功能基因本21 538个; Trembl数据库注释功能基因32 354个。各个数据库功能注释Venn图(图 2), 其中5个数据库能够共同注释到的基因有18 345个。5个数据库分别注释出了本数据库特有基因, 其中Trembl数据库注释出的特有基因最多, 为426个, KEGG数据库注释出的特有基因最少, 有15个(图 2)。
|
| 图 2 转录组不同数据库注释结果Venn图 |
分别使用EdgeR.R和DEseq2.R进行差异分析, 并求出差异基因。再将两种方法得到的差异基因求交集, 得到最终的差异基因(图 3)。最终得到小地老虎成虫头部样本与小地老虎胸部样本转录组差异基因4 069个, 小地老虎成虫胸部样本与小地老虎腹部样本转录组差异基因6 905个, 小地老虎成虫腹部样本与小地老虎头部样本转录组差异基因7 777个, 小地老虎幼虫与小地老虎成虫头部样转录组差异基因5 768个, 小地老虎幼虫与小地老虎成虫胸部样转录组差异基因4 836个, 小地老虎幼虫与小地老虎成虫腹部样转录组差异基因8 917个。小地老虎成虫头部与腹部差异基因最多, 小地老虎成虫头部与胸部差异基因最少。小地老虎幼虫与成虫头、胸、腹三部分相比, 小地老虎幼虫与成虫腹部的差异基因最多, 与胸部的差异基因最少(图 3)。
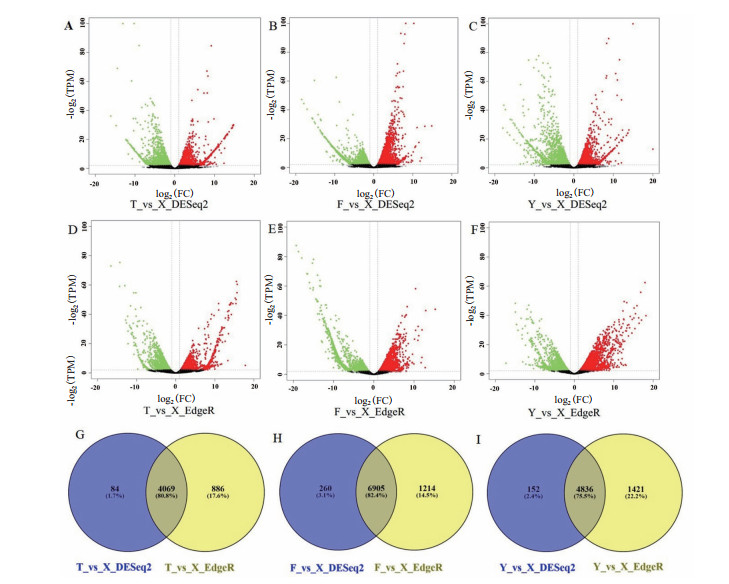
|
| T代表小地老虎成虫头部样本, X代表小地老虎胸部样本, F代表小地老虎腹部样本, Y代表小地老虎幼虫样本, vs代表差异, DESeq2与EdegR分别代表使用DESeq2和EdegR分析。图A、B、C、D、E、F为样本间基因差异表达火山图, 图G、H、I为DESeq2和EdegR分析的差异基因Venn图。例如, 图A中T_vs_X_DESeq2代表小地老虎成虫头部样本与小地老虎胸部样本转录组使用DESeq2分析得出的基因差异表达火山图 图 3 各组样本DESeq2与EdegR基因差异表达火山图和差异基因Venn图 |
通过对小地老虎转录组KEGG通路富集分析, 结合Interperscan结构域预测和其他注释信息得到小地老虎翅发育机制相关通路基因, 并进行表达量分析与表达量差异比较分析, 对差异表达基因绘制小地老虎Wnt信号通路基因表达热图(图 4);并根据KEGG通路差异基因富集分析绘制小地老虎Wnt信号通路基因图(图 5)。由小地老虎Wnt相关基因KEGG通路基因富集分析和表达量差异比较分析可知, Notum、Frizzled、CaN等53个元件均被注释出, 但是PRP、GBp、ICAT等17个元件没有在小地老虎转录组中发现(图 4-5)。Wnt信号通路包含经典Wnt通路, 非经典Wnt /钙通路以及非经典planar cell polarity通路, 其中非经典Wnt /钙通路以及非经典planar cell polarity通路总基因数目较少, 但是在小地老虎转录组中注释出的基因较多, 非经典Wnt /钙通路只有一个元件未注释出, 非经典planar cell polarity通路只有两个元件未注释出; 非经典Wnt /钙通路, 调控细胞内的钙, 非经典planar cell polarity通路调控细胞骨架, 骨架决定细胞的形状。
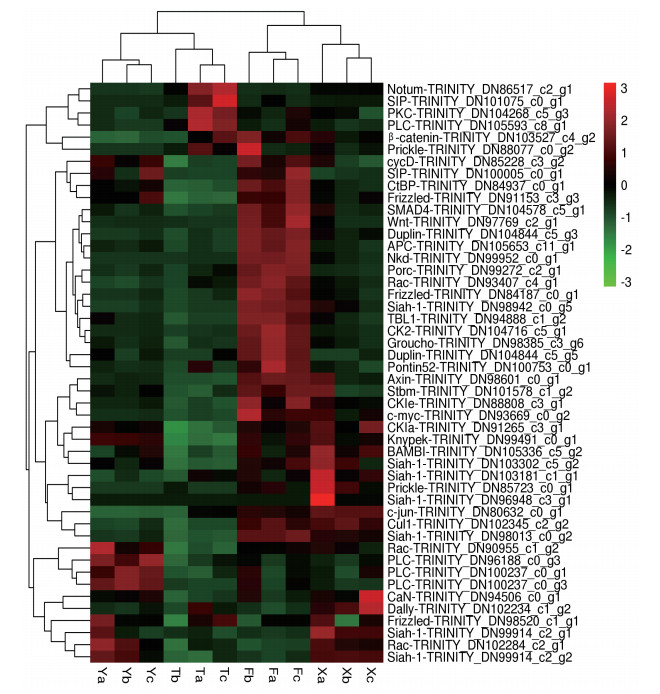
|
| 图中每列表示一个样本, 每行表示一个基因, 图中的颜色表示基因在该组样本中表达量的大小(log10TPM), 红色代表该基因在该样本中表达量较高, 绿色代表表达量较低, 具体表达量大小变化趋势请见右上方颜色条下的数字标注。左侧为基因聚类的树状图, 右侧为基因的名称, 两个基因分支离得越近, 说明它们的表达量越接近; 上方为样本聚类的树状图, 下方为样本的名称, 两个样本分支离的越近, 说明这两个样本所有基因的表达模式越接近, 即基因表达量变化趋势越接近 图 4 小地老虎Wnt信号通路基因表达热图 |
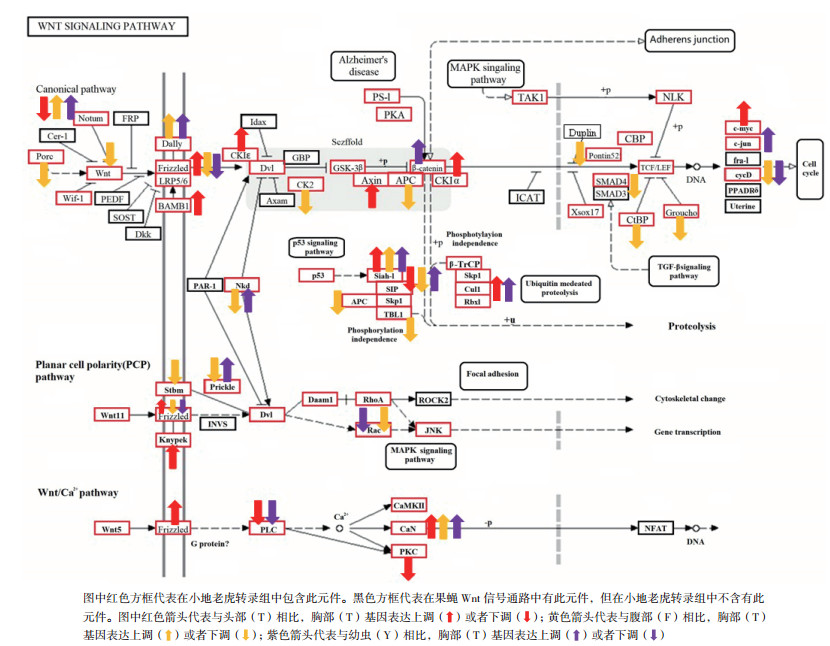
|
| 图 5 小地老虎Wnt信号通路基因图 |
小地老虎胸部组织与头部组织相比, Frizzled(Fz)、BMP and activin membrane-bound inhibitor(BAMBI)、(CKIα)、casein kinase 1(CKIε)、Axin、Myc proto-oncogene protein(c-myc)、E3 ubiquitin-protein ligase(Siah-1)、glypican 4(Knypek)、serine/threonine-protein phosphatase 2B catalytic subunit(CaN)、cullin 1(Cul1)等在小地老虎胸部组织中都上调表达, Notum、calcyclin binding protein(SIP)、classical protein kinase C alpha type(PKC)在胸部组织中下调表达。小地老虎胸部组织与腹部组织相比, Notum、Dally、Siah-1、CaN等在小地老虎胸部组织中都上调表达, wingless-type MMTV integration site family、Frizzled、Rac等在小地老虎胸部组织中下调表达。小地老虎胸部组织与小地老虎幼虫组织相比, Frizzled、Dally、SIP、Cul1、Siah-1等上调表达, Rac、phosphatidylinositol phospholipase C beta(PLC)、Frizzled等下调表达(图 5)。
小地老虎胸部组织相对于头部、腹部及幼虫组织, 只有Siah-1和CaN全部上调表达, Notum虽然在小地老虎胸部组织相对于头部、幼虫对比中上调表达, 但在小地老虎胸部组织相对腹部组织中下调表达。SIP同时在小地老虎胸部相对于头部和腹部比较中下调表达。
非经典planar cell polarity通路中, 小地老虎胸部相对于头部有表达差异的基因全部为表达上调, 小地老虎胸部相对于腹部有表达差异的基因全部为表达下调。非经典planar cell polarity通路中, PLC在小地老虎胸部相对于头部和幼虫的基因表达中均为下调, 下游元件CaN表现为表达上调。
3 讨论Wnt信号通路相关基因在小地老虎幼虫、小地老虎成虫头、胸、腹部均有表达。Wnt信号通路包含3种Wnt信号传导通路:经典Wnt通路, 非经典Wnt /钙通路以及非经典planar cell polarity通路。非经典planar cell polarity通路是通过Wnt与Fz及其共受体结合而激活的。然后受体结合Dishevelled(Dsh), Dsh利用其PDZ和DIX结构域与morphogenesis 1(Daam1)的Dishevelled-associated激活因子1形成复合物。然后Daam1通过鸟嘌呤交换因子激活G-蛋白Rho[24]。Rho激活Rho相关激酶Rho-associated kinase(ROCK), 这是细胞骨架的主要调节因子之一。Dsh还与Ras-related C3 botulinum toxin substrate 1(Rac1)形成复合物, 并介导profilin与肌动蛋白的结合[25]。Rac1激活JNK, 也可导致肌动蛋白聚合。丙氨酸与肌动蛋白结合可导致细胞骨架和原肠胚的重组。经典Wnt通路相关基因有一些在小地老虎转录组中未被注释到, 这可能与小地老虎的发育有关, 有些基因没有表达, 也有可能与物种有关, 小地老虎可能不含有某些基因。同时这些在小地老虎腹部表达的基因可能参与一些基因转录的调控, 这也是此信号通路的主要功能。
Wnt信号通路相关基因在小地老虎幼虫和成虫不同组织表达差异揭示小地老虎可能的翅发育机制, 非经典planar cell polarity通路调控细胞骨架, 决定骨架细胞的形状, Rac在小地老虎腹部相对小地老虎胸部上调表达, Rac也能间接影响JNK, 从而影响一些基因的转录。非经典Wnt /钙通路中, 小地老虎胸部相对于头部、腹部和小地老虎幼虫, CaN、Siah-1都表现为上调表达, CaN在肌肉形成和功能的基本过程中起着重要的作用[26], 因此小地老虎成虫胸部CaN的表达可能与其肌肉功能有关。本次试验的小地老虎成虫为处于迁飞状态的小地老虎成虫, 胸部肌肉相关基因表达较高与其生物性状相适应。
Wnt信号通路相关基因在小地老虎幼虫、成虫头、胸、腹部的表达存在差异, 小地老虎胸部组织与头部组织、腹部组织和小地老虎幼虫组织相比, CaN、Siah-1相关基因都显著上调表达。这些基因有可能在其他昆虫中也会有类似现象。对于昆虫翅发育相关基因的研究主要集中于昆虫羽化之前, 本试验表明, 在昆虫羽化后一些翅发育相关基因会继续在成虫胸部表达, 也可能与翅部肌肉等组织的发育和代谢有关。这些对于昆虫或者对于迁飞昆虫的研究可能提供一些研究思路。近年来, 随着illumina、pacbio、nonapore等高通量测序技术的迅猛发展, 以及CRISPR-Cas9技术的不断发展, 越来越多的科学问题通过基因组测序成功解决, 也为研究昆虫迁飞相关基因功能解析提供了新的技术方法[27]。
另外, 影响昆虫翅发育相关通路还有Decapen-taplegic(Dpp)、Hedgehog、Hippo信号通路等。Dpp是参与果蝇发育的关键形态发生素, Dpp也是第一个经过验证的分泌形态发生素[12-13]。Hedgehog(Hh)信号通路在控制细胞增殖、组织模式形成、干细胞维持和发育方面发挥着多种作用[15-18]。Hippo信号通路也被称为Salvador或者Warts通路, 通过调控细胞增殖和凋亡来控制动物的器官大小。该通路的名字来源于它的一个关键信号元件——蛋白激酶Hippo(Hpo)[19-20]。该基因的突变导致组织过度生长, 或类似“河马”表型。
昆虫翅的进化对昆虫的意义重大, 探究影响昆虫翅生长发育的相关通路, 有助于了解昆虫翅的进化及发育的过程。在众多的昆虫中, 有相当一部分为农业害虫, 有些如棉铃虫、小地老虎等还是可进行迁飞的害虫, 探究昆虫翅的发育机制, 可以为揭开昆虫迁飞面纱并研发新型防治农业害虫方法提供帮助。本研究通过对小地老虎转录组的组装及注释, 结合影响果蝇翅发育相关通路基因, 找到许多潜在与小地老虎翅发育相关通路基因, 为后续研究小地老虎翅发育相关基因提供了良好的基础。
4 结论通过对小地老虎转录组的组装及注释, 最终组装得到转录组243.4 Mb, 组装出转录本429 288个, 独立基因37 744个。小地老虎Wnt相关通路中, Notum、Frizzled、CaN等53个相关元件被注释出。Wnt信号通路中的非经典planar cell polarity通路中, PlC在小地老虎胸部相对于头部和幼虫的基因表达中均为下调, 下游元件CaN、Siah-1在小地老虎胸部相对于头部、腹部及幼虫组织的基因表达中均表现为表达上调, 可能与其肌肉功能有关。
| [1] |
陈永林, 宋绍宗, 邓小山. 中国渤海及黄海海面迁飞昆虫的初步观察[J]. 昆虫学报, 1963, 7(2): 137-148. |
| [2] |
刘辉, 李克斌, 尹姣, 等. 群居型与散居型东亚飞蝗飞行能力的比较研究[J]. 植物保护, 2007, 33(2): 34-37. DOI:10.3969/j.issn.0529-1542.2007.02.008 |
| [3] |
付晓伟, 吴孔明. 迁飞性昆虫对全球气候变化的响应[J]. 中国农业科学, 2015, 48(S1): 1-15. |
| [4] |
Guerra PA, Reppert SM. Sensory basis of lepidopteran migration:focus on the monarch butterfly[J]. Curr Opin Neurobiol, 2015, 34: 20-28. DOI:10.1016/j.conb.2015.01.009 |
| [5] |
Sappington TW. Migratory flight of insect pests within a year-round distribution:European corn borer as a case study[J]. Journal of Integrative Agriculture, 2018, 17(7): 1485-1505. DOI:10.1016/S2095-3119(18)61969-0 |
| [6] |
Dreyer D, Frost B, et al. The earth's magnetic field and visual landmarks steer migratory flight behavior in the nocturnal Australian bogong moth[J]. Curr Biol, 2018, 28(13): 2160-2166. DOI:10.1016/j.cub.2018.05.030 |
| [7] |
Frazier MR, Harrison JF, Kirkton SD, et al. Cold rearing improves cold-flight performance in Drosophila via changes in wing morphology[J]. J Exp Biol, 2008, 211(13): 2116-2122. DOI:10.1242/jeb.019422 |
| [8] |
Prokop J, Pecharova M, Nel A, et al. Paleozoic nymphal wing pads support dual model of insect wing origins[J]. Current Biology, 2017, 27(2): 263-269. DOI:10.1016/j.cub.2016.11.021 |
| [9] |
Rothbacher U, Laurent MN, Blitz IL, et al. Functional conservation of the Wnt signaling sathway revealed by ectopic expression of Drosophila dishevelled in Xenopus[J]. Developmental Biology, 1995, 170(2): 717-721. DOI:10.1006/dbio.1995.1249 |
| [10] |
Wang JN, Li L, Li LY, et al. Emerging role and therapeutic implication of Wnt signaling pathways in liver fibrosis[J]. Gene, 2018, 674: 57-69. DOI:10.1016/j.gene.2018.06.053 |
| [11] |
Zhang HW, Zhang H, Zhang YQ, et al. Dishevelled-DEP domain interacting protein(DDIP)inhibits Wnt signaling by promoting TCF4 degradation and disrupting the TCF4/beta-catenin complex[J]. Cell Signal, 2010, 22(11): 1753-1760. DOI:10.1016/j.cellsig.2010.06.016 |
| [12] |
Gui J, Huang Y, Montanari M, et al. Coupling between dynamic 3D tissue architecture and BMP morphogen signaling during Drosophila wing morphogenesis[J]. Proc Natl Acad Sci USA, 2019, 116(10): 4351-4361. |
| [13] |
Matsuda S, Harmansa S, Affolter M. BMP morphogen gradients in flies[J]. Cytokine Growth Factor Reviews, 2016, 27: 119-127. DOI:10.1016/j.cytogfr.2015.11.003 |
| [14] |
Matsuda S, Yoshiyama N, et al. Dpp/BMP transport mechanism is required for wing venation in the sawfly Athalia rosae[J]. Insect Biochem Mol Biol, 2013, 43(5): 466-473. DOI:10.1016/j.ibmb.2013.02.008 |
| [15] |
Ye X, Liu A. Hedgehog signaling:mechanisms and evolution[J]. Frontiers in Biology, 2011, 6(6): 504-521. DOI:10.1007/s11515-011-1146-2 |
| [16] |
Jacob L, Lum L. Hedgehog signaling pathway in Drosophila[J]. Sci STKE, 2007, 2007(407): cm7. |
| [17] |
Villarreal CM, Darakananda K, et al. Hedgehog signaling regulates imaginal cell differentiation in a basally branching holometabolous insect[J]. Dev Biol, 2015, 404(2): 125-135. |
| [18] |
Lee RT, Zhao Z, Ingham PW. Hedgehog signalling[J]. Development, 2016, 143(3): 367-372. |
| [19] |
Gou J, Lin L, Othmer HG. A model for the Hippo pathway in the Drosophila wing disc[J]. Biophys J, 2018, 115(4): 737-747. DOI:10.1016/j.bpj.2018.07.002 |
| [20] |
Meng ZP, Moroishi T, Guan KL. Mechanisms of Hippo pathway regulation[J]. Gene Dev, 2016, 30(1): 1-17. |
| [21] |
Showers WB. Migratory ecology of the black cutworm[J]. Annual Review of Entomology, 1997, 42: 393-425. DOI:10.1146/annurev.ento.42.1.393 |
| [22] |
Haug JT, Haug C, Garwood RJ. Evolution of insect wings and development - new details from Palaeozoic nymphs[J]. Biol Rev Camb Philos Soc, 2016, 91(1): 53-69. DOI:10.1111/brv.12159 |
| [23] |
Brisson JA, Ishikawa A, Miura T. Wing development genes of the pea aphid and differential gene expression between winged and unwinged morphs[J]. Insect Mol Biol, 2010, 19(Suppl 2): 63-73. |
| [24] |
Swarup S, Pradhan-Sundd T, Verheyen EM. Genome-wide identification of phospho-regulators of Wnt signaling in Drosophila[J]. Development, 2015, 142(8): 1502-1515. DOI:10.1242/dev.116715 |
| [25] |
Girardi F, Le Grand F. Wnt signaling in skeletal muscle development and regeneration[J]. Prog Mol Biol Transl Sci, 2018, 153: 157-179. DOI:10.1016/bs.pmbts.2017.11.026 |
| [26] |
Gajewski KM, Wang JB, Schulz RA. Calcineurin function is required for myofilament formation and troponin Ⅰ isoform transition in Drosophila indirect flight muscle[J]. Developmental Biology, 2006, 289(1): 17-29. |
| [27] |
Ohde T, Takehana Y, Shiotsuki T, et al. CRISPR/Cas9-based heritable targeted mutagenesis in Thermobia domestica:A genetic tool in an apterygote development model of wing evolution[J]. Arthropod Struct Dev, 2018, 47(4): 362-369. DOI:10.1016/j.asd.2018.06.003 |