2. 山西省农业科学院高粱研究所, 晋中 030600;
3. 山西省农业科学院小麦研究所, 临汾 041000
2. Sorghum Institute of Shanxi Academy of Agricultural Sciences, Jinzhong 030600;
3. Wheat Research Institute, Shanxi Academy of Agricultural Sciences, Linfen 041000
受到全球工业飞速发展及人为活动的影响, 温室气体引起的全球变化成为学术界研究可持续发展的重点研究领域[1]。造成温室效应的主要气体是CO2, 合理固定CO2也可使无机碳转化为巨大的可再生碳资源[2-3], 在提倡减排的同时, 国内外学者探索了化学固碳、物理固碳和生物固碳等多种固碳技术[4-10]。生物固碳是通过植物与微生物的固碳能力来实现的[11], 在多种固定CO2的途径中, 因其绿色经济且无污染等特点而备受关注[12-13]。而微生物以其环境适应性强、生长迅速、繁殖快等优势, 成为目前固定CO2研究的焦点。从整个生物圈的物质流、能量流而言, 微生物固定CO2在环境、能源和资源方面具有极其重要的意义[14-15]。
在国内外的各种研究中可固定CO2的微生物主要为自养菌, 广泛分布在如海洋深处[15-16]、火山口[17]、湖泊盆地[18]、土壤[19-21]等多种生态环境下, 从以上研究中也分离获得了一些固碳微生物, 大多为古细菌、蓝细菌、光合细菌以及一些混合培养的菌群[15-21], 多数为兼性厌氧菌, 在菌种纯化、保存和应用方面存在一定难度。因此, 如何既能利用微生物繁殖快、对环境适应性强、生长周期短的特点, 又能分离和筛选到易培养, 好保存的固碳菌株, 成为本研究的目的。
本研究尝试从自养微生物含量较高的活性污泥、沼液及设施土壤中分离筛选固CO2菌, 并对其进行形态观察和生理生化反应测定和16S rDNA测序分析, 通过对比进行鉴定, 并对其生长情况和RubisCO酶活性进行研究, 同时检测固碳酶RubisCO中cbbL基因的特异性条带, 最后将高效固碳菌株施入土壤中, 检测其对土壤RubisCO酶活性的影响。以期通过利用固碳微生物的作用, 减少空气中CO2气体的浓度, 为减缓"温室效应"提供更加高效的方法。
1 材料与方法 1.1 材料 1.1.1 样品采集于活性污泥(太原市东太堡污水处理厂)、沼液、设施土壤等多种生态环境中, 样品采集后将其保存于保鲜袋中并带回实验室进行处理。
1.1.2 无碳源无机培养基组成成分[15, 19](1) MnSO40.02 g、Na2HPO4 1 g、KH2PO4 1.8 g、MgSO4 0.4 g、CaCl2 0.05 g、NaCl 1 g、NH4Cl 0.5 g、FeCl3 0.02 g、Na2S2O3 10 g, 蒸馏水加至1 000 mL, 121℃灭菌20 min, 备用。(2)MnSO4 1 g、Na2HPO4 0.5 g、KH2PO4 0.5 g、MgSO4 1g、CaCl2 0.2 g、NaHCO3 1 g、NH4Cl 0.5 g、KNO3 1 g、NaCl 0.4 g、微量元素溶液2 mL, 蒸馏水加至1 000 mL, 121℃灭菌20 min, 备用。其中, 微量元素溶液的配制为:FeCl3 0.3 g, FeSO4·7H2O 0.3 g, MnSO4·H2O 0.15 g, ZnSO4 0.14 g, CoCl2 0.2 g, 定容至1 000 mL, 过滤除菌后备用。
1.2 方法 1.2.1 固CO2菌株分离纯化及筛选将10 g样品加入装有90 mL无菌水的三角瓶中充分混合, 置于30℃, 150 r/min摇床振荡约20 min, 制成悬液, 进行梯度稀释。在无菌操作下, 选取10-5、10-6、10-7稀释浓度, 吸取1 mL样品悬液注入盛有无碳源固体培养基的无菌培养皿中, 涂匀, 28℃倒置培养7-10 d, 挑取单菌落, 进行标记, 划线分离纯化多次, 于4℃保存[22]。筛选生长速度较快的菌株进行进一步研究。
1.2.2 固CO2菌株固碳功能基因cbbL的克隆 1.2.2.1 细菌基因组DNA的提取及固CO2菌株cbbL基因的扩增按照Ezup柱式细菌基因组DNA抽提试剂盒(上海生工)的提取步骤, 提取不同菌株的基因组DNA。置-20℃冰箱中保存备用。以提取的细菌组总DNA为模板, 进行cbbL基因的扩增[23], 扩增引物的上游引物为k2F:ACCAY CAAGC CSAAG CTSGG, 下游引物为:v2F:GCCTT CSAGC TTGCC SACCRC, 引物由上海生工技术有限公司合成。PCR反应体系参照试剂盒(生工)采用25 μL体系:DNA模板1 μL, 上游引物0.5 μL, 下游引物0.5 μL, 2×Taq PCR Master mix 12.5 μL。PCR反应程序:95℃预变性5 min, 95℃变性45 s, 57℃退火45 s, 72℃延伸60 s(35次循环), 72℃延伸10 min。PCR产物在100 V电压下用1.0%琼脂糖凝胶电泳30 min, 经染色后在凝胶成像仪下拍照, 确定含cbbL基因的固碳菌菌株。
1.2.2.2 cbbL基因测序将492 bp目标条带的25 µL体系的PCR产物全部进行切胶回收, 按照SanPrep柱式DNA胶回收试剂盒(上海生工)对DNA进行回收, 得到纯化后的DNA条带。PCR产物的连接载体用pGEM-TEasy, 连接体系用10 µL体系:pGEM-TEasy Vector 1 µL, PCR Products 4 µL, Ligation Mix 5 µL。连接反应条件为4℃反应过夜。加入50 µL competent cells, 置于冰上静置30 min后, 42℃水浴90 s, 冰上放置2 min。加入500 µL的LB稀释液, 轻轻摇匀。涂布于含有X-Gal、IPTG、Amp的LB琼脂平板上, 37℃过夜培养, 通过蓝白筛选挑选出白色菌落。挑取白色单菌落于3 mL液体LB培养基中, 37℃, 165 r/min恒温箱中振荡培养6 h。PCR检测阳性克隆, 送测序公司测序。
1.2.3 固CO2菌株的鉴定对分离筛选到的固CO2菌株进行菌种鉴定。
1.2.3.1 固CO2菌株形态特征将分离筛选到的菌株进行菌落特征、生长情况的观察, 并进行革兰氏染色, 镜检[24]。
1.2.3.2 固CO2菌株生理生化特性将分离筛选到的菌株进行淀粉水解、明胶水解、乳酸产生、甲基红反应、吲哚反应等生理生化反应, 观察记录反应结果[25]。
1.2.3.3 16SrDNA基因的扩增设计16S rDNA基因序列引物[26]:上游引物16S A5′-AGTTT GATCC TGGCT CA-3′, 下游引物16S B 5′-TACCT TGTTA CGACT TCA-3′。PCR反应体系参照上海生工的PCR扩增试剂盒, 采用25 µL体系:DNA模板1 µL, 上游引物1 µL, 下游引物1 µL, 2×PCR mix 12.5 µL, ddH2O 9.5 µL。PCR反应条件:94℃预变性3 min; 35个循环为94℃变性1 min, 50℃退火1 min, 72℃延伸3 min; 72℃最终延伸10 min。PCR产物在100 V电压下用1.0%的琼脂糖凝胶电泳30 min检测扩增片段, 经染色后置于凝胶成像仪下拍照记录。
1.2.3.4 16SrDNA基因与载体pMD20-T的连接将单一条带的25 μL体系的PCR产物全部进行切胶回收, 按照SanPrep柱式DNA胶回收试剂盒(上海生工)对DNA进行回收, 得到纯化后的DNA条带。PCR产物的连接载体用pMD20-T Vector System, 连接体系用10 µL体系:pMD20-T Vector 1 µL, PCR Products 1 µL, Ligation Mix 5 µL, ddH2O 3 µL。连接反应条件为16℃反应30 min。加入50 μL competent cells, 置于冰上静置20 min后, 水浴60 s, 放置2 min。加入500 μL的LB稀释液, 轻轻摇匀。涂布于含有X-Gal、IPTG、Amp的LB琼脂平板上, 37℃过夜培养, 通过蓝白筛选挑选出白色菌落[27]。
1.2.3.5 重组子的筛选将过夜培养的平板拿出后, 观察菌落情况, 置于4℃冰箱以便更明显的分辨蓝白菌落。挑取白色单菌落于3 mL液体LB培养基中, 37℃, 165 r/min恒温箱中振荡培养过夜。按照质粒DNA抽提试剂盒的步骤, 对菌液进行质粒DNA的提取。在插入位点的上游和下游分别选择一个高酶位点, 对质粒DNA进行酶切验证。上游选择Hind Ⅲ, 下游选择Eco RI。
1.2.3.6 序列测定与分析重组质粒送上海生工进行序列测定, 序列测定采用双向测定, 测序结果在GenBank中进行BlAST比对, 并构建系统发育树[28]。
1.2.4 固碳菌菌体生长及菌体含量测定选取分离筛选到的固碳菌菌株进行液体培养, 通过测定菌液OD值及离心后菌体的干重, 选择生长快、菌体含量高的固碳菌菌株进行土壤培养试验。
1.2.5 土壤中固碳菌培养试验在田间采集土壤, 风干, 去除石块、植株根系等杂质后, 过2 mm筛, 测定土壤基础养分含量(pH 8.73, EC 610 μs/cm, 全N 0.28 g/kg, 有效磷1.5 mg/kg, 速效钾97.16 mg/kg, 有机质3.65 g/kg), 备用。采用内径20 cm, 深度30 cm的塑料盆, 每盆装土5 kg。随水施入固碳菌菌液, 按菌液原液0.5 mL/kg土施用, 每15 d补施一次菌液。定期向土壤补充水分, 培养结束后测定土壤的RubisCO酶活性。以不施菌液为空白处理, 施用灭菌固碳菌为对照。每个处理重复5盆。
1.2.6 RubisCO酶活性测定 1.2.6.1 样品处理土壤样品采集后置于液氮中带回实验室后-80℃超低温冰箱保存。测定前称取样品1 g加9 mL匀浆液在研钵中进行匀浆, 匀浆液为PBS(pH7.2-7.4, 浓度为0.01 mol/L)。土壤样本需进行含水率的测定。菌液样品加PIPA裂解液进行处理。
1.2.6.2 RubisCO酶酶联免疫分析测定菌样、植株及土壤样品的RubisCO酶活性利用上海生工提供的1, 5-二磷酸核酮糖羧化酶/加氧酶(RubisCO)酶联免疫分析(ELISA)试剂盒进行测定[29]。测定方法参照试剂盒说明书, 方法如下:(1)标准品的稀释与加样:在酶标包被板上设标准品孔10孔, 在第一、第二孔中分别加标准品100 μL, 然后在第一、第二孔中加标准品稀释液50 μL, 混匀; 然后从第一孔、第二孔中各取100 μL分别加到第三孔和第四孔, 再在第三、第四孔分别加标准品稀释液50 μL, 混匀; 然后在第三孔和第四孔中先各取50 μL弃掉, 再各取50 μL分别加到第五、第六孔中, 再在第五、第六孔中分别加标准品稀释液50 µL, 混匀; 混匀后从第五、第六孔中各取50 μL分别加到第七、第八孔中, 再在第七、第八孔中分别加标准品稀释液50 μL, 混匀后从第七、第八孔中分别取50 μL加到第九、第十孔中, 再在第九、第十孔分别加标准品稀释液50 μL, 混匀后从第九、第十孔中各取50 μL弃掉。(稀释后各孔加样量都为50 μL, 浓度分别为150 ng/L、100 ng/L、50 ng/L、25 ng/L和12.5 ng/L)。(2)加样:分别设空白孔(空白对照孔不加样品及酶标试剂, 其余各步操作相同)、待测样品孔。在酶标包被板上待测样品孔中先加样品稀释液40 μL, 然后再加待测样品10 μL(样品最终稀释度为5倍)。将样品加于酶标板孔底部, 尽量不触及孔壁, 轻轻晃动混匀。(3)温育:用封板膜封板后置37℃温育30 min。(4)配液:将30倍浓缩洗涤液用蒸馏水30倍稀释后备用。(5)洗涤:小心揭掉封板膜, 弃去液体, 甩干, 每孔加满洗涤液, 静置30 s后弃去, 如此重复5次, 拍干。(6)加酶:每孔加入酶标试剂50 μL, 空白孔除外。(7)温育:操作同3。(8)洗涤:操作同5。(9)显色:每孔先加入显色剂A50 μL, 再加入显色剂B50 μL, 轻轻震荡混匀, 37℃避光显色15 min。(10)终止:每孔加终止液50 μL, 终止反应(此时蓝色立转黄色)。(11)测定:加终止液后15 min以内进行测定, 以空白调零, 450 nm波长依序测量各孔的吸光度(OD值)。
2 结果 2.1 菌株分离筛选利用无碳源无机培养基从3种样品中分离到24株菌株, 采用划线分离纯化后, 对其生长情况进行观察, 筛选出生长速度较快的8株菌, 分别编号为:C2-8R、CX-9R、C5-6R、C2-8W、K01R、S01W、K01W和01W-1。
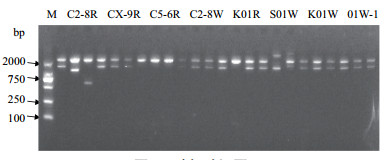
2.2 cbbL基因分析对生长速度较快的8株菌进行cbbL固碳基因的鉴定。用cbbL基因的兼并引物对细菌总DNA进行扩增, 扩增结果见图 1, 图中结果显示, C2-8R、CX-9R、C5-6R、K01R、S01W和01W-1菌株在500 bp附近出现cbbL基因目的片段条带。

|
| 图 1 8株菌cbbL基因PCR扩增结果 |
将测序公司测序结果进行比对, 8株菌可归为两大类, 结果见图 2。

|
| 图 2 8株菌cbbL基因比对结果 |
对筛选的生长速度较快的8株固CO2菌株进行菌落形态观察及革兰氏染色镜检。菌落形态均为黏着突起, 直径在1-5 mm之间, 菌落颜色为红色的是:C2-8R、CX-9R、C5-6R、K01R;菌落颜色为白色的是:C2-8W、S01W、K01W、01W-1;革兰氏染色为阴性菌的是:C2-8R、CX-9R、C5-6R、C2-8W、K01R和01W-1。革兰氏染色为阳性菌的是:S01W和K01W。
2.3.2 固CO2菌株生理生化反应对筛选到的生长较快的8株菌株进行生理生化反应实验, 其淀粉水解、明胶水解、乳酸产生、甲基红反应和吲哚反应等生理生化反应均呈阴性。
2.4 16S rDNA序列分析结果 2.4.1 细菌基因组DNA的提取及扩增对生长较好的8株菌株进行DNA测序分析。利用0.8%琼脂糖凝胶电泳进行检测提取到细菌基因组DNA大小约为20 000 bp。以不同菌株基因组DNA为模板进行PCR反应, 产物经0.8%琼脂糖凝胶电泳检测, 只有一条扩增带, 特异性较高。
2.4.2 重组质粒的的分子验证PCR扩增产物与pMD20-TVector载体重组后转入感受态DH5α细胞中, 经筛选后, 提取3个重复菌落的质粒DNA, 进行限制性内切酶酶切验证(图 3)。从得到的图中可以明显观察到泳道经酶切后会出现新的DNA片段, 可以发现有2个新片段出现, 导致这种现象的原因可能是目的片段上具有2个限制性内切酶位点。实验结果表明:重组质粒上有连接片段, 16S rDNA已克隆到pMD20-T载体上, 重组质粒构建成功。
|
| 图 3 酶切验证图 |
得到的重组质粒由上海生工公司进行序列测定, 测序结果在GenBank中进行BlAST比对, 构建系统发育树见图 4。筛选的菌株分别聚类假单胞菌属和嗜甲基菌属。
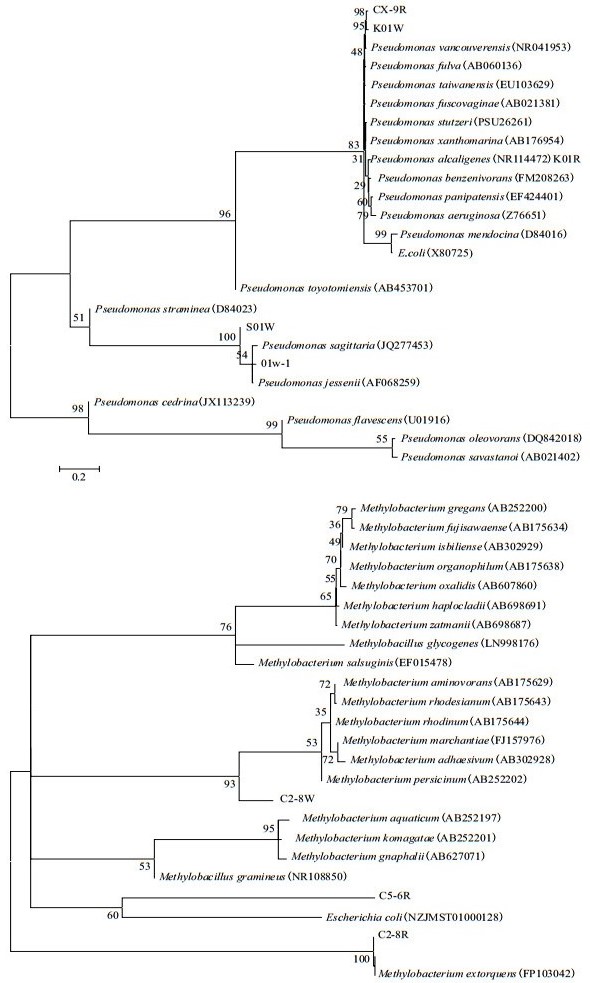
|
| 图 4 固碳菌假单胞菌属和嗜甲基菌属系统发育树 |
通过2.3, 2.4结果, 选取C2-8R、CX-9R、C5-6R、S01W及01W-1菌株进行液体培养, 对菌液OD值和菌体干重进行测定, 结果见图 5、图 6。从这两个图可以看出, C2-8R菌株的生长速度及菌体含量都优于其他菌株。
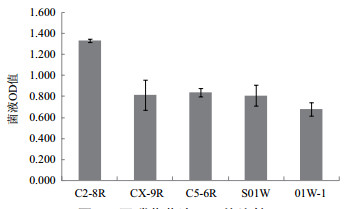
|
| 图 5 固碳菌菌液OD值比较 |

|
| 图 6 固碳菌菌体含量比较 |
将2.4中液体培养的C2-8R、CX-9R、C5-6R、S01W和01W-1菌株的菌液进行RubisCO酶活性测定, 利用上海生工提供的1, 5-二磷酸核酮糖羧化酶/加氧酶(RubisCO)酶联免疫分析试剂盒进行测定, 测定结果见图 7。C2-8R菌株RubisCO酶活性最高, 因此, 选取C2-8R菌株进行土壤施用试验。
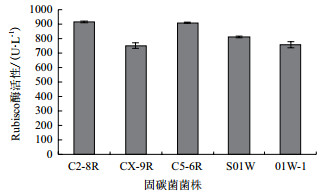
|
| 图 7 固碳菌菌液RubisCO酶活性比较 |
土壤样品的RubisCO酶活性利用上海生工提供的1, 5-二磷酸核酮糖羧化酶/加氧酶(RubisCO)酶联免疫分析试剂盒进行测定, 测定结果见图 8。由图 8可见, 施入固碳菌C2-8R菌液后土壤样品的RubisCO酶活性均比不施用菌液和灭菌菌液的活性高, 初步认为固碳菌C2-8R菌液施入土壤后可提高土壤的RubisCO酶活性。
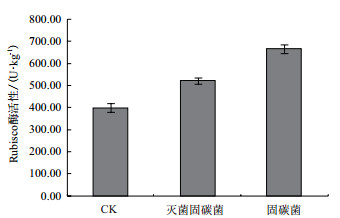
|
| 图 8 施固碳菌菌液后土壤样品的RubisCO酶活性比较 |
目前生物固碳主要是通过植物和微生物的循环途径将CO2转化为有机物质[30], 植物是通过光合作用来固定CO2, 微生物固定CO2则大多是通过自养微生物来实现的, 自养微生物从能量获得的途径不同分为光能自养微生物和化能自养微生物。光能自养微生物主要包括微藻类和光合细菌, 化能自养微生物包括严格化能自养菌和兼性化能自养菌[31]。目前固碳菌的研究在微藻和光合细菌方面涉及较多, 化能自养微生物的研究集中在氢氧化细菌和固碳微生物菌群方面, 此类自养微生物需要提供电子供体来进行繁殖[15, 21, 32]。本研究利用无碳源无机培养基分离筛选的固碳菌株可以直接利用CO2作为碳源生长, 不需要外加碳源和电子供体, 降低了固碳成本。
卡尔文(Calvin)循环是化能自养微生物固定CO2的主要途径, RubisCO酶是催化自养微生物利用卡尔文循环进行CO2固定的限速酶, cbbL基因则是编码RubisCO酶的碳同化功能基因, 含有cbbL基因的菌株具有编码RubisCO酶的能力, 较高的RubisCO酶活性则说明自养微生物具有较高的碳同化能力[33]。本研究筛选的可利用CO2生长的菌株, 通过cbbL基因和RubisCO酶活性的检测, 优选出固碳能力强的菌株。下一步将对固碳菌菌株进行驯化和发酵条件优化, 使其应用到生产实践中, 吸收空气中的CO2减轻温室效应, 最终造福人类。
4 结论本研究利用以空气中CO2为唯一碳源的无碳源培养基, 从多种生态环境中分离筛选到生长较快的固CO2菌8株, 并对其进行形态结构特征和生理生化特性研究。
通过16S rDNA序列测定, 对部分生长较快的菌株进行分子鉴定并构建系统发育树, 确定分离纯化的固碳菌的分子生物学地位, 筛选的菌株分别聚类假单胞菌属和嗜甲基菌属。
通过对5株生长快、含cbbL基因的固CO2菌株进行生长曲线、菌体含量和RubisCO酶活性的测定, 筛选确定固碳效率高的菌株C2-8R(Methanotrophs extorquens)进行应用研究。
通过C2-8R菌液施入土壤试验发现, 施用C2-8R菌液的土壤中RubisCO酶活性均比不施用菌液和灭菌菌液的活性高, 初步认为固碳菌C2-8R菌液施入土壤后可提高土壤的RubisCO酶活性。
| [1] |
Adams JM, Piovesan G. Uncertainties in the role of land vegetation in the carbon cycle[J]. Chemosphere, 2002, 49: 805-819. DOI:10.1016/S0045-6535(02)00382-X |
| [2] |
Oexmann J, Hensel C, Kather A. Post-combustion CO-capture from coal-fired power plants:Preliminary evaluation of an integrated chemical absorption process with piperazine-promoted potassium carbonate[J]. International Journal of Greenhouse Gas Control, 2008, 2(4): 539-552. DOI:10.1016/j.ijggc.2008.04.002 |
| [3] |
Rodríguez N, Alonso M, Grasa G. Process for capturing CO2 arising from the calcination of the CaCO3 used in cement manufacture[J]. Environ Sci Technol, 2008, 42(18): 69-80. |
| [4] |
张炜, 李义连. 二氧化碳储存技术的研究现状和展望[J]. 环境污染与防治, 2006, 28(12): 950-953. DOI:10.3969/j.issn.1001-3865.2006.12.021 |
| [5] |
Grntzis T. Susurface sequestration of carbon dioxide-an overview from an Alberta(Canada)perspective[J]. International Journal of Coal Geology, 2000, 43(1/4): 287-305. |
| [6] |
Freund P, Ormerod WG. Progress toward storage of carbon dioxide[J]. Energy Conversion and Management, 1997, 38(S1): 199-204. |
| [7] |
Lal R. Carbon sequestration[J]. Philosophical Transactions of the Royal Society B, 2008, 363(4): 815-830. |
| [8] |
Dixon RK, Brown S, Houghton RA, et al. Carbon pools and flux of global forest ecosystems[J]. Science, 1994, 263(14): 185-190. |
| [9] |
Masakazu M, Masahiro I. The biological CO2 fixation and utilization project by rite(2)-screening and breeding of microalgae with high capability in fixing CO2[J]. Energy Conversion and Management, 1997, 38(5): 493-497. DOI:10.1016/S0196-8904(96)00066-0 |
| [10] |
Grimston MC, Karakoussis V, Fouquet R, et al. The European and global potential of carbon dioxide sequestration in tackling climate change[J]. Climate Policy, 2001, 1(2): 155-171. DOI:10.3763/cpol.2001.0120 |
| [11] |
Miltner A, Kopinke FD, Kindler R. Non-phototrophic CO2 fixation by soil microorganisms[J]. Plant and Soil, 2005, 269(1-2): 193-203. DOI:10.1007/s11104-004-0483-1 |
| [12] |
Shively JM, English RS, Baker SH, et al. Carbon cycling:the prokaryotic contribution[J]. Current Opinion in Microbiology, 2001, 4: 301-306. DOI:10.1016/S1369-5274(00)00207-1 |
| [13] |
Richarda KR. A brief overview of carbon sequestration economics and policy[J]. Environmental Management, 2004, 33(4): 545-558. |
| [14] |
袁红朝, 秦红灵, 刘守龙, 等. 长期施肥对稻田土壤固碳功能菌群落结构和数量的影响[J]. 生态学报, 2012, 32(1): 0183-0189. |
| [15] |
胡佳俊, 王磊, 李艳丽, 等. 非光合CO2同化微生物菌群的选育/优化及其群落结构分析[J]. 环境科学, 2009, 30(8): 2438-2444. DOI:10.3321/j.issn:0250-3301.2009.08.043 |
| [16] |
Xu HH, Abita FR. Ribulose-1, 5-bisphosphatecarboxylase /oxygenasegene expression and diversity of Lake Eric planktonic microorganisms[J]. Appl Environ Microbiol, 1996, 62: 1913-1921. |
| [17] |
Nanba K, King GM, Dunfield K. Analysis of facultative lithotrophic distribution and diversity on volcanic deposits by use of the large subunit of ribulose-1, 5-bisphosphatecarboxylase / oxygenase[J]. Appl Environ Microbiol, 2004, 70: 2245-2253. DOI:10.1128/AEM.70.4.2245-2253.2004 |
| [18] |
Elsaied H, Naganuma T. Phylogenetic diversity of ribulose-1, 5-bisphosphate carboxylase/oxygenase large-subunit genes from deep-sea microorganisms[J]. Appl Environ Microbiol, 2001, 67: 1751-1765. DOI:10.1128/AEM.67.4.1751-1765.2001 |
| [19] |
周盛, 韦彬勤, 张琼, 等. 一种能同时固定CO2和N2的微生物——兼性固CO2、N2菌的分离鉴定及其验证实验[J]. 环境科学学报, 2013, 33(4): 1043-1050. |
| [20] |
吴小红, 简燕, 陈晓娟, 等. 自养微生物同化CO2的分子生态研究及同化碳在土壤中的转化[J]. 生态学报, 2014, 34(3): 701-709. |
| [21] |
王竞, 周集体, 张晶晶, 等. 固定CO2氢细菌的筛选及其培养条件优化[J]. 应用与环境生物学报, 2000, 6(3): 271-275. DOI:10.3321/j.issn:1006-687X.2000.03.014 |
| [22] |
范秀容, 李广武, 沈萍. 微生物学实验[M]. 第2版. 北京: 高等教育出版社, 1995: 107-112.
|
| [23] |
Tolli J, King GM. Diversity and structure of bacterial chemoautotrophic communities in pine forest and agroecosystem soils[J]. Applied & Environmental Microbiology, 2005, 71(12): 8411-8418. |
| [24] |
布坎南RE, 等. 伯杰氏细菌鉴定手册[M]. 第8版. 北京: 科学出版社, 1984: 729-797.
|
| [25] |
东秀珠, 蔡妙英. 一般细菌分类手册[M]. 北京: 科学出版社, 2001: 145-155.
|
| [26] |
Follett RF. Soil management concepts and carbon sequestration in cropland soils[J]. Soils and Tillage Research, 2001, 61(1/2): 77-92. |
| [27] |
Smith P. Carbon sequestration in croplands:the potential in Europe and the global context[J]. European Jourmal of Agronomy, 2004, 20(3): 229-236. DOI:10.1016/j.eja.2003.08.002 |
| [28] |
Yang HQ, Xu ZH, Fan MH. Progress in carbon dioxide separation and capture:A review[J]. Journal of Environmental Sciences, 2008, 20: 14-27. DOI:10.1016/S1001-0742(08)60002-9 |
| [29] |
李娟, 赵秉强, 李秀英, 等. 长期不同施肥制度下几种土壤微生物学特征变化[J]. 植物生态学报, 2008, 32(4): 891-899. DOI:10.3773/j.issn.1005-264x.2008.04.018 |
| [30] |
Boyle NR, Morgan JA. Computation of metabolic fluxes and efficiencies for biological carbon dioxide fixation[J]. Metabolic Engineering, 2011, 13(2): 150-158. DOI:10.1016/j.ymben.2011.01.005 |
| [31] |
袁红朝, 秦红灵, 刘守龙, 等. 固碳微生物分子生态学研究[J]. 中国农业科学, 2011, 44(14): 2951-2958. DOI:10.3864/j.issn.0578-1752.2011.14.012 |
| [32] |
徐敏, 刘国祥, 胡征宇. 耐受极高浓度CO2藻类的研究及其在固碳领域的应用[J]. 中国科学院研究生院学报, 2005, 22(5): 529-535. DOI:10.3969/j.issn.1002-1175.2005.05.001 |
| [33] |
Ragsdales SW, Wood HG. Enzymology of the acetyl-CoA pathway of CO2 fixation[J]. Critical Reviews in Biochemistry and Molecular Biology, 1991, 26(3/4): 261-300. |



