2. 北京食品营养与人类健康高精尖创新中心 农业部农业转基因生物安全评价(食用)重点实验室 中国农业大学, 北京 100083
2. Beijing Advanced Innovation Center for Food Nutrition and Human Health, Key Laboratory of Safety Assessment of Genetically Modified Organism(Food Safety)in Ministry of Agriculture, China Agricultural University, Beijing 100083
荧光铜纳米簇生物传感器大约在10年前兴起, 这种传感器的工作原理是以DNA为模板, 以Cu2+为前体、以抗坏血酸等为还原剂, 在DNA片段的大沟结构上生成纳米级铜颗粒, 再通过聚合酶链式扩增反应(Polymerase chain reaction, PCR)实现待测信号的放大, 最后依赖铜纳米簇的荧光特性实现信号输出。荧光铜纳米簇生物传感器初期发展主要以反应原理、荧光铜纳米簇性质、模板多样性等内容为主, 自2012年, 研究开始向荧光铜纳米簇生物传感器的应用方向延伸, 其重要的发现如图 1所示。
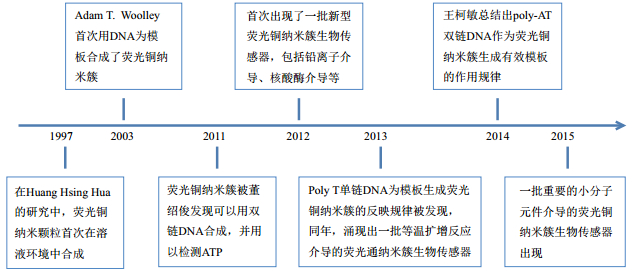
|
| 图 1 荧光铜纳米簇生物传感器重要研究进展时间轴 |
根据靶标性质分化设计, 荧光铜纳米簇生物传感器可以检测生物体内特定酶的活性、某一段基因序列、环境中重金属污染等多种靶标分子, 在医学、生物学、环境科学等多个领域都有较好的发展前景。
目前, 对于荧光铜纳米簇的生成原理、性质等方面的研究框架已经比较完善, 应用于靶物质的信号捕捉、信号放大、信号输出方面也分别演化出了许多设计精巧的方法。这些设计经过优化和扩展, 在未来将可能实际服务于各领域的检测工作。
1 荧光铜纳米簇的基本性质 1.1 荧光铜纳米簇的形成机制荧光铜纳米簇的形成要经历铜离子与DNA分子结合、二价铜离子在DNA相应位点上还原为零价铜原子两个过程。二价铜离子还原形成纳米颗粒是在抗坏血酸的作用下完成的。硫酸铜或硝酸铜溶液中的二价铜离子被抗坏血酸还原为一价铜, 再由一价铜还原为零价金属后, 在DNA上形成纳米级颗粒[1]。CuNCs的荧光原理是由于其具有很少的表面缺陷, 导致非辐射电子弛豫效果较差, 形成荧光性提高。
目前, DNA模板影响荧光铜纳米簇形成的具体原理尚不是完全清楚, 其生成的有效模板一般是采用双链poly(AT)序列和单链polyT序列两种。
根据现有的研究, 一个普遍接受的双链poly(AT)作为模版的荧光铜纳米簇反应原理是, 鸟嘌呤中的N7/O6以及胞嘧啶中的N3都由于在中性条件下的静电作用和氧化作用而形成了Cu2+有较高的亲和性, 导致G-C对Cu2+具有较强的配位络合作用从而不利于Cu2+的还原反应, 所以A-T碱基互补配对可以增强Cu2+的还原作用[2]。当铜离子被还原为铜原子后, 这些铜原子会在dsDNA的大沟中形成荧光铜纳米簇。
而对于单链polyT序列, 推测polyT介导CuNCs的形成是由于胸腺嘧啶和Cu2+之间的结合相互作用, 被胸腺嘧啶复合的Cu2+沿着poly T模板被抗坏血酸还原成CuO。而polyT序列只支持单链形态作为模版的原因, 推断是由于dsDNA和Cu原子之间的强烈相互作用, 导致dsDNA几何结构使生成的CuNCs发生了光学变化。
由于DNA分子可以通过人为编码碱基序列的方式杂交形成线性或立体的复杂结构, 所以用Cu2+在DNA分子上沉降覆盖, 形成涂膜, 就可实现利用DNA二级结构而诱导荧光同纳米簇的可控生成。
1.2 基于核酸生成的荧光铜纳米簇的作用规律要得到生成不同金属纳米粒子的模板, 需要从大量随机序列DNA片段当中分别筛选。对于金属铜而言, 并非所有随机的DNA序列都可以成为荧光铜纳米簇的模板, 能有效生成铜纳米簇的模板区段主要有polyT单链序列和poly(AT)双链序列两种。模板的性质对产生铜纳米簇的效果起着很大影响, 二者之间的大致规律可以描述为[3-4]:(1)模板区段越长, 产生铜纳米荧光越强; 但当CuNCs达到一定尺寸后, 形成的静电屏障作用太大, 会阻止CuNCs的继续形成; (2)模板区段聚合度越高, 产生铜纳米荧光越强。
1.3 荧光铜纳米簇的表征荧光铜纳米簇可以用荧光分光光度计来测量它的荧光特性。其激发光在340 nm, 最大发射光的波长与生成铜纳米簇的模板序列以及其他实验条件有关, 通常位于500-660 nm。通过高分辨率透射电子显微镜(Transmission electron microscope, TEM), 可以观察荧光铜纳米簇的大小、形状等表征, 探究反应条件和荧光铜纳米簇性质之间的关系; 也可研究其在DNA模板上的形成位置和特点, 了解荧光铜纳米簇形成机制。
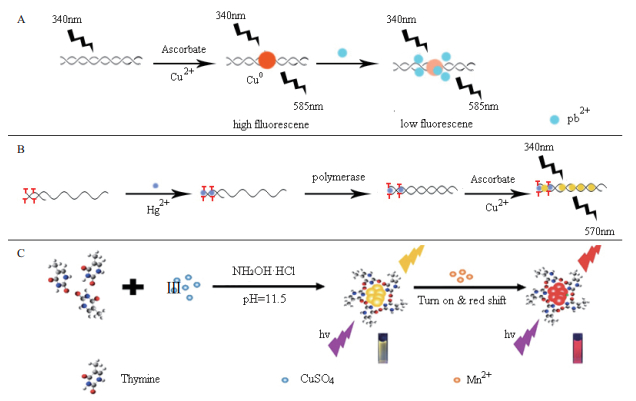
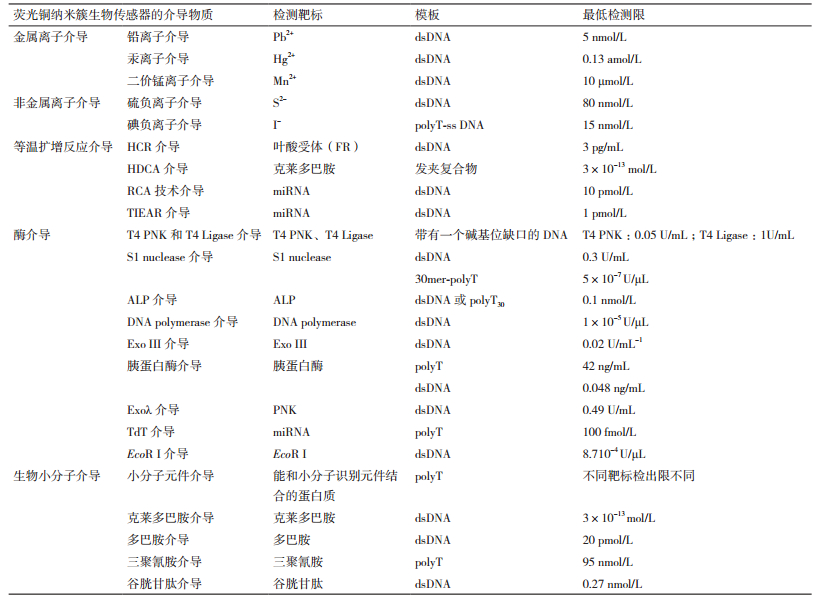
2 基于不同物质与核酸作用方式的荧光铜纳米簇生物传感器 2.1 金属离子介导的荧光铜纳米簇生物传感器 2.1.1 铅离子介导荧光铜纳米簇生物传感器Chen等[5]在研究铅离子检测技术的过程中, 发现铅离子通过与荧光铜纳米簇形成的中间形态一价铜反应, 可以导致二价铜离子无法正常转化为零价铜, 从而淬灭dsDNA-CuNCs的荧光, 反应过程, 如图 2-A所示。通过测定铜纳米簇荧光的减弱程度, 即可反映体系中铅离子的浓度, 检出限为5 nmol/L。
2.1.2 汞离子介导的荧光铜纳米簇生物传感器Qing等[6]利用Hg2+离子与胸腺嘧啶络合形成T-Hg2+-T错配碱基对的性质, 设计出DNA-CuNCs为探针检测Hg2+的方法。如图 2-B所示, 对引物进行特殊设计, 使其3'端与母链形成T-T错配, 则错配的末端无法在DNA聚合酶的作用下正常延伸生成互补链, 无法得到荧光铜纳米粒子的模板dsDNA, 不产生荧光。但在Hg2+存在时, T-Hg2+-T错配碱基能够允许DNA聚合酶催化互补链的形成, 从而得到dsDNA, 随后在CuSO4和抗坏血酸的反应下得到荧光铜纳米簇, 检测到荧光。此外, 利用这个原理, 也可检测其他与Hg2+特异结合的分子, 如半胱氨酸、谷胱甘肽和同型半胱氨酸, 它们与汞离子结合后, 破坏T-Hg2+-T碱基配对, 使通过汞离子结合的两条单链polyT-ssDNA分子形成的dsDNA解离, 游离出polyT序列, 发生荧光铜纳米簇生成反应, 检测到荧光信号[7]。
2.1.3 二价锰离子介导的荧光铜纳米簇生物传感器Mn2+可以使CuNCs的最大发射波长发生红移。如图 2-C所示, Han等[8]据此设计的传感器利用单个碱基T生成CuNCs, 发射黄色荧光, Mn2+存在时, 变色为红色荧光。此方法对Mn2+的检出限可达10 μmol/L。
三种金属离子介导的生物传感器各有优势, 而且原理都比较简单, 操作简便, 不需耗费较长的时间。其中Hg2+介导的生物传感器由于应用较广泛, 在近几年的研究中被较频繁地应用。Mn2+介导的生物传感器则可以实现可视化检测, 也有其独特的优势。
2.2 非金属介导的荧光铜纳米簇生物传感器许多非金属可以通过与荧光铜纳米簇的反应原料Cu2+或CuNCs本身发生反应, 淬灭其荧光。例如, Liu等[9]利用图 3所示的S2-和CuNCs结合后淬灭荧光的性质, 发展出以dsDNA-CuNCs作为探针检测环境和水体中S2-含量的方法。荧光减弱的程度与检体中S2-的含量程正相关。检测可在5 min内完成, 检出限80 nmol/L。又如, CuNCs作为纳米级颗粒具有很大的表面积, 并有双电层, 能够吸引I-, 在反应体系中过量Cu2+的存在下, 发生反应Cu2++Cu+2I- →2CuI。Chen等[10]利用I-瞬时淬灭CuNCs荧光的性质, 发展出一个以polyT-ss DNA分子为模板的荧光铜纳米簇生物传感器以检测I-的含量, 检出限可达15 nmol/L。此外, 曲酸(Kojic acid)与Cu2+之间存在较强结合。据此, 可以利用其阻碍CuNCs形成的性质, 以荧光信号消失作为曲酸存在的检测标志。

|
| 图 3 非金属介导的荧光铜纳米簇生物传感器 |
非金属离子介导的生物传感器原理简单, 检出限低, 可以实现精准检测, 但由于其本身原理的局限性, 应用广泛性不是很强, 想要将其延伸至其他物质的检测, 仍需更加精巧和周密的设计。
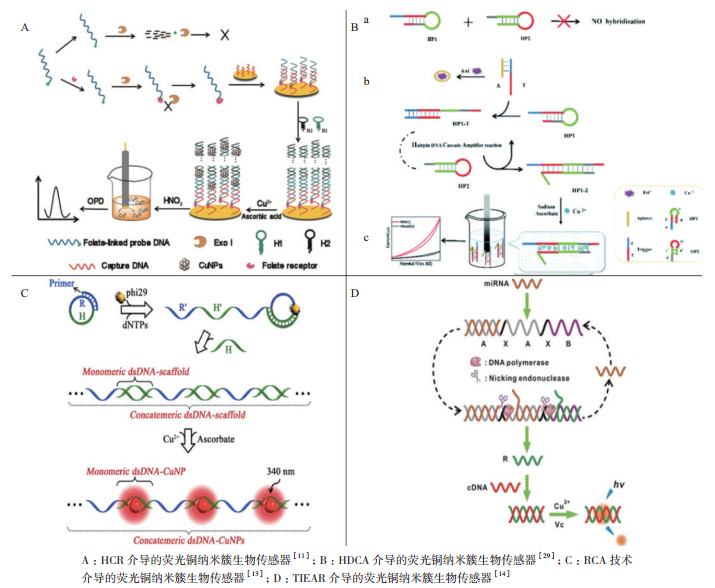
2.3 基于等温扩增反应的荧光铜纳米簇生物传感器 2.3.1 HCR(Hybridization chain reaction杂交链式反应)介导的荧光铜纳米簇生物传感器HCR的基本过程主要包括captureDNA的固定、探针DNA与captureDNA部分杂交结合、探针DNA粘性末端触发HCR反应的开始, 并形成一个由发夹结构H1、H2共存而成的dsDNA, 以及dsDNA聚合物作为模板生成CuNCs的过程。
Zhao等[11]设计了一个复杂机理的HCR触发反应体系, 如图 4-A所示。此HCR介导的生物传感器是以叶酸受体作为靶标, 并通过电化学输出信号工作的。在单链探针DNA 3'端修饰一个叶酸分子。在叶酸受体FR的保护下, DNA免受Exo Ⅰ的水解, 于是与电极上的captureDNA部分杂交结合固定。然后单链探针5'端再通过触发HCR反应结合发夹H1、H2, 在此电极表面形成的dsDNA并生成CuNCs。之后CuNCs酸水解, 解离出的铜离子催化OPD氧化成DAP, 释放电化学信号。Zhao等通过对电化学信号的检测, 证实了由探针DNA触发的HCR反应是信号变化的关键。
Song等[12]利用HCR, 将captureDNA通过生物素-链霉亲和素的相互作用结合到用链霉亲和素修饰的磁柱上, 然后用靶标DNA触发HCR反应并生成CuNCs, 最后通过荧光传感法输出信号。这种方法能够有效放大待检DNA分子的信号, 并可以应用于单核苷酸错配的检验。
2.3.2 发夹-DNA联级放大系统(Hairpin-DNA cascade amplification, HDCA)介导的荧光铜纳米簇生物传感器HDCA系统也是一种信号放大系统, 主要利用DNA的递推链置换的原理实现。如图 4-B所示, 少量靶标可以将HDCA系统的启动因子trigger从复合物中释放出来, 解放的trigger与其中一个发夹结构部分区域结合并逐渐引发连置换, 杂交成为一个中间体。中间体又可以与另一个发夹结构链置换并结合, 形成发夹复合物并将tigger从中间体中置换下来, 引发另一轮链置换, 由此实现一个单一trigger分子触发的多轮HDCA循环。其中, 发夹结构之间的结合形式可能成为某种信号的输出形式, 如设计发夹复合物作为CuNCs的模板, 以荧光信号输出。
2.3.3 滚环复制(Rolling circle replication, RCA)技术介导的荧光铜纳米簇生物传感器Xu等[13]为解决用普通单链模板生成荧光铜纳米簇时稳定性不好、荧光弱的问题, 引入RCA复制模式, 如图 4-C所示。RCA是一种等温复制技术, 应用于荧光铜纳米簇生物传感器时, 将模板设计为包含R、H两个功能区的环状单链DNA分子。其中R区包括引物识别、结合序列; H区是杂交区域, 其序列与互补链形成dsDNA后可以作为CuNCs的生成模板。引物可连续复制环状DNA模板, 形成一个由许多R、H短片段交替首尾串联而成的长链ssDNA分子。随后H区段全部与互补链杂交, 并参与后续CuNCs的生成反应。由此可实现几千个dsDNA单元的串联, 显著提高荧光水平, 从而为开发基于CuNCs的、针对可直接或间接引发RCR的各种靶标的检测方法提供机会。
2.3.4 靶触发等温指数扩增反应(Target- triggered isothermal exponential amplification reaction, TIEAR)介导的荧光铜纳米簇生物传感器TIEAR是一种等温扩增系统, 它以具有5个区域(AXAXB)的单分子DNA寡聚体作为扩增模板, 聚合酶和切口内切核酸酶作为催化剂, 并且使用miRNA靶标作为触发因子。两个重复A区是靶T的结合序列。两个重复X区是切口内切核酸酶识别位点。B区是荧光铜纳米簇合成区域。如图 4-D所示, Wang等[14]据此设计的荧光铜纳米簇生物传感器可以检测在靶标miRNA。靶标出现后, TIEAR被miRNAs与区域A的结合启动, 然后在polymerase/dNTPs环境下双链形成并延伸, 形成一个稳定的含有两个内切酶识别位点的dsDNA。第一个位点的切割导致了第二轮复制循环的开始, 而第二个位点的切割使大量reporterR合成, R与cDNA结合成复合物, 经过二价铜离子的还原生产荧光性的铜纳米颗粒, 由此实现扩增循环和信号放大。
用等温扩增反应代替传统的PCR, 可以大大提升信号放大的倍数以降低检出限, 而且免去PCR的变温过程, 对于体系中含有对温度敏感的靶标或修饰分子, 此方法有较强的利用价值。尤其是HCR技术, 对其中的探针可做不同修饰, 就可以实现针对多种靶标对检测, 应用性非常好, 在许多研究中都发挥了重要作用。但是等温扩增反应的原理也相对较为复杂, 要求设计和操作的相对精准。
2.4 核酸酶介导的荧光铜纳米簇生物传感器 2.4.1 S1核酸外切酶(S1 nuclease)介导的荧光铜纳米簇生物传感器利用随机序列的双链DNA可以生成荧光铜纳米簇而单链DNA无法生成的简单原理, 体系中nuclease的存在使母链DNA分解成小片段, 无法与互补链杂交为荧光铜纳米簇的双链模板, 即无法检测到荧光[15]检出限0.3 U/ mL。也可以利用30mer-polyT作为荧光铜纳米簇模模板, 当nuclease将模板水解为小片段后, 也无法检测到荧光, 实现将nuclease的存在转化为荧光消失信号。检出限5 × 10-7 units/μL。
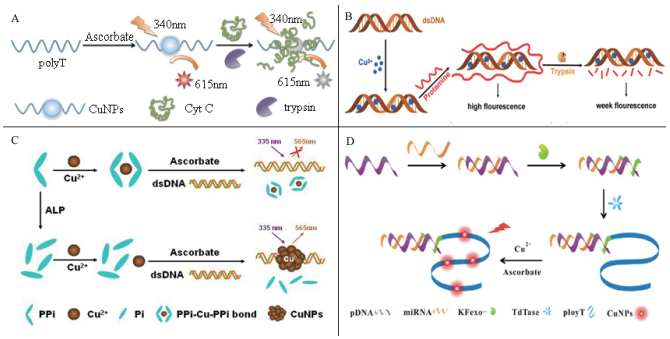
2.4.2 外切核酸酶Ⅲ(Exonuclease, Exo Ⅲ)介导的荧光铜纳米簇生物传感器Zang等[16]利用Exo Ⅲ从3'-5'切割DNA的特性, 用荧光铜纳米簇生物传感器定量检测Exo Ⅲ。当检测体系中存在Exo Ⅲ时, 作为模板的dsDNA被酶解成小分子, 阻止了荧光铜纳米的产生。此方法检出限可达0.02 U/mL(图 5-A)。
λ外切酶可逐步切去双链DNA 5'单核苷酸, 沿5'-3'方向催化双链DNA 5'端单核苷酸的移除。但Exoλ的作用对于5'端羟基作用无效, 只能催化5'端带有磷酸基团的核苷酸移除。
Zhang等[17]利用Exoλ的性质, 设计图 5-B所示的Exoλ介导的基于荧光铜纳米簇的生物传感器, 以检测靶标PNK。PNK的作用是将5'-OH转化为5'-P。双链DNA既作为荧光铜纳米簇的生成模板, 又作为酶的作用底物。如果无PNK存在, 则Exoλ无法消化5'端羟基化的DNA分子, 在dsDNA上可正常形成荧光铜纳米簇; 当存在PNK将dsDNA模板磷酸化后, Exoλ即可消化dsDNA模板, 由于缺少dsDNA模板而无法形成荧光铜纳米簇, 因此不能检测到明显的荧光。
2.4.4 EcoR I(限制性内切酶)介导的荧光铜纳米簇生物传感器限制性核酸内切酶是可以识别特定的核苷酸序列, 并在每条链中特定部位的两个核苷酸之间的磷酸二酯键进行切割的一类酶。在EcoRI存在下, 双链DNA被切割, 由于缺少双链DNA模板, Cu2+和抗坏血酸不能形成荧光CuNCs。如果存在限制性内切酶抑制剂, 阻碍其水解DNA的作用, 则能够检测到荧光。
Zhao等[18]利用此原理设计了如图 5-C所示的荧光铜纳米簇生物传感器, 并检验了其工作性能。其中, 他们用到的限制性内切酶抑制剂是α4肽。抑制剂是各种抗微生物和抗病毒药物的潜在候选物, 因此, EcoR I介导的荧光铜纳米簇生物传感器也可以应用于检测其抑制剂, 在医学上有较强的应用性。
2.5 复合酶介导的荧光铜纳米簇生物传感器 2.5.1 T4 PNK(T4 Polynucleotide kinas:T4多核苷酸激酶)和T4 Ligase(T4连接酶)介导的荧光铜纳米簇生物传感器用带有一个碱基位缺口的DNA为模板, 无法产生荧光铜纳米簇, 此缺陷模板在T4 PNK的作用下使缺口处5'-OH磷酸化, 并在T4 Ligase的作用下连接缺口, 从而修复缺裂的DNA, 使之成为完整的双链模板, 能正常生成荧光铜纳米簇[19], 其原理如图 6-A所示。两种酶也可分别介导荧光铜纳米簇生物传感器, 利用其原理也可以实现两种酶的抑制剂的测定。
2.5.2 双链特异性核酸酶(Duplex-specific nuclease, DSN)和末端脱氧核苷酸转移酶(Terminal deoxynuc-leotidyl transferase, TdT)介导的荧光铜纳米簇生物传感器DSN是一种能高效识别并酶切完全互补配对的DNA双链或者DNA/RNA杂交双链中的DNA链, 而对单链DNA和单/双链RNA几乎没有作用的核酸。TdT是一种无需模板的DNA聚合酶, 催化脱氧核苷酸结合到DNA分子的3'羟基端, 但对于磷酸化的末端则没有活性。Xu等[20]利用DSN和TdT两种酶设计的荧光铜纳米簇生物传感器检测miRNA的工作原理如图 6-B所示, 3'端磷酸化的的小型单链随机序列DNA探针既无法被DSN酶识别, 又无法被TdT识别, 所以在抗坏血酸和Cu2+的作用下无法生成荧光铜纳米簇显荧光。但当靶标miRNA存在时, 探针的3'端磷酸基团与靶标RNA结合, 形成复合物, 成为DSN的底物。复合物被DSN切割后一方面触发了更多靶标-探针的结合反应, 形成循环, 同时也游离出带有3'端羟基的小型DNA片段。此片段在TdT酶的作用下延伸, 形成一段可以作为荧光铜纳米簇生物传感器模版的单链polyT长链, 从而在抗坏血酸和Cu2+的存在下发生反应, 显示荧光。
2.6 其他酶介导的荧光铜纳米簇生物传感器 2.6.1 DNA聚合酶(DNA polymerase)介导的荧光铜纳米簇生物传感器Qing等[21]利用双链DNA可作为荧光铜纳米簇生成模板的性质, 发展了一种以铜纳米的荧光信号作为显色物质输出信号的DNA聚合酶检验方法。体系中含有单链DNA分子、对应的引物和脱氧核糖核苷酸(dNTPs)。当DNA聚合酶存在与检体中时, 互补链合成反应被启动, 形成的双链DNA分子可以作为后续荧光铜纳米的生成模板。
2.6.2 胰蛋白酶介导的荧光铜纳米簇生物传感器Ou等[22]发现, Cytc c淬灭CuNCs荧光的原理除了过去已经认识到的与CuNCs之间电子转化的原理, 还可以是因为其被胰蛋白酶水解而暴露出了游离的半胱氨酸残基, 残基上的硫原子与荧光铜纳米簇的铜原子通过金属-配体键结合为一个非荧光的络合物, 从而造成荧光信号减弱或消失, 反应原理如图 7-A所示。据此可检测胰蛋白酶, 检出限为42 ng/mL。
Wang等[23]发现, 使用dsDNA作模板生成荧光铜纳米簇时, 加入鱼精蛋白可以形成精蛋白/DNA复合物, 从而增强荧光, 其原理如图 7-B所示。如果加入胰蛋白酶水解精蛋白, 则荧光明显淬灭。用这种方法检测胰蛋白酶, 检出限可达0.048 ng/mL。
2.6.3 碱性磷酸酶(Alkaline phosphatase, ALP)介导的荧光铜纳米簇生物传感器Zhang等[24]受焦磷酸(Pyrophosphoric acid, 简称PPi)与Cu2+具有强亲和力的事实的启发, 推测PPi和Cu2+之间的螯合会导致Cu2+合成CuNCs过程中Cu2+向CuO的转化, 导致低荧光。如图 7-C所示, 经AlP处理后, PPi水解为Pi, 使Cu2+和PPi之间的络合失活, 以dsDNA或polyT30[25]为模板, Cu2+正常被抗坏血酸经Cu+还原为零价铜产生荧光铜纳米簇。因此, 可通过测定荧光强度的恢复和增强反映ALP浓度。检出限可达0.1 nmol/L。
2.6.4 TdT介导的荧光铜纳米簇生物传感器Chi等[26]利用KF外切酶(Klenow fragment exo, KFexo)和TdT, 设计图 7-D所示的以miRNA(Micro ribonu-cleic acid)为靶标的荧光铜纳米生物传感器。首先, 靶标miRNA与探针DNA杂交形成引物(miRNA)-模板复合物。然后, KFexo催化引物延伸, 产生与探针DNA互补的短DNA链。随后TdT直接催化短DNA链的3'-OH延伸, 形成polyT模板。至此, 一个短的miRNA可以被有效地转换成一个长的polyT序列, 它可以作为模板形成荧光铜纳米簇。Chen等[27]利用TdT, 设计以核酸酶为靶标的荧光铜纳米簇生物传感器。该传感器用具有3'端磷酸化的发夹DNA作为底物, 其无法在TdT的作用下延伸出polyT序列, 无法生成CuNCs。但当核酸酶存在时, 发夹DNA被水解为大量单链片段。该片段作为引物, TdT引发的链延伸启动, 形成一长链polyT序列, 作为CuNCs生成的模板, 显示荧光。利用这个生物传感器, 将发夹DNA设计为不同核酸酶的适当底物, 可以检测不同种类的核酸酶。Chen等已用Exo Ⅲ和EcoR V作为靶标验证。
以酶介导的铜纳米核酸生物传感器最基础的应用就是在细胞提取物体系中检测靶标酶的活性, 由于其伤害小, 不引入污染, 未来在医学领域具有很强的应用前景, 可能直接发展为一种体内检测酶活性的无损方法, 这对癌症的提前发现、DNA损害原因鉴别都有较大帮助。此外, TdT介导的纳米核酸生物传感器在近年来应用十分广泛, 利用其直接诱导脱氧核苷酸在模板末端结合延伸的性质可以方便可控地制作荧光铜纳米簇模板, 原理简单, 非常具有突破性。另外, 以酶介导的同纳米核酸生物传感器在工作时, 要注意不同酶工作的条件范围和最适条件, 避免酶失活。
2.7 生物小分子介导的荧光铜纳米簇生物传感器 2.7.1 小分子元件介导的荧光铜纳米簇生物传感器Wang等[28]在实验中以polyT为模板, 在其3'端连接一个小分子识别元件, 此时加入硫酸铜溶液和抗坏血酸进行反应, 模板正常产生荧光铜纳米簇。如图 8-A所示, 若体系中没有和小分子识别元件结合的蛋白质, 则在Exo I酶的作用下, polyT模板水解, 荧光消失。若体系中存在这种结合蛋白, 就会形成蛋白质/小分子-polyT的杂合结构, 由于蛋白质的空间位阻作用, 阻碍了Exo I酶靠近模板, 保护polyT模板不被水解, 铜纳米粒子正常产生, 可以检测到荧光。
Sheng等[29]以克莱多巴胺为测定靶标, 巧妙地设计了一个由RAC介导的循环扩增-信号放大系统(图 4-B), 其中包括一个A-T复合物, 它是由一个能触发HDCA循环系统的trigger序列和一个能与靶标克莱多巴胺结合以捕捉靶标信号的适体A组成的。
两个DNA发夹结构HP1和HP2最初不互相杂交, 但在加入靶标RAC后, 适体(A)和靶标RAC之间的特异性结合导致A-T复合物的解离。随后, HP1的暴露的突出结构a和Ttigger的f区域杂交逐渐引发链置换, 从而通过结构域杂交(a, b和f, g)产生HP1-T中间体。HP1-T具有暴露的ssDNA结构域d, 能够与HP2中的结构域d *杂交。因此, d和d *杂交后, 序列b将发生分支迁移并置换T序列(g)形成HP1-2双链体(bcd和b * c * d *), T被HP1置换下来后, HP1和HP2的进一步杂交可实现下一轮催化循环。poly(AT)序列设计在HP1-HP2的结构域b c d和b * c * d *上。在Cu2+和抗坏血酸钠的存在下, 在HP1-HP2的dsDNA结构上可以形成CuNP, 最后以电化学信号的输出形式来反映检体中克莱多巴胺的含量。
RAC靶标可以在重复循环中催化多个HP1-HP2双链体的再生, 从而实现信号的循环放大。Sheng等已用加标动物尿样来检验HDCA系统对靶标RAC的检测效果, 检出限低至0.3 pmol/L。
2.7.3 多巴胺介导的荧光铜纳米簇生物传感器如图 8-B所示, Wang等[30]利用多巴胺与CuNCs之间存在光诱导的电子转移从而淬灭荧光的作用, 使用随机序列双链DNA分子作为模板发展出多巴胺的检测方法。检出限可达20 pmol/L。
2.7.4 三聚氰胺介导的荧光铜纳米簇生物传感器Zhu等[31]发现三聚氰胺可以和胸腺嘧啶通过氢键稳定结合, 由此他们利用单链polyT序列作为模板生成荧光铜纳米簇, 如图 8-C所示, 如果检测体系中存在三聚氰胺, 则可以将两条单链polyT序列用氢键结合成双链, 此时荧光成倍增长。检出限可达95 nmol/L。
2.7.5 谷胱甘肽(Glutathione, GSH)介导的荧光铜纳米簇生物传感器谷胱甘肽和Cu2+之间有强结合作用。谷胱甘肽的存在会导致Cu2+无法与dsDNA模版结合, 从而无法生成CuNCs。Wang等[32]基于谷胱甘肽介导的荧光铜纳米生物传感器, 并用电化学输出信号检测谷胱甘肽的含量。如图 8-D所示, 作为模板的dsDNA被通过金-硫化学键固定在金电极上, 如果有CuNCs生成, 则会引发DPV显著信号响应。当谷胱甘肽存在时, 谷胱甘肽-Cu2+复合物形成, 导致能够到达电极上的dsDNA的Cu2+数量急剧下降, 电化学信号随之减弱。
小分子介导的荧光铜纳米簇生物传感器一般利用小分子与反应体系中模板或产物之间的特异性反应工作, 针对小分子本身为靶标的检测具有灵敏度极高(可达pmol/L级)、响应迅速、定量准确的特点, 但由于工作原理是利用二者之间的特异性反应, 所以难以应用到其他靶标上。
2.8 基于电化学信号的荧光铜纳米簇生物传感器由于CuNCs在酸性环境下有水解的特性, 可释放出铜离子, 能通过电信号输出并测量。其中, 由于大部分检体中待检物质含量均不高, 无法保证每个检测体系最终都能生成足够的CuNCs, 释放足够的铜离子并直接引发可观察到的电信号变化。因此, 荧光铜纳米簇介导的生物传感器用电化学信号表达检体含量时, 为了保证少量的检体生成的CuNCs可以引发程度足够大的电化学信号变化, 我们一般不以酸水解释放的铜离子直接作为电极检测的目标, 而是将铜离子作为催化剂, 催化某一种可以较剧烈引发电化学信号变化的物质(如将铜离子作为OPD氧化成DAP的催化剂), 再测定生成物的电信号, 并可采用等温扩增反应, 以此实现信号的逐级放大, 提高传感器的灵敏度。
目前基于CuNCs酸水解的电化学信号检测, 主要包括电化学阻抗信号谱(Electrochemical impedance signal spectrum, EIS)法、循环伏安(Cyclic voltammetry, CV)法、差分脉冲伏安(Differential pulse volt-ampere, DPV)法等。
2.9 基于荧光信号的荧光铜纳米簇生物传感器荧光信号是荧光铜纳米簇生物传感器最常用、最传统的信号输出方式。CuNCs的生成可实现在室温下、15 min内完成, 直接以反应体系中的溶液为样品, 通过荧光分光光度计即可测定CuNCs的荧光吸收、发射波长及荧光强度, 具有反应简易、定量方便、响应迅速、操作简单的优势。
2.10 可视化的荧光铜纳米簇生物传感器通过紫外灯或紫外透照器可以直接观测CuNCs体系的荧光特性, 适于定性地检测靶标物质, 可视化检测可以粗略地比对荧光相对强弱, 并且在一些通过靶标使荧光发生颜色变化来实现检测工作的传感器上有独特的应用, 近年来的研究也比较多。可视化操作由于具有裸眼观测、操作方便的优势, 也有较好的实际应用价值, 但在研究中, 由于无法精准定量, 只能作为辅助手段检验荧光铜纳米簇生物传感器工作效果。
3 不同物质介导的荧光铜纳米簇生物传感器工作性能对比现将各生物传感器的工作性能总结如表 1, 可以看出, 等温扩增反应介导的荧光铜纳米簇生物传感器由于具有很强的信号放大功能, 故而明显较其他传感器灵敏, 检出限至少可达pmol/L级。此外, 小分子元件介导的生物传感器也有较为灵敏的检测效果。
由于形成荧光铜纳米簇的反应所需时间短、对人体和环境毒性小、无需使用特殊标记的配体或复杂的配体设计, 故用荧光铜纳米簇作为检测物质的输出信号, 是一种非常经济、安全的方法, 满足环保和便捷检验的发展方向。尤其是对于医学方面细胞内跟踪和体内检测、定制药物的设计等方面有很好的应用前景。
4.1 氨基酸及蛋白质检测 4.1.1 半胱氨酸半胱氨酸及其他含硫原子氨基酸可以通过与铜原子络合而淬灭荧光铜纳米簇的荧光。通过这个原理可以检测这些含硫氨基酸以及其他能够产生含硫氨基酸的物质, 由于这类物质非常多, 如细胞色素C等, 所以据此设计的生物传感器应用十分广泛。此外, 半胱氨酸可以与汞离子结合, 通过由汞离子介导的荧光铜纳米簇生物传感器检测。
4.1.2 端粒配体G-四链体是在人体端粒DNA中形成的、可抑制端粒酶活性的结构。其配体是一种蛋白质。因此, 检测G-四联体的配体在治疗癌症方面具有潜在的应用价值。
Yang等[33]据此设计出的荧光铜纳米簇生物传感器可以检测端粒配体的存在。如图 9, 在不存在配体的情况下, 人端粒DNA(GDNA)与其互补DNA(cDNA)杂交形成双链DNA(dsDNA), 其可以作为形成DNA-CuNP的有效模板, 导致高荧光的发生。在配体存在下, GDNA折叠成G-四联体。单链cDNA不支持DNA-CuNP的形成, 导致低荧光的发生。因此, 可以通过监测DNA-CuNCs的荧光变化来筛选端粒结合配体。

|
| 图 9 荧光铜纳米簇生物传感器检测端粒配体 |
利用荧光铜纳米簇作为输出信号检验酶的原理十分多样。各种酶系可能参与DNA分子的合成或水解, 从而导致荧光产生或淬灭; 可能参与催化降解或合成铜纳米颗粒的络合物, 从而导致荧光产生或淬灭。如前文所提到的多种内切酶、聚合酶、核酸酶、磷酸酶等都可通过这些原理进行检测。在DNA的扩增、荧光铜纳米簇产生的过程中, 有很多物质(金属、非金属离子、生物小分子等)均可介导, 凡是能与这些物质发生反应的酶, 理论上都可以作为荧光铜纳米簇的检测靶标, 如各种蛋白酶、水解酶等。
4.3 核酸分子的检测 4.3.1 识别DNA含有高效生产荧光铜纳米簇的DNA序列(如poly-AT双链序列)经扩增后作为荧光铜纳米簇的反应模板, 产生出有荧光性的铜纳米颗粒, 将自身存在的信号转化为荧光信号或电信号输出, 可以被直接检测到。无法高效产生荧光铜纳米簇的DNA序列通过改造其引物, 使扩增产物中引物序列高效产生荧光铜纳米簇, 也可以实现检测。
4.3.2 识别错配如果荧光铜纳米簇的模板发生错配突变, 则生成的铜纳米簇荧光性质受到突变位点、错配种类的影响。通过测定铜纳米簇荧光强度, 可以区分完整或变异序列, 并识别变异类型。这种技术广泛应用于单核苷酸错配的检测。
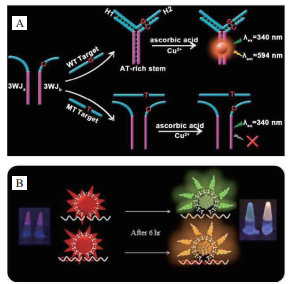
还可以利用错配设计如图 10-A所示的特殊DNA结构, 如Sun等[34]设计的3WJ结构检测碱基错配时, 两条富含互补的连续AT序列的探针可以同时于第三条靶DNA形成一个Y字形结构, 作为CuNCs的生成模板。但当靶DNA存在突变时, 就会与探针DNA形成单核苷酸错配并阻碍CuNCs的形成, 荧光信号消失。
4.3.3 识别RNAmiRNAs是内源性的小型非编码RNA分子。以它作为引物扩增一段带有荧光铜纳米簇生成模板的DNA分子, 可以用铜纳米的荧光性作为输出信号反映RNA的存在。靶标信号可以通过等温扩增反应放大。另外, Borghei等[35]以polyT为模板, 如图 10-B所示设计生物传感器。在没有靶miRNA-155的情况下DNA-CuNCs的荧光在510 nm处有最大发射, 荧光为绿色, 在靶miRNA-155存在的情况下, 发生60 nm的红移, 荧光呈橙色, 创新性地以颜色变化作为靶标存在的检测标志。
4.4 金属离子的检测利用铅离子淬灭dsDNA介导的铜纳米簇荧光的能力, 可检测水体、人体尿样中的铅离子含量, 并应用于医学、环境科学等方面的其他样品检测。检出限为5 nmol/L。此外, 荧光铜纳米簇生物传感器最直接的应用就是检测铜离子, 荧光铜纳米簇的生成所需铜离子浓度很低, 目前较为成功的是Qing等[36]发展的polyT40检测体系, 可实现在1 min内、检出限为5.6 μmol/L的快速检测。利用Hg2+离子与胸腺嘧啶络合形成T-Hg2+-T错配碱基对的性质、Mn2+离子使荧光铜纳米簇的荧光发生红移的性质, 均可通过荧光铜纳米簇生物传感器检测, 具体原理不做重复说明。
4.5 化学基团、化学分子的检测荧光铜纳米簇在化学基团方面的应用十分广泛。这些化学分子在荧光铜纳米簇的形成过程中可以影响多个环节。有些化学分子本身对模板DNA分子或CuNCs分子产生破坏, 淬灭CuNCs的荧光; 有些则可以将自身的配体修饰到模板DNA分子上, 并利用其与配体的结合, 使DNA分子的性质发生改变, 如破坏其特定结构或使酶的其的作用失活, 从而使荧光信号改变。
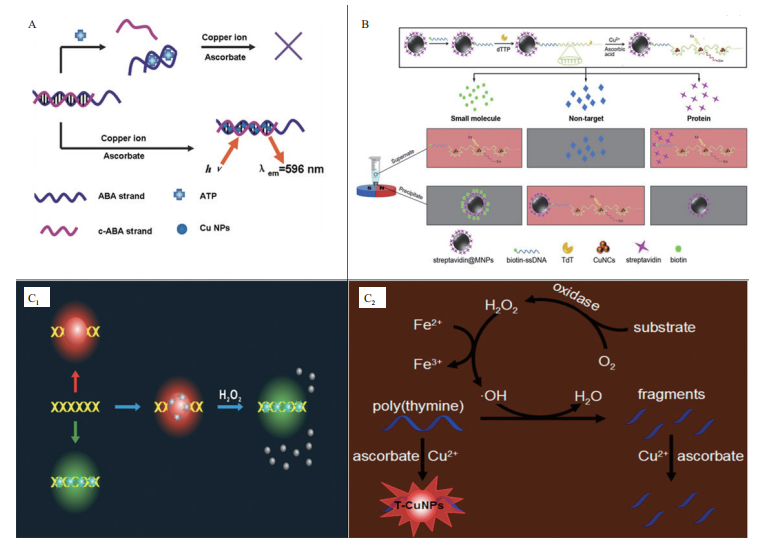
4.5.1 ATP的检测如图 11-A, Zhou等[37]发现ATP分子能够诱导DNA反平行四联体结构的形成, 使得作为荧光铜纳米簇生成模板的双链DNA分子结构发生破坏, 一条链松散脱落而无法成功发生铜纳米簇生成的反应。从而将ATP的存在转变为荧光消失的信号, 设计成一个“turn-off”型的生物传感器, 检出限为0.1 μmol/L。同时, 利用ATP诱导DNA反平行四联体结构形成的性质, Song等[38]令其保护ss-polyT DNA模板免受ExoI的水解, 在抗坏血酸和Cu2+的存在下形成CuNCs, 从而设计出一个“turn-on”型的以ATP为靶标的荧光铜纳米簇生物传感器, 更加清晰地观察到靶标的存在。
4.5.2 生物素等其他小分子除了Song等用HCR等温扩增技术、磁珠修饰技术测定的生物素和链霉亲和素以外, 很多小分子与特定蛋白质都有类似生物素和链霉亲和素之间的结合关系(如叶酸分子和叶酸受体等)。利用这个原理, 可以同时检测许多小分子和相应结合蛋白。将磁珠(一般为磁性Fe3O4纳米颗粒)用结合蛋白涂覆包被, 再用小分子修饰CuNCs模板DNA分子(如ss-polyT序列)。如果体系中没有检体, 则在小分子和结合蛋白的特异相互作用下, CuNCs模板DNA分子将结合到磁珠之上, 并随磁珠通过磁力沉淀, 发生CuNCs生成反应, 赋予沉淀较高的荧光信号。如果体系中存在检体, 则磁珠将选择与体系中游离的检体结合, 通过磁力沉淀将其带入下层, 而模板DNA则留在上清夜, 通过CuNCs生成反应赋予上清夜较高的荧光信号。Cao等[39]已用此原理, 并用生物素-链霉亲和素作为模型物质进行实验, 并引入TdT介导的polyT合成系统制作CuNCs的模板DNA分子, 由此设计的如图 11-B所示的生物传感器在同一系统中生物素的检出限为3.1 nmol/L; 链霉亲和素的检出限为0.47 nmol/L。
4.5.3 H2O2的检测H2O2具有氧化性, 可以生成羟基自由基, 既能够攻击DNA分子, 又可以氧化破坏CuNCs。利用H2O2的性质, 将其淬灭CuNCs荧光性质的能力通过荧光铜纳米簇生物传感器反映出来, 就可以测知其存在并测定其含量。
例如, Mao等[40]利用单链polyT-DNA作为荧光铜纳米粒子生成模版时对模板长度的要求, 设计出如图 11-C中的检测H2O2的办法。在H2O2存在时, 加入二价铁离子会使反应体系产生羟基自由基, 攻击ss-polyT-DNA片段, 使其分解成小片段, 无法作为荧光铜纳米簇的模板, 并以荧光的减弱或消失作为被检物质消失的信号。此方法还可应用于检测其他可被氧化酶催化氧化并产生H2O2的物质, 如葡萄糖、胆碱、胆固醇、黄嘌呤和乳酸等。其中, Mao等已在实验中证实了方法用于检验血清样品中葡萄糖含量的可行性。
Chen等[41]利用H2O2破坏CuNCs的能力, 设计了其基于荧光铜纳米簇生物传感器的检测方法。如图 11-C2所示, dsDNA诱导的CuNCs本身显示红色荧光, 在CuNCs的环境中加入SYBR Green I, 由于SYBR Green I只吸附在CuNCs表面, 而不嵌入dsDNA, 故此时显示CuNCs的红色荧光。但当H2O2存在时, CuNCs解构并被淬灭荧光; 与此同时, SYBR Green I与dsDNA结合的抑制被消除, 显示绿色荧光。由此, 即实现将绿色荧光作为H2O2含量的输出信号。
5 总结与展望首先, 目前对荧光铜纳米簇的了解还只局限在10年的研究基础上。虽然对于荧光铜纳米簇的生成形成了基本认知, 对于它的反应体系也达成了基本的共识, 但是荧光铜纳米簇的反应原理和机制仍有很多细节问题需要继续研究。例如, 在模版方面, 用随机序列、双链polyAT序列和单链polyT序列反应有何异同之处; 在荧光铜纳米簇的形成方面, 荧光铜纳米簇在DNA分子中生成的具体位置、其与DNA分子结合的具体方式; 荧光铜纳米簇反应过程的具体步骤、反应物添加的最佳顺序等都需要更加深入的讨论。
其次, 虽然荧光铜纳米簇生物传感器已经拓展出成百上千种应用, 但是在未来能否将它们发展得应用性、操作性更好, 能否服务于实际生活, 还需要对荧光铜纳米簇本身更加深入的研究, 解决这些细节问题。铜是人体内的一种必需微量元素。在正常范围内对人体有重要作用, 但缺乏或过量都会导致相应的病理改变。所以利用铜元素的体内检测、利用铜元素进行细胞成像是未来医学发展的必然趋势。然而在此之前我们必须继续研究荧光铜纳米簇在体内生成的稳定性, 如反应过程中产物会否被体内核酸酶切割等, 要首先在体外实验中搞清基本的操作可行性。
最后, 荧光铜纳米簇生物传感器在现阶段主要还是采用荧光信号输出, 电化学输出信号法操作性不如荧光法简便; 而可视化信号输出又需要特殊的反应特性, 所以在未来的工作中需要有新传感方法的建立。生物传感器离不开材料学的应用, 今后可能需要荧光铜纳米簇生物传感器与新型纳米材料、复合纳米材料等学科进行交叉, 如近年开展的金纳米簇、银纳米簇等, 探究二者结合是否具备更多优势, 能否创造出新的传感方法。另外, 荧光铜纳米簇可应用的领域虽多, 但是一般一种传感器无法做到多重靶标检测, 在未来的研究工作中, 仍需要筛选出多能性比较好的传感器, 以赋予荧光铜纳米簇生物传感器实际的应用价值。
| [1] |
Rotaru A, Dutta S, Jentzsch E, et al. Selective dsDNA-templated formation of copper nanoparticles in solution[J]. Angewandte Chemie, 2010, 49(33): 5665-5667. DOI:10.1002/anie.v49:33 |
| [2] |
Song QW, Shi Y, He DC, et al. Sequence-dependent dsDNA-templated formation of fluorescent copper nanoparticles[J]. Chemistry, 2015, 21(6): 2417-2422. DOI:10.1002/chem.201405726 |
| [3] |
Qing ZH, He XX, He DM, et al. Poly(thymine)-templated selective formation of fluorescent copper nanoparticles[J]. Angewandte Chemie, 2013, 52(37): 9719-9722. DOI:10.1002/anie.201304631 |
| [4] |
Qing ZH, Qing TP, Mao ZG, et al. dsDNA-templated fluorescent copper nanoparticles:poly(AT-TA)-dependent formation[J]. RSC Advances, 2014, 4(105): 61092-61095. DOI:10.1039/C4RA11551A |
| [5] |
Chen JH, Liu J, Fang ZY, et al. Random dsDNA-templated formation of copper nanoparticles as novel fluorescence probes for label-free lead ions detection[J]. Chem Comm, 2012, 48(7): 1057-1059. DOI:10.1039/C2CC16668B |
| [6] |
Qing ZH, Qing TP, Mao ZG, et al. dsDNA-specific fluorescent copper nanoparticles as a'green'nano-dye for polymerization-mediated biochemical analysis[J]. Chem Comm, 2014, 50(84): 12746-12748. DOI:10.1039/C4CC05101G |
| [7] |
Zhang L, Cai QY, Li J, et al. A label-free method for detecting biothiols based on poly(thymine)-templated copper nanoparticles[J]. Biosensors and Bioelectronics, 2015, 69: 77-82. DOI:10.1016/j.bios.2015.02.012 |
| [8] |
Han BY, Xiang RC, Hou XF, et al. One-step rapid synthesis of single thymine-templated fluorescent copper nanoclusters for "turn on" detection of Mn2+[J]. Analytical Methods, 2017, 9(17): 2590-2595. DOI:10.1039/C7AY00625J |
| [9] |
Liu J, Chen JH, Fang ZY, et al. A simple and sensitive sensor for rapid detection of sulfide anions using DNA-templated copper nanoparticles as fluorescent probes[J]. Analyst, 2012, 137(23): 5502-5505. DOI:10.1039/c2an35885a |
| [10] |
Chen ZZ, Niu YX, Cheng GY, et al. Fast, highly sensitive and selective assay of iodide ions with single-stranded DNA-templated copper nanoparticles as a fluorescent probe for its application in Kunming mice samples[J]. Analyst, 2017, 142(15): 2781-2785. DOI:10.1039/C7AN00595D |
| [11] |
Zhao J, Hu SS, Cao Y, et al. Electrochemical detection of protein based on hybridization chain re-action-assisted formation of copper nanoparticles[J]. Biosensors and Bioelectronics, 2015, 66: 327-331. DOI:10.1016/j.bios.2014.11.039 |
| [12] |
Song CX, Yang XH, Wang KM, et al. label-free and non-enzymatic detection of DNA based on hybridization chain reaction amplification and dsDNA-templated copper[J]. Analytica Chimica Acta, 2014, 827(3): 74-79. |
| [13] |
Xu FZ, Shi H, He XX, et al. Concatemeric dsDNA-templated copper nanoparticles strategy with improved sensitivity and stability based on rolling circle replication and its application in microRNA detection[J]. Anal Chem, 2014, 86(14): 6976-6982. DOI:10.1021/ac500955r |
| [14] |
Wang XP, Yin BC, Ye BC. A novel fluorescence probe of dsDNA-templated copper nanoclusters for quantitative detection of microsRNAs[J]. RSC Advances, 2013, 3(23): 8633-8636. DOI:10.1039/c3ra23296d |
| [15] |
Hu R, Liu YR, Kong RM, et al. Double-strand DNA-templated formation of copper nanoparticles as fluorescent probe for Label free nuclease enzyme detection[J]. Biosensors and Bioelectronics, 2013, 42(4): 31-35. |
| [16] |
Zhang H, Lin ZH, Su XG. label-free detection of exonuclease Ⅲ by using dsDNA-templated copper nanoparticles as fluorescent probe[J]. Talanta, 2015, 131: 59-63. DOI:10.1016/j.talanta.2014.07.065 |
| [17] |
Zhang ll, Zhao JJ, Zhang H, et al. Double strand DNA-templated copper nanoparticle as a novel fluorescence indicator for label-free detection of polynucleotide kinase activity[J]. Biosensors and Bioelectronics, 2013, 44(18): 6-9. |
| [18] |
Zhao HZ, Dong JJ, Zhou FL, et al. One facile fluorescence strategy for sensitive detection of endonuclease activity using DNA-templated copper nanoclusters as signal indicators[J]. Sensors and Actuators B, 2017, 238: 828-833. DOI:10.1016/j.snb.2016.07.083 |
| [19] |
Qing TP, He XX, He DG, et al. Dumbbell DNA-templated CuNCs as a nano-fluorescent probe for detection of enzymes involved in ligase-mediated DNA repair[J]. Biosensors and Bioelectronics, 2017, 94: 456-463. DOI:10.1016/j.bios.2017.03.035 |
| [20] |
Xu FZ, Luo L, Shi H, et al. label-free and sensitive microRNA detection based on a target recycling amplification-integrated superlong poly(thymine)-hosted copper nanoparticle strategy[J]. Analytica Chimica Acta, 2018, 1010: 54-61. DOI:10.1016/j.aca.2018.01.010 |
| [21] |
Qing ZH, Qing TP, Mao ZG, et al. dsDNA-specific fluorescent copper nanoparticles as a'green'nano-dye for polymerization-mediated biochemical analysis[J]. Chem Comm, 2014, 50: 12746-12748. DOI:10.1039/C4CC05101G |
| [22] |
Ou LJ, Li XY, Li LJ, et al. A sensitive assay for trypsin using poly(thymine)-templated copper nanoparticles as fluorescent probes[J]. Analyst, 2015, 140(6): 1871-1875. DOI:10.1039/C4AN01994F |
| [23] |
Wang L, Shi FP, Li Y. An ultra-sensitive and label-free fluorescent probe for trypsin and inhibitor based on DNA hosted Cu nanoclusters[J]. Sensors and Actuators B:Chemical, 2016, 222: 945-951. DOI:10.1016/j.snb.2015.09.024 |
| [24] |
Zhang ll, Zhao JJ, Duan M, et al. Inhibition of dsDNA-templated copper nanoparticles by pyrophosphate as a label-free fluorescent strategy for alkaline phosphatase assay[J]. Anal Chem, 2013, 85(8): 3797-3801. DOI:10.1021/ac4001942 |
| [25] |
Li JY, Si L, Bao JC, et al. Fluorescence regulation of poly(thymine)-templated copper nanoparticles via an enzyme-triggered reaction towards sensitive and selective detection of alkaline phosphatase[J]. Anal Chem, 2017, 89(6): 3681-3686. DOI:10.1021/acs.analchem.6b05112 |
| [26] |
Chi BZ, Liang RP, Qiu WB, et al. Direct fluorescence detection of microRNA based on enzymatically engineered primer extension poly-thymine(EPEPT)reaction using copper nanoparticles as nano-dye[J]. Biosensors and Bioelectronics, 2107, 87: 216-221. |
| [27] |
Chen J, Xu Y, Ji XH, et al. Enzymatic polymerization-based formation of fluorescent copper nanoparticles for the nuclease assay[J]. Sensors and Actuators B, 2017, 239: 262-269. DOI:10.1016/j.snb.2016.08.017 |
| [28] |
Wang HB, Zhang HD, Chen Y, et al. A fluorescent biosensor for protein detection based on poly(thymine)- templated copper nanoparticles and terminal protection of small molecule-linked DNA[J]. Biosensors and Bioelectronics, 2015, 74(2): 581-586. |
| [29] |
Sheng FF, Zhang XJ, Wang GF. Novel ultrasensitive homogeneous electrochemical aptasensor based on dsDNA-templated copper nanoparticles for the detection of ractopamine[J]. Journal of Materials Chemistry B, 2017, 5(1): 53-61. |
| [30] |
Wang HB, Zhang HD, Chen Y, et al. A label-free and ultrasensitive fluorescent sensor for dopamine detection based on double-stranded DNA templated copper manoparticles[J]. Sensors and Actuators B:Chemical, 2015, 220: 146-153. DOI:10.1016/j.snb.2015.05.055 |
| [31] |
Zhu HW, Dai WX, Yu XD, et al. Poly thymine stabilized copper nanoclusters as a fluorescence probe for melamine sensing[J]. Talanta, 2015, 144: 642-647. DOI:10.1016/j.talanta.2015.07.022 |
| [32] |
Wang ZX, Han P, Mao XX, et al. Sensitive detection of glutathione by using DNA-templated copper nanoparticles as electrochemical reporters[J]. Sensors and Actuators B, 2017, 238: 325-330. DOI:10.1016/j.snb.2016.07.078 |
| [33] |
Yang LZ, Wang YJ, Li BX, et al. High-throughput identification of telomere-binding ligands based on the fluorescence regulation of DNA-copper nanoparticles[J]. Biosensors and Bioelectronics, 2017, 87: 915-917. DOI:10.1016/j.bios.2016.09.055 |
| [34] |
Sun FF, You Y, Liu J, et al. DNA three-way junction for differentiation of single nucleotide polymorphisms with fluorescent copper nanoparticles[J]. Chemistry-A European Journal, 2017, 23(29): 6979-6982. DOI:10.1002/chem.201701361 |
| [35] |
Borghei Y, Hosseini M, Ganjali MR, et al. Label-free fluorescent detection of microRNA-155 based on synthesis of hairpin DNA-templated copper nanoclusters by etching(top-down approach)[J]. Sensors and Actuators B, 2017, 248: 133-139. DOI:10.1016/j.snb.2017.03.148 |
| [36] |
Qing ZH, Zhu LX, Yang S, et al. In situ formation of fluorescent copper nanoparticles for ultrafast zero-background Cu2+ detection and its toxicides screening[J]. Biosensors and Bioelectronics, 2016, 78: 471-476. DOI:10.1016/j.bios.2015.11.057 |
| [37] |
Zhou ZX, Du Y, Dong SJ. Double-strand DNA-templated formation of copper nanoparticles as fluorescent probe for label-free aptamer sensor[J]. Anal Chem, 2011, 83(13): 5122-5127. DOI:10.1021/ac200120g |
| [38] |
Song QW, Wang RH, Sun FF, et al. A nuclease-assisted label-free aptasensor for fluorescence turn-on detection of ATP based on the in situ formation of copper nanoparticles[J]. Biosensors and Bioelectronics, 2017, 87: 760-763. DOI:10.1016/j.bios.2016.09.029 |
| [39] |
Cao JP, Wang W, Bo B, et al. A dual-signal strategy for the solid detection of both small molecules and proteins based on magnetic separation and highly fluorescent copper nanoclusters[J]. Biosensors and Bioelectronics, 2017, 90: 534-541. DOI:10.1016/j.bios.2016.10.021 |
| [40] |
Mao ZG, Qing ZH, Qing TP, et al. Poly(thymine)-templated copper nanoparticles as a fluorescent indicator for hydrogen peroxide and oxidase-based biosensing[J]. Anal Chem, 2015, 87(14): 7454-7460. DOI:10.1021/acs.analchem.5b01700 |
| [41] |
Chen J, Ji XH, He ZK. Smart composite reagent composed of double-stranded DNA- templated copper nanoparticle and SYBR green I for hydrogen peroxide related biosensing[J]. Anal Chem, 2017, 89(7): 3988-3995. DOI:10.1021/acs.analchem.6b04484 |