2. 农业部农业转基因生物安全评价(食用)重点实验室,北京 100083
2. Key Laboratory of Safety Assessment of Genetically Modified Organism(Food Safety), Ministry of Agriculture, Beijing 100083
核酸作为重要的生物大分子,在生物生命活动中具有重要的作用。核酸分子除了能够携带遗传信息以外,单链DNA还能自我折叠成复杂的三级结构,成为具有特异性识别能力以及催化活性的功能核酸,其中包括核酸适配体(Aptamer)、脱氧核酶(DNAzyme)等。功能核酸的发现,为生物传感、生物成像提供了广阔的发展空间,已经在包括检测、分离、材料科学、纳米技术、材料合成、体内成像及医疗领域得到了广泛的应用[1-8]。
大多数的脱氧核酶具有金属离子依赖性的特点,只有在特定金属离子存在的情况下才展现出催化活性,使DNA片段在特定位置上发生剪切。第一个脱氧核酶是于1994年被筛选出来,Pb2+存在下能够特异切割RNA[9]。随后其他金属离子依赖性的核酶也逐渐被人们筛选出来,如镉离子脱氧核酶(Cd16 DNAzyme)[10]、铬离子脱氧核酶(Ce13d DNAzyme)[11]、铜离子脱氧核酶(PSCu10 DNAzyme)[12]、铅离子脱氧核酶(GR5 DNAzyme,8-17 DNAzyme)[13]及10-23脱氧核酶等。
10-23脱氧核酶(10-23 DNAzyme,DZ13)是一种能特异结合并切割RNA分子的功能核酸DNA,具有高效的催化降解能力。1997年,Santoro等[14]建立了体外筛选系统,从随机的DNA分子库中筛选出对与Mg2+依赖性的脱氧核酶,由于选自第10轮扩增的第23个克隆,故被称为10-23脱氧核酶。由于DZ13高效识别与切割能力,其介导的生物传感器得到了广泛的应用,主要涉及体内的靶向治疗、生物成像及生物传感等方面。本文将介绍DZ13介导的生物传感器的研究进展,旨在为未来使用10-23脱氧核酶搭建新型快捷生物传感器奠定理论基础。
1 DZ13介绍 1.1 DZ13筛选技术DZ13的筛选技术是指数富集的配基系统进化技术(Systematic evolution of Ligands by exponential enrichment,SELEX)。首先需要建一个包含1013-1016随机序列的DNA库,这些序列中间会嵌入一小段RNA作为剪切位点,位点两边的结合序列为随机区域。通过将这些序列与靶标金属离子一起孵育,一小部分序列会与金属离子结合并且切割RNA位点。切掉的序列通过两次PCR反应获得全长。1995年,Breaker和Joyce[15]想通过建立体外筛选系统筛选出Mg2+特异性的脱氧核酶,在原有的基础上,筛选出能够切割RNA的通用型DZ13,经过实验筛选出了DZ13能够在Mg2+、Mn2+、Zn2+和Pb2+存在进行特异位点切割,而且催化过程能够在细胞内环境下进行,为DZ13在体内的应用奠定了理论基础。
1.2 DZ13催化结构DZ13包括催化核心和侧臂两部分(图 1),催化核心是由15个脱氧核糖核苷酸组成的环状结构,侧臂通过Waston-Crick碱基配的形式与靶标序列进行特异性结合,将环状催化核心固定在底物分子催化位点上。10-23脱氧核酶切割位点为R(嘌呤核糖核苷酸)与Y(嘧啶核糖核苷酸)之间的磷酸二酯键,在与酶链结合成复合结构时,R不进行碱基配对,Y必须与酶链形成碱基配对结构(图 1)。当切割位点是A与U时,接近生理条件下(2 mmol/L MgCl2,150 mmol/L KCl,pH 7.5,37℃)催化效率最高kcat≈0.1 min-1。通过对侧臂的设计,能够实现对含有核糖核苷酸位点的DNA进行特异性识别与切割。
1.3 DZ13活性影响因素DZ13的催化活性受到多方面因素的影响,为了提高催化效率,研究人员从反应条件到对脱氧核酶内部碱基以及机构的改造等方面进行大量的研究。
(1) 反应体系的pH值。在37℃,10 mmol/L Ca2+,3种不同缓冲液(BIS-TRIS丙烷缓冲液、PIPES缓冲液、EPPS缓冲液)的条件下,10-23脱氧核酶催化活性在pH 6.5-8.5之间呈现对数-直线关系,R2达到0.94[16]。(2)二价金属阳离子。是10-23脱氧核酶启动反应的关键因素,起到稳定活性过渡状态以及帮助折叠的作用,活性影响排序为:Mn2+(EPPS) > Pb2+、Mg2+、Ca2+ > Cd2+(Tris) > Sr2+、Ba2+ > > Zn2+、Co2+[16]。(3)底物结合区的长度与碱基组成。底物结合区过长及GC含量越多,切割产物不易脱落,会导致剪切速率的降低。过短底物不容易结合,也会导致剪切效率降低。常用7+7,8+8,9+9等结合长度[17]。(4)缓冲液类型。不同类型的缓冲液会影响二价金属阳离子对10-23脱氧核酶的作用,Mn2+存在的N-(2羟乙基)哌嗪-N-丙磺酸缓冲液(EPPS)相对于其他金属离子能够使10-23脱氧核酶具有较好的剪切活性,但在三羟甲基氨基甲烷(Tris)缓冲液中几乎没有剪切活性。Cd2+在Tris缓冲液中可使10-23脱氧核酶有适度活性,但在EPPS缓冲液中对活性的影响极微[16, 18]。(5)温度。大多数研究显示温度在37℃条件下10-23脱氧核酶可发挥较好的活性。
1.4 DZ13结构改造对酶活性影响Wang等[19]发现用核苷类似物替换到DZ13 A9位置上,能够明显的提高剪切率,并且得出活性影响关系:氨基酸 > 羟基 > 羟基苯甲酸 > 苯基。将LNA(Locked nucleic acid)加入10-23脱氧核酶的结合臂中形成LNAzyme,活性会显著提高[20-22]; 若对脱氧核酶以及结合区域进行硫代修饰,稳定性会显著提高但会使剪切活性大大降低,在酶链两端进行少量硫代修饰可在一定程度上提高稳定性的同时保持大部分的剪切活性[22-23]; 若对3' 末端引物倒置连接的胸腺嘧啶脱氧核苷,或者在酶链两端进行2-甲氧基修饰可以保持酶链具有良好的稳定性,末端修饰同时适当延长结合区域可提高剪切活性[22-24]。
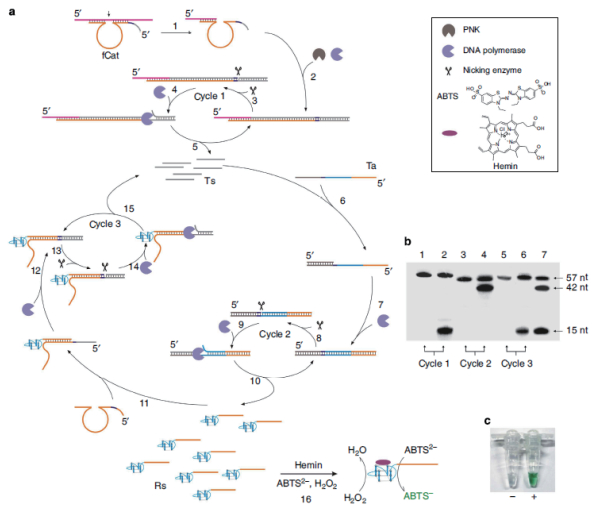
2 DZ13介导的光学传感器 2.1 比色传感器Zhao等[25]报道了利用DZ13建立了一种恒温条件下对RNA进行扩增以及检测的新方法(图 2)。10-23酶链包含两个与靶标RNA结合臂,发生剪切后,5' 端RNA片段会作为引物对10-23酶链进行复制,产生双链DNA序列。由于在10-23酶链的5' 端设计了一段含有切刻内切酶识别位点的片段,被内切酶剪切之后会形成新的黏性末端,通过DNA聚合酶进行链置换反应,通过3个循环可以进行信号放大。产生的单链DNA富含G序列,反应结果可以通过与ABTS2-与双氧水显色的手段达到检测的目的,检测限为1 fmol/L-100 pmol/L。
Carter等[26]利用DZ13修饰的纳米金离子建立DDZ-AuNP传感器检测登革热病毒(Dengue viras,DENV)。10-23酶链的结合臂是与DENV基因组RNA特异性序列的互补序列,能够与靶标RNA特异性结合。整个反应可以分为3个阶段:DDZ识别并剪切DENA RNA基因组; AuNPs活化; AuNPs聚沉检测。当存在靶标RNA时,DDZ特异性结合,在37℃Mg2+存在的情况下进行G149位点的剪切,使AuNPs在NaCl存在情况下减少表面斥力作用产生聚集效果,颜色由红色变成无色。反之,如果没有靶标RNA存在时,则没有颜色的变化呈红色。
2.2 荧光传感器荧光检测法作为常用检测方法,具有操作简单、信号检测方便、原理简单以及反应快等特点[27]。因此,基于DZ13的荧光传感器发展出多种不同类型。
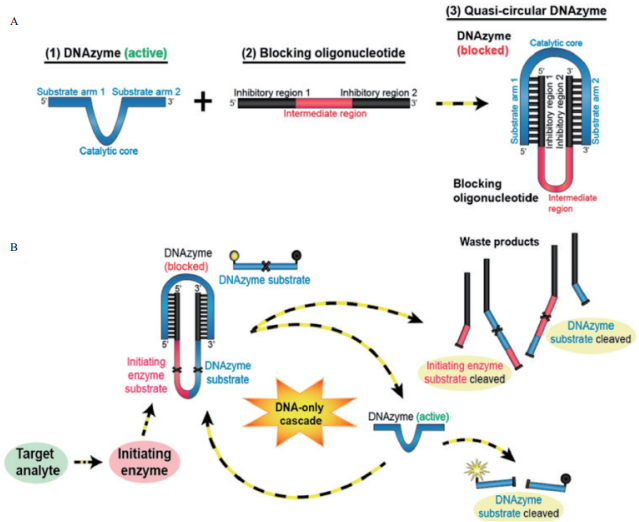
2.2.1 基于荧光探针荧光传感器Bone等[28]设计了一套DNA串联结构(DNA-only cascade,DoC),实现了对包括金属离子(Pb2+)、小分子物质(dATP)以及靶标核酸序列的多重检测(图 3)。DoC结构中,10-23酶链被辅助连封闭,不具有切割活性。辅助链上具有起始反应的酶位点,当溶液中存在靶标物质时,会催化酶对辅助链上的位点进行切割,产生具有活性的10-23酶链,会对溶液中含有荧光基团与淬灭基团的底物链进行切割实现信号的输出。该方法能够识别单碱基突变的核酸链,并且能够短时间内对靶标进行定量检测,该方法检测限能够达到铅离子5 nmol/L,核酸5 pmol/L。
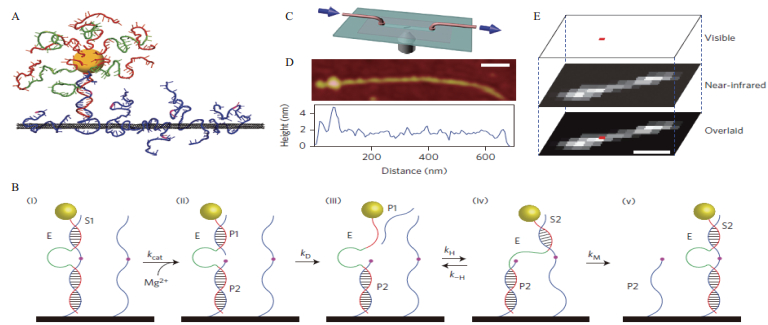
Cha等[29]利用DZ13建立了DZ13介导的DNA马达,能够在碳纳米管内对纳米材料(CdS)进行运输(图 4)。DNA马达移动的能量来源是通过10-23酶链对RNA底物链的切割产生。当10-23酶链与修饰在碳纳米管上的RNA底物链进行结构后,在镁离子存在的条件下,对RNA进行切割,产生切割片段,并随后与酶链脱离,10-23酶链会继续识别下一条RNA底物,完成一个循环。通过不断的重复循环,实现了沿碳纳米管的移动。该方法整个移动过程是可控的,能够随时停止随时开始,通过选择具有荧光性质的“纳米货物”可以进行实时监测。实验环境下,观测到的DNA马达单方向移动能够达到3 μm,速度能够达到1 nm/min。
Fan等[30]建立了DZ13-MnO2纳米传感器,高效的实现了细胞内基因沉默(图 5)。MnO2纳米片吸附上标记有Ce6的脱氧核酶,能够保护酶链不被消化也能有效的进入胞内。当胞内存在谷胱甘肽(Glutathione r-glutamyl cysteingl+glycine,GSH)时,MnO2还原成Mn2+作为激活离子使10-23脱氧核酶具有活性,对靶标RNA进行有效的切割。通过检测荧光实现对10-23脱氧核酶递送过程的监测。
Todd等[31]建立了DzyNA-PCR方法,运用DZ13对核酸序列进行实时荧光检测(图 6)。该方法设计了特殊的酶链引物,其包含靶标特异性序列以及10-23核酶的反义互补序列。PCR扩增过程中,会产生具有活性的10-23核酶扩增产物,再与含有荧光基团淬灭基团的底物结合并且切割产生荧光信号,通过PCR不断进行信号积累实现检测靶标的目的。运用该方法检测质粒DNA能够实现至少6个数量级的扩增(R2=0.992)。涉及到人类基因组作为靶标时,最低能够检测到10个拷贝。
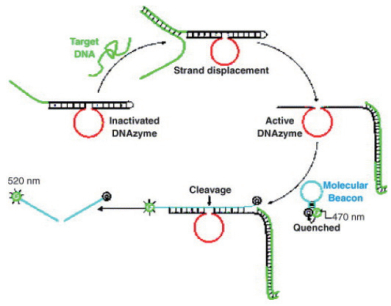
Tian等[32]设计了新型10-23封闭结构,对靶标DNA进行检测(图 7)。该方法设计了长链结构,其中包括10-23酶链核心序列、10-23互补序列和靶标结合序列3个部分,通过自组装的方式,DZ13催化核心与互补序列结合进行封闭。当存在靶标DNA序列时,会与酶链上的互补序列结合,通过链置换反应打开封闭结构,产生活性中心。进一步结合自淬灭的分子信标,并进行位点切割释放荧光信号。对于靶标DNA检测限能够达到10 pmol/L,比之前报道的提高了3个数量级。
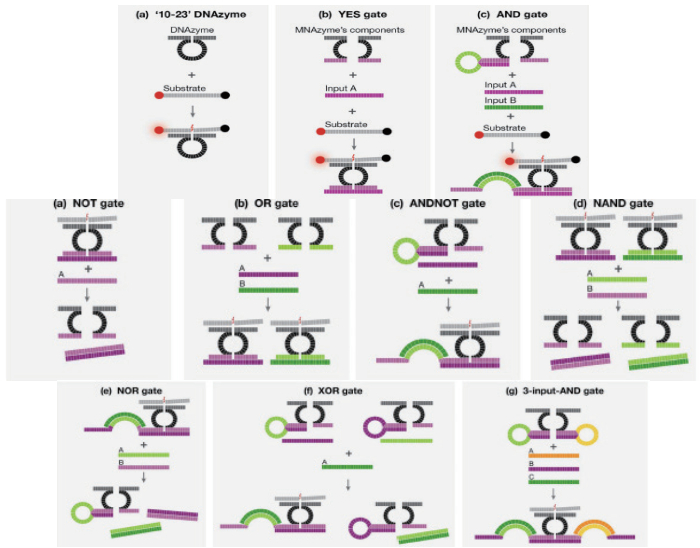
Kahan-Hanum等[33]建立关于DZ13用来检测或诊断miRNA介导mRNA的传感器库(图 8),其中逻辑涉及YES、AND、NOT、OR、NAND、ANDNOT、XOR和NOR等,每一个逻辑门都只有在靶标序列出现的情况下才会发生10-23酶链的切割,产生荧光信号。而且,该方法在细胞溶解物及哺乳动物细胞也得到了验证,为10-23核酶相关的传感器结构设计奠定了大量的理论基础。
|
| 图 8 DZ13介导的生物传感器逻辑门种类 |
Zhao等[25]建立的DZ13温条件下对RNA进行扩增以及检测(图 9)。该方法通过对其中探针的变更,设计出DZ13介导的实时荧光RNA检测技术。通过DZ13、DNA聚合酶和核酸内切酶的作用下,实现RNA的循环方法以及G4序列的富集。通过收集荧光信号值实现靶标RNA实时荧光检测。
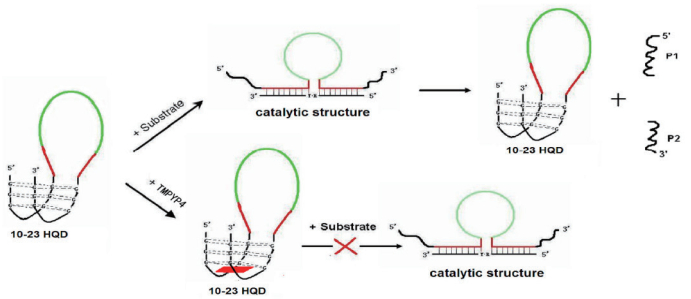
Wu等[34]设计了一种新型包含G4序列的10-23发卡结构,并实现了对该酶活性的控制(图 10)。酶结构中通过加入TMPyP4实现控制,实验表明DZ13以及G4结构的稳定性得到极大的提升,并且TMPyP4的加入降低了10-23酶的催化活性,甚至失去剪切活性。这种结构能够实现对于靶标序列剪切的可控性,在基因治疗方面具有较大潜力。
2.2.3 金纳米材料荧光传感器Yehl等[35]将DZ13修饰在金纳米颗粒的表面形成金纳米酶结构,通过光控作用在胞内实现对mRNA的切割以及相关基因的调控(图 11)。该方法构建的DZ13-Gold结构具有很高的稳定性,对核酸酶有较高的抗性,并且对细胞具有很小的毒性。DZ13-Gold结构进入细胞内后,通过532 nm脉冲激光器的照射,使得10-23核酶与金纳米颗粒分离到细胞液中,对靶标进行特意性识别并剪切。整个过程可以通过荧光显微镜进行观察。
3 DZ13介导的电化学传感器近几十年来,电化学传感器的研究取得的了巨大的进步。相比光学传感器,电化学传感器在检测极限以及准确性上有很大的优势[36],并且在设备小型化以及低背景信号方面有更多的发展空间[37]。
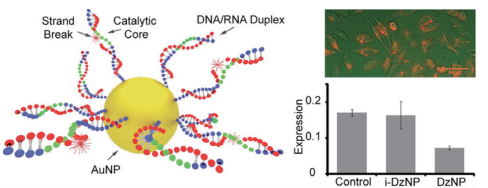
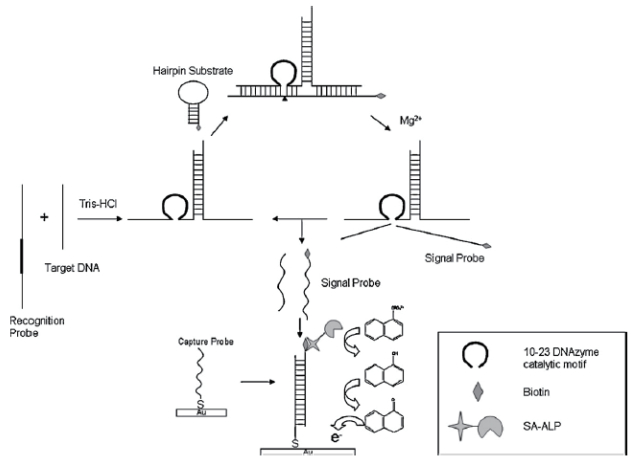
Sun等[38]利用DNA连接靠近的原则设计了由DZ13引起的靶标DNA电化学检测方法(图 12)。该方法中存在两种探针,识别探针和捕获探针。识别探针上含有10-23核酶序列,当靶标DNA出现时,在Tris-HCl条件下,两者通过碱基互补配对原则结合,产生10-23活性结构。接着,环状发卡底物开链与DZ13活性中心结合,并在Mg2+条件下发生对发卡底物链的切割,释放出含有生物素标记的片段。该片段被电极表面的捕获探针结合,通过电信号进行输出。释放的核酶链可以进一步结合其他的靶标序列,实现循环放大。该方法检测可以达到50 fmol/L,并且能够检测到单碱基的差异。
Gao等[39]构建了10-23DZ电化学生物传感器检测血清样本中的Mg2+。首先将5' 端修饰硫醇的10-23酶链修饰在金电极的表面,溶液中5' 端修饰二茂铁的底物链通过碱基互补配对原则靠近金电极表面。当溶液中不存在Mg2+时,由于二茂铁的电子传递作用会产生明显的电流信号。当存在Mg2+时,底物链被10-23酶链切割,使二茂铁远离金电极,电信号减弱。整个检测范围0.2-5.0 mmol/L,检测极限位0.05 mmol/L。
4 DZ13介导传感器在基因治疗方面应用由于10-23DZ对于RNA序列的高效的剪切作用,其在细菌基因、癌症基因以癌症基因方面的沉默效应得到了广泛的关注。10-23DZ介导的相关传感器具有稳定核酶性质、提高递送效率等特点,在体内基因治疗方面取得了很大的进步[40]。
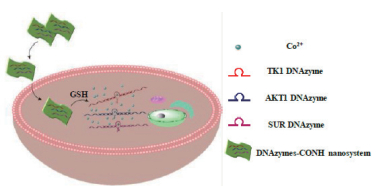
Li等[41]将DZ13与氢氧化钴纳米片(COHN)组成纳米传感器成功的对3个肿瘤相关基因进行了沉默(图 13)。利用氨基活化的氢氧化钴纳米片作为DZ13的传送载体,其中氢氧化钴作为氧化物能够被胞内的谷胱甘肽还原成二价Co2+,Co2+作为诱导离子激活DZ13,完成对肿瘤相关mRNA剪切,实现该基因的沉默。将修饰罗丹明的DZ13-COHN对活细胞进行实验可以通过共聚焦荧光成像观测传感器在胞内情况。
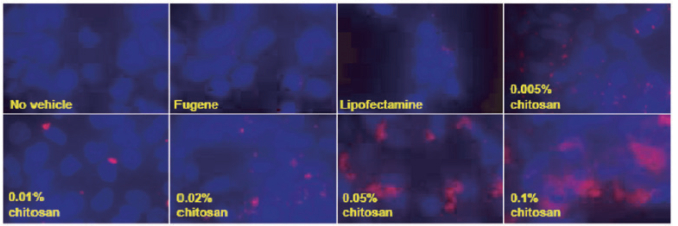
Dass等[42]通过将DZ13与壳聚糖结合在一起组成了DZ13-壳聚糖纳米传感器,成功建立一套关于DZ13高效安全的递送系统,完成了骨肉瘤细胞中相关基因的沉默(图 14)。其中DZ13-壳聚糖纳米传感器大小为350 nm,进入细胞内部时间为48 h。将罗丹明标记在DZ13上,通过荧光电镜观察可以确定传感器进入细胞的效率以及DZ13保持的完整性,整个体系室温下就可以进行,传感器稳定性良好,室温条件一个月内保持活性不变。
5 结语DZ13作为一种功能性核酸分子,基本性质是RNA特异性切割工具,具有高效的催化能力、特异性的底物结合能力和较为稳定的化学性质等。利用其本身性质加上与纳米材料相结合,组成具有多功能的纳米工作,可以在生物成像、生物传感以及相关疾病治疗方面广泛应用。
现阶段,DZ13为主的应用主要涉及新型基因治疗药物开发方面,递送方式主要是通过构建质粒的方式。未来借助DZ13功能化的纳米材料,可进行纳米靶向药物递送,推进癌症、肿瘤等相关疾病的治疗。然而,DZ13介导的生物传感器应用相对较少,信号输出的方式也较为单一,电化学方面、光磁方面应用较少。通过将DZ13与电化学以及纳米材料相结合搭建新型传感器,可以有效的扩大靶物质检测范围,提高检测以及检测极限。并且,DZ13与靶物质结合的空间构象机制尚不清楚,后续可探讨其结合方式是诱导形成还是刚性构,通过比较不同靶物质的结合构象的差异机制,以此为依据进行DZ13的序列裁剪以开发适合不同靶物质的特异性更高、亲和力更强的DZ13衍生核酶。
| [1] |
Sullenger BA, Gilboa E. Emerging clinical applications of RNA[J]. Nature, 2002, 418(6894): 252. DOI:10.1038/418252a |
| [2] |
Cho EJ, Rajendran M, Ellington AD. Aptamers as Emerging Probes for Macromolecular Sensing[M]. US: Springer, 2005.
|
| [3] |
Ravelet C, Grosset C, Peyrin E. Liquid chromatography, electrochromatography and capillary electrophoresis applications of DNA and RNA aptamers[J]. Journal of Chromatography A, 2006, 1117(1): 1-10. |
| [4] |
Lu Y, Liu J. Smart Nanomaterials inspired by biology:dynamic assembly of error-free nanomaterials in response to multiple chemical and biological stimuli[J]. Accounts of Chemical Research, 2007, 40(5): 315-323. DOI:10.1021/ar600053g |
| [5] |
Storhoff JJ, Mirkin CA. ChemInform Abstract:Programmed materials synthesis with DNA[J]. Cheminform, 1999, 30(39): 1849-1862. |
| [6] |
Silverman SK. Catalytic DNA(deoxyribozymes)for synthetic applications-current abilities and future prospects[J]. Chemical communications(Cambridge, England), 2008, 39(30): 3467. |
| [7] |
Famulok M, Mayer G, Blind M. Nucleic acid aptamers-from selection in vitro to applications in vivo[J]. Accounts of Chemical Research, 2000, 33(9): 591. DOI:10.1021/ar960167q |
| [8] |
Song S, Wang L, Li J, et al. Aptamer-based biosensors[J]. Trac Trends in Anal Chem, 2008, 27(2): 108-117. DOI:10.1016/j.trac.2007.12.004 |
| [9] |
Breaker RR, Joyce GF. A DNA enzyme that cleaves RNA[J]. Chemistry & Biology, 1994, 1(4): 223. |
| [10] |
Huang PJ, Liu J. Rational evolution of Cd2+-specific DNAzymes with phosphorothioate modified cleavage junction and Cd2+ sensing[J]. Nucleic Acids Research, 2015, 43(12): 6125-6133. DOI:10.1093/nar/gkv519 |
| [11] |
Zhou W, Vazin M, Yu T, et al. In vitro selection of chromium-dependent DNA zymes for sensing chromium(Ⅲ)and chromium(Ⅵ)[J]. Chemistry, 2016, 22(28): 9835. DOI:10.1002/chem.201601426 |
| [12] |
Huang PJ, Liu J. An Ultrasensitive light-up Cu2+ biosensor using a new DNAzyme cleaving a phosphorothioate modified substrate[J]. Anal Chem, 2016, 88(6): 3341. DOI:10.1021/acs.analchem.5b04904 |
| [13] |
Lan T, Furuya K, Lu Y. A highly selective lead sensor based on a classic lead DNAzyme[J]. Chem Commun, 2010, 46(22): 3896. DOI:10.1039/b926910j |
| [14] |
Santoro SW, Joyce GF. A general purpose RNA-cleaving DNA enzyme[J]. Proc Natl Acad Sci USA, 1997, 94(9): 4262-4256. DOI:10.1073/pnas.94.9.4262 |
| [15] |
Breaker RR, Joyce GF. A DNA enzyme with Mg2+-dependent RNA phosphoesterase activity[J]. Chemistry & Biology, 1995, 2(10): 655. |
| [16] |
Santoro SW, Joyce GF. Mechanism and utility of an RNA-cleaving DNA enzyme[J]. Biochemistry, 1998, 37(38): 13330-13342. DOI:10.1021/bi9812221 |
| [17] |
Cairns M J, Hopkins TM, Witherington C, et al. The influence of arm length asymmetry and base substitution on the activity of the 10-23 DNA enzyme[J]. Antisense and Nucleic Acid Drug Development, 2000, 10(5): 323-332. DOI:10.1089/oli.1.2000.10.323 |
| [18] |
Pan WH, Devlin HF, Kelley C, et al. A selection system for identifying accessible sites in target RNAs[J]. RNA, 2001, 7(4): 610-621. DOI:10.1017/S1355838201001595 |
| [19] |
Wang Q, Zhang D, Liu Y, et al. A structure-activity relationship study for 2'-deoxyadenosine analogs at A9 position in the catalytic core of 10-23 DNAzyme for rate enhancement[J]. Nucleic Acid Therapeutics, 2012, 22(6): 423-427. DOI:10.1089/nat.2012.0365 |
| [20] |
Vester B, Lundberg LB, S Rensen MD, et al. LNAzymes:incorporation of LNA-type monomers into DNAzymes markedly increases RNA cleavage[J]. J Am Chem Soc, 2002, 124(46): 13682-13683. DOI:10.1021/ja0276220 |
| [21] |
Wengel J, Vester B, Lundberg LB, et al. LNA and alpha-L-LNA:towards therapeutic applications[J]. Nucleosides Nucleotides & Nucleic Acids, 2003, 22(5-8): 601-614. |
| [22] |
Asahina Y, Ito Y, Wu CH, et al. DNA ribonucleases that are active against intracellular hepatitis B viral RNA targets[J]. Hepatology, 1998, 28(2): 547. DOI:10.1002/(ISSN)1527-3350 |
| [23] |
Sioud M, Leirdal M. Design of nuclease resistant protein kinase calpha DNA enzymes with potential therapeutic application[J]. Journal of Molecular Biology, 2000, 296(3): 937-947. DOI:10.1006/jmbi.2000.3491 |
| [24] |
Yen L, Strittmatter SM, Kalb RG. Sequence-specific cleavage of Huntingtin mRNA by catalytic DNA[J]. Annals of Neurology, 1999, 46(3): 366-373. DOI:10.1002/(ISSN)1531-8249 |
| [25] |
Zhao Y, Li Z, Tang Z. Cleavage-based signal amplification of RNA[J]. Nature Communications, 2013, 4(2): 1493. |
| [26] |
Carter JR, Balaraman V, Kucharski CA, et al. A novel dengue virus detection method that couples DNAzyme and gold nanoparticle approaches[J]. Virology Journal, 2013, 10: 201. DOI:10.1186/1743-422X-10-201 |
| [27] |
Li Y, Xu J, Wang L, et al. Aptamer-based fluorescent detection of bisphenol A using nonconjugated gold nanoparticles and CdTe quantum dots[J]. Sensors & Actuators B Chemical, 2016, 222: 815-822. |
| [28] |
Bone SM, Hasick NJ, Lima NE, et al. DNA-only cascade:a universal tool for signal amplification, enhancing the detection of target analytes[J]. Anal Chem, 2014, 86(18): 9106-9113. DOI:10.1021/ac501811r |
| [29] |
Cha TG, Pan J, Chen H, et al. A synthetic DNA motor that transports nanoparticles along carbon nanotubes[J]. Nat Nanotechnol, 2014, 9(1): 39-43. |
| [30] |
Fan H, Zhao Z, Yan G, et al. A smart DNAzyme-MnO2 nanosystem for efficient gene silencing[J]. Angewandte Chemie International Edition, 2015, 54(16): 4801-4805. DOI:10.1002/anie.201411417 |
| [31] |
Todd AV, Fuery CJ, Impey HL, et al. DzyNA-PCR:use of DNAzymes to detect and quantify nucleic acid sequences in a real-time fluorescent format[J]. Clinical Chemistry, 2000, 46(5): 625-630. |
| [32] |
Tian Y, Mao C. DNAzyme amplification of molecular beacon signal[J]. Talanta, 2005, 67(3): 532-537. DOI:10.1016/j.talanta.2005.06.044 |
| [33] |
Kahan-Hanum M, Douek Y, Adar R, et al. A Library of programma-ble DNAzymes that operate in a cellular environment[J]. Sci Rep, 2013, 3: 1535. DOI:10.1038/srep01535 |
| [34] |
Wu L, Tong Q, Li HC, et al. Design of a new hairpin DNAzyme:The activity controlled by TMPyP 4[J]. African Journal of Biotechnology, 2011, 10(40): 7902-7910. DOI:10.5897/AJB |
| [35] |
Yehl K, Joshi JP, Greene Bl, et al. Catalytic deoxyribozyme-modif-ied nanoparticles for RNAi-independent gene regulation[J]. ACS Nano, 2012, 6(10): 9150-9157. DOI:10.1021/nn3034265 |
| [36] |
Elbaz J, Moshe M, Shlyahovsky B, et al. Cooperative multicomponent self-assembly of nucleic acid structures for the activation of DNAzyme cascades:a paradigm for DNA sensors and aptasensors[J]. Chemistry, 2009, 15(14): 3411-3418. DOI:10.1002/chem.v15:14 |
| [37] |
Gong H, Zhong T, Gao L, et al. Unlabeled hairpin DNA probe for electrochemical detection of single-nucleotide mismatches based on MutS-DNA interactions[J]. Anal Chem, 2009, 81(20): 8639-8643. DOI:10.1021/ac901371n |
| [38] |
Sun C, Zhang L, Jiang J, et al. Electrochemical DNA biosensor based on proximity-dependent DNA ligation assays with DNAzyme amplification of hairpin substrate signal[J]. Biosens Bioelectron, 2010, 25(11): 2483-2489. DOI:10.1016/j.bios.2010.04.012 |
| [39] |
Gao X, Huang H, Niu S, et al. Determination of magnesium ion in serum samples by a DNAzyme-based electrochemical biosensor[J]. Analytical Methods, 2012, 4(4): 947-952. DOI:10.1039/c2ay05846d |
| [40] |
Fokina AA, Stetsenko DA, Franois JC. DNA enzymes as potential therapeutics:towards clinical application of 10-23 DNAzymes[J]. Expert Opinion on Biological Therapy, 2015, 15(5): 689-711. DOI:10.1517/14712598.2015.1025048 |
| [41] |
Li N, Li Y, Gao X, et al. Multiplexed gene silencing in Living cells and in vivo using a DNAzymes-CoOOH nanocomposite[J]. Chem Commun, 2017, 53(36): 4962-4965. DOI:10.1039/C7CC00822H |
| [42] |
Dass CR, Friedhuber AM, Khachigian LM, et al. Biocompatible chitosan-DNAzyme nanoparticle exhibits enhanced biological activity[J]. Journal of Microencapsulation, 2008, 25(6): 421-425. DOI:10.1080/02652040802033673 |