S-腺苷甲硫氨酸(S-adenosyl methionine,SAM)在DNA甲基转移酶的催化下把甲基(-CH3)转移到DNA分子中特定碱基上的过程称为DNA甲基化的过程[1]。多数发生在CpG岛的胞嘧啶第5位碳原子(C5)上,通常发生在对称序列CG中,但是在CHG和CHH(H=A、C或T)序列中也有发生[2]。DNA甲基化是一种广泛存在的表观遗传过程,包括转座子的抑制、基因组印记[3]、X染色体失活[4]、细胞分化[5]和胚胎发育[6]。在胁迫条件下植物通过DNA甲基化过程,诱导一些与抗逆相关的基因表达,增强植物的抗逆性,维持植物的生长发育和进化过程[7]。DNA甲基化是一种植物响应逆境胁迫的分子机制[8]。植物中存在2种DNA甲基化方式:一种是维持甲基化(Maintenance methylation),通过半保留复制把双链DNA分子的一条链存在甲基化,另一条链没被甲基化的亲代甲基化模式传递给子代[9];另一种是从头甲基化(De novo methylation),通过不同的DNA甲基转移酶催化将不存在甲基化的DNA双链形成甲基化[10]。植物中的C5-DNA甲基转移酶有4种,维持DNA甲基转移酶(Methyltransferase,MET)家族[11];染色质甲基化酶(Chromomethylase,CMT)家族[12];Dnmt2;结构域重排甲基转移酶(Domains rearranged methyltransferase,DRM)家族可以在RNA指导下,催化胞嘧啶从头甲基化,以及维持非CpG位点的甲基化[13];MET、CMT与动物的DNMT1为同源[14];DRM与动物的DNMT3同源[15]。
非生物胁迫严重影响和制约着农作物的产量和品质。当烟草发生渗透胁迫时(Nicotiana L.)组织培养细胞的异染色质区会发生DNA超甲基化[16]。CHROMOMETHYLASE3(SLCMT3)基因可以使Chr番茄果实的成熟,提高控制成熟关键基因的表达[17];当萝卜(Raphanus sativus L.)受到重金属铬胁迫时DNA的甲基化水平会升高[18];玉米(Zea mays L.)受到冷胁迫时,根系通过降低核小体中心DNA甲基化水平诱导ZmMI1表达,受到盐胁迫时玉米幼苗zmPP2C甲基化将下调和zmGST表达量也下调[19];大豆受干旱、冷、盐胁迫时,根中CaDRM1和CaDRM2的表达量上调,以应对胁迫压力[20];苜蓿种子发育和结瘤的早期阶段MtDRM1和MtDRM2高度表达,MtDRM3在种子和结节发育阶段表达量高[20]。DNA甲基转移酶与抗逆性密切相关,深入对DNA甲基转移酶的作用机制进行研究,对进一步挖掘抗逆基因具有重要意义。
棉花作为重要的经济作物,其棉纤维可以制作成织物,棉籽可以榨油,在人们的日常生活中应用广泛。胁迫条件下,棉花产量会大幅度减产;DNA甲基转移酶可以增强植物的耐受性,GhDMT3是DRM家族的一员,在水稻中鉴定出2个DRM家族相关基因、苜蓿中鉴定出4个DRM家族的成员、在大豆中发现5个DRM家族相关基因、在玉米和拟南芥中各含有2个DRM家族的成员,而棉花中的DNA甲基转移酶研究较少。本研究通过对棉花GhDMT3进行生物信息学分析,并构建pYL156:GhDMT3沉默载体,对其进行功能验证,为提高棉花抗逆性奠定基础。
1 材料与方法 1.1 材料陆地棉TM-1由中国农业科学院棉花研究所品种资源研究室保存。所用菌种为大肠杆菌E.coli DH5α,农杆菌LB3101、pYL156、pYL192(辅助载体)和pYL156:GhDMT3(阳性对照载体)载体由中国农业科学院棉花研究所基因组测序课题组提供。
1.2 方法 1.2.1 RNA的提取及cDNA的制备按照北京艾德莱生物科技的RN38 EASYspin Plus植物RNA快速提取试剂盒说明书提取RNA,按照北京全式金的TransScript ΙΙ All-in -One First-Strand cDNA Synthe-sis SuperMix for qPCR(One-Step gDNA Removal)说明书制备cDNA。
1.2.2 GhDMT3的克隆用Primer Premier 5软件设计GhDMT3引物(F:ATGGTTGACAATAATTCGGG-TGG;R:TCAGATAATCATTGATTTTGTG),以cDNA为模板进行扩增,PCR程序为94℃ 5 min;94℃ 20 s,54℃ 20 s,72℃ 70s,34个循环;72℃ 10 min,4℃保存。PCR产物用1%的琼脂糖凝胶电泳进行检测,回收、纯化,与T载连接,并转入E.coli DH5α感受态细胞,筛选阳性单克隆并测序,获得正确的t-GhDMT3序列。
1.2.3 构建pYL156:GhDMT3的VIGS载体用EcoRⅠ和XmaⅠ双酶切PYL156,获得线性pYL156载体,以克隆载体t-GhDMT3为模板用引物in-V-GhDMT3进行扩增,获得VIGS片段。用in-FuSion技术将GhDMT3的VIGS沉默片段插入到线性的PYL156载体上,转入大肠杆菌,挑取阳性单克隆测序,获得正确的单克隆,并用EcoRⅠ和XmaⅠ双酶切验证,成功构建pYL156:GhDMT3载体。
1.2.4 pYL156:GhDMT3的VIGS沉默及表达量检测种植TM-1到两片子叶平展时,用一次性的医用注射器在叶片的背面,进行VIGS注射,注射面积在75%以上。然后置于25℃黑暗培养24 h,处理结束后,在适宜的温度(28℃ 14 h/25℃ 10 h)下保证幼苗正常生长。当出现白化后,对植株分别进行冷(4℃)和干旱(自然干旱)处理,当与对照出现表型差异时,测定GhDMT3的相对表达量。
2 结果 2.1 GhDMT3蛋白的理化性质及亲水性/疏水性分析通过ExPASY网站的生物软件ProtParam程序对GhDMT3编码的蛋白进行组分分析,该蛋白质相对分子质量为71 107.02 kD,等电点为4.85,该基因编码的蛋白质分子式为C3151H4897N849O981S23,共计9 901个原子,编码640个氨基酸,其中65个带正电的氨基酸(Arg +Lys),96个带负电的氨基酸(Asp+Glu),不稳定系数为36.33,脂肪系数为81.81,总平均亲水性为-0.364,预测该蛋白为酸性蛋白,不含有赖氨酸和丝氨酸,色氨酸含量最低为1.6%,亮氨酸含量最高为10.2%。
2.2 GhDMT3蛋白质的亲疏水性分析利用ProtScale网站对GhDMT3蛋白的亲疏水性进行预测,结果(图 1)表明,在256位的S亲水性最强分值为-2.357,在517位的F疏水性最强分值为1.210,亲水性总平均值为-1.157,推测该基因的整个氨基酸序列中亲水性氨基酸比疏水性氨基酸多,所以该蛋白为亲水性蛋白。综上所述,该基因编码的蛋白具有一定的亲水能力但不稳定。
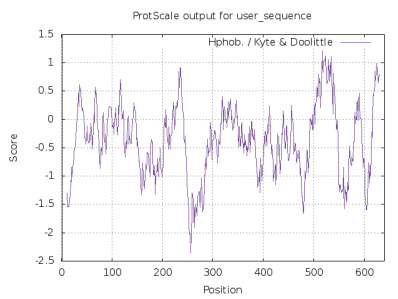
|
| 图 1 GhDMT3蛋白的疏水性/亲水性检测 |
通过TMHMM网站对GhDMT3蛋白的跨膜结构域进行分析(图 2),GhDMT3蛋白不存在跨膜结构域,属于非跨膜蛋白,与该蛋白的疏水性区域分析结果基本一致。利用SignalP-4.0网站对GhDMT3蛋白的信号肽预测结果为C值(剪切位点)为0.114,S值(信号肽)为0.109,Y值(综合剪切点)为0.105(图 3)。GhDMT3蛋白并不是分泌型蛋白,不在细胞中发生迁移。
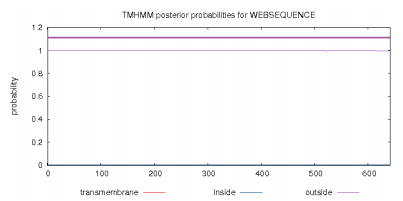
|
| 图 2 GhDMT3蛋白的跨膜结构域分析 |

|
| 图 3 GhDMT3蛋白的信号肽预测 |
通过软件预测分析GhDMT3蛋白的二级结构。结果(图 4)表明,无规则卷曲包含298个氨基酸残基,占46.56%,α螺旋包含266个氨基酸残基,占41.11%,延伸链包含67个氨基酸残基,占10.47%,β-转角包含33个氨基酸,占5.12%。
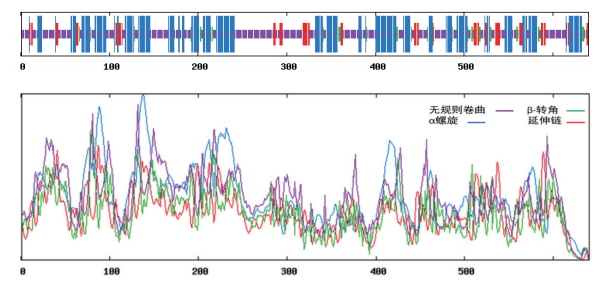
|
| 图 4 GhDMT3基因编码蛋白的二级结构 |
使用软件SWISS-MODEL预测GhDMT3蛋白的三维结构,GhDMT3蛋白三维结构以α螺旋为主(图 5),符合GhDMT3蛋白的二级结构分析。
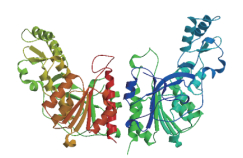
|
| 图 5 GhDMT3蛋白的三级结构 |
利用RT-PCR技术扩增GhDMT3,转T载体,获得t-GhDMT3;以克隆载体t-GhDMT3为模板,对in-v-GhDMT3进行扩增,获得VIGS片段(图 6-A)。用in-FuSion技术将GhDMT3的VIGS沉默片段插入到pYL156载体上,并用EcoRⅠ和XmaⅠ双酶切验证(图 6-B),即成功构建pYL156:GhDMT3载体。
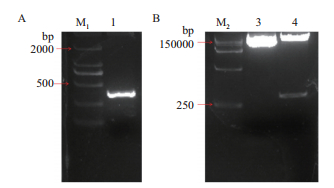
|
| 图 6 VIGS载体pYL156:GhDMT3的构建与验证 M1:2 kb DNA Marker;M2:15 kb DNA marker;1:in-v-GhDMT3的PCR产物;3:pYL156载体;4:pYL156:GhDMT3载体EcoRⅠ和XmaⅠ双酶切产物 |
VIGS侵染棉花植株15 d左右,阳性植株叶片白化明显,其他正常生长,用没有侵染的棉花和用pYL156侵染的棉花作对照,对沉默植株进行冷、干旱的胁迫处理。冷处理36 h后植株表型差异明显(图 7-A),注射pYL156的植株与野生型的新生真叶枯萎卷曲,注射pYL156:GhDMT3的植株正常生长,表型变化不明显。pYL156侵染的棉花中在不同胁迫下表达量无显著变化,与对照相比,用pYL156:GhDMT3侵染过的棉花GhDMT3转录水平明显下降。根部下降最多;茎部次之;叶片下降最少(图 7-B)。

|
| 图 7 VIGS侵染后的棉花表型和冷胁迫36 h的棉花表型(A);VIGS侵染成功冷胁迫处理后植株的相对表达量(B) |
自然干旱6 d后植株表型差异明显,注射pYL156的植株和野生型的子叶脱落,新生真叶发黄枯萎,严重失水,植株死亡,注射pYL156:GhDMT3的植株真叶没有枯萎失水现象(图 8-A)。检测pYL156:GhDMT3侵染过的棉花中GhDMT3的相对表达量,由图可知pYL156侵染的棉花中在不同胁迫下表达量没有变化,用pYL156:GhDMT3侵染过的棉花中GhDMT3转录水平与对照相比叶片中下降最多;茎部次之;根部下降最少(图 8-B)。
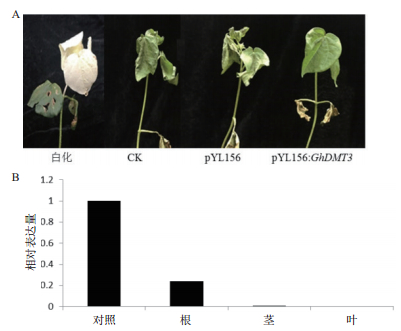
|
| 图 8 VIGS侵染后的表型和干旱胁迫6 d的表型(A)及VIGS侵染成功干旱胁迫处理后植株的相对表达量(B) |
DNA甲基转移酶是DNA甲基化过程中的关键酶,在拟南芥[10]、水稻[21]、玉米[22]、大豆[20]、葡萄[23]、番茄[24]及蓖麻[25]等植物中鉴定出DNA甲基转移酶。GhDMT3与蓖麻中的DRM家族基因编码蛋白一致,该家族基因编码蛋白为不稳定的亲水性蛋白,不具有跨膜结构域,二级结构以无规则卷曲、α螺旋为主为。通过分析GhDMT3三维结构与DNA甲基转移酶的典型结构类似,但是有一定的区别,具体原因需要后续实验探明。
大豆的DRM基因在非生物胁迫期间转录水平较高参与诱导DNA甲基化改变[20],当拟南芥drm1和drm2双突变时会造成所有已知模式的胞嘧啶从头甲基化均有缺失[10],DRM家族基因对于FWA和SUP的基因沉默是重要的,可以通过不同的机制使直接重复和反向重复发生甲基化[26]。通过沉默GhDMT3基因,证明该基因与抗逆性相关,为明确该基因的功能奠定基础,而该基因的具体功能需要进一步研究。
4 结论成功构建了GhDMT3的VIGS沉默载体,获得转基因植株,在冷、干旱胁迫条件下,转基因植株抵抗力增强。
| [1] |
Pavlopoulou A, Kossida S. Plant cytosine-5 DNA methyltransferases: structure, function, and molecular evolution[J]. Genomics, 2007, 90(4): 530-541. |
| [2] |
Li E, Zhang Y. DNA methylation in mammals[J]. Cold Spring Harb Perspect Biol, 2014, 6(5): a19133. |
| [3] |
Macdonald WA. Epigenetic mechanisms of genomic imprinting: common themes in the regulation of imprinted regions in mammals, plants, and insects[J]. Genet Res Int, 2012, 2012: 585024. |
| [4] |
Bala TN, Brahmachary M, Garg P, et al. DNA methylation profiling in X; autosome translocations supports a role for L1 repeats in the spread of X chromosome inactivation[J]. Hum Mol Genet, 2014, 23(5): 1224-1236. DOI:10.1093/hmg/ddt553 |
| [5] |
Choi Y, Gehring M, Johnson L, et al. DEMETER, a DNA glycosylase domain protein, is required for endosperm gene imprinting and seed viability in Arabidopsis[J]. Cell, 2002, 110(1): 33-42. |
| [6] |
Gehring M, Bubb KL, Henikoff S. Extensive demethylation of repetitive elements during seed development underlies gene imprinting[J]. Science, 2009, 324(5933): 1447-1451. DOI:10.1126/science.1171609 |
| [7] |
Krasilnikova J, Lauberte L, Stoyanova E, et al. Oregonin from Alnus incana bark affects DNA methyltransferases expression and mitochondrial DNA copies in mouse embryonic fibroblasts[J]. J Enzyme Inhib Med Chem, 2018, 33(1): 1055-1063. DOI:10.1080/14756366.2018.1476504 |
| [8] |
Zhong S, Fei Z, Chen YR, et al. Single-base resolution methylomes of tomato fruit development reveal epigenome modifications associated with ripening[J]. Nat Biotechnol, 2013, 31(2): 154-159. |
| [9] |
Chen T, Li E. Structure and function of eukaryotic DNA methyltransferases[J]. Curr Top Dev Biol, 2004, 60: 55-89. DOI:10.1016/S0070-2153(04)60003-2 |
| [10] |
Jullien PE, Susaki D, Yelagandula R, et al. DNA methylation dynamics during sexual reproduction in Arabidopsis thaliana[J]. Curr Biol, 2012, 22(19): 1825-1830. DOI:10.1016/j.cub.2012.07.061 |
| [11] |
Du J. Structure and mechanism of plant DNA methyltransferases[J]. Adv Exp Med Biol, 2016, 945: 173-192. DOI:10.1007/978-3-319-43624-1 |
| [12] |
Pavlopoulou A, Kossida S. Plant cytosine-5 DNA methyltransfera-ses: structure, function, and molecular evolution[J]. Genomics, 2007, 90(4): 530-541. DOI:10.1016/j.ygeno.2007.06.011 |
| [13] |
Cao X, Aufsatz W, Zilberman D, et al. Role of the DRM and CMT3 methyltransferases in RNA-directed DNA methylation[J]. Curr Biol, 2003, 13(24): 2212-2217. DOI:10.1016/j.cub.2003.11.052 |
| [14] |
Hermann A, Gowher H, Jeltsch A. Biochemistry and biology of mammalian DNA methyltransferases[J]. Cell Mol Life Sci, 2004, 61(19-20): 2571-2587. DOI:10.1007/s00018-004-4201-1 |
| [15] |
Garg R, Kumari R, Tiwari S, et al. Genomic survey, gene expression analysis and structural modeling suggest diverse roles of DNA methyltransferases in legumes[J]. PLoS One, 2014, 9(2): e88947. DOI:10.1371/journal.pone.0088947 |
| [16] |
Kovar˘ik A, Koukalová B, Bezde˘k M, et al. Hypermethylation of tobacco heterochromatic loci in response to osmotic stress[J]. Theoretical and Applied Genetics, 1997(1/2): 301-306. |
| [17] |
Chen W, Kong J, Qin C, et al. Requirement of CHROMOMETHYL-ASE3 for somatic inheritance of the spontaneous tomato epimutation Colourless non-ripening[J]. Sci Rep, 2015, 5: 9192. DOI:10.1038/srep09192 |
| [18] |
Dai H, Yang Z. Variation in Cd accumulation among radish cultivars and identification of low-Cd cultivars[J]. Environmental Science & Pollution Research, 2017, 24(17): 15116-15124. |
| [19] |
Steward N, Ito M, Yamaguchi Y, et al. Periodic DNA methylation in maize nucleosomes and demethylation by environmental stress[J]. J Biol Chem, 2002, 277(40): 37741-37746. DOI:10.1074/jbc.M204050200 |
| [20] |
Garg R, Kumari R, Tiwari S, et al. Genomic survey, gene expression analysis and structural modeling suggest diverse roles of DNA methyltransferases in legumes[J]. PLoS One, 2014, 9(2): e88947. DOI:10.1371/journal.pone.0088947 |
| [21] |
Teerawanichpan P, Chandrasekharan MB, Jiang Y, et al. Characterization of two rice DNA methyltransferase genes and RNAi-mediated reactivation of a silenced transgene in rice callus[J]. Planta, 2004, 218(3): 337-349. DOI:10.1007/s00425-003-1112-6 |
| [22] |
Qian Y, Xi Y, Cheng B, et al. Genome-wide identification and expression profiling of DNA methyltransferase gene family in maize[J]. Plant Cell Rep, 2014, 33(10): 1661-1672. DOI:10.1007/s00299-014-1645-0 |
| [23] |
Ren F, Yang L, Ling SU, et al. Genome-wide identification and analysis of DNA methyltransferases in grape[J]. Agricultural Science & Technology, 2017(10): 7-13+20. |
| [24] |
Kumar R, Chauhan PK, Khurana A. Identification and expression profiling of DNA methyltransferases during development and stress conditions in Solanaceae[J]. Funct Integr Genomics, 2016, 16(5): 513-528. DOI:10.1007/s10142-016-0502-3 |
| [25] |
代梦媛, 高梅, 郭丽芬, 等. 蓖麻DNA甲基转移酶序列与基因表达分析[J]. 云南农业大学学报:自然科学版, 2016(4): 604-614. |
| [26] |
Cao X, Jacobsen SE. Role of the Arabidopsis DRM methyltransfera-ses in de novo DNA methylation and gene silencing[J]. Curr Biol, 2002, 12(13): 1138-1144. DOI:10.1016/S0960-9822(02)00925-9 |




