2. 农业部农业转基因生物安全评价(食用)重点实验室,北京 100083
2. Key Laboratory of Safety Assessment of Genetically Modified Organism(Food Safety), Ministry of Agriculture, Beijing 100083
在传统生物学的概念中,DNA的主要功能是储存遗传信息。然而早在1982年,Seeman[1]就提出,DNA所特有的分子识别性质或许可以用于完全非生物的环境中。人们从1990年开始对包括适配体和Ribozyme在内的功能核酸进行研究和探索[2]。从此,功能核酸成为感知应用领域中极具吸引力的检测工具,可源于自然,或是通过随机的核酸库人工筛选出来[3]。功能核酸作为靶物质的识别分子,与信号转换和输出系统构成生物传感器的3大组成要素[4]。与传统的分析方法相比,功能核酸生物传感器因其灵敏度高、选择性好、所需时间短、操作简单和检测成本低等特点,极大地推动了医学临床诊断、环境监测和药物筛选的发展。
近年来,为了实现超灵敏检测,人们将不同的信号放大策略整合到传感器的设计中,包括连接酶链式反应[5]、杂交链式反应(Hybridization chain reaction,HCR)[6]、滚环扩增(Rolling circle amplif-ication,RCA)[7]、链置换反应[8]和酶催化反应[9]等。尽管这些技术的运用能够将传感器的灵敏性提高到一定程度,但也免不了存在些不足,如灵敏性有限、程序繁琐等。但是,工具酶的高灵敏性和特异性、操作简单以及反应条件温和的特性能很好的弥补这些不足,其介导的信号放大技术在生化分析中得到了快速发展,这些工具酶包括序列依赖的限制性内切酶、切刻酶,以及非序列依赖的RNase H、外切酶、双链特异性核酸酶、AP(Apurinic/apyrimidinic,脱嘌呤/脱嘧啶)内切酶和DNase Ⅰ等[10]。尤其是Exo Ⅲ,作为一种不需要特殊识别位点,并且有着高效稳定的切割活性的核酸酶,已经被广泛用于靶物质的信号循环放大过程。Exo Ⅲ的最佳底物是dsDNA平末端或3'凹端,对ssDNA和3'端含有4个以上单核苷酸凸起的dsDNA活性很低,早期被用于DNA测序[11-12],后来随着功能核酸生物传感器的不断发展,将Exo Ⅲ与功能核酸搭配使用,可将非核酸信号转换成核酸信号,搭载各种信号输出和放大方式,实现靶物质的快速精准检测。因此,Exo Ⅲ在功能核酸生物传感器的研究应用中占据十分重要的地位。本文对Exo Ⅲ的不同性质进行介绍,并根据靶物质不同对Exo Ⅲ介导的生物传感器进行分类综述。此外,还对Exo Ⅲ结合其他信号放大策略或纳米材料的生物传感器进行较为详细的介绍,旨在使人们更多地了解Exo Ⅲ在生物传感器中的应用特点和发展现状,为今后所用。
1 Exo Ⅲ与核酸相互作用的酶活性质Exo Ⅲ是分子量为28 askD的单体蛋白质,沉降系数(S20,w)为2.92并且是球形的(f/fo = 1.15)[13]。Mol等[14]报道了Exo Ⅲ的1.7 A高分辨率晶体结构,该结构具有2倍对称、4层αβ折叠结构,与DNase Ⅰ和RNase H的结构有相似之处。Exo Ⅲ可以催化dsDNA中几种磷酯键的水解,包括3'-5'外切酶活性、3'磷酸酶活性和AP内切酶活性(图 1)。另外,还具有RNase H活性。下面将对这几种主要的活性进行较为详细的介绍。
1.1 磷酸酶活性Exo Ⅲ在大肠杆菌中被大量纯化,最开始被鉴定为一种磷酸酶。在磷酸钾缓冲液中,Exo Ⅲ发挥磷酸酶活性的最佳pH为6.8-7.4;在Tris-马来酸盐缓冲液中,最佳pH为6.7-7.0,两种缓冲液达最大磷酸酶活性的pH均为7左右[15]。此外,Mg2+对Exo Ⅲ磷酸酶的活性具有十分重要的作用,在缺少Mg2+的条件下,磷酸酶活性只达最大酶活的5%,其他二价阳离子如Ca2+和Zn2+无法取代Mg2+的作用;且当Mg2+处于浓度最优时,Zn2+的添加会导致磷酸酶的酶活抑制[15]。Richardson等[16]证明Exo Ⅲ只有将dsDNA的3'磷酸基团移除之后,才能从3'端逐步释放出5'核苷酸,发挥外切酶的活性。
1.2 外切酶活性Exo Ⅲ对dsDNA具有水解专一性,当dsDNA存在时,Exo Ⅲ催化dsDNA分子从3'末端进行逐步水解[16]。该酶的最适底物为平末端或者3'凹端的dsDNA[17]。在消化过程中,部分水解的dsDNA产生凸出的5'尾巴,双链区域会越来越短,直到没有足够的碱基数量维持碱基对以保持双链结构。由于ssDNA链内存在氢键,所以Exo Ⅲ对ssDNA也具有一定的降解作用,但降解效率远低于dsDNA[18]。在dsDNA消化40%-50%之后,由于剩余的DNA几乎都是单链的,因此水解速率会突然减缓[18]。Wu等[19]证明,在23℃时,一个Exo Ⅲ分子在10 min内能够从DNA的3'端水解100个核苷酸。5℃时,在高盐环境中,一个Exo Ⅲ只能催化一个dsDNA分子的3'端约6个核苷酸同步水解;若Exo Ⅲ与dsDNA分子3'端之比提高到2倍时,每个3'末端将有约12个核苷酸水解[20]。
当合成双链由均聚物如(dA)n·(dT)n或交替共聚物如d(A-T)n组成时,此时相关的动力学与天然来源的DNA不同,因其会通过氢键的自发断裂和重组使链在彼此之间移动,从而维持最大限度的碱基配对,直到底物被消化的程度接近100%[18]。
1.3 内切酶活性DNA的烷基化主要发生在鸟嘌呤的N7和腺嘌呤的N3位,在中性或酸性环境中,DNA会因为失去烷基化的鸟嘌呤残基导致单链断开[21]。1969年,Friedberg和Goldthwait[22]纯化出内切酶Ⅱ,该酶不仅能够降解烷基化的DNA,对天然的T4和T7噬菌体DNA还能够造成有限的单链断裂[23]。此外,在高纯度的内切酶Ⅱ样品中还发现了外切酶活性,但当时被误认为是受到外切酶污染的缘故[22]。Yajko和Weiss[24]发现,Exo Ⅲ缺陷型的大肠杆菌突变体,其内切酶Ⅱ的活性也同样受到影响,反之亦然,即Exo Ⅲ和内切酶Ⅱ的活性具有一定的相关性。随后Weiss将纯化好的内切酶Ⅱ样品分别进行聚丙烯酰胺凝胶电泳、蔗糖密度梯度离心和凝胶过滤层析,发现每个实验中,只要是含有内切酶Ⅱ活性的部分都同样包含外切酶和DNA-3'-磷酸酶活性,这些活性均来自同一个单体蛋白Exo Ⅲ,可能是因为该蛋白的某个活性位点能够通过脱嘌呤或DNA双链自发的末端解旋从而识别链间区域[13]。
1.4 RNase H活性[18]将Exo Ⅲ的纯化产物进行凝胶过滤时,无法将RNase的活性与Exo Ⅲ的其他酶活区分开。Poly(rA)·poly(dT)的降解速率是poly(dA)·poly(dT)的1/5。杂交链中RNA链的降解比DNA链快100倍,同时缺少任何一条链,将无法从另一条链检测出酶活,这种现象可能是由于Exo Ⅲ识别与切割链互补的DNA链上的脱氧核糖残基。
2 Exo Ⅲ介导的功能核酸生物传感器由于Exo Ⅲ的最佳底物是dsDNA平末端或3'凹端,对ssDNA和3'端含有4个以上单核苷酸凸起的dsDNA活性很低,所以在传感器发生酶催化反应时,为了保证靶物质的完整性,信号探针参与杂交后应为平末端或3'凹端[10]。
2.1 Exo Ⅲ介导的microRNA功能核酸生物传感器的研究进展研究表明,microRNAs可以成为某些疾病如恶性肿瘤的生物标志物,microRNAs的表达与肿瘤的发生、发展和治疗反应等具有紧密的联系,因此检测肿瘤细胞的microRNAs对于癌症的机制探索、控制和治疗具有很重要的意义[25]。Huang等[26]利用Exo Ⅲ和石墨烯介导的信号放大体系设计了一个检测microRNAs的生物传感器(图 2-A)。miRNA-34a与probe DNA互补杂交成pDNA/miRNA-34a,之后Exo Ⅲ将probe DNA消化成短的寡核苷酸片段,同时释放出的miRNA-34a再与完整的probe DNA杂交参与信号放大过程,而这些短的寡核苷酸(1-4 mer)能够增强石墨烯上罗丹明6G的电离效率,使信号增强,这种方法测得的microRNA浓度能达到fmol/L水平。
Min等[27]根据聚集诱导发光的原理设计了一个具有TPEPy黄色荧光基团的超级耐光DNA探针,对尿液和活细胞中的microRNAs进行了监测(图 2-B)。反应开始时,microRNA 21与探针的DNA部分杂交形成双链DNA-RNA,由于3'为平末端,Exo Ⅲ对该双链进行识别和切割,释放出microRNA 21继续与完整的探针杂交开始下一个循环。即一个microRNA会引起多个循环并释放出许多疏水的TPEPy,聚集在一起产生黄色荧光,检测限为1 pmol/L。将FAM/Cy3及其相应的淬灭基团作为对照,同样可以进行microRNAs的检测,但是由于荧光漂白现象,这种方法并不适合长期的细胞示踪。
2.2 Exo Ⅲ介导的DNA功能核酸生物传感器的研究进展Cai等[28]利用两个3'端凸出的P1、P2分子信标进行Exo Ⅲ介导的双重循环放大反应来检测人血色素沉着症基因(T)(图 3-A)。由于这两个分子信标P1和P2为3'端凸出,可抵抗Exo Ⅲ的消化,因此可在Exo Ⅲ条件下稳定存在,背景信号低。T存在时,先与P1杂交形成双链,由于TP1双链中P1为平末端,因此其3'端被Exo Ⅲ部分消化,释放出T和X。由于Exo Ⅲ对单链T有很弱的消化活性,因此这一步的放大反应会一直持续到所有的T被缓慢降解完,此时已经积累大量的X。X的设计比较特殊,可以抵抗住Exo Ⅲ的消化。P2两端修饰了荧光和淬灭基团,由于P2的发夹结构,处于荧光淬灭状态。当与X杂交时,发夹打开,荧光增强,双链中的P2产生了3'凹端,被Exo Ⅲ消化,而X可以循环消化P2,导致很强的放大反应。根据DNA浓度与荧光信号的关系,可对T进行定量检测,最低检测限达0.3 pmol/L DNA。Zuo等[29]也利用5'端修饰荧光基团,分子内部修饰淬灭基团的分子信标对DNA进行了检测。
Huang等[30]将Exo Ⅲ介导的靶物质循环和鸟嘌呤纳米线的信号放大方法进行巧妙结合,对人体免疫缺损病毒(即艾滋病毒HIV)基因进行电化学检测(图 3-B)。当靶DNA存在的时候,会与HP1杂交生成平末端,引发Exo Ⅲ的消化,释放出靶基因进入Exo Ⅲ介导的消化循环。每个消化循环都会得到一个help DNA产物,该产物与HP2杂交能够将HP2的富G区域c-myc释放,在K+存在情况下形成平行的G-四链体(G4)结构。由于Mg2+能够识别和促发G4序列沉淀并且形成高聚物结构[31],因此最后一步加入c-myc和Mg2+孵育可产生鸟嘌呤纳米线,与氯高铁血红素(Hemin)形成复合物,催化H2O2氧化四甲基联苯胺(3, 3, 5, 5-tetramethylbenz-idine,TMB),可观察到电化学信号的增强,检测限为3.6 pmol/L。Chen等[32]也报道了一种利用Exo Ⅲ检测HIV基因的生物传感器(图 3-C)。首先将DNA探针的3'端修饰了羧基四甲基罗丹明(Carboxytetramethyl rhodamine,TAMRA),与此同时为了与上转换纳米颗粒(Upconversion nanoparticles,UCNPs)表面的羧基可以共价结合,在其5'端修饰了氨基。当靶DNA与标记了TAMRA的DNA探针杂交时,Exo Ⅲ识别并降解dsDNA,释放出靶DNA继续该循环,同时释放出游离TAMRA。事实上,UCNPs只有与完整的含TAMRA标签的DNA探针共价结合,才会发生从UCNPs到TAMRA的能量共振转移,由此对HIV基因进行检测,检测限为15 pmol/L。Luo等[33]还利用Exo Ⅲ介导的循环放大优势对编码肠杆菌β-半乳糖苷酶的Lac Z基因进行检测,最低检测限为8.7 fmol/L。
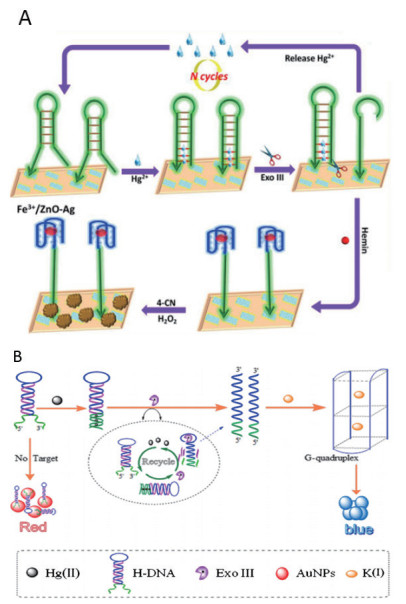
2.3 Exo Ⅲ介导的金属离子功能核酸生物传感器Zhang等[34]采用Fe3+/ZnO-Ag光催化剂结合Exo Ⅲ建立了一种灵敏的光电化学传感器来检测Hg2+的浓度(图 4-A)。Fe3+/ZnO-Ag在可见光照射下能够提供光电流。加入Hg2+后,根据T-Hg2+-T的原理可以使发夹DNA形成平末端供Exo Ⅲ发挥切割功能,切割后释放的Hg2+重新与3'凸起端的发夹结合进入循环;而切割后产物暴露出的G4序列与hemin组成复合物hemin/G4作为DNAzyme发挥活性,使4-氯-1-萘酚(4-CN)被氧化为不溶物苯基-4-氯己二烯并沉积在电极表面,阻隔界面电子传递过程从而影响系统光电流。基于此生物催化沉积法,可以实现对Hg2+的光电化学法检测,线性范围是0.5-100 nmol/L,检测限是0.1 nmol/L。Gan等[35]也根据T-Hg2+-T与Exo Ⅲ介导的循环信号放大组合设计了一个电化学传感器,最后利用[Ru(NH3)6]3+与DNA骨架的静电作用力特异性检测Hg2+的浓度,检测限低至1 pmol/L。
Hong等[36]采用Exo Ⅲ和金纳米颗粒(gold nanoparticles,AuNPs),设计了一种基于DNA构象变化的比色传感器(图 4-B)。无Hg2+时,发夹DNA(H-DNA)结合到AuNPs上,使其能够在高离子浓度的溶液中稳定存在。Hg2+存在时,由于T-Hg2+-T的原理,H-DNA的凸出端会完全形成3'平末端,Exo Ⅲ则开始从H-DNA的3'端逐步移除单核苷酸,于是Hg2+被释放继续进行循环反应。H-DNA被降解后可形成稳定的G4结构,此时AuNPs在高离子浓度的环境中发生聚集,导致颜色由红变蓝。该方法的线性检测范围为10 pmol/L-100 nmol/L,检测限低至3.2 pmol/L。
2.4 Exo Ⅲ介导的蛋白质功能核酸生物传感器凝血酶不仅是止血和凝血过程中很重要的丝氨酸蛋白酶,而且还是一种指示肿瘤生长和转移的生物标志物[37-38]。Yang等[39]利用凝血酶的适配体发明了一种“DNA多重开关”的电化学传感器以检测凝血酶(图 5-A)。第一步,先将双链S1/S2组装到金电极表面,之后S1的部分序列会与凝血酶的适配体(TBA)杂交产生新的dsDNA(S1/S2-TBA)。该dsDNA具有3'凸起端,能够抵抗Exo Ⅲ的消化。作为电化学发光指示剂,Ru(phen)32+能有效插入到双链中产生强烈的电化学信号。第二步,加入凝血酶后,与TBA结合形成TBA-凝血酶的复合物,释放TBA,导致电极表面固定的双链由3'端凸起变成凹陷,产生了Exo Ⅲ的切割位点,之后Exo Ⅲ消化了S2的红色部分序列,因此蓝色序列S2*得到释放。由于释放的S2*与TBA存在部分互补,因此S2*与TBA进一步杂交,使S1/S2-TBA由3'端凸起变成S1/S2/S2*-TBA平末端,Exo Ⅲ又能发挥降解作用,降解TBA和S2的红色序列,导致更多S2*的释放。释放的S2*再次与TBA杂交,不断启动dsDNA的循环降解,这将大大减少Ru(phen)32+的插入,抑制电化学发光效应。这种由Exo Ⅲ介导的dsDNA循环对凝血酶的检测具有很高灵敏度,检测范围是0.1-200 pmol/L,检测限是45 fmol/L。Zhao等[40]也将Exo Ⅲ的信号放大原理运用到检测凝血酶的生物传感器中。凝血酶存在时,两个TBA亚基能自组装成完整的适配体以识别和结合凝血酶,由此使相连的短双链区域稳定存在,Exo Ⅲ发挥降解作用,然后借助T4连接酶和铜纳米颗粒的性质实现信号检测,使传感器表现出较高的灵敏性,检测范围是100 fmol/L-1 nmol/L,检测限为20.3 fmol/L。
癌胚抗原(Carcinoembryonic antigen,CEA)是一种与肺癌、乳腺癌和结肠癌相关的广谱肿瘤生物标志物[41]。由于在患病早期,生物标志物的浓度比较低,因此需要可靠的、灵敏度较高的方法对其进行定量检测[42]。He等[43]首次将Exo Ⅲ介导的信号放大策略与“DNA walker”纳米机器巧妙结合,开发出一种检测癌胚抗原的生物传感器(图 5-B)。H1和H2是可通过自身杂交成茎环的序列,癌胚抗原存在时,会与H1中的适配体部分结合导致H1的构象变化,从茎环结构转变为CEA@H1复合体结构,暴露出的a、b区域,于是与H2的3'凸出端开始杂交,形成平末端,使Exo Ⅲ从H2的3'端开始降解,释放出CEA@H1和walker DNA。CEA@H1将与新的H2结合参与新的循环,而walker DNA则通过与硅微球表面修饰的H3-e*发生作用形成H3@walker DNA,从而暴露i,因此i可与H4的i*杂交引发链置换反应最终形成H3@H4@walker DNA三重复合体。由于H3@H4复合体倾向于形成热力学上最有利的构象,因此walker DNA会被释放参与下一个反应循环。H3@H4复合体中,k会形成G4结构,与NMM染料结合发出强烈的荧光信号。该传感器具有很高的靶标特异性,并实现了1.2 pg/mL的低检测限,线性范围是10 pg/mL-100 ng/mL。
Lu等[44]先将转录因子NF-κB p50的信号转换成ssDNA信号,再利用Exo Ⅲ对该ssDNA进行循环信号放大,对NF-κB p50进行检测(图 5-C)。首先,ON1和ON2杂交形成dsDNA,且包括了一段NF-κB p50识别位点。不存在NF-κB p50时,由于dsDNA为平末端因此会被Exo Ⅲ完全降解,无法产生后续反应。存在NF-κB p50时,它会结合到dsDNA上,保护ON2的3'端抵抗Exo Ⅲ的降解,而由于ON1的3'端没有保护,因此被降解,从而释放出ON3。以上属于NF-κB p50到ssDNA的信号转换部分。修饰亚甲蓝的ON4能够与ON3杂交,并在Exo Ⅲ的作用下开启循环反应。NF-κB p50的检测范围是10-5 000 pmol/L,检测限是10 pmol/L。Li等[45]还使用了银纳米簇分子信标(AgMBs)的特点设计了荧光开关,同时利用Exo Ⅲ的切割和信号循环性质,对多种转录因子进行定量分析。除了用Exo Ⅲ检测凝血酶、抗原和转录因子外,Yang和Gao[46]还将AuNPs的聚沉性质与Exo Ⅲ联合使用,设计了一种检测叶酸受体的可视生物传感器。
2.5 Exo Ⅲ介导的酶活检测功能核酸生物传感器Xue等[47]将DNA固定在磁珠上,协同Exo Ⅲ的循环指数放大效应,建立了一种检测甲基转移酶(DNA adenine methyltransferase,DAM)活性的荧光磁性生物传感器(图 6-A)。首先将含有G4序列的单链(绿色)与分子信标杂交成dsDNA,组装在磁珠上(图 6-Aa)。其次,由于发夹探针具有GATC识别位点,因此根据DAM和Dpn Ⅰ内切酶的性质,联合使用对发夹探针进行先甲基化再切割,释放出TSP单链(图 6-Ab),杂交到磁珠上的dsDNA,形成平末端,此时Exo Ⅲ从3'端开始降解,释放出TSP和新TSP,TSP继续循环Ⅰ反应,新TSP进入循环Ⅱ(图 6-Ac)。这两种循环的原理类似,且每轮循环后的降解产物即磁珠上残留的G4序列(绿色),会与ZnPPIX形成ZnPPIX/G4复合物,发出强烈的荧光信号,从而对DAM的酶活进行定量检测,最低检测限为2.0×10-4 U/mL。Li等[48]和Liu等[49]也同样利用了DAM和Dpn Ⅰ的性质,结合电化学或2-氨基嘌呤的荧光特性对DAM的活性进行了测定。
端粒酶是一种能够将(TTAGGG)n串联重复序列添加到染色体末端维持端粒长度的核糖核蛋白[50]。大多数正常的细胞中,端粒酶活性处于抑制状态,但人类85%癌症细胞中,端粒酶活性出现上调或者再激活,因此端粒酶活性的检测对于癌症诊断,筛选抗癌药物或者癌症治疗的评估具有十分重要的意义[51-54]。Min等[55]通过点击反应将疏水的TPE-Py与亲水的线性DNA序列连接形成TPE-Py-DNA,作为荧光报告基因(图 6-B)。端粒酶存在时,能够将重复单元(TTAGGG)n添加到TS primer末端,此延长产物会与TPE-Py-DNA杂交形成dsDNA,随后被Exo Ⅲ消化释放出延长产物和TPE-Py。释放后的产物会继续与新的TPE-Py-DNA杂交开始下一个循环,而疏水的TPE-Py将聚集在一起产生与端粒酶活性相关的荧光信号。该方法不仅可以对不同的细胞系进行胞内端粒酶的活性检查,而且能根据尿液样品将癌症病人与正常人区准确地区分开,具有很高的临床应用价值。除了DAM和端粒酶,还可将Exo Ⅲ用于T4多聚核苷酸激酶[56-57]和糖苷酶[58]等活性的检测。
2.6 Exo Ⅲ介导的其他靶物质功能核酸生物传感器2012年,Niu等[59]利用一条标记有荧光基团的DNA单链发夹探针,通过Exo Ⅲ介导的信号循环放大反应对可卡因进行了简单高效的定量检测。2013年及2016年,Zhu等[9]和Zhao等[60]根据ATP依赖的T4连接酶的酶活性质,以及Exo Ⅲ对底物的切割特性,设计了特异性检测ATP浓度的简单、灵敏的生物传感器,检测限分别为0.2 nmol/L和20 pmol/L。2016年,Ramezani等[61]将AuNPs淬灭荧光的特性,结合卡那霉素的适配体及其互补链,设计了一种Exo Ⅲ介导的信号循环放大生物传感器,可对血清和牛奶中的卡那霉素含量进行检测。Fu等[62]通过3条DNA中的脱碱基位点可特异性识别三聚氰胺的原理,设计了一个由Exo Ⅲ介导的三聚氰胺生物传感器,线性范围是1 nmol/L-0.5 μmol/L,检测限是0.43 nmol/L。Sun等[63]将正电荷的磁珠与ssDNA通过非共价静电吸引作用连接在一起,由于致病菌带有大量负电荷表面,因此与磁珠会发生相互作用,DNA则被取代释放到溶液中参与Exo Ⅲ介导的DNA信号放大过程,从而对致病菌实现定量检测的目的。
3 Exo Ⅲ与其他信号放大策略相结合的生物传感器为了提高生物传感器的灵敏性,信号放大技术被广泛运用到传感器的设计当中,从而提高输出信号的强度。PCR是最普遍的DNA扩增技术之一,可是PCR有一些限制因素,如需要热循环和高温变性[64]。而RCA和HCR属于等温技术,不需要热循环,能够在更广阔的领域进行应用。将Exo Ⅲ与RCA或者HCR相结合的双重信号放大策略,比单纯的一重信号放大具有更强的信号输出和更高的灵敏性,甚至可以将检测限降低至amol/L级。
3.1 Exo Ⅲ与RCA相结合的生物传感器RCA是一种利用扩增酶(如phi29、Bst、Klenow等)进行高效等温扩增的过程[65]。在RCA过程中,聚合酶将单核苷酸持续地添加到与环形模板退火杂交后的引物上,产生具有重复序列的长单链,甚至能够使靶物质的检测达到一个单分子的水平,并且在溶液[7]、固相载体[66]或者复杂的生理环境中[67](如细胞表面或者细胞内)均可进行。
Zhou等[68]将Exo Ⅲ介导的靶标循环体系与RCA技术合二为一,设计出一种具有超高灵敏性的电化学生物传感器,用来检测tDNA(即一种与阿尔茨海默病相关的生物标志物)(图 7-A)。先将hDNA通过Au-SH键修饰在电极表面,当tDNA与hDNA杂交后,Exo Ⅲ通过切割hDNA,释放出tDNA与更多的hDNA杂交、切割及释放,形成一个往复循环。同时,hDNA被切割后的剩余片段能够促发RCA,产生大量富G4序列的长链,与hemin形成的hemin/G4复合物具有拟过氧化物酶活性并产生电化学信号。这种双重信号放大的方法具有很高的灵敏性,检测限为3.3 amol/L。
双酚A是用于合成聚碳酸酯和环氧树脂的单体,在人体内具有雌激素的活性[69]。聚碳酸酯和环氧树脂的应用十分广泛,可被用作奶瓶、杯子和医疗器械等的原材料。暴露在双酚A的环境中,可能引发各种疾病包括心血管疾病、癌症、出生缺陷或者生殖障碍等,即使非常低的含量也会对人体造成危害[70],因此对双酚A的定量检测方法要求具备较高的灵敏性和选择性。Li等[71]利用抗双酚A的适配体设计了一种RCA/Exo Ⅲ级联放大生物传感器(图 7-B)。首先,抗双酚A的适配体(深蓝色,用于识别双酚A)与触发序列(绿色,用于促发RCA)形成双链DNA。双酚A存在时,抗双酚A的适配体会打开构象与双酚A结合,同时释放出触发序列,引发RCA反应,作为初级信号放大。此时,RCA产物在发夹的辅助下促发Exo Ⅲ介导二级信号放大,累积丰富的灯笼式G4结构与ZnPPIX结合,产生较强的荧光信号,呈现出极高的灵敏度,检测限为5.4×10-17 mol/L。此外,基于Exo Ⅲ-RCA的信号放大优势,Xue等[72]对DNA进行了检测,检测限为0.51 amol;Liu等[73]对核酸酶S1(检测限为5×10-7 U/μL)和ATP(检测限为5×10-4 μmol/L)进行了检测。
3.2 Exo Ⅲ与HCR相结合的生物传感器2004年,Dirks和Pierce首次提出HCR的概念[74],这是一种无酶的体外核酸等温信号放大技术。该技术需要两条可杂交互补且含有黏性末端的发夹探针,以及引发探针。两条发夹探针可以稳定存在于溶液中,一旦加入引发探针,就会触发HCR反应的开始,使两条发夹探针相继打开,形成具有多个重复单元的长链dsDNA,直到原料耗尽。与其他核酸扩增方法相比,HCR最大的优势在于无酶参与,为生物传感器的研究工作提供了很大便利。
Sun等[75]利用HCR、Exo Ⅲ结合表面增强拉曼光谱(Surface-enhanced Raman spectroscopy,SE-RS)的方法设计了一个双重信号放大的生物传感器用于心肌梗塞基因的检测(图 8-A)。靶基因存在时,会打开发夹probe 1互补杂交成dsDNA,Exo Ⅲ从probe 1的3'平末端开始切割,释放出靶基因进入下个循环,同时剩余的切割产物作为促发子打开Hp1继而启动HCR反应,生成线性长dsDNA。由于Hp1和Hp2末端带有Tamra标签,因此可以呈现出具有特征性的SERS指纹信号。此方法实现了1 fmol/L-10 nmol/L的线性关系,检测限为1 fmol/L。
Yu等[76]不仅利用Exo Ⅲ和HCR进行双重信号放大,而且还利用了石墨烯和荧光嵌入剂SYBR GREEN Ⅰ,建立了一种检测Hg2+荧光生物传感器(图 8-B)。首先利用T-Hg2+-T的原理使HP DNA和assisted DNA之间通过链置换反应形成dsDNA,随后Exo Ⅲ从其3'端平末端开始降解导致Hg2+和assisted DNA循环利用,同时释放出DNA触发链(Trigger strand),开始HCR形成长链双螺旋结构。SYBR GREEN Ⅰ可以嵌入刚性dsDNA中发出较强的荧光,而不被石墨烯吸附。通过条件优化,该传感器对Hg2+的最低检测限为7.37 pmol/L,线性范围是0-1.5 nmol/L,并且对Hg2+具有较高的选择性。Bao等[77]和Xiong等[78]也将Exo Ⅲ和HCR联用设计了能够检测Hg2+的电化学传感器。Sun等[79]利用Exo Ⅲ介导的DNA循环和HCR检测了腺苷含量。
4 Exo Ⅲ与纳米材料相结合的生物传感器纳米材料指的是由无机或有机材料制备合成,并且在三维空间中至少有一维在100 nm以下的功能材料,由于粒子尺寸非常小,纳米材料产生了不同于传统材料的一些物理、化学特性,如量子尺寸效应和表面效应等[80]。如今各种新型的纳米材料得到了大力发展和应用,包括AuNPs、半导体量子点、碳纳米管、石墨烯和氧化石墨烯、金属纳米簇、碳点和上转换纳米颗粒等,这些纳米材料都可以与适当的分析方法学和检测技术搭载,设计成多样化的生物传感器以检测生物样本或者环境污染中的相关靶物质[81]。
AuNPs不仅稳定性高,具有独特的光学、热学和较好的生物相容性质,还可与荧光基团发生荧光共振能量转移成为极强的荧光淬灭剂[82]。Zhao等[83]利用赭曲霉毒素A的适配体,采用Exo Ⅲ的信号放大手段结合AuNPs的荧光淬灭原理对赭曲霉毒素A进行定量检测,检测限低至4.82 nmol/L。AuNPs不仅能够产生颜色和荧光淬灭效应促进信号转导,还能够通过加载大量信号标签使信号识别产生放大效应,从而提高生物传感器的灵敏度和特异性。例如,Huang等[84]设计了一种多重标记的电化学生物传感器,将3种含有不同Redox标签的信号探针修饰在AuNPs上作为生物条形码放大策略,与Exo Ⅲ介导的酶催化反应组成了双重信号放大系统检测,可以同时对转基因样本中的3种DNA组分进行检测。
量子点(Quantum dots,QDs)是一种很有前景的荧光纳米颗粒,与其他有机染料和荧光蛋白相比,具有荧光强度大、光稳定性高和波长可调的优点[85]。Jie等[85]利用磁珠@Au复合材料的磁性分离特性以及Exo Ⅲ介导的靶标循环放大策略,使用两种可区分的CdSe/ZnS和CdTe QDs荧光探针,对miRNA-141和miRNA-21进行了同步荧光检测,检测范围在5 pmol/L-50 nmol/L之间,检测限是1.5 pmol/L。同年,Jie等[86]在Au-氧化石墨烯纳米复合材料修饰电极上逐级组装CdSe-NH2和CdSe-COOH QDs层,根据双层QDs与Au@Ag之间的电化学发光能量共振转移导致电化学信号变化的原理,结合Exo Ⅲ信号循环放大体系设计了一个检测凝血酶的生物传感器。Zhang等[87]通过一步法合成DNA-ZnS:Mn2+量子点,建立了一种以QDs和WS2纳米片之间的荧光共振能量转移为研究基础的生物传感器,用于检测链霉亲和素。
Wang等[88]将金纳米簇/石墨烯(AuNCs/GR)纳米杂化物标记上SH-DNA和碱性磷酸酶(Alkaline phosphatase,ALP)形成AuNCs/GR-DNA-ALP复合物,作为催化界面。靶DNA引发Exo Ⅲ循环后产生的切割产物,与金电极上的抓捕探针和AuNCs/GR-DNA-ALP中的SH-DNA均部分互补杂交,从而使AuNCs/GR-DNA-ALP固定在电极表面,最终AuNCs/GR-DNA-ALP催化银沉积反应生成银纳米颗粒产生可检测的电化学信号。此外,除了本节提到的金纳米颗粒、量子点、石墨烯及金纳米簇,还有前面提到的银纳米簇分子信标、上转换纳米颗粒等均可与Exo Ⅲ的信号放大策略相结合,大大提高传感器的灵敏度和特异性。
5 总结和展望综上,Exo Ⅲ介导的信号放大策略已被广泛应用于生物传感器的检测领域,其最显著的优势在于Exo Ⅲ酶切速度快,能够在较短的时间内完成信号放大过程;其次,常温即可反应,活性不易丢失,不需热启动步骤;可与各种常见的信号输出方式(如荧光法、比色法和电化学法等)或其他信号放大方式(RCA或HCR)相结合,以及能够借助磁珠或者AuNPs等新兴纳米材料提高检测效果,具有较好的兼容性。另外,还能够将不同种类的靶物质均转换成核酸信号,再进行Exo Ⅲ介导的信号放大,即不受靶物质种类的约束,这些特点充分满足了生物传感器快速、简便的要求。需要注意的是,Exo Ⅲ是一种具有多种活性的核酸酶,在使用过程中需要考虑该酶的切割性质,同时溶液中金属离子的作用可能会影响酶活,尽量避免错误切割、酶活受抑制等问题的出现。
目前Exo Ⅲ介导的生物传感器主要应用于DNA、蛋白质及小分子物质的检测,在实际应用方面,偏向于与疾病相关的生物标志物检测,以及环境中重金属的检测等。未来Exo Ⅲ介导的信号放大策略在生化分析的发展趋势可能包括以下几方面:(1)简便生物传感器的大量应用:传感器的设计可繁可简,最终目的是实际应用,争取用更短的时间更低的成本实现利益最大化。与复杂的传感器相比,设计简单的传感器不仅节约成本,而且完全可以通过Exo Ⅲ介导一重或双重信号放大的方式达到较低的检测限,为开拓应用市场提供了有利的条件保障。(2)纳米材料的多样化搭载:随着新型纳米材料的不断更新和应用,使生物传感器在灵敏度和分析方法的应用方面有很大提升。然而与Exo Ⅲ搭载的新型纳米材料主要集中在石墨烯、金纳米颗粒和量子点等,在其他类型纳米材料如水凝胶等的应用还有很大空间。(3)胞内检测:目前由Exo Ⅲ介导的信号循环放大检测主要集中在体外实验,对胞内的检测虽有文献报道,但这方面的应用较少,对于临床诊断来说,胞内检测和胞内成像无疑将成为未来的研究重点。(4)多重检测:Exo Ⅲ价格合理、应用简便,可通过核酸链的精心设计或者多酶联用,进行多靶标物质的检测,因此由Exo Ⅲ介导的多功能生物传感器或将成为未来的发展方向。(5)不同金属离子的检测:目前关于Exo Ⅲ检测金属离子的文献基本局限于利用T-Hg2+-T的原理来检测Hg2+,因此对其他金属离子的检测还需进一步探索和研究。(6)在病毒、细菌感染以及肉品掺假等实际应用领域将取得更多进展。由此可见,Exo Ⅲ高效、简便的特点充分满足了生物传感器的检测需求,具有十分可观的应用前景。
| [1] |
Seeman NC. Nucleic acid junctions and lattices[J]. Journal of Theoretical Biology, 1982, 99(2): 237-247. DOI:10.1016/0022-5193(82)90002-9 |
| [2] |
Wilson DS, Szostak JW. In vitro selection of functional nucleic acids[J]. Annu Rev Biochem, 1999, 68(1): 611-647. DOI:10.1146/annurev.biochem.68.1.611 |
| [3] |
Navani NK, Li Y. Nucleic acid aptamers and enzymes as sensors[J]. Curr Opin Chem Biol, 2006, 10(3): 272-281. DOI:10.1016/j.cbpa.2006.04.003 |
| [4] |
Perumal V, Hashim U. Advances in biosensors:Principle, architecture and applications[J]. Journal of Applied Biomedicine, 2014, 12(1): 1-15. DOI:10.1016/j.jab.2013.02.001 |
| [5] |
Gibriel AA, Adel O. Advances in ligase chain reaction and ligation-based amplifications for genotyping assays:Detection and applications[J]. Mutat Res, 2017, 773: 66-90. DOI:10.1016/j.mrrev.2017.05.001 |
| [6] |
Cheng N, Zhu P, Xu Y, et al. High-sensitivity assay for Hg(Ⅱ)and Ag(Ⅰ)ion detection:a new class of droplet digital PCR logic gates for an intelligent DNA calculator[J]. Biosens Bioelectron, 2016, 84: 1-6. DOI:10.1016/j.bios.2016.04.084 |
| [7] |
Jiang H, Liang Z, Ma Y, et al. G-quadruplex fluorescent probe-mediated real-time rolling circle amplification strategy for highly sensitive microRNA detection[J]. Anal Chim Acta, 2016, 943: 114-122. DOI:10.1016/j.aca.2016.09.019 |
| [8] |
Zhu G, Li Y, Zhang C. Simultaneous detection of mercury(Ⅱ)and silver(Ⅰ)ions with picomolar sensitivity[J]. Chem Commun, 2014, 50(5): 572-574. DOI:10.1039/C3CC46884D |
| [9] |
Zhu W, Zhao Z, Li Z, et al. A label free exonuclease Ⅲ-aided fluorescence assay for adenosine triphosphate based on graphene oxide and ligation reaction[J]. New J Chem, 2013, 37(4): 927-932. DOI:10.1039/c2nj41055a |
| [10] |
Gerasimova YV, Kolpashchikov DM. Enzyme-assisted target recycling(EATR)for nucleic acid detection[J]. Chem Soc Rev, 2014, 43(17): 6405-6438. DOI:10.1039/C4CS00083H |
| [11] |
Henikoff S. Unidirectional digestion with exonuclease Ⅲ creates targeted breakpoints for DNA sequencing[J]. Gene, 1984, 28(3): 351-359. DOI:10.1016/0378-1119(84)90153-7 |
| [12] |
Guo L, Wu R. New rapid methods for DNA sequencing based on exonuclease Ⅲ digestion followed by repair synthesis[J]. Nucleic Acids Research, 1982, 10(6): 2065-2084. DOI:10.1093/nar/10.6.2065 |
| [13] |
Weiss B. Endonuclease Ⅱ of Escherichia coli is exonuclease Ⅲ[J]. J Biol Chem, 1976, 251(7): 1896-1901. |
| [14] |
Mol CD, Kuo CF, Thayer MM, et al. Structure and function of the multifunctional DNA-repair enzyme exonuclease Ⅲ[J]. Nature, 1995, 374(6520): 381-386. DOI:10.1038/374381a0 |
| [15] |
Richardson CC, Kornberg A. A deoxyribonucleic acid phosphatase-exonuclease from Escherichia coli[J]. J Biol Chem, 1964, 239: 242-250. |
| [16] |
Richardson CC, Lehman I, Kornberg A. A deoxyribonucleic acid phosphatase-exonuclease from Escherichia coli Ⅱ. Characterization of the exonuclease activity[J]. J Biol Chem, 1964, 239(1): 251-258. |
| [17] |
Wen L, Xu F, He X, et al. Application progress of exonuclease-assisted signal amplification strategies in biochemical analysis[J]. Chinese Journal of Anal Chem, 2015, 43(11): 1620-1628. DOI:10.1016/S1872-2040(15)60874-X |
| [18] |
Rogers SG, Weiss B. Exonuclease Ⅲ of Escherichia coli K-12, an AP endonuclease[J]. Methods Enzymol, 1980, 65: 201-211. DOI:10.1016/S0076-6879(80)65028-9 |
| [19] |
Wu R, Ruben G, Siegel B, et al. Synchronous digestion of SV40 DNA by exonuclease Ⅲ[J]. Biochemistry, 1976, 15(4): 734-740. DOI:10.1021/bi00649a003 |
| [20] |
Donelson JE, Wu R. Nucleotide sequence analysis of deoxyribonuc-leic acid Ⅶ. characterization of Escherichia coli exonuclease Ⅲ activity for possible use in terminal nucleotide sequence analysis of duplex deoxyribonucleic acid[J]. J Biol Chem, 1972, 247(14): 4661-4668. |
| [21] |
Lawley PD. Effects of some chemical mutagens and carcinogens on nucleic acids[J]. Progress in Nucleic Acid Research and Molecular Biology, 1966, 5: 89-131. DOI:10.1016/S0079-6603(08)60232-9 |
| [22] |
Friedberg EC, Goldthwait DA. Endonuclease Ⅱ of E. coli, Ⅰ. Isolation and Purification[J]. Proc Natl Acad Sci USA, 1969, 62(3): 934-940. DOI:10.1073/pnas.62.3.934 |
| [23] |
Friedberg EC, Hadi SM, Goldthwait DA. Endonuclease Ⅱ of Escherichia coli Ⅱ. Enzyme properties and studies on the degradation of alkylated and native deoxyribonucleic acid[J]. J Biol Chem, 1969, 244(21): 5879-5889. |
| [24] |
Yajko DM, Weiss B. Mutations simultaneously affecting endonuclease Ⅱ and exonuclease Ⅲ in Escherichia coli[J]. Proc Natl Acad Sci, 1975, 72(2): 688-692. DOI:10.1073/pnas.72.2.688 |
| [25] |
Iorio MV, Croce CM. MicroRNA dysregulation in cancer:diagnostics, monitoring and therapeutics. A comprehensive review[J]. EMBO Molecular Medicine,, 2012, 4(3): 143-159. DOI:10.1002/emmm.201100209 |
| [26] |
Huang R, Chiu W, Li Y, et al. Detection of microRNA in tumor cells using exonuclease Ⅲ and graphene oxide-regulated signal amplification[J]. ACS Appl Mater Interfaces, 2014, 6(24): 21780-21787. DOI:10.1021/am500534g |
| [27] |
Min X, Zhang M, Huang F, et al. Live cell microRNA imaging using exonuclease Ⅲ-aided recycling amplification based on aggregation-induced emission luminogens[J]. ACS Appl Mater Interfaces, 2016, 8(14): 8998-9003. DOI:10.1021/acsami.6b01581 |
| [28] |
Cai Z, Chen Y, Lin C, et al. A dual-signal amplification method for the DNA detection based on exonuclease Ⅲ[J]. Biosens Bioelectron, 2014, 61: 370-373. DOI:10.1016/j.bios.2014.05.046 |
| [29] |
Zuo X, Xia F, Xiao Y, et al. Sensitive and selective amplified fluorescence DNA detection based on exonuclease Ⅲ-aided target recycling[J]. J Am Chem Soc, 2010, 132(6): 1816-1818. DOI:10.1021/ja909551b |
| [30] |
Huang Y, Gao Z, Luo H, et al. Sensitive detection of HIV gene by coupling exonuclease Ⅲ-assisted target recycling and guanine nanowire amplification[J]. Sensors and Actuators B:Chemical, 2017, 238: 1017-1023. DOI:10.1016/j.snb.2016.07.144 |
| [31] |
Lin J, Yan Y, Ou T, et al. Effective detection and separation method for G-quadruplex DNA based on its specific precipitation with Mg2+[J]. Biomacromolecules, 2010, 11(12): 3384-3389. DOI:10.1021/bm100862k |
| [32] |
Chen Y, Duong H, Wen S, et al. Exonuclease Ⅲ-assisted upconversion resonance energy transfer in a wash-free suspension DNA assay[J]. Anal Chem, 2018, 90: 663-668. DOI:10.1021/acs.analchem.7b04240 |
| [33] |
Luo C, Tang H, Cheng W, et al. A sensitive electrochemical DNA biosensor for specific detection of Enterobacteriaceae bacteria by Exonuclease Ⅲ-assisted signal amplification[J]. Biosens Bioelectron, 2013, 48: 132-137. DOI:10.1016/j.bios.2013.03.084 |
| [34] |
Zhang B, Meng H, Wang X, et al. Fe3+ doped ZnO-Ag photocatalyst for photoelectrochemical sensing platform of ultrasensitive Hg2+ detection using exonuclease Ⅲ-assisted target recycling and DNAzyme-catalyzed amplification[J]. Sensors and Actuators B:Chemical, 2018, 255: 2531-2537. DOI:10.1016/j.snb.2017.09.058 |
| [35] |
Gan X, Zhao H, Chen S, et al. Electrochemical DNA sensor for specific detection of picomolar Hg(Ⅱ)based on exonuclease Ⅲ-assisted recycling signal amplification[J]. Analyst, 2015, 140(6): 2029-2036. DOI:10.1039/C5AN00082C |
| [36] |
Hong M, Zeng B, Li M, et al. An ultrasensitive conformation-dependent colorimetric probe for the detection of mercury(Ⅱ)using exonuclease Ⅲ-assisted target recycling and gold nanoparticles[J]. Microchimica Acta, 2018, 185(1): 72. DOI:10.1007/s00604-017-2536-1 |
| [37] |
Franchini M, Mannucci PM. Thrombin and cancer:from molecular basis to therapeutic implications[J]. Semin Thromb Hemost, 2012, 38(1): 95-101. DOI:10.1055/s-0031-1300955 |
| [38] |
Marchetti M, Diani E, Ten Cate H, et al. Characterization of the thrombin generation potential of leukemic and solid tumor cells by calibrated automated thrombography[J]. Haematologica, 2012, 97(8): 1173-1180. DOI:10.3324/haematol.2011.055343 |
| [39] |
Yang M, Chen Y, Xiang Y, et al. Target-induced structure switching of DNA for label-free and ultrasensitive electrochemiluminescent detection of proteins[J]. Chem Commun, 2014, 50(24): 3211-3213. DOI:10.1039/C4CC00694A |
| [40] |
Zhao J, Xin M, Cao Y, et al. An electrochemical aptasensor for thrombin detection based on the recycling of exonuclease Ⅲ and double-stranded DNA-templated copper nanoparticles assisted signal amplification[J]. Anal Chim Acta, 2015, 860: 23-28. DOI:10.1016/j.aca.2014.12.026 |
| [41] |
Rong Z, Wang C, Wang J, et al. Magnetic immunoassay for cancer biomarker detection based on surface-enhanced resonance Raman scattering from coupled plasmonic nanostructures[J]. Biosens Bioelectron, 2016, 84: 15-21. DOI:10.1016/j.bios.2016.04.006 |
| [42] |
Petricoin EF, Belluco C, Araujo RP, et al. The blood peptidome:a higher dimension of information content for cancer biomarker discovery[J]. Nature Reviews Cancer, 2006, 6(12): 961-967. DOI:10.1038/nrc2011 |
| [43] |
He M, Wang K, Wang W, et al. Smart DNA machine for carcinoembryonic antigen detection by exonuclease Ⅲ-assisted target recycling and DNA walker cascade amplification[J]. Anal Chem, 2017, 89(17): 9292-9298. DOI:10.1021/acs.analchem.7b02073 |
| [44] |
Lu L, Su H, Li F. Ultrasensitive homogeneous electrochemical detection of transcription factor by coupled isothermal cleavage reaction and cycling amplification based on exonuclease Ⅲ[J]. Anal Chem, 2017, 89(16): 8328-8334. DOI:10.1021/acs.analchem.7b01538 |
| [45] |
Li B, Xu L, Chen Y, et al. Sensitive and label-free fluorescent detection of transcription factors based on DNA-Ag nanoclusters molecular beacons and exonuclease Ⅲ-assisted signal amplification[J]. Anal Chem, 2017, 89(14): 7316-7323. DOI:10.1021/acs.analchem.7b00055 |
| [46] |
Yang X, Gao Z. Gold nanoparticle-based exonuclease Ⅲ signal amplification for highly sensitive colorimetric detection of folate receptor[J]. Nanoscale, 2014, 6(6): 3055-3058. DOI:10.1039/C3NR06139F |
| [47] |
Xue Q, Zhang Y, Xu S, et al. Magnetic nanoparticles-cooperated fluorescence sensor for sensitive and accurate detection of DNA methyltransferase activity coupled with exonuclease Ⅲ-assisted target recycling[J]. Analyst, 2015, 140(22): 7637-7644. DOI:10.1039/C5AN01546D |
| [48] |
Li X, Xie Z, Wang W, et al. Rapid detection of Dam methyltransferase activity based on the exonuclease Ⅲ-assisted isothermal amplification cycle[J]. Analytical Methods, 2016, 8(13): 2771-2777. DOI:10.1039/C5AY03397G |
| [49] |
Liu H, Ma C, Zhou M, et al. Quencher-free fluorescence strategy for detection of DNA methyltransferase activity based on exonuclease Ⅲ-assisted signal amplification[J]. Analytical and BioAnal Chem, 2016, 408(28): 8111-8116. DOI:10.1007/s00216-016-9914-0 |
| [50] |
Morin GB. The human telomere terminal transferase enzyme is a ribonucleoprotein that synthesizes TTAGGG repeats[J]. Cell, 1989, 59(3): 521-529. DOI:10.1016/0092-8674(89)90035-4 |
| [51] |
Kim NW, Piatyszek MA, Prowse KR, et al. Specific association of human telomerase activity with immortal cells and cancer[J]. Science, 1994, 266(5193): 2011-2015. DOI:10.1126/science.7605428 |
| [52] |
Hiyama E, Yokoyama T, Tatsumoto N, et al. Telomerase activity in gastric cancer[J]. Cancer Res, 1995, 55(15): 3258-3262. |
| [53] |
Sommerfeld HJ, Meeker AK, Piatyszek MA, et al. Telomerase activity:a prevalent marker of malignant human prostate tissue[J]. Cancer Res, 1996, 56(1): 218-222. |
| [54] |
Kinoshita H, Ogawa O, Kakehi Y, et al. Detection of telomerase activity in exfoliated cells in urine from patients with bladder cancer[J]. J Natl Cancer Inst, 1997, 89(10): 724-730. DOI:10.1093/jnci/89.10.724 |
| [55] |
Min X, Xia L, Zhuang Y, et al. An AIEgens and exonuclease Ⅲ ai-ded quadratic amplification assay for detecting and cellular imaging of telomerase activity[J]. Science Bulletin, 2017, 62(14): 997-1003. DOI:10.1016/j.scib.2017.06.008 |
| [56] |
Wang Y, Wu Y, Wang Y, et al. A sensitive immobilization-free electrochemical assay for T4PNK activity based on exonuclease Ⅲ-assisted recycling[J]. RSC Advances, 2015, 5(92): 75348-75353. DOI:10.1039/C5RA12849H |
| [57] |
Sun N, Kong R, Qu F, et al. An amplified fluorescence detection of T4 polynucleotide kinase activity based on coupled exonuclease Ⅲ reaction and a graphene oxide platform[J]. Anal, 2015, 140(6): 1827-1831. DOI:10.1039/C4AN01953A |
| [58] |
Zhao J, Ma Y, Kong R, et al. Tungsten disulfide nanosheet and exonuclease Ⅲ co-assisted amplification strategy for highly sensitive fluorescence polarization detection of DNA glycosylase activity[J]. Anal Chim Acta, 2015, 887: 216-223. DOI:10.1016/j.aca.2015.07.006 |
| [59] |
Niu S, Lou X, Jiang Y, et al. A novel fluorescence sensor for cocaine with signal amplification through cycling exo-cleaving with a hairpin probe[J]. Anal Lett, 2012, 45(13): 1919-1927. DOI:10.1080/00032719.2012.677972 |
| [60] |
Zhao T, Lin C, Yao Q, et al. A label-free electrochemiluminescent sensor for ATP detection based on ATP-dependent ligation[J]. Talanta, 2016, 154: 492-497. DOI:10.1016/j.talanta.2016.03.091 |
| [61] |
Ramezani M, Danesh NM, Lavaee P, et al. A selective and sensitive fluorescent aptasensor for detection of kanamycin based on catalytic recycling activity of exonuclease Ⅲ and gold nanoparticles[J]. Sensors and Actuators B:Chemical, 2016, 222: 1-7. DOI:10.1016/j.snb.2015.08.024 |
| [62] |
Fu C, Liu C, Li Y, et al. Homogeneous electrochemical biosensor for melamine based on DNA triplex structure and exonuclease Ⅲ-assisted recycling amplification[J]. Anal Chem, 2016, 88(20): 10176-10182. DOI:10.1021/acs.analchem.6b02753 |
| [63] |
Sun Y, Zhao C, Yan Z, et al. Simple and sensitive microbial pathogen detection using a label-free DNA amplification assay[J]. Chem Commun, 2016, 52(47): 7505-7508. DOI:10.1039/C6CC02672A |
| [64] |
Gill P, Ghaemi A. Nucleic acid isothermal amplification technologies:a review[J]. Nucleosides, Nucleotides, and Nucleic Acids, 2008, 27(3): 224-243. DOI:10.1080/15257770701845204 |
| [65] |
Ali MM, Li F, Zhang Z, et al. Rolling circle amplification:a versatile tool for chemical biology, materials science and medicine[J]. Chem Soc Rev, 2014, 43(10): 3324-3341. DOI:10.1039/c3cs60439j |
| [66] |
Chen A, Ma S, Zhuo Y, et al. In situ electrochemical generation of electrochemiluminescent silver naonoclusters on target-cycling synchronized rolling circle amplification platform for microRNA detection[J]. Anal Chem, 2016, 88(6): 3203-3210. DOI:10.1021/acs.analchem.5b04578 |
| [67] |
Deng R, Zhang K, Sun Y, et al. Highly specific imaging of mRNA in single cells by target RNA-initiated rolling circle amplification[J]. Chem Sci, 2017, 8(5): 3668-3675. DOI:10.1039/C7SC00292K |
| [68] |
Zhou X, Guo S, Gao J, et al. Cascade dual-signal enhancement by integrating exonuclease Ⅲ-assisted target-recycling and rolling circle amplification for ultrasensitive electrochemical detection of DNA[J]. J Electrochem Soci, 2017, 164(13): B603-B609. DOI:10.1149/2.0511713jes |
| [69] |
Ragavan K, Rastogi NK, Thakur M. Sensors and biosensors for analysis of bisphenol-A[J]. TrAC Trends in Anal Chem, 2013, 52: 248-260. DOI:10.1016/j.trac.2013.09.006 |
| [70] |
Rezg R, El-Fazaa S, Gharbi N, et al. Bisphenol A and human chronic diseases:current evidences, possible mechanisms, and future perspectives[J]. Environ Int, 2014, 64: 83-90. DOI:10.1016/j.envint.2013.12.007 |
| [71] |
Li X, Song J, Xue Q, et al. A label-free and sensitive fluorescent qualitative assay for bisphenol a based on rolling circle amplifica-tion/exonuclease Ⅲ-combined cascade amplification[J]. Nanomaterials, 2016, 6(10): 190. DOI:10.3390/nano6100190 |
| [72] |
Xue Q, Lv Y, Cui H, et al. A DNA nanomachine based on rolling circle amplification-bridged two-stage exonuclease Ⅲ-assisted recycling strategy for label-free multi-amplified biosensing of nucleic acid[J]. Anal Chim Acta, 2015, 856: 103-109. DOI:10.1016/j.aca.2014.11.035 |
| [73] |
Liu C, Kong X, Yuan J, et al. A dual-amplification fluorescent sensing platform for ultrasensitive assay of nuclease and ATP based on rolling circle replication and exonuclease Ⅲ-aided recycling[J]. RSC Advances, 2015, 5(92): 75055-75061. DOI:10.1039/C5RA13301G |
| [74] |
Dirks RM, Pierce NA. Triggered amplification by hybridization chain reaction[J]. Proc Natl Acad Sci USA, 2004, 101(43): 15275-15278. DOI:10.1073/pnas.0407024101 |
| [75] |
Sun Y, Peng P, Guo R, et al. Exonuclease Ⅲ-boosted cascade reactions for ultrasensitive SERS detection of nucleic acids[J]. Biosens Bioelectron, 2018, 104: 32-38. DOI:10.1016/j.bios.2017.12.047 |
| [76] |
Yu L, Lan W, Xu H, et al. Label-free detection of Hg2+ based on Hg2+-triggered toehold binding, Exonuclease Ⅲ assisted target recycling and hybridization chain reaction[J]. Sensors and Actuators B:Chemical, 2017, 248: 411-418. DOI:10.1016/j.snb.2017.03.153 |
| [77] |
Bao T, Wen W, Zhang X, et al. An exonuclease-assisted amplification electrochemical aptasensor for Hg2+ detection based on hybridization chain reaction[J]. Biosens Bioelectron, 2015, 70: 318-323. DOI:10.1016/j.bios.2015.03.065 |
| [78] |
Xiong E, Zhang X, Liu Y, et al. An electrochemical biosensor for sensitive detection of Hg2+ based on exonuclease Ⅲ-assisted target recycling and hybridization chain reaction amplification strategies[J]. Analytical Methods, 2016, 8(9): 2106-2111. DOI:10.1039/C5AY02793D |
| [79] |
Sun J, Jiang W, Zhu J, et al. Label-free fluorescence dual-amplified detection of adenosine based on exonuclease Ⅲ-assisted DNA cycling and hybridization chain reaction[J]. Biosens Bioelectron, 2015, 70: 15-20. DOI:10.1016/j.bios.2015.03.014 |
| [80] |
Zhong W. Nanomaterials in fluorescence-based biosensing[J]. Analytical and BioAnal Chem, 2009, 394(1): 47-59. DOI:10.1007/s00216-009-2643-x |
| [81] |
Liu J, Hu Y, Yang Y, et al. Emerging functional nanomaterials for the detection of food contaminants[J]. Trends in Food Science & Technology, 2018, 71: 94-106. |
| [82] |
Chen J, Li Z, Ge J, et al. An aptamer-based signal-on bio-assay for sensitive and selective detection of kanamycin A by using gold nanoparticles[J]. Talanta, 2015, 139: 226-232. DOI:10.1016/j.talanta.2015.02.036 |
| [83] |
Zhao Y, Liu R, Sun W, et al. Ochratoxin A detection platform based on signal amplification by exonuclease Ⅲ and fluorescence quenching by gold nanoparticles[J]. Sensors and Actuators B:Chemical, 2018, 255: 1640-1645. DOI:10.1016/j.snb.2017.08.176 |
| [84] |
Huang L, Zheng L, Chen Y, et al. A novel GMO biosensor for rapid ultrasensitive and simultaneous detection of multiple DNA components in GMO products[J]. Biosens Bioelectron, 2015, 66: 431-437. DOI:10.1016/j.bios.2014.12.005 |
| [85] |
Jie G, Zhao Y, Wang X, et al. Multiplexed fluorescence detection of microRNAs based on novel distinguishable quantum dot signal probes by cycle amplification strategy[J]. Sensors and Actuators B:Chemical, 2017, 252: 1026-1034. DOI:10.1016/j.snb.2017.06.107 |
| [86] |
Jie G, Lu Z, Zhao Y, et al. Quantum dots bilayers/Au@ Ag-based electrochemiluminescence resonance energy transfer for detection of thrombin by autocatalytic multiple amplification strategy[J]. Sensors and Actuators B:Chemical, 2017, 240: 857-862. DOI:10.1016/j.snb.2016.09.055 |
| [87] |
Zhang C, Ding C, Zhou G, et al. One-step synthesis of DNA functionalized cadmium-free quantum dots and its application in FRET-based protein sensing[J]. Anal Chim Acta, 2017, 957: 63-69. DOI:10.1016/j.aca.2016.12.024 |
| [88] |
Wang W, Bao T, Zeng X, et al. Ultrasensitive electrochemical DNA biosensor based on functionalized gold clusters/graphene nanohybrids coupling with exonuclease Ⅲ-aided cascade target recycling[J]. Biosens Bioelectron, 2017, 91: 183-189. DOI:10.1016/j.bios.2016.12.017 |