2. 农业部农业转基因生物安全评价(食用)重点实验室,北京 100083
2. Key Laboratory of Safety Assessment of Genetically Modified Organism(Food Safety), Ministry of Agriculture, Beijing 100083
在人类正常的生命活动中,作为遗传物质的DNA经常会遭受各种各样的损伤,其中非常重要的一种损伤形式即为碱基缺失损伤。碱基缺失位点(AP位点)会在机体正常状态下自发形成,也可以由外源因子及氧化损伤诱导产生。在正常的生理状态下,相较于脱嘧啶位点,脱嘌呤位点更为常见。每天每个细胞会产生1-2万个左右脱嘌呤位点,500个左右脱嘧啶位点,根据细胞类型不同,生成的AP位点的数量也会有所不同[1]。在对于AP位点的碱基切除修复(Base excision repair,BER)过程中,脱嘌呤/脱嘧啶核酸内切酶(Apurinic/apyrimidinic endonuclease,APE)扮演着重要的角色,而APE1又是APE家族中非常重要的一种。研究表明,哺乳动物细胞中AP位点的切除主要是在APE1的作用下实现的(占到了整个BER途径的95%以上)[2],其能够特异性地在AP位点处切开DNA链,并与其他酶,如DNA修复酶(PARP)、DNA聚合酶β等,先后作用实现对于碱基缺失损伤的切除修复[3]。
在目前已有的许多快速检测手段中,很多都利用到了各种酶的特异性切割能力。同时,以包括核酸探针、适配体、G四联体在内的功能核酸为基础的生物传感器因其灵活多样的特点在过去几十年间也得到了广泛的应用。而由于APE1对于AP位点有着特异性作用,因此可以利用其与功能核酸联用设计功能核酸生物传感器用于各种靶物质的检测。如设计一段带有AP位点的功能核酸探针,利用APE1对其进行特异性切割,得到的产物能够发出荧光或与其他物质发生特异性结合生成电化学信号。到目前为止,研究者已经制备出了一些与APE1活性相关的功能核酸生物传感器,其中既有APE1介导的用于检测其他酶如DNA糖基化酶的活性,又有对APE1本身的活性进行检测。根据它们的信号输出原理,可以将其分为电化学生物传感器、荧光生物传感器等。同时,其中还有一些实现了与纳米材料如金纳米粒子、二氧化硅纳米粒子的结合,未来有着应用于体内检测的价值。
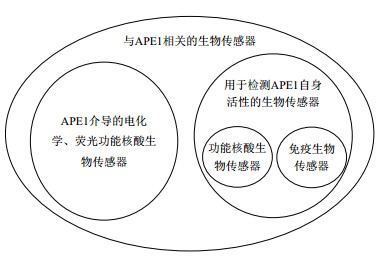
目前与APE1相关的功能核酸生物传感器主要可以分为以下两类:利用APE1特殊的酶活性构建功能核酸生物传感器,通过输出电化学或荧光信号实现对一些靶物质或酶活性的精确检测[4-5];以APE1及其酶活性为目标,将构建好的特殊的核酸探针分别递送至正常细胞及癌症细胞,通过生成的荧光信号证明APE1作为癌症生物标志物的利用价值并实现对于癌症的早期诊断,或者将APE1特异性抗体(anti-APE1)与金纳米粒子(gold nanoparticles,Au NPs)和石墨烯等一些纳米元件结合构建免疫生物传感器,以APE1自身为靶物质对其进行免疫定量检测(图 1)[6-7]。
|
| 图 1 与APE1相关的生物传感器 |
AP位点是一种常见的会严重威胁人体基因组稳定的DNA损伤类型。机体在正常状态下,可能会通过自发脱嘌呤/嘧啶、损伤特异性DNA糖苷酶水解脱氧核糖和受损碱基之间的N-糖苷键或糖苷键的自发水解生成AP位点。由于嘧啶与嘌呤相比与脱氧核糖相连的糖苷键更为稳定,因此脱嘌呤的形式更为常见。常见的水解N-糖苷键生成AP位点的DNA酶有甲基嘌呤DNA糖苷酶(MPG)、人类8-羟基-鸟嘌呤DNA糖苷酶(hOGG1)等。另外,S-腺苷甲硫氨酸(SAM)是一种内源性烷化剂,能将DNA碱基烷化使其带负电,使得生成的N-糖苷键稳定性减弱,从而脱掉碱基,生成AP位点[8-10]。
1.2 APE1基因与蛋白人类的APE1基因定位于14号染色体长臂(14q11.2-12),是目前已知的人类基因组中最短的序列之一,从转录起始位点到polyT尾部的总长度约为2.6 kb,其中含有4个内含子、5个外显子(第一个外显子不编码),有4个外显子的长度都小于200 bp,另一个约为800 bp[11]。这个较大的外显子包含有54%的APE1蛋白编码区和所有的3’非编码区。另外,APE1基因的启动子内不存在TATA盒,而是含有多功能转录起始位点[12]。
人类APE1的相对分子质量为37 000,由318个氨基酸组成(图 2),其C端具有修复活性,N端含有核定位序列,是APE1氧化还原酶活性所必需的(图 3)。其中212位的天冬酰胺(Asn-212)和α8环内的第222-229位氨基酸对于APE1与DNA上的AP位点结合并在AP位点处发挥其酶活性切开DNA链有着十分重要的作用[13-14]。人类APE1的氨基酸序列高度保守,与其他哺乳动物甚至果蝇、细菌之间都存在着高度同源性[9]。研究表明,APE1在人体内普遍存在且总体表达水平都较高,但是在一些恶性肿瘤,尤其是卵巢癌、进展期宫颈癌、非小细胞肺癌和胃癌细胞中的表达有明显增高[15-19],Wen等[20]对APE1与卵巢癌之间的关系进行的研究发现,APE1的过量表达明显促进了卵巢癌症状的发展。Chen等[21]通过体外试验证明,降低APE1的活性有助于提高放射疗法杀灭人类胰腺癌细胞的效果。另外,Ströbel等[22]还发现,APE1介导的DNA修复过程会提高恶性胶质瘤对于抗癌药物的耐受性。这些研究成果证明,APE1可以作为一种生物标志物用于癌症的早期诊断。因此,构建生物传感器实现对APE1活性的高灵敏、高特异性检测对于癌症的早期诊断有着非常重要的意义。

|
| 图 2 人类APE1的氨基酸序列 |
|
| 图 3 人类APE1的三维结构 |
AP位点可以在插入酶的作用下,以另一条完整链为模板根据碱基互补配对原则补充正确的碱基进行修复;也可以在APE1的作用下,从5'-3'方向在缺失碱基片段处将DNA链切开,然后在DNA聚合酶作用下互补合成,最后在DNA连接酶的作用下完成修复[2]。
2 APE1介导的功能核酸电化学生物传感器DNA甲基化是一种常见的DNA损伤形式,在正常状况下不易被生物传感器所检测。利用APE1能够在AP位点处切开DNA的特殊性质,将其与另外一些酶和信号分子联用,Wu等[4]实现了对于DNA甲基化损伤的电化学形式的检测。在DNA发生甲基化以后,首先利用人类烷基腺嘌呤DNA糖基化酶(hAAG)切除甲基化的腺嘌呤,在DNA上生成AP位点,之后APE1能够在AP位点处切开DNA链生成不同长度的小的DNA片段。与正常的长链DNA相比,信号分子Ru(bpy)2(dppz)2+与短链DNA片段的结合能力降低,导致其与DNA结合生成的电化学信号出现明显的减弱(图 4)。实验表明,光电信号减弱的程度与造成DNA甲基化损伤的物质——甲基甲磺酸的量呈现良好的线性关系,该种生物传感器能够检测到低至1 mmol/L的甲基甲磺酸造成的碱基甲基化,在此浓度下传感器表面的DNA中的3-甲基腺嘌呤的量约为42.5 fmol。
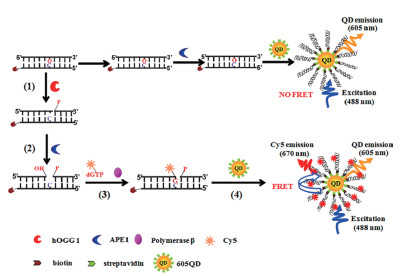
3 APE1介导的功能核酸荧光生物传感器利用包括APE1在内的一些具有特殊活性的酶,将它们与具有荧光特性的物质结合,能够实现对于一些靶物质的荧光信号检测。Wang等[5]利用APE1和DNA聚合酶β以及量子点和荧光基团实现了对于hOGG1活性的检测。他们首先通过链霉亲和素-生物素的连接方式将反义链上修饰有8-氧鸟嘌呤(8-oxoG)的双链DNA固定在量子点表面,当hOGG1存在时,其能识别8-oxoG并切断脱氧核糖和受损碱基之间的N-糖苷键,释放受损碱基形成AP位点。之后APE1在AP位点处发挥作用打开DNA的一条链,随后在DNA聚合酶β的作用下,将标记有荧光基团Cy5的dGTP填补在AP位点处,生成带有荧光标记的DNA双链(图 5)。随后,由于量子点与Cy5之间的荧光能量共振转移(Fluorescence resonance energy transfer,FRET)效应,Cy5的荧光信号即可指示hOGG1的活性。实验证明,当hOGG1的比活在2×10-6 U/μL-2×10-3 U/μL之间时,Cy5的荧光信号与其比活的对数呈现出良好的线性关系。
4 APE1介导的功能核酸纳米生物传感器除了仅仅使用APE1与其他具有特异活性的酶以及一些信号分子外,将APE1与一些纳米材料联用能够使APE1更好地发挥其特异性功能用于一些靶物质的检测。
2015年,Zhang等[23]将APE1与二氧化硅-金纳米粒子结合,实现了对DNA糖基化酶的活性检测。与之前介绍的APE1介导的电化学传感器的基本原理类似,他们将标记有电化学信号分子的DNA探针连接在固定于电极上的二氧化硅-金纳米粒子上,提高了传感器中DNA探针的稳定性及电化学信号转导的灵敏度,同时,他们还将信号分子Ru(bpy)2(dppz)2+修饰在纳米材料单壁碳纳米管(Single-walled carbon nanotube,SWCNT)上,之后再与DNA探针连接,这使得DNA探针上能够标记较多的信号分子,实现了信号放大的功能。当hOGG1的比活在2-200 U/mL之间时,该生物传感器的信号与酶的比活呈现良好的线性关系,同时该生物传感器对于hOGG1的比活的检出限能够低至0.225 U/mL。
5 以APE1为靶物质的生物传感器 5.1 APE1功能核酸生物传感器目前,对APE1活性进行检测的功能核酸生物传感器主要是利用荧光信号对APE1的活性进行表征。通过设计带有AP位点并标有荧光基团的发卡状功能核酸探针,Fang等[24]实现了对于APE1酶活性的定量检测。另外,利用G四联体与特异性物质结合发荧光的原理,Huang等[25]也实现了对APE1酶活性的荧光信号检测。他们首先设计了在茎部具有AP位点的DNA发卡结构。APE1能够在该处切开DNA的一条链生成一个新的3'-OH使得克列诺聚合酶能够在此处开始引发聚合反应打开发卡结构形成双链DNA,在形成的DNA双链中包含有一段Nt.BbvCI核酸内切酶能够识别的序列,Nt.BbvCI核酸内切酶会在此处切断DNA的一条链,此时克列诺聚合酶重新发挥聚合酶作用使得富含鸟嘌呤的DNA单链片段脱落下来,通过不断的循环过程生成大量的富含鸟嘌呤的DNA单链(图 6)。随后,富含鸟嘌呤的DNA单链与荧光染料N-甲基卟啉二丙酸Ⅸ(NMM)发生特异性结合形成G4结构生成强烈的荧光信号。基于这一原理,实现了对于APE1活性检测的信号放大。实验证明,当APE1的比活在0.01 U/mL-1.0 U/mL之间时,荧光信号强度与其有着良好的线性关系,该检测方法对APE1活性的检出限能够低至0.006 U/mL。
2017年,Zhai等[6]利用APE1的特性,结合二氧化硅-金纳米粒子,根据简单的切割原理实现了对于活细胞中APE1活性的荧光检测。同时,该方法有望应用于针对癌症细胞的靶向药物递送。在该传感器中,研究者将标有荧光基团以及修饰有AP位点的DNA纳米探针通过链霉亲和素-生物素的特异结合连接在二氧化硅-金纳米粒子表面,由于此时荧光基团与纳米粒子距离较近,荧光处于被淬灭状态。当APE1具有活性时,能将DNA探针切断释放出标记有荧光基团的DNA单链,荧光基团重新发出荧光。如果将荧光基团替换为治疗癌症的药物,由于APE1是一种典型的癌症细胞的生物标志物,故该方法也具有应用于靶向递送治疗癌症药物的潜力。
5.2 APE1免疫生物传感器除了检测APE1的酶活性,目前还有一些研究以APE1为靶物质直接进行检测。如Han等[26]利用金和镍纳米粒子的信号放大功能实现了对痕量APE1的检测,Zhao等[27]利用抗体及信号分子Ru(bpy)32+实现了对于APE1的电化学信号免疫定量检测。Zhuo等[28]利用与ELISA相似的原理,实现了对APE1的检测,该方法的亮点在于其构建了包括碳纳米管和金纳米粒子在内的多种材料的复合微球。之后将APE1的特异性抗体以及信号分子标记在其表面,提高了APE1抗体的稳定性,实现了对于APE1的电化学信号检测。当APE1的浓度在1 fg/mL-1 pg/mL之间时,该检测方法生成的电化学信号与APE1的活性呈现良好的线性关系,同时该方法对于APE1的的检出限能够低至0.3 fg/mL。与之类似,Zhong等[7]也制作了由金纳米粒子和石墨烯组成的纳米载体,以APE1为靶物质对其进行了双抗体夹心法的电化学检测。但总起来讲,尽管直接以APE1为靶物质进行检测能够得到非常低的检出限值,但无法直观地反映其活性大小,而实际上APE1的活性大小才是其作为癌症生物标志物反映机体状况的关键。因此在对APE1进行检测时,还是应当主要侧重于利用其特异性设计生物传感器,检测其酶活性的大小。
6 APE家族其他成员的特性及传感器检测应用APE家族共有4种类型的酶,它们在修复核酸损伤方面的的作用是类似的,都是在AP位点处的磷酸骨架上制造缺口。根据制造出的缺口的种类不同可以将它们分为Ⅰ型和Ⅱ型。
6.1 APE家族成员的分类及其特性研究者主要将APE家族的成员分为两大类:Ⅰ型APE在AP位点的3'端通过β-消除作用切开核酸链,在AP位点处生成3'-OH以及带有5'-磷酸基团的正常核酸链;Ⅱ型APE在AP位点的5'端通过水解作用切开核酸链,在AP位点处生成5'-磷酸基团以及带有3'-OH的正常核酸链[29]。目前APE家族的成员共有4位,它们都能识别并作用于AP位点,但作用机理和生成的切口略有不同。绝大多数APE都属于属于Ⅱ型。人体内具有两种APE,分别为APE1和APE2,不过APE1的活性远高于APE2,在人体内的碱基切除修复过程中发挥了95%以上的作用,因此APE1是人体内最主要的APE[30]。人体内的APE2属于Ⅱ型APE,同时其活性十分依赖于人体内的金属离子。
6.2 与APE4相关的功能核酸生物传感器与APE1相似,利用其他APE家族成员的特异性活性同样能够构建各种各样的功能核酸生物传感器。目前除APE1之外研究最多、应用最广泛的是APE4。
利用APE4的活性以及滚环放大(Rolling circle amplification,RCA)技术得到带有许多G四联体结构的RCA产物,Wu等[31]实现了对于DNA糖基化酶活性的荧光信号放大。另外,Leung等[32]利用G四联体同特异性信号分子结合发荧光的特性,同样构建出了检测APE4活性的荧光功能核酸生物传感器。此外,将APE4与RCA相结合,同时利用了核酸外切酶Ⅲ的特殊性质,Kong等[33]构建了带有循环过程的功能核酸生物传感器实现了对于荧光信号的放大。首先,APE4在AP位点处切开DNA单链,生成的大片段能够作为引物探针与锁式探针特异性结合开启滚环扩增。而滚环扩增得到的产物能够有间隔地与许多标记有荧光基团和淬灭基团的信号探针结合。之后,核酸外切酶Ⅲ发挥作用,从信号探针的3'末端开始将信号探针切碎,生成的游离的标记有荧光基团的核酸重新发出荧光。而剩下的滚环扩增产物则可以进入下一个循环。该方法对检测APE4或APE1的活性有较大的应用潜力。
7 结语总体来说,目前APE1在功能核酸检测领域的应用还是较少且比较单一的,在未来的研究中应着力充分发挥APE1的仿生学功能,使APE1成为功能核酸检测中常用的酶工具之一。同时,APE1介导的功能核酸检测技术的检测对象还应进一步扩大,目前该类型的生物传感器检测对象绝大多数为DNA糖基化酶的活性,未来可以利用APE1的特异性切割活性,得到类型丰富的、能与更多靶物质发生特异性反应的功能核酸,使其在功能核酸重金属及小分子检测领域发挥更大的功能。另外,利用APE1的功能核酸生物传感器的检测手段还应进一步丰富,如将其与更多的荧光信号分子、电化学信号分子结合,使APE1能够应用于更广泛的检测领域。
另外,由于APE1是一种比较典型的癌症生物标志物,借鉴功能核酸生物传感器进行检测的原理,同样可以将APE1的活性与标有荧光基团的功能核酸结合实现对于癌症细胞的特异性体内成像以及治疗癌症药物的靶向递送,这对于癌症的早期诊断和治疗有着十分重要的意义。
| [1] |
王军彩, 谢劲心, 林斯星, 等. 人类脱嘌呤核酸内切酶的研究进展[J]. 中华预防医学杂志, 2004(5): 57-59. |
| [2] |
Laev SS, Salakhutdinov NF, Lavrik OI. Inhibitors of nuclease and redox activity of apurinic/apyrimidinic endonuclease 1/redox effector factor 1(APE1/Ref-1)[J]. Bioorganic & Medicinal Chemistry, 2017, 25(9): 2531-2544. |
| [3] |
Dianov GL, Sleeth KM, Dianova Ⅱ, et al. Repair of abasic sites in DNA[J]. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, 2003, 531(1): 157-163. |
| [4] |
Wu Y, Zhang B, Guo L H. Label-free and selective photoelectroche-mical detection of chemical DNA methylation damage using DNA repair enzymes[J]. Anal Chem, 2013, 85(14): 6908-6914. DOI:10.1021/ac401346x |
| [5] |
Wang L, Ma F, Tang B, et al. Base-Excision-Repair-Induced Construction of a Single Quantum-Dot-Based Sensor for Sensitive Detection of DNA Glycosylase Activity[J]. Anal Chem, 2016, 88(15): 7523-7529. DOI:10.1021/acs.analchem.6b00664 |
| [6] |
Zhai J, Liu Y, Huang S, et al. A specific DNA-nanoprobe for tracking the activities of human apurinic/apyrimidinic endonuclease 1 in living cells[J]. Nucleic Acids Res, 2017, 45(6): e45. DOI:10.1093/nar/gkw1205 |
| [7] |
Zhong Z, Li M, Qing Y, et al. Signal-on electrochemical immunoassay for APE1 using ionic liquid doped Au nanoparticle/graphene as a nanocarrier and alkaline phosphatase as enhancer[J]. Analyst, 2014, 139(24): 6563-6568. DOI:10.1039/C4AN01712A |
| [8] |
Barzilay G, Mol CD, Robson CN, et al. Identification of critical active-site residues in the multifunctional human DNA repair enzyme HAP1[J]. Nat Struct Mol Biol, 1995, 2(7): 561-568. DOI:10.1038/nsb0795-561 |
| [9] |
Barzilay G, Walker LJ, Robson CN, et al. Site-directed mutagenesis of the human DNA repair enzyme HAP1:identification of residues important for AP endonuclease and RNase H activity[J]. Nucleic Acids Res, 1995, 23(9): 1544-1550. DOI:10.1093/nar/23.9.1544 |
| [10] |
Barzilay G, Hickson ID. Structure and function of apurinic/apyrimidinic endonucleases[J]. Bioessays, 1995, 17(8): 713-719. DOI:10.1002/(ISSN)1521-1878 |
| [11] |
Sander M, Lowenhaupt K, Rich A. Drosophila Rrp1 protein:an apurinic endonuclease with homologous recombination activities[J]. Proc Natil Acad Sci, 1991, 88(15): 6780-6784. DOI:10.1073/pnas.88.15.6780 |
| [12] |
Harrison L, Ascione A G, Wilson DM, et al. Characterization of the promoter region of the human apurinic endonuclease gene(APE)[J]. J Biol Chem, 1995, 270(10): 5556-5564. DOI:10.1074/jbc.270.10.5556 |
| [13] |
Kanazhevskaya LY, Koval VV, Lomzov AA, et al. The role of Asn-212 in the catalytic mechanism of human endonuclease APE1:stopped-flow kinetic study of incision activity on a natural AP site and a tetrahydrofuran analogue[J]. DNA Repair, 2014, 21: 43-54. DOI:10.1016/j.dnarep.2014.06.008 |
| [14] |
Shen JC, Loeb LA. Mutations in the α8 loop of human APE1 alter binding and cleavage of DNA containing an abasic site[J]. J Biol Chem, 2003, 278(47): 46994-47001. DOI:10.1074/jbc.M309362200 |
| [15] |
吕小慧, 郭欣, 李佳, 等. APE1/Ref-1在卵巢癌中的功能研究进展[J]. 中国妇幼健康研究, 2016, 27(8): 1027-1029. DOI:10.3969/j.issn.1673-5293.2016.08.042 |
| [16] |
刘传, 应明真, 王雅杰. APE1/Ref-1基因结构及功能的研究进展[J]. 医学研究杂志, 2013, 42(3): 176-178. DOI:10.3969/j.issn.1673-548X.2013.03.055 |
| [17] |
徐海燕, 辛晓燕. APE1/Ref-1与肿瘤的研究进展[J]. 现代肿瘤医学, 2012, 20(11): 2426-2429. DOI:10.3969/j.issn.1672-4992.2012.11.75 |
| [18] |
Wang Z, Xu W, Lin Z, et al. Reduced apurinic/apyrimidinic endonuclease activity enhances the antitumor activity of oxymatrine in lung cancer cells[J]. International Journal of Oncology, 2016, 49(6): 2331-2340. DOI:10.3892/ijo.2016.3734 |
| [19] |
Qing Y, Li Q, Ren T, et al. Upregulation of PD-L1 and APE1 is associated with tumorigenesis and poor prognosis of gastric cancer[J]. Drug Des Devel Ther, 2015, 9: 901. |
| [20] |
Wen X, Lu R, Xie S, et al. APE1 overexpression promotes the progression of ovarian cancer and serves as a potential therapeutic target[J]. Cancer Biomarkers, 2016, 17(3): 313-322. DOI:10.3233/CBM-160643 |
| [21] |
Chen S, Xiong GS, Wu S, et al. Downregulation of apurinic/apyrimidinic endonuclease 1/redox factor-1 enhances the sensitivity of human pancreatic cancer cells to radiotherapy in vitro[J]. Cancer Biothe Radiopharm, 2013, 28(2): 169-176. |
| [22] |
Ströbel T, Madlener S, Tuna S, et al. Ape1 guides DNA repair pathway choice that is associated with drug tolerance in glioblastoma[J]. Sci Rep, 2017, 7(1): 9674. DOI:10.1038/s41598-017-10013-w |
| [23] |
Zhang T, Zhao H, Quan X, et al. An electrochemiluminescence sensing for DNA glycosylase assay with enhanced host-guest recognition technique based on α-cyclodextrin functionalized gold/silica cell-shell nanoparticles[J]. Electrochimica Acta, 2015, 157: 54-61. DOI:10.1016/j.electacta.2015.01.075 |
| [24] |
Fang S, Chen L, Zhao M. Unimolecular chemically modified DNA fluorescent probe for one-step quantitative measurement of the activity of human apurinic/apyrimidinic endonuclease 1 in biological samples[J]. Anal Chem, 2015, 87(24): 11952-11956. DOI:10.1021/acs.analchem.5b03939 |
| [25] |
Huang Y, Ma Y, Li Y, et al. Sensitive and label-free fluorescence detection of apurinic/apyrimidinic endonuclease 1 activity based on isothermal amplified-generation of G-quadruplex[J]. New J Chem, 2017, 41(5): 1893-1896. DOI:10.1039/C6NJ03477B |
| [26] |
Han J, Zhuo Y, Chai Y, et al. Ultrasensitive electrochemical strategy for trace detection of APE-1 via triple signal amplification strategy[J]. Biosens Bioelectron, 2013, 41: 116-122. DOI:10.1016/j.bios.2012.07.082 |
| [27] |
Zhao M, Chai XD, Han J, et al. A reagentless electrochemilumine-scent immunosensor for apurinic/apyrimidinic endonuclease 1 detection based on the new Ru(bpy)32+/bi-arginine system[J]. Anal Chim Acta, 2014, 846: 36-43. DOI:10.1016/j.aca.2014.07.017 |
| [28] |
Zhuo Y, Liao N, Chai YQ, et al. Ultrasensitive apurinic/apyrimidi-nic endonuclease 1 immunosensing based on self-enhanced electrochemiluminescence of a Ru(Ⅱ)complex[J]. Anal Chem, 2013, 86(2): 1053-1060. |
| [29] |
Levin JD, Demple B. Analysis of class Ⅱ(hydrolytic)and class Ⅰ(β-lyase)apurinic/apyrimidinic endonucleases with a synthetic DNA substrate[J]. Nucleic Acids Res, 1990, 18(17): 5069-5075. DOI:10.1093/nar/18.17.5069 |
| [30] |
Burkovics P, Szukacsov V, Unk I, et al. Human Ape2 protein has a 3'-5' exonuclease activity that acts preferentially on mismatched base pairs[J]. Nucleic Acids Res, 2006, 34(9): 2508-2515. DOI:10.1093/nar/gkl259 |
| [31] |
Wu Y, Yan P, Xu X, et al. A unique dual recognition hairpin probe mediated fluorescence amplification method for sensitive detection of uracil-DNA glycosylase and endonuclease Ⅳ activities[J]. Analyst, 2016, 141(5): 1789-1795. DOI:10.1039/C5AN02483H |
| [32] |
Leung KH, He HZ, Wang W, et al. Label-free luminescent switch-on detection of endonuclease Ⅳ activity using a G-quadruplex-selective iridium(Ⅲ)complex[J]. ACS Appl Mater Interfaces, 2013, 5(23): 12249-12253. DOI:10.1021/am404314p |
| [33] |
Kong XJ, Wu S, Cen Y, et al. Endonuclease Ⅳ cleaves apurinic/apyrimidinic sites in single-stranded DNA and its application for biosensing[J]. Analyst, 2016, 141(14): 4373-4380. DOI:10.1039/C6AN00738D |