“民以食为天,食以安为先”。食品安全是社会广泛关注的话题,也是关乎国计民生的大事。随着我国经济水平的提升,食品的流通量加大、加工方式更加多样,这对食品安全检测技术提出了更高的要求,如灵敏度更高、特异性更好、检测时间更短、成本更低和现场检测等。
食品安全风险因子检测方法中,高效液相色谱法、气相色谱法等大型仪器检测方法灵敏度好、操作简单,同时也存在检测成本高、无法实现高通量检测等缺陷;免疫检测法特异性好、检测原理相对简单,但抗体的成本较高,并且性质不够稳定;功能核酸生物传感器种类多样,无需大型仪器设备,通过信号扩增技术可以提高检测灵敏度,检测成本相对较低。功能核酸能够通过特定核酸结构发挥相应功能,功能核酸生物传感器应用功能核酸实现信号识别、信号放大或信号输出。
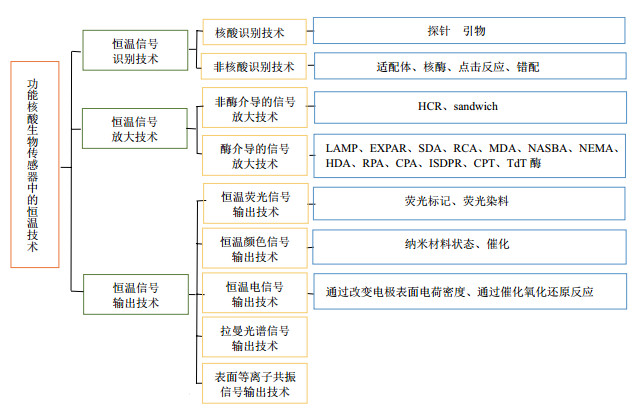
恒温技术是无需变换温度即可进行相关反应,达到目的效果的技术,因此无需变温设备,能够在一定程度上节省检测时间、降低检测成本。恒温技术仅需恒温仪器,部分恒温技术甚至无需温度控制相关仪器,在室温下即可进行。为了降低对仪器设备的依赖度,实现现场检测,恒温技术在功能核酸生物传感器中得到发展和应用。通常,生物传感器由3部分组成:信号识别元件、信号放大元件和信号输出元件。根据恒温技术在传感器中的不同作用,可将恒温技术划分为恒温介导的信号识别技术、恒温介导的信号放大技术、恒温介导的信号输出技术3大类(图 1)。
|
| 图 1 功能核酸生物传感器中的恒温技术分类图 |
使用功能核酸生物传感器进行核酸靶物质检测时,最常用的就是引物和探针。引物和探针都是基于碱基互补配对原则来识别特定核酸序列的。不同之处在于,引物与核酸结合后要进行延伸,所以引物的3'端必须有羟基,为引物延伸做准备。探针通过多年的发展和改进,已经与多种技术进行结合,可以实现信号识别和信号输出双重功能。最常用的是荧光探针,在核酸两端分别标记荧光基团和淬灭基团,通过改变标记的基团之间的距离产生荧光信号的变化。例如,taqman探针、小沟结合(Minor groove binder,MGB)探针和分子信标等。引物和探针在进行核酸识别中最为经典,但是其特异性有待提升,未来可以与新兴纳米材料进行结合,在生物传感器中实现更多功能。
1.2 非核酸靶物质识别技术 1.2.1 适配体适配体是一类能够特异性结合靶物质的脱氧核糖核酸(Deoxyribonucleic acid,DNA)或者核糖核酸(Ribonucleic acid,RNA)片段。适配体的特异性主要由核酸序列决定。相比于抗体,适配体具有稳定性高、成本低、不存在批次差异和便于保存等特点。此外,适配体在实际检测应用中便于被修饰和固定,并且适配体信号识别模式容易与核酸扩增相结合,实现信号放大,提高检测灵敏度。
适配体通常是由体外筛选得到的,其中指数富集的配基系统进化技术(Systematic evolution of ligands by exponential enrichment,SELEX)筛选方法应用最为广泛。SELEX方法是Ellington和Szostak等[1]于1990年发明的。在此基础上,发展多种SELEX的改进方法,如转换SELEX[2]、毛细管电泳SELEX[3]和微流控SELEX[4]。
目前适配体在多个检测方法中有广泛应用,如光学、电化学和质量测定。此外,其可以作为治疗剂[5]、抗感染药[6]和药物递送分子[7],并且可以用于癌症检测[8]。适配体的性能优越,更多物质的适配体将会出现,筛选特异性高、亲和力强的适配体的相关研究将会增加。
1.2.2 脱氧核酶第一个脱氧核酶是于1994年用Pb2+筛选的RNA切割核酶[9]。切割核酶由酶链和底物链两部分组成。当特定的物质存在时,与酶链相互作用,其切割特性被激活,底物链在特定位点被切割。RNA切割核酶的底物链中插入了核糖核苷酸,并在核糖核苷酸处被切割。切割核酶识别靶物质的特异性主要是由酶链的序列决定的。RNA切割核酶相比于DNA切割核酶切割效率更高,所以应用更加广泛。
根据激活切割活性物质的不同,切割核酶可以分为金属核酶和非金属核酶。以金属离子作为切割活性激活物质的RNA切割核酶被广泛应用于功能核酸生物传感器中。多种金属离子的切割核酶被筛选,如Mg2+[10]、Zn2+[11]等。另外,微生物分泌物[12]、组氨酸[13]等也可以激活核酶的切割活性。部分RNA切割核酶能够被不同物质激活切割活性,今后对RNA切割核酶特异性的研究需进一步加强。
1.2.3 点击反应点击化学,又译为“链接化学”、“动态组合化学”、“速配接合组合式化学”,是在2001年引入的一个合成概念,主旨是通过小单元的拼接来快速可靠地完成形形色色分子的化学合成[14]。点击化学的代表反应为铜催化的叠氮-炔基环加成反应,此原理广泛应用于铜离子的检测。
Ge等[15]开发了一种基于点击反应的铜离子检测方法。作者将G-四联体序列进行劈裂,将两段G-四联体序列的3'端和5'端分别修饰叠氮基和炔基。当没有铜离子时,不能发生点击反应,G-四联体无法发挥过氧化物酶催化活性。当铜离子存在时,在铜离子的催化作用下,发生点击反应,形成完整的G-四联体序列,在氯化血红素和过氧化氢存在时,催化TMB产生有色物质。借助酶标仪,最低检测限可达5.9 nmol/L。此外,还可以通过金纳米颗粒的颜色特性,利用铜离子催化点击反应的性质检测铜离子[16]。点击反应原理简单,容易实现,今后的研究中可以将其与更多功能核酸结合应用于传感器检测中。
1.2.4 错配通常,单链DNA可以基于碱基互补配对原则通过形成氢键产生稳定的DNA双链结构。但是,近年来研究发现,若两条链都为胸腺嘧啶(Thymine,T),在汞离子(Hg2+)存在时,可以形成T-Hg2+-T错配结构,稳定DNA双链结构。能够形成相似结构的还有胞嘧啶(Cytosine,C)和银离子(Ag+),形成C-Ag+-C结构。基于上述原理,多种银离子和汞离子的检测方法被开发。
错配结构可以与不同的核酸结构结合,如发卡和G-四联体。Stobiecka等[17]利用T-Hg2+-T错配开发了一种由汞离子和半胱氨酸作为开关的检测方法。作者将发卡颈部的序列插入一个T-T错配,发卡序列两端分别标记荧光基团和淬灭基团。起始为发卡打开的状态,有荧光信号,当汞离子存在时,发卡结构形成,荧光淬灭。当再加入半胱氨酸时,半胱氨酸能够与汞离子结合,导致发卡结构被破坏,荧光信号再次产生。Li等[18]将C-C错配设计在G-四联体序列中,银离子的存在与否会影响G-四联体结构的形成。当环境中存在游离的银离子,G-四联体可以在氯化血红素存在时催化ABTS反应,实现可视化检测。
2 恒温技术介导的信号放大技术 2.1 非酶依赖的信号放大技术 2.1.1 杂交链式反应杂交链式反应(Hybridization chain reaction,HCR)是一个无酶参与的扩增过程,于2004年被Dirks和Pierce[19]报道。首先,需要设计合理的发卡引物(H1和H2)和核酸促发因子。发卡通常由三部分构成:颈、环及toehold序列。toehold序列的长度是HCR的决定性因素,如果太短,则不足以启动HCR反应。研究表明,当toehold序列为6-10个碱基对时,HCR反应的速率比较稳定[20]。基于toehold原理,触发子可以破坏发卡1结构,将发卡1部分单链暴露出来,继而打开发卡2,实现相互杂交,得到长双链DNA产物。通过与荧光染料、G-四联体、纳米颗粒、电化学信号物质等结合,可以实现HCR扩增产物的荧光信号输出、电化学信号输出、化学发光信号输出、可视化信号输出等。目前HCR已经应用于多种靶物质的检测,如核酸(DNA或RNA)、小分子、金属离子甚至癌细胞。
2.1.2 三明治结构三明治结构(Sandwich)在生物检测、临床分析、环境监测等领域发挥重要作用。相比于普通的识别模式,sandwich需要分析物质与两个识别物结合,这大大提高了其特异性。在核酸检测中,普通sandwich只有一个信号分子,为了实现信号放大的目的,核酸检测中可以在sandwich的基础上增加了多重信号探针,这种技术即为supersandwich。Xia等[21]率先开展了相关研究工作。他们采用了标记亚甲基蓝的探针,当靶物质存在时,可与信号探针杂交形成黏性末端,暴露出的单链可继续结合靶物质,进而结合信号探针,如此循环形成supersandwich结构。这种方法的检测限(100 fmol/L)明显低于普通sandwich的检测限(100 pmol/L)。在此基础上,Chen等[22]引入辅助探针,与信号探针的两个部分互补配对,当靶物质存在时,结合信号探针形成粘性末端,结合辅助探针,进而再次结合信号探针,如此循环,实现仅一个靶物质分子就可以生成带有多个信号探针的DNA长双链。此方法检测限低至100 amol/L。
2.2 酶依赖的信号扩增技术 2.2.1 环介导的等温扩增技术环介导的等温扩增技术(Loop-mediated isothermal amplification,LAMP)最早由Notomi等[23]在2000年提出,是一种简单(一步式)、快速、高灵敏(6拷贝数)的核酸扩增技术。LAMP扩增的实现依赖于其特殊的引物(每次扩增需4-6个引物)和有链置换作用的Bst DNA聚合酶。在扩增过程中,引物识别模板链特定的DNA片段。扩增初期,4条引物被使用,分别为上下游内外引物,在Bst DNA聚合酶的作用下延伸,形成哑铃形的DNA结构,然后进行延伸和链替代,得到双链DNA产物[24]。整个扩增过程在60-65℃环境下,45-60 min内即可完成。
LAMP扩增灵敏度高,可以扩增6拷贝数的DNA片段,但是也容易被污染,产生假阳性。扩增产物可以通过浊度检测、电泳检测、荧光检测(荧光染料)等多种方式表征。随着LAMP技术的发展,多重LAMP扩增方法目前已与多种技术结合,应用于检测领域,如多重微流控LAMP[25]、实时多重LAMP[26]。Chen等[27]将多重LAMP与核酸侧流层析传感器进行结合。LAMP扩增过程中采用2条内引物做半抗原标记,靶标物质的两端通过“半抗原标记-抗体”识别体系进行检测,最后通过金纳米粒子进行信号输出。使用该方法,可在50 min内完成铜绿假单胞菌和其毒素基因的检测,最低检测限可达20 CFU/mL。
2.2.2 依赖解旋酶扩增技术(Helicase-dependent amplification,HDA)该技术在2004年被首次报道,是一种类似于体内DNA复制的扩增方式[28]。HDA利用解旋酶在三磷酸腺苷存在情况下的解旋活性,将DNA双链打开,无需热熔解步骤。为了防止DNA链再次结合,加入单链结合蛋白与解旋的DNA单链结合。上游引物和下游引物与DNA单链结合,在DNA聚合酶的作用下延伸形成新链,新链又会被解旋酶打开并被单链结合蛋白结合,从而进入新一轮的解旋扩增循环,如此反复循环。在60-65℃的单一温度下,60-120 min内即可达到指数扩增。HDA系统中使用的热稳定UvrD解旋酶极大地提高了该方法在更高温度(60-65℃)下的特异性和灵敏度。但是解旋酶UvrD的解旋速度(20 bp/s)和持续性(每个结合解旋少于100 bp)导致HDA不适用于长链扩增[28]。
HDA已经被应用于多种靶物质的检测,如致病菌、埃博拉病毒RNA及单碱基突变等。在与功能核酸侧流层析传感器联用时,通常将HDA反应体系中的上、下游引物分别做半抗原标记,通过HDA扩增获得两端有不同半抗原标记的双链DNA产物[29]。Tong等[30]结合限制性切克内切酶来提高检测过程中的特异性、减少扩增反应所需的时间。
2.2.3 重组酶聚合酶扩增技术(Recombinase polym-erase amplification,RPA)2006年Piepenburg等[31]首次报道该技术。在核酸扩增技术中,重组酶聚合酶扩增技术引起格外的注意RPA。RPA没有初始DNA双链熔解步骤,在37-42℃之间,20 min内即可完成扩增。重组酶聚合酶技术包括3种核心成分:结合单链核酸的重组酶、单链结合蛋白及具有链置换活性DNA聚合酶。在扩增过程中,重组酶与引物结合形成复合物定向寻找同源序列。当找到靶序列时,在重组酶的作用下双链DNA被打开,引物插入。为了防止双链重新结合,单链结合蛋白与被打开的单链DNA结合。在DNA聚合酶的作用下,引物延伸得到新链,完成DNA模板链的指数型扩增。
RPA可以有效扩增DNA和RNA模板,已经广泛应用于多种目标的检测,包括细菌、真菌、寄生虫、癌细胞、病毒和转基因。检测和扩增时间的限制不同,可能与靶物质序列、样品种类、引物和扩增子长度等有关系。RPA在终点检测和实时检测中都有应用,其中具有代表性的是试纸条检测[32]和实时荧光检测[33]。试纸条提供了一种低成本、可视的定性/半定量检测方法。相反,实时荧光检测能够实现扩增过程的实时检测,但需要特定的信号读取设备。
2.2.4 交叉引物扩增技术交叉引物扩增技术(Cro-ss-priming amplification,CPA)是由杭州优思达公司独立研发成功的一种新的核酸恒温扩增技术,也是中国首个具有自主知识产权的核酸扩增技术。CPA扩增体系中除包含具有链置换功能的Bst DNA聚合酶大片段外,还包括多条扩增引物,在Bst DNA聚合酶在作用下,不同位置的引物发生连续的循环扩增[34]。该反应通常在65℃恒温下进行,得到双链DNA产物。反应时间约1 h。
CPA扩增成本低,操作简单,在致病菌检测、转基因检测、病毒检测等都有应用。CPA扩增产物可以通过浊度、扩增子电泳、双链DNA特异性荧光染料或环磷酸荧光试剂等进行检测。另外,也可以与试纸条检测传感器联合使用。Wang等[35]报道的多重CPA结合多重核酸侧流层析传感器,通过引物设计和侧流层析传感器检测线数量调整实现了多重检测。
2.2.5 等温链置换聚合酶扩增技术(Isothermal stra-nddisplacement polym-erase reaction,ISDPR)ISDPR基于一条发夹模板、一条线性引物和具有链置换活性的DNA聚合酶,发夹与单链靶标DNA杂交后被打开暴露出单链模板,线性引物进一步与模板DNA杂交并在DNA聚合酶延伸后替代靶标DNA,得到双链DNA产物,而被替代的靶标DNA可以继续打开新的发夹模板实现循环扩增,从而达到大量扩增的目的,该反应通常在42℃恒温下进行。
此恒温扩增方式设计简单,操作方便。He等[36]将ISDPR反应与试纸条联合使用,实现核酸的可视化检测。作者将发卡和引物分别标记生物素和地高辛,所以扩增产物的两端分别被标记了生物素和地高辛。在试纸条检测线标记生物素抗体,扩增产物可以被固定在试纸条上,然后标记地高辛抗体的金纳米颗粒与扩增产物结合,实现颜色信号的输出。
2.2.6 切克内切酶介导等温扩增技术(Nicking endo-nuclease mediated isothermal amplification,NEMA)NEMA是利用切克内切酶和Bst DNA聚合酶实现的扩增反应。切克内切酶能够识别DNA双链特定的序列,并在特定位置切割其中的一条DNA链。DNA链首先被普通引物和带有切割位点的引物扩增,得到带有切割位点的DNA双链产物。双链DNA的其中一条链被切克内切酶切割,在其他引物的作用下延伸,发生链替代扩增反应。NEMA的扩增产物为DNA单链。
通常切克内切酶的最适孵育温度为55-65℃。另外,NEMA可以扩增400-500 bp的DNA序列,可以满足日常分子诊断需求。有报道NEMA可以用于定量检测。例如,Xu等[37]开发出NEMA-SMB扩增方式,将NEMA与分子信标进行结合,分子信标使扩增过程以荧光信号的方式被表征出来,实现高特异性和高灵敏度的定量检测。虽然NEMA用于全基因组扩增时反应体系不够稳定,但是仍可作为链置换扩增反应的一个替代选择。
2.2.7 核酸序列依赖扩增技术(Nucleic acid sequen-ce-based amplification,NASBA)NASBA于1991年被首次报道[38],NASBA技术通过AMV逆转录酶、T7RNA聚合酶、RNase H三种酶和一对引物(下游引物5'端带有T7RNA聚合酶启动子序列)得到RNA单链产物。NASBA被设计用于RNA扩增,但是某些情况下也可用于DNA扩增。NASBA反应温度通常为4℃,反应前需要双链高温分离步骤,若为DNA双链,则需95℃处理,若为RNA,则需65℃处理。反应时间从1.5-3 h不等。NASBA技术能直接使用RNA单链做模板,有效降低核酸污染。
多重NASBA在1999年被首次报道,研究人员在一个NASBA反应中使用生物素和ECL标记定量检测两个mRNAs[39]。另外,通过使用不同的荧光素基团标记的分子信标,可以实现多重实时NASBA[40],可以使用特定的激发光和发射光滤光片同时测量不同的荧光团。
2.2.8 滚环扩增技术(Rolling cycling amplification,RCA)RCA通过使用环状DNA模板和高度持续的DNA或RNA聚合酶实现短核酸靶标的扩增,扩增产物为含有与环状模板互补的串联序列的长单链核酸。当短核酸链与环状DNA模板结合时,在聚合酶的作用下开始延伸,并替代已合成的链,形成长单链产物,长度可达105碱基。用于扩增RNA靶标时,通常使用T7 RNA聚合酶;用于DNA靶标时,通常使用噬菌体Φ29酶、Bst和Vent exo-DNA聚合酶。RCA扩增通常在30-65℃下进行,在30-90 min内即可完成扩增[41],并且在10拷贝数DNA存在时即可启动反应。为了提高RCA反应的扩增效率,超支化RCA和网状RCA已经被开发[42]。
1998年首次将RCA应用于生物检测[43]。目前被应用于DNA或者RNA的单碱基突变检测、miRNA、蛋白质、三磷酸腺苷等的检测。人们尝试将大多数检测技术应用于检测RCA扩增产物,如伏安法、荧光法、表面增强拉曼光谱、比色法和化学发光法。
2.2.9 等温指数扩增技术(Exponential amplification reaction,EXPAR)EXPAR首次于2003年被报道,此扩增方式能够实现高灵敏度检测,其扩增的特异性取决于引物延伸和替代[44]。等温指数扩增技术以单链靶标为引物,通过基于切克内切酶和Bst DNA聚合酶实现的链置换反应。根据靶标自主设计含有酶切位点的单链模板,引物经过DNA聚合酶延伸后与模板形成完整的切克内切酶识别位点,单链切割后发生链置换反应。该反应通常在65℃恒温下进行。
Wang等[45]运用EXPAR进行mRNA扩增时,将第一轮EXPAR产物设计为新的EXPAR的引物,实现进一步的扩增。但是EXPAR不能用于长核酸链的扩增,这限制了其应用。
2.2.10 链置换扩增反应(Strand displacement ampli-fication,SDA)SDA最早是Walker等[46]于1992年报道。SDA扩增需要热前处理解开双链DNA。SDA扩增需要HincⅡ限制性切割酶和具有链置换作用的聚合酶。SDA扩增过程中所需引物5'端含有HincⅡ限制酶识别的特定序列。因为合成中使用巯基修饰的脱氧腺苷三磷酸(Deoxyadenosine triphosphate,dATP)代替dATP,所以新合成的DNA链具有硫代磷酸酯修饰,不能被HincⅡ切割。原有引物在切割位点处被切割,在聚合酶作用下延伸3'末端并取代下游DNA链,以此达到扩展目的。反应温度通常为30-55℃。
SDA被应用于检测多种靶物质。SDA可以与荧光基团结合用于致病菌的检测,如结核分枝杆菌,扩增过程中,荧光标记探针与扩增产物结合,使荧光强度随扩增的进行增加。另外,通过与适配体结合,也被应用于可卡因的检测[47]。基于SDA扩增原理,利用SYBR Green Ⅰ表征扩增产物,同样可以实现miRNA的检测。
2.2.11 循环探针技术(Cyclic probe technology,CPT)CPT与上述信号放大方式不同,CPT不扩增靶序列,而是通过探针和酶的作用原理实现信号放大[48]。循环探针为25-30 bp的单链多核苷酸,包括3部分:3'端DNA、中间4-6碱基的RNA和5'端DNA。循环探针可以与靶物质杂交形成双链。RNase H酶仅能探针-靶物质杂合物的切割磷酸-核糖核苷酸化学键,所以探针被切割为两段序列,靶物质可以重新和未切割探针结合,循环后获得大量切割后的探针片段DNA。此方法仅能应用于DNA靶物质。CPT反应温度为55-65℃。CPT产物可以通过多种方式检测,最常见的是通过使用放射性同位素标记探针的聚丙烯酰胺凝胶电泳,抗体介导的比色酶测定和试纸条也可以实现CPT产物检测。CPT可以在某些条件下显示线性响应,便于定量。
2.2.12 末端脱氧核苷酸转移酶(Terminal deoxynuc-leotidyl transferase,TdT)TdT是一种催化脱氧核苷酸结合到DNA或RNA分子的3'羟基端的无需模板的DNA聚合酶,最早于1958年在牛胸腺中发现[49]。TdT可以催化dNTP或标记了小分子的dNTP,如Cy3-dNTP,biotin-dNTP,聚合于RNA或单双链DNA的3'-OH末端的反应,该反应不需要特定模板,但引物必须是至少有3个以上碱基的寡核苷酸。TdT酶通常在37℃下使用。
当被用于检测中信号放大时,TdT酶通常与dGTPs相结合,增加G-四联体的含量。Liu等[50]通过TdT酶聚合制备富含G的随机DNA序列,并证明了这种随机富含G的DNA能够形成G-四联体。他们研发了两种检操作单且无标记的检测TdT酶活性的策略,包括基于脱氧核糖核酸酶的比色测定法和实时荧光测定法。该实验的实时检测方法为临床诊断中关键酶的生化分析提供了一种可行的方法,且操作相对于传统方法更加简便。
3 恒温技术介导的信号输出技术 3.1 恒温技术介导的荧光信号输出技术荧光信号是比较灵敏的信号输出方式,在功能核酸生物传感器中应用广泛。荧光产生机理有所不同,最为经典的是荧光共振能量转移原理。荧光共振能量转移是指一个体系含有能量供体与能量受体两个荧光团,在供体被激发光所激发后可以向受体发生能量转移,进而受体被激发,发出荧光,该种能量的属性是非辐射的[51]。
根据其与核酸的作用方式不同,荧光信号分子可以大致分为标记型和免标记型。标记型是指将荧光信号分子通过共价键的方式,标记在核酸链上,通过核酸结构或位置的改变,对荧光信号产生影响。可以标记在核酸链上的荧光基团有罗丹明类、荧光素类及多环芳烃类等。非标记型中最具有代表性的是核酸染料,如单链核酸结合染料SYBR Gold、SYBR Green Ⅱ等,双链核酸结合染料SYBR Green Ⅰ、TOTO-3等。纳米材料在荧光信号输出方面也有不同形式的应用,如金属纳米簇、量子点等。
3.2 恒温技术介导的可视化信号输出技术可视化检测主要是通过颜色的变化来实现的,无需借助任何仪器设备就可以获得检测结果。也可以借助酶标仪等仪器,进行更加准确的实验结果分析和定量检测。颜色信号一方面可以通过纳米材料在溶液中的状态产生变化;另一方面可以通过催化底物产生有色物质发生变化。
通过在溶液中的状态来产生颜色变化最具有代表性的是金纳米颗粒。金纳米颗粒有灵敏度高、颜色变化明显、便于修饰核酸链等优势。DNA单链可以稳定金纳米颗粒的分散状态,呈现红色。而DNA双链可以促进金纳米颗粒聚集呈现蓝色。除此之外,还可以通过金硫键将核酸链修饰到金纳米颗粒的表面,通过核酸链的碱基互补,促进金纳米颗粒形成聚集状态。
目前发现G-四联体和氯化血红素复合物及很多纳米材料有催化活性,通过催化底物产生有色物质也可以实现可视化检测。Wu等[52]利用此原理进行L-半胱氨酸的检测。首先使用富胞嘧啶序列的DNA制备了DNA-Ag/Pt纳米簇,尺寸小、比表面积大的纳米结构能有更多的活性位点,更强的催化活性。当加入L-半胱氨酸后,可以促进纳米簇聚集,催化活性降低,催化TMB产生的有色物质变少,实现L-半胱氨酸的检测,检测限为2.0 nmol/L。
3.3 恒温技术介导的电信号输出技术电化学生物传感器具有灵敏度高、操作简便、便于携带等优点。根据电化学信号产生的机理不同,可以大致分为两大类:一是通过改变电极表面的电荷分布产生电信号;二是通过催化氧化还原反应产生电信号。通常,第一种机理所使用的电信号物质通过直接标记在核酸末端、与单双链核酸结合、嵌入到具有特殊构象的核酸中、游离于溶液中检测等方式表征电化学信号的产生,包括亚甲基蓝、六氨合钌、二茂铁和铁氰化钾等。第二种则通过与底物相互作用,促进氧化还原反应和电子转移产生电化学信号,常用的有G-四联体、金属有机框架等。
3.4 拉曼光谱信号输出技术1928年,Raman等[53]发现了拉曼散射现象。拉曼光谱属于分子振动光谱,可以反映分子的特征结构。但是拉曼散射效应是个非常弱的过程,目前应用比较多的是表面增强拉曼光谱,用通常的拉曼光谱法测定吸附在胶质金属颗粒如银、金或铜表面的样品,或吸附在这些金属片的粗糙表面上的样品,其拉曼光谱的强度可提高103-106倍。关于增强机理的本质,学术界目前仍未达成共识,大多数学者认为SERS增强主要由物理增强和化学增强两个方面构成,并认为前者占主导地位,而后者在增强效应中只贡献1-2个数量级。
Qian等[54]利用玻璃基质标记肽核酸发卡,当靶物质存在时,发卡结构被打开,并且复合物带有负电荷,可以与银纳米粒子静电吸附,增强拉曼光谱信号。检测限可以达到34 pmol/L。
3.5 表面等离子共振信号输出技术表面等离子共振(Surface plasmon resonance,SPR)现象在传感器中作为信号输出方式被应用。光在棱镜与金属膜表面上发生全反射现象时,会形成消逝波进入到光疏介质中,而在介质(假设为金属介质)中又存在一定的等离子波,当两波相遇时可能会发生共振。当消逝波与表面等离子波发生共振时,检测到的反射光强会大幅度地减弱。当反射光完全消失时入射角就是SPR角。SPR角随金属表面折射率变化而变化,而折射率的变化又与金属表面结合的分子质量成正比。因此可以通过对生物反应过程中SPR角的动态变化获取生物分子之间相互作用的特异信号。Wang等[55]通过基于表面等离子体共振原理利用金纳米粒子作为信号放大策略检测microRNA。首先,他们将DNA探针标记在金芯片上,当靶物质存在时可以引发sandwich结构的形成,与标记核酸链的金纳米颗粒杂交。标记金纳米颗粒的核酸链可以在此基础上不断结合,实现信号放大的效果,此方法检测限达到45 pmol/L。
4 总结与展望恒温技术介导的功能核酸生物传感器发展迅速,在信号识别、信号放大和信号输出方面都展现了巨大的优势。在信号识别方面,恒温技术介导的功能核酸生物传感器通过适配体、RNA切割核酶、点击反应等方式实现靶物质的高特异性识别;在信号扩增方面,恒温技术发挥了无需变温设备的优势,同时兼具扩增时间短、扩增产物多样等特点,无酶扩增方式不仅降低了检测成本,同时也避免了酶活抑制剂对扩增效果的影响;在信号输出方面,荧光信号、可视化信号、电化学信号等灵敏度较高,可以提高传感器检测性能。恒温介导的功能核酸生物传感器具有巨大的发展潜力,需要更深入的研究。
一方面,恒温技术介导的功能核酸生物传感器需要强有力的理论支持。相关恒温技术需要得到更加深入的理论研究,以便为后续的优化和应用提供基础。另一方面,恒温技术介导的功能核酸生物传感器需要进一步与实际应用相结合,并且从实际应用的角度出发,进行相关性能的优化。
最后,交叉学科是一个重要的发展趋势。通过将生物学、纳米材料学、化学等学科综合应用,可以开发性能更加优越的检测技术。如应用纳米材料具有尺寸效应、磁性、酶催化活性等特点,将其与功能核酸相结合,可以实现信号扩增或信号输出的目的。通过将其他学科的技术与恒温技术介导的功能核酸生物传感器结合,可以实现更高灵敏度和更高特异性的检测。
| [1] |
Ellington AD, Szostak JW. In vitro selection of RNA molecules that bind specific ligands[J]. Nature, 1990, 346(6287): 818. DOI:10.1038/346818a0 |
| [2] |
White R, Rusconi C, Scardino E, et al. Generation of species cross-reactive aptamers using "toggle" SELEX[J]. Molecular Therapy, 2001, 4(6): 567-573. DOI:10.1006/mthe.2001.0495 |
| [3] |
Mendonsa SD, Bowser MT. In vitro evolution of functional DNA using capillary electrophoresis[J]. J Am Chem Soc, 2004, 126(1): 20-21. DOI:10.1021/ja037832s |
| [4] |
Qian J, Lou X, Zhang Y, et al. Generation of highly specific aptamers via micromagnetic selection[J]. Anal Chem, 2009, 81(13): 5490-5495. DOI:10.1021/ac900759k |
| [5] |
Lee JF, Stovall GM, Ellington AD. Aptamer therapeutics advance[J]. Curr Opin Chem Biol, 2006, 10(3): 282-289. DOI:10.1016/j.cbpa.2006.03.015 |
| [6] |
Ulrich H, Magdesian MH, Alves MJM, et al. In vitro selection of RNA aptamers that bind to cell adhesion receptors of trypanosoma cruzi and inhibit cell invasion[J]. J Biol Chem, 2002, 277(23): 20756-20762. DOI:10.1074/jbc.M111859200 |
| [7] |
Bagalkot V, Zhang L, Levy-Nissenbaum E, et al. Quantum dot-aptamer conjugates for synchronous cancer imaging, therapy, and sensing of drug delivery based on bi-fluorescence resonance energy transfer[J]. Nano Lett, 2007, 7(10): 3065-3070. DOI:10.1021/nl071546n |
| [8] |
Tang Z, Shangguan D, Wang K, et al. Selection of aptamers for molecular recognition and characterization of cancer cells[J]. Anal Chem, 2007, 79(13): 4900-4907. DOI:10.1021/ac070189y |
| [9] |
Breaker RR, Joyce GF. A DNA enzyme that cleaves RNA[J]. Chemistry and Biology, 1994, 1(4): 223-229. DOI:10.1016/1074-5521(94)90014-0 |
| [10] |
Zheng H, Shabalin IG, Handing KB, et al. Magnesium-binding architectures in RNA crystal structures:validation, binding preferences, classification and motif detection[J]. Nucleic Acids Res, 2015, 43(7): 3789-3801. DOI:10.1093/nar/gkv225 |
| [11] |
Li J, Zheng W, Kwon AH, et al. In vitro selection and characteriza-tion of a highly efficient Zn(Ⅱ)-dependent RNA-cleaving deoxyribozyme[J]. Nucleic Acids Res, 2000, 2: 481-488. |
| [12] |
Ali MM, Aguirre SD, Lazim H, et al. Fluorogenic DNAzyme probes as bacterial indicators[J]. Angew Chem Int Ed Engl, 2011, 50(16): 3751-3754. DOI:10.1002/anie.v50.16 |
| [13] |
Ye S, Guo Y, Xiao J, et al. A sensitive SERS assay of L-histidine via a DNAzyme-activated target recycling cascade amplification strategy[J]. Chem Commun, 2013, 49(35): 3643-3645. DOI:10.1039/c3cc41619d |
| [14] |
Kolb HC, Finn MG, Sharpless KB. Click chemistry:diverse chemical function from a few good reactions[J]. Cheminform, 2001, 32(35): 2004-2021. |
| [15] |
Ge C, Luo Q, Wang D, et al. Colorimetric detection of copper(Ⅱ)ion using click chemistry and hemin/G-quadruplex horseradish peroxidase-mimicking DNAzyme[J]. Anal Chem, 2014, 86(13): 6387-6392. DOI:10.1021/ac501739a |
| [16] |
Shen Q, Li W, Tang S, et al. A simple "clickable" biosensor for colorimetric detection of copper(Ⅱ)ions based on unmodified gold nanoparticles[J]. Biosens Bioelectron, 2013, 1: 663-668. |
| [17] |
Stobiecka M, Molinero AA, Chałupa A, et al. Mercury/Homocyste-ine ligation-induced ON/OFF-switching of a T-T mismatch-based oligonucleotide molecular beacon[J]. Analytical Ahemistry, 2012, 84(11): 4970-4978. |
| [18] |
Li T, Shi L, Wang E, et al. Silver-ion-mediated DNAzyme switch for the ultrasensitive and selective colorimetric detection of aqueous Ag+ and cysteine[J]. Chemistry, 2010, 15(14): 3347-3350. |
| [19] |
Dirks RM, Pierce NA. Triggered amplification by hybridization chain reaction[J]. Proc Natl Acad Sci USA, 2004, 101(43): 15275-15278. DOI:10.1073/pnas.0407024101 |
| [20] |
Zhang DY, Winfree E. Control of DNA strand displacement kinetics using toehold exchange[J]. J Am Chem Soc, 2009, 131(47): 17303. DOI:10.1021/ja906987s |
| [21] |
Xia F, White RJ, Zuo X, et al. An electrochemical supersandwich assay for sensitive and selective DNA detection in complex matrices[J]. J Am Chem Soc, 2010, 41: 14346-14348. |
| [22] |
Chen X, Lin Y-H, Li J, et al. A simple and ultrasensitive electrochemical DNA biosensor based on DNA concatamers[J]. Chem Commun, 2011, 47(44): 12116-12118. DOI:10.1039/c1cc15695k |
| [23] |
Notomi T, Okayama H, Masubuchi H, et al. Loop-mediated isothermal amplification of DNA[J]. Nucleic Acids Res, 2000, 28(12): e63-e63. DOI:10.1093/nar/28.12.e63 |
| [24] |
Tomita N, Mori Y, Kanda H, et al. Loop-mediated isothermal amplification(LAMP)of gene sequences and simple visual detection of products[J]. Nature Protocols, 2008, 3(5): 877. DOI:10.1038/nprot.2008.57 |
| [25] |
Fang X, Chen H, Yu S, et al. Predicting viruses accurately by a multiplex microfluidic loop-mediated isothermal amplification chip[J]. Anal Chem, 2010, 83(3): 690-695. |
| [26] |
Tanner NA, Zhang Y, Evans Jr TC. Simultaneous multiple target detection in real-time loop-mediated isothermal amplification[J]. Biotechniques, 2012, 53(2): 81-89. |
| [27] |
Chen Y, Cheng N, Xu Y, et al. Point-of-care and visual detection of P. aeruginosa and its toxin genes by multiple LAMP and lateral flow nucleic acid biosensor[J]. Biosens Bioelectron, 2016, 81: 317-323. DOI:10.1016/j.bios.2016.03.006 |
| [28] |
Vincent M, Xu Y, Kong H. Helicase-dependent isothermal DNA amplification. EMBO Rep. 5:795-800[J]. Embo Reports, 2004, 5(8): 795-800. DOI:10.1038/sj.embor.7400200 |
| [29] |
Kolm C, Mach R, Krska R, et al. A rapid DNA lateral flow test for the detection of transgenic maize by isothermal amplification of the 35S promoter[J]. Analytical Methods, 2014, 7(1): 129-134. |
| [30] |
Tong Y, Lemieux B, Kong H. Multiple strategies to improve sensitivity, speed and robustness of isothermal nucleic acid amplification for rapid pathogen detection[J]. Bmc Biotechnology, 2011, 11(1): 50. DOI:10.1186/1472-6750-11-50 |
| [31] |
Piepenburg O, Williams CH, Stemple DL, et al. DNA detection using recombination proteins[J]. PLoS Biol, 2006, 7: e204. |
| [32] |
Tu PA, Shiu JS, Lee SH, et al. Development of a recombinase polymerase amplification lateral flow dipstick(RPA-LFD)for the field diagnosis of caprine arthritis-encephalitis virus(CAEV)infection[J]. J Virol Methods, 2017, 243: 98-104. DOI:10.1016/j.jviromet.2017.01.023 |
| [33] |
Ahmed A, Linden HVD, Hartskeerl RA. Development of a recombinase polymerase amplification assay for the detection of pathogenic leptospira[J]. Int J Environ Res Public Health, 2014, 11(5): 4953-4964. DOI:10.3390/ijerph110504953 |
| [34] |
Fang R, Li X, Hu L, et al. Cross-priming amplification for rapid detection of Mycobacterium tuberculosis in sputum specimens[J]. J Clin Microbiol, 2009, 47(3): 845-847. DOI:10.1128/JCM.01528-08 |
| [35] |
Wang Y, Wang Y, Zhang L, et al. Visual and multiplex detection of nucleic acid sequence by multiple cross displacement amplification coupled with gold nanoparticle-based lateral flow biosensor[J]. Sensors and Actuators B:Chemical, 2016, 241: 1283-1293. |
| [36] |
He Y, Zeng K, Zhang S, et al. Visual detection of gene mutations based on isothermal strand-displacement polymerase reaction and lateral flow strip[J]. Biosens Bioelectron, 2012, 31(1): 310-315. DOI:10.1016/j.bios.2011.10.037 |
| [37] |
Xu W, Wang C, Zhu P, et al. Real-time quantitative nicking endonuclease-mediated isothermal amplification with small molecular beacons[J]. Analyst, 2016, 141(8): 2542-2552. DOI:10.1039/C6AN00145A |
| [38] |
Compton J. Nucleic acid sequence-based amplification[J]. Nature, 1991, 350(6313): 91-92. DOI:10.1038/350091a0 |
| [39] |
van Deursen PB, Gunther AW, Spaargaren-van Riel CC, et al. A novel quantitative multiplex NASBA method:application to measuring tissue factor and CD14 mRNA levels in human monocytes[J]. Nucleic Acids Res, 1999, 27(17): e15-i. DOI:10.1093/nar/27.17.e15 |
| [40] |
Greijer AE, Adriaanse HM, Dekkers CA, et al. Multiplex real-time NASBA for monitoring expression dynamics of human cytomegalovirus encoded IE1 and pp67 RNA[J]. Journal of Clinical Virology, 2002, 24(1): 57-66. |
| [41] |
Kaocharoen S, Wang B, Tsui KM, et al. Hyperbranched rolling circle amplification as a rapid and sensitive method for species identification within the Cryptococcus species complex[J]. Electrophoresis, 2008, 29(15): 3183-3191. |
| [42] |
Zhu X, Feng C, Zhang B, et al. A netlike rolling circle nucleic acid amplification technique[J]. Analyst, 2015, 140(1): 74-78. DOI:10.1039/C4AN01711K |
| [43] |
Lizardi PM, Huang X, Zhu Z, et al. Mutation detection and single-molecule counting using isothermal rolling-circle amplification[J]. Nature Genetics, 1998, 19(3): 225. DOI:10.1038/898 |
| [44] |
Van Ness J, Van Ness LK, Galas DJ. Isothermal reactions for the amplificmation of oligonucleotides[J]. Proc Natl Acad Sci, 2003, 100(8): 4504-4509. DOI:10.1073/pnas.0730811100 |
| [45] |
Wang H, Wang H, Duan X, et al. Sensitive detection of mRNA by using specific cleavage-mediated isothermal exponential amplification reaction[J]. Sensors and Actuators B:Chemical, 2017, 252: 215-221. DOI:10.1016/j.snb.2017.06.008 |
| [46] |
Walker GT, Fraiser MS, Schram JL, et al. Strand displacement amplification—an isothermal, in vitro DNA amplification technique[J]. Nucleic Acids Res, 1992, 20(7): 1691-1696. DOI:10.1093/nar/20.7.1691 |
| [47] |
He JL, Wu ZS, Zhou H, et al. Fluorescence aptameric sensor for strand displacement amplification detection of cocaine[J]. Anal Chem, 2010, 82(4): 1358-1364. DOI:10.1021/ac902416u |
| [48] |
Dickinson LT, Mah DC, Poirier RT, et al. Genomic DNA detection using cycling probe technology and capillary gel electrophoresis with laser-induced fluorescence[J]. Molecular & Cellular Probes, 2004, 18(5): 341. |
| [49] |
Bollum F, Potter VR. Incorporation of thymidine into deoxyribonucleic acid by enzymes from rat tissues[J]. J Biology Chemistry, 1958, 233(2): 478-482. |
| [50] |
Liu Z, Li W, Nie Z, et al. Randomly arrayed G-quadruplexes for label-free and real-time assay of enzyme activity[J]. Chem Commun, 2014, 50(52): 6875-6878. DOI:10.1039/c4cc03103b |
| [51] |
Clegg RM. Fluorescence resonance energy transfer[J]. Current Opinion in Biotechnology, 1995, 6(1): 103-110. DOI:10.1016/0958-1669(95)80016-6 |
| [52] |
Wu LL, Wang LY, Xie ZJ, et al. Colorimetric assay of l-cysteine based on peroxidase-mimicking DNA-Ag/Pt nanoclusters[J]. Sensors and Actuators B:Chemical, 2016, 235: 110-116. DOI:10.1016/j.snb.2016.05.069 |
| [53] |
Raman CV, Krishnan KS. Polarisation of scattered light-quanta[J]. Nature, 1928, 122(3066): 169. |
| [54] |
Qian Y, Fan T, Yao Y, et al. Label-free and Raman dyes-free surface-enhanced Raman spectroscopy for detection of DNA[J]. Sensors and Actuators B:Chemical, 2018, 254: 483-489. DOI:10.1016/j.snb.2017.07.112 |
| [55] |
Wang Q, Liu R, Yang X, et al. Surface plasmon resonance biosensor for enzyme-free amplified microRNA detection based on gold nanoparticles and DNA supersandwich[J]. Sensors and Actuators B:Chemical, 2016, 223: 613-620. DOI:10.1016/j.snb.2015.09.152 |




