2. 西华大学食品与生物工程学院,成都 610039
2. College of Food Science and Biological Engineering, Xihua University, Chengdu 610039
随着人类社会的发展,新型人类疾病不断出现、病原微生物对临床药物耐药性不断增加以及动植物病害严重发生等问题日益严重,促使人们呼唤新结构新活性抗生素的发现,目前,新抗生素中50%-78%由放线菌产生[1-2]。稀有放线菌是一类出现频率比链霉菌低得多的放线菌类群,有着产生新的天然活性物质的极大潜力[3-4],其研究一直备受关注[5]。
塔里木盆地是我国最大内陆盆地,气候极端干旱,植被稀少,土壤荒漠化和盐碱化严重,环境较为特殊,可能存在独特的稀有放线菌[6-7]。微生物的数量和组成与其生存环境(水、热、pH、植被、土壤等生态条件)密切相关[8-9],为了给研究者有目标的分离获得该地区稀有放线菌资源提供参考,本研究根据倪健等[10]对塔里木盆地生态小区的划分,选择5个代表性生态小区,使用选择性培养基分离稀有放线菌,分析不同生态小区稀有放线菌分布情况。
为提高产活性物质潜力菌的筛选效率,本项目组还对抗生素合成相关基因进行了筛选,主要包括Ⅰ型PKS、Ⅱ型PKS、NRPS、APH和HMG-CoA。它们是相应抗生素类代谢产物产生的关键催化物,其中聚酮合酶(Polyketide synthase,PKS)能够催化小分子羧酸类物质合成具有重要药用价值的次级代谢产物——聚酮类化合物。放线菌主要含有Ⅰ型PKS和Ⅱ型PKS,Ⅰ型PKS催化合成的抗生素主要包括大环内脂类、聚醚类、多烯类和安莎类;Ⅱ型PKS催化合成的抗生素主要包括蒽环类和四环类。非核糖体多肽合成酶(Nonribosomal Peptide -synthetase,NRPS)是一些重要多肽类抗生素的合成系统中起关键作用的酶,能识别特定的氨基酸并将其直接相连形成多肽链。氨基糖苷磷酸转移酶(Aminoglycoside Phosphotransferases,APH)与氨基糖苷类抗生素合成密切相关[11],氨基糖苷类抗生素主要包括链霉素、新霉素等。3-羟基-3-甲基戊二酰辅酶A(3-Hydroxy-3-Methylglutaryl Coenzyme A,HMG-CoA)还原酶与类异戊二烯的生物合成密切相关,后者有极大的临床应用价值[12]。这些基因是筛选生产相应抗生素放线菌的重要依据。本研究根据基因筛选评估稀有放线菌的抗生素类活性物质的产生潜力,旨在为进一步开发利用稀有放线菌提供菌种基础。
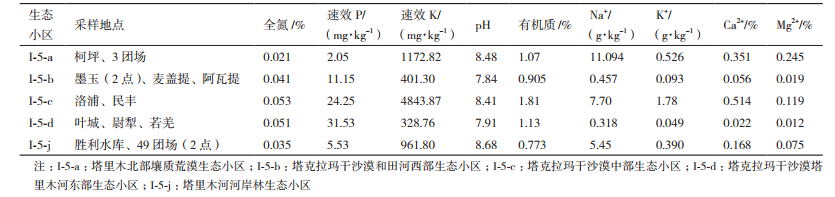
1 材料与方法 1.1 材料试验选择塔里木盆地具代表性的5个生态小区,根据各小区的面积,每小区选择2-4个点进行采样,采样取土下5-20 cm深度的土壤,装入灭菌袋,编号注明采集信息,4℃低温保藏带回实验室后,将各小区样品以四分法均匀混合并检测理化性质,以5份混合土样为材料进行分离。各样品理化性质见表 1。
(1) 预处理。干热处理:土样风干,研磨过筛,120℃干热处理1 h,称取1 g样品到10 mL的无菌水中,120 r/min振荡10 min。苯酚处理:取1 g样品加入到1.5%苯酚的无菌水中,30℃水浴30 min。SDS处理:称取1 g土样,用0.05%的SDS(5 mmol/l磷酸盐缓冲液配制),加入6%的酵母膏,40℃下200 r/min振荡20 min[13]。
(2) 分离培养:采用稀释法涂板分离,使用改良HV培养基;ATCC172培养基[14];海藻糖-脯氨酸培养基;改良脯氨酸培养基;改良高氏二号培养基[15];葡萄糖-天门冬酰氨培养基[16]和YIM-3培养基[17]等7种培养基进行复合筛选。培养基加入终浓度为60 μg/mL的重铬酸钾、50 μg/mL的放线菌酮和25 μg/mL的制霉菌素来抑制细菌和霉菌的生长。以预处理和培养基为因素,设置21个处理进行分离,28℃培养2周[18]。
(3) 纯化:根据菌落形态挑菌纯化,纯化培养基为ISP4培养基。ISP 4培养基:淀粉10 g,(NH4)2SO44g,CaCO32 g,MgSO42 g,K2HPO42 g,琼脂16-18 g,水1 000 mL,pH调至7.0-7.5。
1.2.2 菌种鉴定采用16S rRNA基因序列分析方法鉴定菌种。以酶小量法提取待测菌株基因组DNA[16],利用通用引物(正向PA:5'-CAGA GTT-TGATCCTGGCT-3'和反向PB:5'-AGGAGGTGA TC-CAGCCGCA -3')进行16S rDNA基因的扩增,PCR扩增产物送至上海生工生物工程技术服务有限公司进行序列测定。获得的16S rRNA基因序列在NCBI上采用Blast Search法进行相似性检索,从GenBank和EMBL/DDBJ数据库中搜集到相关放线菌的16S rRNA基因序列进行比较鉴定。最后使用显微镜观察形态特征并与分子鉴定结果比照进行综合鉴定。
1.2.3 生态分布及多样性分析分析各生态小区菌种分布及多样性,计算不同小区的物种均匀度(Evenness)、丰富度(Shannon)和多样性(Simpson)指数,其中Simpson指数用如下公式计算:
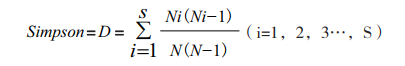
其中,N为个体总数,S属数,Ni为i属的个体数。
1.2.4 抗生素合成相关基因扩增采用5对引物扩增菌株的功能基因。引物K1F(5'-TSAAGTCSAACA TCGGBCA-3')和M6R(5'-CGCAGGTTSCSGTAC C-AGTA-3')用来扩增PKS-Ⅰ基因[19],引物KSa(5'-TSGRCTACRTCAACGGSCAC GG-3')和KSβ(5'-TA-CSAGTCSWTCGCCTG GTTC-3')用来扩增PKS-Ⅱ基因[20],引物A3F(5'-GCSTACSYS ATSTACACSTCS-GG-3')和A7R(5'-SASGTCVCCSGTSCGG TAS-3')用来扩增NRPS基因,引物STR-F(5'-CGGCTGCT-CGACCACGAC-3')和STR-R(5'-GTCCTCGATGTC-CCACAG-3')用来扩增APH基因[21],引物HMGF(5'-GGGCATCGCCGCGACCCTCGTCGACGAGCG-3')和HMGR(5'-GCGATGACGGCG AGGC GGCGGGCG-TTCTC-3')用来扩增HMG-CoA基因[12]。扩增产物进行电泳检测,目的条带进行割胶回收纯化,克隆测序,通过Blast比对进行验证。
1.2.5 拮抗活性初筛采用抑菌圈法检测分离到的放线菌对金黄色葡萄球菌(Staphylococcus aureus,菌种编号ATCC 25923)、大肠杆菌(Escherichia coli,菌种编号ATCC 25922)的拮抗活性(本部分实验病原菌均购于中国农业菌种保存中心)。使用血球计数板将病原菌调制成108个/ mL浓度的菌悬液,将金黄色葡萄球菌、大肠杆菌菌悬液取100 μL涂布到LB培养基上,再在平板上接种放线菌的菌饼(琼脂块直径0.6 cm)。LB培养平板37℃培养24-48 h。采用平板对峙培养法检测分离菌对给新疆主要经济作物造成危害的棉花枯萎病原菌(Fusarium oxysperiumsp. Vasinfectum,菌种编号ACCC 31038)、棉花黄萎病原菌(Verticillium dahliae,菌种编号ACCC 36211)和辣椒疫霉病原菌(Phytophthoracapsici,菌种编号ACCC 36278)的拮抗活性,将棉花枯、黄萎病原菌和辣椒疫霉病原菌菌悬液取100 μL涂布到PDA培养基上,接种放线菌(悬液浓度及方法同上)PDA培养平板28℃培养5 d。测量以上两组实验的抑菌圈直径(病原菌的菌落/丝与放线菌之间形成的隔离带外圈的直径),每个菌种做3组重复,取3组重复平均值,判断放线菌对病原菌的拮抗活性。
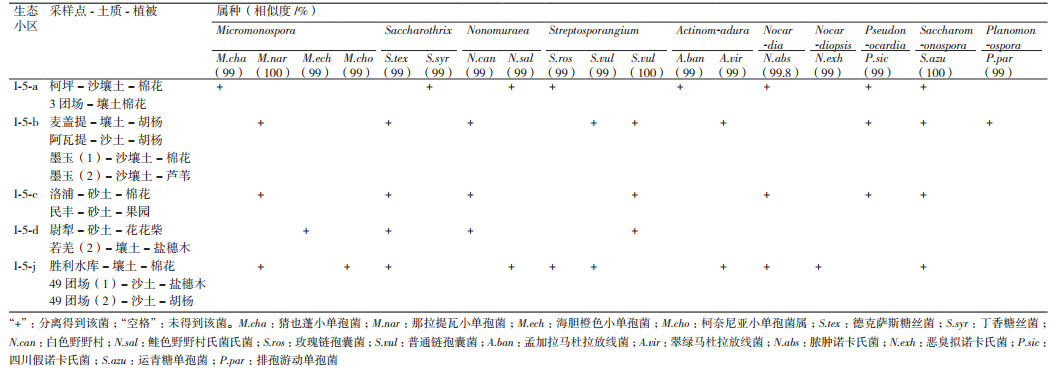
2 结果 2.1 生态小区稀有放线菌分布分析 2.1.1 生态小区稀有放线菌组成状况对分离到的放线菌菌株先观察其菌落形态特征,选择形态差异明显的稀有放线菌菌株,并在ISP 4培养基上插片培养1周,取片进行显微观测,记录其菌体、基丝形态,比照《放线菌的分类和鉴定》(阎逊初,1992),并结合16S rRNA基因序列在Blast上的比对结果进行菌属鉴定。结果(表 2)显示分离得18株稀有放线菌,属于放线菌的10个属。
不同生态小区稀有放线菌分布种类由多到少依次为:塔里木河河岸林生态小区(I-5-j)10株,分别属于8属;塔克拉玛干沙漠和田河西部生态小区(I-5-b)9株,分别属于8属;塔里木北部壤质荒漠生态小区(I-5-a)8株,分别属于8个属;塔克拉玛干沙漠中部生态小区(I-5-c)7株,分别属于7属;塔克拉玛干沙漠塔里木河东部生态小区(I-5-d)4株,分别属于4个属。
从稀有放线菌在生态小区的分布情况看,三项指数丰富度、均匀度、多样性均为壤质土壤小区较高(表 3),反映出壤质土壤较为适宜微生物生长,其种类也很丰富。此外,从总体上看,不同植被土样放线菌分布,以耕作土多样性较为丰富,这一点与其它研究者结论基本相符,原因可能为耕作土水分充足、施肥较多、熟化程度高,利于稀有放线菌繁衍。
依据每个生态小区稀有放线菌组成、菌数(CFU计,数据未列出),计算不同小区的物种均匀度(Evenness)、丰富度(Shannon)和多样性(Simpson)指数。结果(表 3)显示丰富度指数以塔里木北部壤质荒漠生态小区最大,为2.382 1,该小区物种所涵盖属的数目最多(属数相同情况下,各属的个体数目分配均匀性最高小区多样性指数最高);物种均匀度指数以塔克拉玛干沙漠塔里木河东部生态小区最大,为0.871 8,说明该小区各个菌种的个体数目分配均匀性最高;Simpson指数是综合均匀度和丰富度的一项多样性指数,以塔里木北部壤质荒漠生态小区最大,为0.779 9,该小区的稀有放线菌属数多且各属的种数较为均匀,多样性最好。
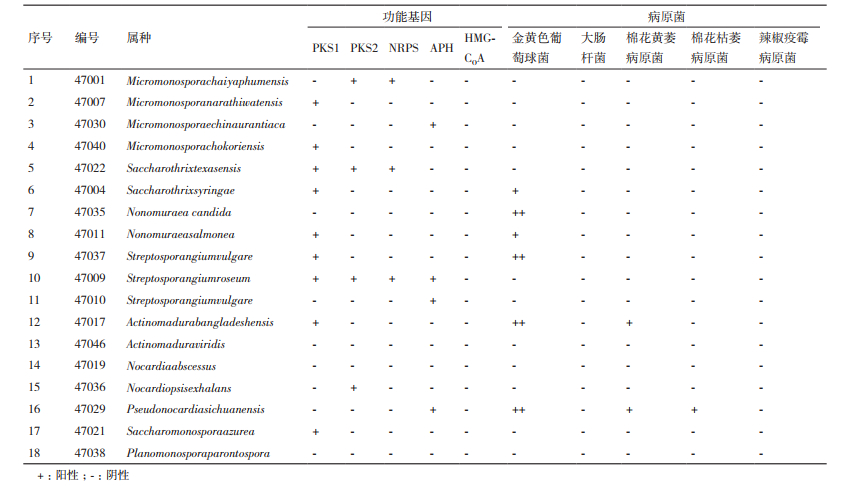
2.2 抗生素合成相关基因及菌株的拮抗效果检测利用5对引物通过PCR扩增菌株抗生素合成相关基因,测得序列通过Blast比对进行验证。验证序列与相关功能基因相符为“阳性(+)”,不符为“阴性(-)”。对分离菌对病原菌的对峙培养平板进行检测,抑菌圈直径 < 0.2 cm为无拮抗性(阴性“-”),0.2 cm≤抑菌圈直径≤0.4 cm为弱拮抗性(阳性“+”),抑菌圈直径>0.4 cm为强拮抗性(++)、对分离菌的5种功能基因的分布及拮抗检测结果见表 4。
18株稀有放线菌中,有14株菌含有1种以上的功能基因,其中的9株菌含有Ⅰ型PKS基因,4株菌含有Ⅱ型PKS基因,3株菌含有NRPS基因,4株菌含有APH基因,没有检测到HMG-COA基因。有一株链孢囊菌含有4种功能基因,有合成多种抗生素的潜力。Ⅰ型PKS在稀有放线菌中的分布较为普遍,在这18株菌能占到有50%的比例,有产生大环内酯类和聚醚类等抗生素的潜力。HMG-COA基因在这18株菌中没有检测到,可能不具有产生异戊二烯化合物的潜力,通过检测,可以为有针对性的筛选产次级代谢产物稀有放线菌提供指导。
观察放线菌的拮抗效果[22]。菌株47017、47029、47035和47037对金黄色葡萄球菌有强的拮抗活性,菌株47004和47011对金黄色葡萄球菌有弱拮抗活性。18株菌对大肠杆菌没有拮抗活性。菌株47017对棉花黄萎病原菌有拮抗活性,菌株47029对棉花枯萎病原菌和棉花黄萎病原菌都有拮抗活性,其它菌株没有拮抗3种植物病原真菌的活性。以含APH基因的47029菌株拮抗种类最多,表现出了广谱抗菌活性。有4株含有PKS Ⅰ型基因的菌对金黄色葡萄球菌表现出拮抗性,菌株所含基因种类与其对病原菌拮抗性效果见表 4。
3 讨论从分离结果来看,几个样点的稀有放线菌种类较多。一些产生物活性物质的重要菌源,如小单孢菌、马杜拉放线菌、诺卡氏菌等都能分离到。但从分离的角度讲,由于本研究采样点集中于塔里木河沿线地区及塔中地区,涉及生态区域仅占整个盆地1/3左右(共计13个生态小区)。加之实验虽选用了一些分离效率比较高的培养基,但实际上很多稀有放线菌培养条件特殊[23]。因此,本实验可能无法分离到一些特殊的微生物。研究者仍需要不断拓展分离源和创新分离手段来开发这些资源。
本次分离菌株含有较为丰富PKS、NRPS及APH基因,基因分布以PKS Ⅰ型最多,未检出HMG-CoA还原酶基因。PKS Ⅰ型基因阳性率为50%,Ⅱ型PKS为22.2%,NRPS为16.7%,APH为22.2%。除PKS Ⅰ型基因外,其它基因检出率与已报道链霉菌的检出率相当[24]。有的菌株同时含有多种功能基因(如链孢囊菌),因而可能产生不同的抗菌或细胞毒活性物质,从这一层面讲,本研究所分离的放线菌菌群具有更为复杂的多样性。此外,两株亲缘关系极近的菌株47010、47037,含有不同的抗生素合成基因,这也说明,菌株的产活性物质能力具有菌株特异性而非菌种特异性,通过分子检测的手段可以忽略菌株之间的相似性而快速的分析菌株的产抗潜力。因此,分子检测不仅能检验菌株的产抗潜力,也是同一物种不同菌株间进行细致分类的研究方法之一。
本研究基因和活性之间的关系不能一一对应,如菌株47035虽然没有抗生素合成基因,但具有拮抗金黄色葡萄球菌的活性。可能原因:一是,这些菌株产生其他种类的抗生素,而我们检测的基因有限,出现“漏”筛。二是,PKS、NRPS等基因本身存在多样性,同一种基因的表达也可能在结构和活性上有差别。
此外,18株放线菌中有14株具有抗生素合成相关基因,但只有6株具有拮抗病原菌的活性,其可能原因:一是所选取靶标种类较少,放线菌产生的抗生素对上述5种靶标菌不具有拮抗性(但可能对其它靶标菌具有作用),需要增加靶标菌检测;二是野生型的菌株可能只产生痕量的次级代谢产物,或者合成基因沉默表达,从而对靶标菌不能表现出拮抗性;三是检测培养基不合适。对于这些菌株,笔者认为不能简单定义为“无用菌株”。因为它们往往含有次级代谢产物的合成基因簇。通过激活其次级代谢产物合成基因的表达,如摸索培养、发酵条件,找出抗生素合成基因表达的调控因素,或者通过基因操作如核糖体工程[25]等手段,激活潜在“有用”次级代谢产物相关基因表达,甚至通过诱变,使菌株突变产生新的化合物,都可能使其功能基因发挥作用。这也是分子手段筛选产抗生素菌的一项优势所在,不仅提高了筛选效率,也能充分挖掘潜在的有用菌源。
4 结论本实验从塔里木盆地5个生态小区土样中分离稀有放线菌,经16S rRNA序列及形态分析,鉴定分离得18种菌株,其中塔里木北部壤质荒漠生态小区菌种丰富度指数最大,为2.3821。通过扩增分离菌株产抗生素相关基因Ⅰ型PKS、Ⅱ型PKS、NRPS、APH及HMG-CoA,并进行电泳分析,有14株菌含有抗生素合成相关基因,其中4株含有多种基因。检测分离菌株对5种主要动植物病原菌的抗菌活性,有6株具有拮抗活性。其中2株具有广谱抗菌活性。本研究基因和活性之间关系并非一一对应。
| [1] |
Kavita T, Gupta RK. Rare actinomycetes:a potential storehouse for novel antibiotics[J]. Critical Reviews in Biotechnology, 2012, 32(2): 108-132. DOI:10.3109/07388551.2011.562482 |
| [2] |
Bérdy J. Thoughts and facts about antibiotics:where we are now and where we are heading[J]. Journal of Antibiotics, 2012, 65(8): 385-395. DOI:10.1038/ja.2012.27 |
| [3] |
Yang J, Zhang W, Chen H, et al. Biocontrol of RhizoctoniasolaniKühn using fermentative solution of Streptomyces corchorusii strain NF0919[J]. Agricultural Biotechnology, 2015, 4(1): 59-61. |
| [4] |
Reddy KRK, Jyothi G, Sowjanya C, et al. Plant growth-promoting actinomycetes:Mass production, delivery systems, and commercialization[M]. Singapore: Springer, 2016: 287-298.
|
| [5] |
阮继生, 黄英. 放线菌快速鉴定与系统分类[M]. 北京: 科学出版社, 2011.
|
| [6] |
Horikoshi K. Extremophiles Handbook[M]. Tokyo: Springer, 2011: 1203-1240.
|
| [7] |
关统伟, 车振明, 向文良, 等. 罗布泊盐湖沉积物放线菌的群落结构[J]. 应用与环境生物学报, 2013, 19(3): 466-470. |
| [8] |
Azman AS, Othman I, Velu SS., et al. Mangrove rare actinobacteria:taxonomy, natural compound, and discovery of bioactivity[J]. Frontiers Microbiology, 2015, 6(856): 856. |
| [9] |
Kumar RR, Jadeja VJ. Isolation of actinomycetes:A complete approach[J]. International Journal Current Microbiology and Applied Sciences, 2016, 5(5): 606-618. DOI:10.20546/ijcmas |
| [10] |
倪健, 郭柯, 刘海江, 等. 中国西北干旱区生态区划[J]. 植物生态学报, 2005, 29(2): 175-184. DOI:10.3321/j.issn:1005-264X.2005.02.001 |
| [11] |
Wagner N, Oβwald C, Biener R, et al. Comparative analysis of transcriptional activities of heterologous promoters in the rare actinomycete Actinoplanesfriuliensis[J]. J Biotechnol, 2009, 142(3): 200-204. |
| [12] |
Sigmund JM, Clark DC, Rainey FA, et al. Detection of eubacterial 3-hydroxy-3-methylglutaryl coenzyme A reductases from natural populations of actinomycetes[J]. Microbial Ecology, 2003, 46(1): 106-112. DOI:10.1007/s00248-002-2029-5 |
| [13] |
Subramani R, Aalbersberg W. Culturable rare actinomycetes:diversity, isolation and marine natural product discovery[J]. Applied Microbiology and Biotechnology, 2013, 97(21): 9291-9321. DOI:10.1007/s00253-013-5229-7 |
| [14] |
李晓华, 龙慈凡, 周秀芬, 邓子新. 链霉菌质粒pSET152电转化稀有放线菌小单孢菌的研究[J]. 微生物学报, 2007, 47(4): 718-720. DOI:10.3321/j.issn:0001-6209.2007.04.030 |
| [15] |
刘琳琳.稀有放线菌的筛选及新种鉴定[D].哈尔滨: 东北农业大学, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10224-1012515493.htm
|
| [16] |
黄路枝.代谢农药活性物质稀有放线菌的筛选[D].杨凌: 西北农林科技大学, 2007. http://cdmd.cnki.com.cn/Article/CDMD-10712-2007189032.htm
|
| [17] |
张姗姗, 来航线, 韦小敏, 等. 红碱淖湖滨土壤微生物及稀有放线菌研究[J]. 西北农林科技大学学报:自然科学版, 2013, 41(8): 1-8. |
| [18] |
蒋莲秀, 吴越, 陈建宏, 等. 具有广谱抗菌活性的红树林稀有放线菌的分离及鉴定[J]. 中国抗生素杂志, 2017, 42(4): 311-317. DOI:10.3969/j.issn.1001-8689.2017.04.009 |
| [19] |
Ayuso A, Genilloud O. New PCR primers for the screening of NRPS and PKS-Ⅰ systems in actinomycetes:detection and distribution of these biosynthetic gene sequences in major taxonomic groups[J]. Microbial Ecology, 2005, 49(1): 10-24. DOI:10.1007/s00248-004-0249-6 |
| [20] |
Ayuso A, Clark D, Salazar O, et al. A novel strain de-replication approach based on PKS and NRPS biosynthetic pathway diversity in actinomycetes[J]. Applied Microbiology and Biotechnology, 2005, 67(6): 795-806. DOI:10.1007/s00253-004-1828-7 |
| [21] |
Anderson AS, Clark DJ, Gibbons PH, et al. The detection of diverse aminoglycoside phosphotransferases within natural populations of actinomycetes[J]. Journal of Industrial Microbiology and Biotechnology, 2002, 29(2): 60-69. DOI:10.1038/sj.jim.7000260 |
| [22] |
吴家法, 吴思婷, 李智鸣, 等. 茅尾海红树林土壤可培养放线菌多样性及其抗尖孢镰刀菌活性分析[J]. 中国抗生素杂志, 2017, 42(4): 294-301. DOI:10.3969/j.issn.1001-8689.2017.04.007 |
| [23] |
王雪, 放线菌新抗生素生物合成基因的发掘[D].武汉: 华中农业大学, 2011. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2004092
|
| [24] |
矫文策, 海洋放线菌的分离鉴定及Ⅰ型聚酮合成酶基因筛选的研究[D].大连: 大连理工大学, 2007. http://cdmd.cnki.com.cn/article/cdmd-10141-2009206664.htm
|
| [25] |
金城. 聚酮类抗生素生物合成基因簇间的关系[J]. 微生物学通报, 2014, 41(7): 1470. |