天然橡胶是不可或缺的工业原料和重要的战略物质,其主要来源是橡胶树(Hevea brasliensi)。受自然环境的制约,我国能种植橡胶树的地区十分有限,且种植在这些区域的橡胶树经常遭受低温和干旱等非生物逆境胁迫。非生物逆境严重影响橡胶树的生长和产胶,但目前对于橡胶树应答非生物胁迫的分子机制知之甚少。克隆并鉴定橡胶树非生物胁迫响应基因的功能将为其抗性遗传改良奠定基础。
Metacaspase属于半胱氨酸蛋白酶家族成员,广泛存在于植物、真菌和原生生物中[1-2]。Metacaspase含有caspase-like结构域,与动物天冬氨酸特异性半胱氨酸蛋白酶(Cysteinyl aspartate-specific proteinases,caspase)结构十分相似[2-3]。Caspases是动物程序性细胞死亡(Programmed cell death,PCD)的关键调节因子[4]。研究表明,与动物caspases相似,植物metacaspases也是各类PCD的重要调节因子。模式植物拟南芥基因组中含有9个metacaspase基因——AtMC1-AtMC9[5]。其中,AtMC1和AtMC2拮抗地调控过敏反应相关的PCD[6];AtMC4正调控生物和非生物胁迫诱导的PCD[7];AtMC8是UVC和H2O2诱导PCD所必需的[8];而AtMC9则是木质部管状分子PCD的重要调控因子[9]。除拟南芥外,其他植物中也克隆和鉴定了一些在PCD过程中发挥作用的metacaspases,如小麦TaMCA1[10]、辣椒Camc9[11]、杨树PttMC13和PttMC14[12]等。
活性氧(Reactive oxygen species,ROS)是非生物胁迫诱导的植物PCD发生的关键信号分子[13]。干旱、高盐和低温等非生物胁迫均会导致ROS产生,ROS的过量积累会导致氧化胁迫,诱发PCD。研究证实,metacaspases在调控氧化胁迫诱导的PCD中发挥重要作用。酵母metacaspase基因YCA1是H2O2诱导PCD所必需的;酵母中敲除YCA1会抑制H2O2诱导的PCD,而超量表达YCA1会增强H2O2诱导的PCD[14]。Hao等[10]发现小麦TaMCA1具有与YCA1相似的功能,在酵母中表达TaMCA1会加速H2O2诱导的细胞死亡,降低了对氧化胁迫的抗性。拟南芥AtMC4和AtMC8也被证实正调控氧化胁迫诱导的PCD[7-8]。拟南芥中超量表达AtMC4加速了氧化胁迫诱导的PCD[7]。拟南芥原生质体中超量表达AtMC8同样增强了H2O2诱导的PCD,而敲除AtMC8则降低了H2O2诱导的PCD[8]。
通过对葡萄、水稻和番茄metacaspase家族基因启动子区的顺式作用元件进行分析发现,大多数metacaspase基因启动子区含有非生物胁迫应答相关的顺式作用元件[15-17],暗示metacaspases可能在植物非生物胁迫应答中发挥重要作用。但有关metacaspases在低温、干旱和盐胁迫等非生物胁迫应答中的具体功能仍不清楚。前期研究已在全基因水平上鉴定到9个橡胶树metacaspase基因——HbMC1-HbMC9;对这些基因进行系统地表达分析发现,HbMC2的表达受干旱和高盐等非生物胁迫调控[18]。为明确HbMC2在非生物胁迫中的作用,本研究利用酵母表达系统初步分析HbMC2在氧化、干旱和盐胁迫中的功能,旨在为进一步揭示HbMC2在橡胶树中的功能奠定基础。
1 材料与方法 1.1 材料橡胶树品系热研7-33-97胶乳cDNA为橡胶树死皮发生机理实验室前期研究准备样品;酵母表达载体pYES2和酿酒酵母菌株INVSc1、反转录试剂盒、限制性内切酶、DNase Ⅰ和T4 DNA连接酶购自Thermo Fisher Scientific公司;琼脂糖凝胶回收试剂盒和质粒提取试剂盒购自OMEGA公司;pEASY-Blunt Simple Cloning Vector、TransStart FastPfu DNA Polymerase、TransStart Taq DNA Polymerase、大肠杆菌Trans1-T1和DH5α感受态细胞购自北京全式金生物技术有限公司;DNA marker和DO Supplement-Ura购自TaKaRa公司;酵母总RNA快速提取试剂盒、半乳糖和葡萄糖等购自生工生物工程(上海)股份有限公司,引物合成及测序也由该公司完成。
1.2 方法 1.2.1 HbMC2酵母表达载体的构建根据橡胶树HbMC2(GenBank登录号:KU188282)序列设计扩增编码区的引物,并在引物5'端分别引入Bam HI和Not Ⅰ酶切位点和保护碱基(HbMC2-F:5'-GCGGATCCATGTTCATGCTCGTCGACTG-3',HbMC2-R:5'-TAGCGGCCGCTCAGAAAGAAAAAGGTCTGG-3')。以橡胶树品系热研7-33-97胶乳cDNA为模板,PCR扩增目标基因。PCR产物经1%琼脂糖凝胶电泳检测后,采用琼脂糖凝胶回收试剂盒回收目标基因片段。将回收片段与pEASY-Blunt Simple Cloning Vector连接,转化大肠杆菌Trans1-T1感受态细胞,涂于含100 mg/L卡那霉素的LB固体培养基上,37℃培养过夜,挑单克隆进行PCR检测,并对阳性克隆进行测序分析。
采用质粒提取试剂盒提取测序正确克隆及酵母表达载体pYES2质粒,Bam HI和Not Ⅰ进行双酶切。酶切产物经1%琼脂糖凝胶电泳后,切胶回收目标条带。采用T4 DNA连接酶将回收的目标基因和pYES2载体骨架连接。连接产物转化大肠杆菌DH5α感受态细胞,并涂于含100 mg/L氨苄青霉素的LB固体平板上,37℃培养过夜。挑取单克隆,利用pYES2载体T7引物(5'-TAATACGACTCACTAT-AGGG-3')和HbMC2-R引物进行PCR鉴定。阳性克隆进一步测序验证,获得酵母表达载体pYES2-HbMC2。
1.2.2 重组载体的转化及阳性检测提取pYES2-HbMC2和空载体pYES2质粒,并通过醋酸锂转化法将它们分别转入酿酒酵母菌株INVSc1感受态细胞中,获得重组酵母INVSc1(pYES2-HbMC2)和对照酵母INVSc1(pYES2)。将转化酵母菌液涂在SC-Ura固体选择培养基上,30℃培养3 d。挑取单克隆,30℃,200 r/min振荡培养过夜。将培养液沸水浴5 min,冰上放置5 min,多次反复以破碎细胞。以细胞破碎液作为模板进行PCR检测。PCR反应体系配制和条件设置参照TransStart Taq DNA Polymerase说明书。PCR产物经1%琼脂糖凝胶电泳检测。
1.2.3 HbMC2在重组酵母中的表达检测将重组酵母INVSc1(pYES2-HbMC2)和INVSc1(pYES2)接种于SC-Ura液体培养基(含2%葡萄糖)中,30℃,200 r/min振荡培养24 h,测OD600值。取一定量的培养物,离心收集菌体,然后用液体诱导培养基(SC-Ura+2%半乳糖)重悬菌体,并调整OD600=0.2,各取50 mL,30℃,200 r/min诱导培养,于诱导培养24、36和48 h时,采用酵母总RNA快速提取试剂盒提取总RNA。DNaseⅠ去除DNA后,反转录合成第一链cDNA。采用半定量RT-PCR检测HbMC2在重组酵母中的表达,以酿酒酵母Actin(GenBank登录号:L00026.1)为内参[19]。产物经1%琼脂糖凝胶电泳检测。
1.2.4 重组酵母的胁迫处理为探究HbMC2在非生物胁迫应答中的作用,在液体培养条件下比较氧化、盐及干旱胁迫下重组酵母INVSc1(pYES2-HbMC2)和对照酵母INVSc1(pYES2)的生长差异。将保存的酵母菌划线活化后接种于液体诱导培养基中,30℃,200 r/min诱导培养36 h。取一定量的培养液,离心收集菌体,然后用含0、3 mmol/L H2O2、0.8 mol/L NaCl或1.2 mol/L山梨醇的液体诱导培养基重悬菌体,并调整OD600=0.2。各取10 mL,30℃,200 r/min振荡培养48 h,测量OD600值。每处理3次重复。
同时比较高浓度H2O2、NaCl或山梨醇胁迫处理后重组酵母和对照酵母的存活差异。取诱导培养36 h的菌液5 mL,离心收集菌体,然后分别重悬于10 mmol/L H2O2、3 mol/L NaCl或3 mol/L山梨醇溶液中,并调整OD600=1.0。混匀后,30℃,160 r/min,处理6 h(H2O2处理)或36 h(NaCl和山梨醇处理)。以菌体重悬于无菌水中处理10 h或36 h作为对照。每处理3次重复。处理后的菌液进行5个梯度的5倍稀释。稀释后,各取5 μL点样在SC-Ura固体培养基上。30℃培养3 d后,比较2种菌落的存活差异。
此外,还比较了重组酵母和对照酵母在含H2O2的固体培养基上的生长差异。取诱导培养36 h的菌液,并调整OD600=1.0。随后将2种菌液进行5个梯度的5倍稀释。稀释后,各取5 μL点样在含有0或3 mmol/L H2O2的固体诱导培养基上,每处理3次重复。30℃培养3 d后,比较2种酵母的生长差异。
2 结果 2.1 转HbMC2重组酵母的获得根据橡胶树HbMC2序列和酵母表达载体pYES2多克隆位点信息设计了添加Bam HI和Not Ⅰ酶切位点的引物,并采用RT-PCR方法从橡胶树中扩增得到与预期片段长度(1 104 bp)一致的条带。将目的条带回收后与克隆载体连接,转化大肠杆菌。对PCR筛选的阳性单克隆进行测序分析。结果表明,扩增目标片段序列与预期完全一致。
抽提测序正确克隆及酵母表达载体pYES2质粒,Bam HI和Not Ⅰ双酶切后,回收目标片段,连接后转化大肠杆菌。PCR检测及测序结果显示,HbMC2已整合到pYES2中,酵母表达载体pYES2-HbMC2构建成功。采用醋酸锂转化法分别将pYES2-HbMC2和pYES2质粒转入酿酒酵母菌株INVSc1中。转化酵母接种在SC-Ura固体选择培养基上培养3 d后,挑选pYES2-HbMC2和pYES2重组酵母单克隆各3个,PCR鉴定阳性转化子。琼脂糖凝胶电泳检测结果(图 1)显示,所有单克隆均扩增与预期片段长度一致的条带,表明pYES2-HbMC2和pYES2质粒已转入酿酒酵母INVSc1,获得重组酵母INVSc1(pYES2-HbMC2)和对照酵母INVSc1(pYES2)。
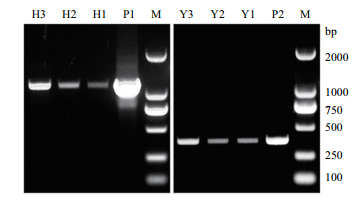
|
| 图 1 重组酵母的PCR检测 M:DL2000 DNA Marker;P1:pYES2-HbMC2质粒;H1-H3:3个pYES2-HbMC2重组酵母单克隆;P2:pYES2质粒;Y1-Y3:3个pYES2重组酵母单克隆 |
采用半定量RT-PCR检测了HbMC2在重组酵母和对照酵母中的诱导表达情况。经半乳糖诱导后,HbMC2在重组酵母INVSc1(pYES2-HbMC2)中明显表达,而对照酵母INVSc1(pYES2)中检测不到HbMC2表达;整个诱导过程中,重组酵母INVSc1(pYES2-HbMC2)中HbMC2的表达呈先升高后降低趋势,在诱导36 h时,HbMC2的表达水平最高(图 2)。因此,后续的功能鉴定中采用诱导培养36 h的菌体用于各种非生物胁迫处理。
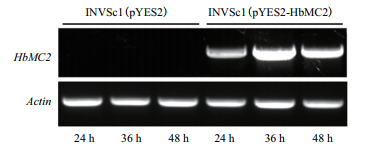
|
| 图 2 HbMC2在重组酵母中的诱导表达模式分析 |
将诱导培养36 h的重组酵母和对照酵母菌液梯度稀释后,点样在含有0(对照)或3 mmol/L H2O2的固体诱导培养基上,30℃培养3 d后,观察比较2种酵母菌的生长差异(图 3),对照(非胁迫)条件下,HbMC2重组酵母和对照酵母的菌斑数及生长状况基本一致,无明显差异,表明酵母中表达HbMC2对其正常生长无明显影响。H2O2诱导的氧化胁迫明显抑制了酵母的生长,但重组酵母INVSc1(pYES2-HbMC2)的生长状况明显差于对照酵母INVSc1(pYES2);在稀释54倍后,HbMC2重组酵母的菌斑数明显少于对照酵母。
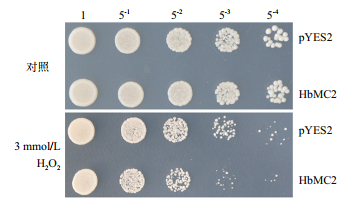
|
| 图 3 HbMC2重组酵母和对照酵母在氧化胁迫下的生长差异 HbMC2:重组酵母INVSc1(pYES2-HbMC2);pYES2:对照酵母INVSc1(pYES2),下同 |
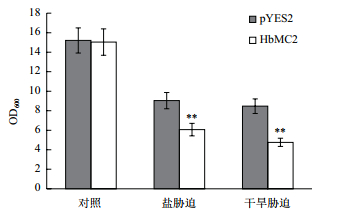
比较HbMC2重组酵母和对照酵母在液体诱导培养基中的生长差异发现,正常条件下,起始菌体浓度一致的两种酵母经48 h震荡培养后,二者的OD600值没有明显差异。但氧化胁迫(3 mmol/L H2O2)条件下培养48 h后,重组酵母INVSc1(pYES2-HbMC2)的OD600值显著低于对照酵母INVSc1(pYES2)(图 4)。以上结果表明,HbMC2的表达抑制了重组酵母在氧化胁迫下的生长,降低了重组酵母对氧化胁迫的抗性。
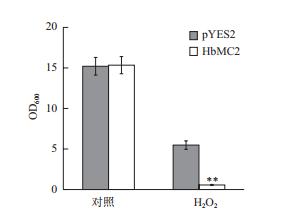
|
| 图 4 氧化胁迫下HbMC2重组酵母和对照酵母菌液OD600值差异分析 对照:无胁迫处理;H2O2:3 mmol/L H2O2处理;**:重组酵母与对照酵母间差异极显著(P < 0.01,t检验),下同 |
比较高浓度H2O2胁迫处理后2种酵母的存活差异发现,10 mmol/L H2O2处理6 h后,INVSc1(pYES2-HbMC2)的菌斑数明显少于INVSc1(pYES2);在稀释53倍后,INVSc1(pYES2)仍有菌斑生长,而INVSc1(pYES2-HbMC2)已无菌斑生长;稀释52倍时,INVSc1(pYES2-HbMC2)的菌斑数不足INVSc1(pYES2)的1/6。而对照组(无胁迫处理)中,两种酵母的生长状况差异不大(图 5)。以上结果表明,氧化胁迫处理后,HbMC2重组酵母的细胞死亡率明显高于对照酵母,HbMC2的高表达会促进氧化胁迫下重组酵母细胞的死亡。
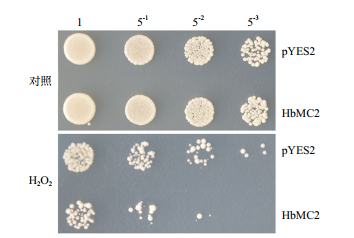
|
| 图 5 氧化胁迫条件下HbMC2重组酵母和对照酵母的存活差异 对照:无胁迫处理;H2O2:10 mmol/L H2O2处理6 h |
橡胶树HbMC2的表达受干旱和盐胁迫的诱导,推测该基因可能在干旱和盐胁迫应答中发挥作用。分别用NaCl和山梨醇胁迫模拟盐和干旱胁迫,比较分析HbMC2重组酵母和对照酵母在含有一定浓度NaCl或山梨醇的液体诱导培养基中的生长差异(图 6)。盐(0.8 mol/L NaCl)或干旱(1.2 mol/L山梨醇)胁迫下培养48 h后,重组酵母INVSc1(pYES2-HbMC2)的OD600值显著低于对照酵母INVSc1(pYES2)。而正常培养条件下,HbMC2重组酵母和对照酵母的OD600值没有明显差异。以上结果表明,HbMC2的表达抑制了重组酵母在盐和干旱胁迫下的生长,降低了其对盐和干旱胁迫的抗性。
|
| 图 6 盐或干旱胁迫下HbMC2重组酵母和对照酵母菌液OD600值差异分析 对照:无胁迫处理;盐胁迫:0.8 mol/L NaCl处理;干旱胁迫:1.2 mol/L山梨醇处理 |
比较高浓度NaCl或山梨醇胁迫处理后HbMC2重组酵母和对照酵母的存活差异发现,3 mol/L NaCl处理36 h后,INVSc1(pYES2-HbMC2)的菌斑数明显少于INVSc1(pYES2);稀释52倍后,INVSc1(pYES2-HbMC2)已无菌斑生长,而INVSc1(pYES2)仍有较多菌斑生长(图 7)。3 mol/L山梨醇处理36 h后,INVSc1(pYES2-HbMC2)的菌斑数也明显少于INVSc1(pYES2);稀释54倍后,INVSc1(pYES2-HbMC2)已无菌斑生长,而INVSc1(pYES2)仍有菌斑生长;53倍稀释条件下,INVSc1(pYES2-HbMC2)的菌斑数不足INVSc1(pYES2)的1/25。而对照(无胁迫处理)组中,2种酵母的生长状况无明显差异(图 7)。以上结果表明,盐和干旱胁迫下,重组酵母INVSc1(pYES2-HbMC2)的死亡率较对照酵母INVSc1(pYES2)明显升高,HbMC2的高表达会促进盐和干旱胁迫下重组酵母细胞的死亡。
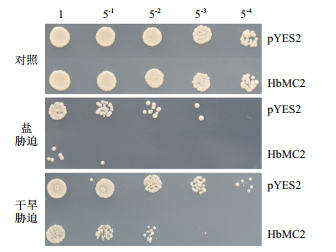
|
| 图 7 盐或干旱处理后HbMC2重组酵母和对照酵母的存活差异 对照:无胁迫处理;盐胁迫:3 mol/L NaCl处理36 h;干旱胁迫:3 mol/L山梨醇处理36 h |
PCD是一种由基因控制的、主动的细胞死亡过程,在植物生长发育、抗病及抗逆等过程中发挥着极其重要的作用[20-21]。研究表明,Metacaspases是植物PCD的重要调节因子。Metacaspases参与的PCD类型有多种,包括生物或非生物胁迫引起的PCD以及发育相关的PCD。鉴于Metacaspases在PCD中重要作用,目前已经对拟南芥[5]、葡萄[15]、水稻[16]、番茄[17]、橡胶树[18]、小麦[22]和黄瓜[23]等物种中的Metacaspase家族基因进行了鉴定分析,明确了一些Metacaspases的生物学功能,但大多数Metacaspase基因的功能还不清楚。橡胶树基因组中含有9个Metacaspase基因——HbMC1-HbMC9[18],但它们的生物学功能均未知。本研究从橡胶树中克隆了HbMC2,并利用酵母表达系统分析了该基因的功能,初步证实在酵母中表达HbMC2会增强氧化、干旱及盐胁迫下重组酵母细胞的死亡,HbMC2是非生物胁迫诱导细胞死亡的一个正调节因子。
植物非生物胁迫应答的分子机制与酵母具有一定的相似性。利用酵母表达系统能快速、准确的鉴定植物逆境响应相关基因在非生物胁迫中的功能。如冯德明等[24]通过将刚毛怪柳ThDREB转入酵母证实该基因过表达能提高抗旱、耐盐和耐重金属等胁迫的能力。此外,酵母表达系统也被成功用于研究植物metacaspase基因的功能,如小麦TaMCA1[10]、拟南芥AtMCP1b和AtMCP2b[25]等。前期研究发现橡胶树HbMC2的表达受干旱和高盐等非生物胁迫调控[18]。为明确HbMC2的功能,本研究同样利用酿酒酵母表达系统分析了该基因在非生物胁迫中作用。结果表明,在酵母中表达HbMC2降低了重组酵母对氧化、干旱及盐胁迫的抗性。目前,有关metacaspase基因在氧化胁迫应答中的功能研究已有不少报道,如小麦TaMCA1[10]、拟南芥AtMC4[7]、AtMC8[8]、AtMCP1b和AtMCP2b[25]等。基因表达分析表明,许多metacaspases的表达受干旱和高盐等非生物胁迫调控,且大多数metacaspase基因启动子区含有非生物胁迫响应相关的顺式作用元件[15-17]。但迄今未见有关metacaspases在干旱和盐胁迫中功能的研究报道。本研究增进了对植物metacaspases在干旱和高盐等非生物胁迫中功能的认识,为进一步探究metacaspases在植物干旱和高盐等非生物胁迫中的功能提供了参考依据。
橡胶树是重要工业原料和战略物资天然橡胶的主要来源,保障我国天然橡胶基本供给能力具有重要意义。我国属于非传统植胶区,橡胶树生长过程中经常遭受低温、台风和季节性干旱等非生物逆境胁迫,这些逆境因子严重制约着我国天然橡胶产业的发展。发掘橡胶树非生物胁迫应答基因并解析其功能将为通过基因工程手段提高橡胶树抗逆性提供候选基因。本研究初步证实在酵母中表达橡胶树HbMC2会降低对非生物胁迫的抗性,HbMC2是非生物胁迫抗性的负调控因子,研究结果为在橡胶树中进一步揭示HbMC2奠定基础。本研究仅在酵母中对HbMC2的功能进行了初步分析,有关HbMC2在橡胶树非生物胁迫应答中的功能仍需进一步鉴定。根据对重组酵母的研究结果,推测敲除或降低HbMC2的表达可能会提高橡胶树对非生物逆境的抗性。
4 结论本研究利用酵母表达系统分析了橡胶树metacaspase基因HbMC2在非生物胁迫应答中的功能,证实酵母中表达HbMC2会抑制氧化、干旱和盐胁迫下重组酵母的生长,增强非生物胁迫诱导的细胞死亡,降低重组酵母对非生物胁迫的抗性。HbMC2负调控非生物胁迫抗性,敲除或抑制HbMC2的表达可能会提高橡胶树的抗逆性。
| [1] |
董京萍, 孔维文, 李斌, 等. 植物Metacaspases的结构和生化特性概述[J]. 安徽农业科学, 2013, 41(28): 11253-11254, 11258. DOI:10.3969/j.issn.0517-6611.2013.28.002 |
| [2] |
Fagundes D, Bohn B, Cabreira C, et al. Caspases in plants:metacaspase gene family in plant stress responses[J]. Funct Integr Genomics, 2015, 15(6): 639-649. DOI:10.1007/s10142-015-0459-7 |
| [3] |
马聪, 孔维文. 植物Metacaspase研究进展[J]. 植物学报, 2012, 47(5): 543-549. |
| [4] |
Shalini S, Dorstyn L, Dawar S, et al. Old, new and emerging functions of caspases[J]. Cell Death Differ, 2015, 22(4): 526-539. DOI:10.1038/cdd.2014.216 |
| [5] |
Tsiatsiani L, Van Breusegem F, Gallois P, et al. Metacaspases[J]. Cell Death Differ, 2011, 18(8): 1279-1288. DOI:10.1038/cdd.2011.66 |
| [6] |
Coll NS, Vercammen D, Smidler A, et al. Arabidopsis type Ⅰ metacaspases control cell death[J]. Science, 2010, 330(6009): 1393-1397. DOI:10.1126/science.1194980 |
| [7] |
Watanabe N, Lam E. Arabidopsis metacaspase 2d is a positive mediator of cell death induced during biotic and abiotic stresses[J]. Plant J, 2011, 66(6): 969-982. DOI:10.1111/tpj.2011.66.issue-6 |
| [8] |
He R, Drury GE, Rotari Ⅵ, et al. Metacaspase-8 modulates programmed cell death induced by ultraviolet light and H2O2 in Arabidopsis[J]. J Biol Chem, 2008, 283(2): 774-783. DOI:10.1074/jbc.M704185200 |
| [9] |
Bollhoner B, Zhang B, Stael S, et al. Post mortem function of AtMC9 in xylem vessel elements[J]. New Phytol, 2013, 200(2): 498-510. DOI:10.1111/nph.12387 |
| [10] |
Hao Y, Wang X, Wang K, et al. TaMCA1, a regulator of cell death, is important for the interaction between wheat and Puccinia striiformis[J]. Sci Rep, 2016, 6: 26946. DOI:10.1038/srep26946 |
| [11] |
Kim SM, Bae C, Oh SK, et al. A pepper(Capsicum annuum L.)metacaspase 9(Camc9)plays a role in pathogen-induced cell death in plants[J]. Mol Plant Pathol, 2013, 14(6): 557-566. DOI:10.1111/mpp.2013.14.issue-6 |
| [12] |
Bollhoner B, Jokipii-Lukkari S, Bygdell J, et al. The function of two type Ⅱ metacaspases in woody tissues of Populus trees[J]. New Phytol, 2018, 217(4): 1551-1565. DOI:10.1111/nph.14945 |
| [13] |
Petrov V, Hille J, Mueller-Roeber B, et al. ROS-mediated abiotic stress-induced programmed cell death in plants[J]. Front Plant Sci, 2015, 6: 69. |
| [14] |
Madeo F, Herker E, Maldener C, et al. A caspase-related protease regulates apoptosis in yeast[J]. Mol Cell, 2002, 9(4): 911-917. DOI:10.1016/S1097-2765(02)00501-4 |
| [15] |
Zhang C, Gong P, Wei R, et al. The metacaspase gene family of Vitis vinifera L. :characterization and differential expression during ovule abortion in stenospermocarpic seedless grapes[J]. Gene, 2013, 528(2): 267-276. DOI:10.1016/j.gene.2013.06.062 |
| [16] |
Huang L, Zhang H, Hong Y, et al. Stress-responsive expression, subcellular localization and protein-protein interactions of the rice metacaspase family[J]. Int J Mol Sci, 2015, 16(7): 16216-16241. DOI:10.3390/ijms160716216 |
| [17] |
Liu H, Liu J, Wei Y. Identification and analysis of the metacaspase gene family in tomato[J]. Biochem Biophys Res Commun, 2016, 479(3): 523-529. DOI:10.1016/j.bbrc.2016.09.103 |
| [18] |
Liu H, Deng Z, Chen J, et al. Genome-wide identification and expression analysis of the metacaspase gene family in Hevea brasiliensis[J]. Plant Physiol Biochem, 2016, 105: 90-101. DOI:10.1016/j.plaphy.2016.04.011 |
| [19] |
Teste MA, Duquenne M, Francois JM, et al. Validation of reference genes for quantitative expression analysis by real-time RT-PCR in Saccharomyces cerevisiae[J]. BMC Mol Biol, 2009, 10: 99. DOI:10.1186/1471-2199-10-99 |
| [20] |
Daneva A, Gao Z, Van Durme M, et al. Functions and regulation of programmed cell death in plant development[J]. Annu Rev Cell Dev Biol, 2016, 32: 441-468. DOI:10.1146/annurev-cellbio-111315-124915 |
| [21] |
Kabbage M, Kessens R, Bartholomay LC, et al. The life and death of a plant cell[J]. Annu Rev Plant Biol, 2017, 68: 375-404. DOI:10.1146/annurev-arplant-043015-111655 |
| [22] |
Bostancioglu SM, Tombuloglu G, Tombuloglu H. Genome-wide identification of barley MCs(metacaspases)and their possible roles in boron-induced programmed cell death[J]. Mol Biol Rep, 2018. DOI:10.1007/s11033-018-4154-3 |
| [23] |
Zhou Y, Hu L, Jiang L, et al. Genome-wide identification, characterization, and transcriptional analysis of the metacaspase gene family in cucumber(Cucumis sativus)[J]. Genome, 2018, 61(3): 187-194. DOI:10.1139/gen-2017-0174 |
| [24] |
冯德明, 温佩颖, 赵畅, 等. 刚毛柽柳ThDREB基因在酵母中的表达及抗逆能力分析[J]. 植物研究, 2017, 37(1): 63-68. |
| [25] |
Watanabe N, Lam E. Two Arabidopsis metacaspases AtMCP1b and AtMCP2b are arginine/lysine-specific cysteine proteases and activate apoptosis-like cell death in yeast[J]. J Biol Chem, 2005, 280(15): 14691-14699. DOI:10.1074/jbc.M413527200 |




