中国是世界上枸杞生产大国,2012年枸杞栽培面积和产量分别为1.454×105 hm2和2.049×105 t,均居世界第一[1]。宁夏是世界枸杞发源地和正宗原产地,品种资源丰富,现已审定枸杞品种有20个,收集保存种质资源2 000份以上[2]。随着枸杞品种选育和推广速度加速,各地品种资源交流更加频繁,造成同物异名和同名异物现象非常普遍。另外,枸杞品种选育主要采用群体选优方式,使得核心骨干亲本反复利用,如从“大麻叶”生产园中先后选育出“宁杞1号”、“宁杞2号”、“宁杞4号”和“精杞4号”等[3-6]品种,造成品种间遗传差异变小,遗传背景狭窄,为枸杞品种的准确鉴定提出更高的要求。
传统的形态学、同工酶等品种鉴定方法易受环境的影响,不仅需要具备扎实的专业理论知识,而且耗时费力。随着DNA分子标记技术快速发展,SSR标记具有共显性、多态性高、扩增结果稳定及重复性好等优点,已被国际植物品种保护联盟(UPOV)纳入农作物品种制定DUS(Distinctness,uniformity,stability)指南内容,确定为植物新品种保护最广泛应用的分子标记体系[7]。目前,利用SSR标记构建了葡萄、梨、棉花、水稻及苹果等[8-12]植物分子身份证。
近年来,基于高通量测序技术开发了大量的枸杞SSR引物,筛选出了一批多态性好的引物。党少飞等[13]对大麻叶枸杞进行简化基因组测序(RAD-seq),分析了SSR基元分布特点,其中三碱基重复单元最丰富,占重复序列总数的66.5%。王瑛等[14]通过对宁夏枸杞和黑果枸杞转录组测序开发了EST-SSR引物,从中筛选出10对EST-SSR引物可将7个枸杞品种逐一区分开来。尹跃等[15]从11对SSR引物中筛选出4对引物可将12个枸杞品种完全区分开来,构建了12个品种指纹图谱。邵千顺等[16]利用4对SSR引物构建了17份枸杞种质的DNA指纹图谱。Chen等[17]基于枸杞果实转录组测序,开发了一批EST-SSR引物并对11份枸杞种质进行遗传多样性分析。上述研究主要是基于转录组和简化基因组测序开发的SSR标记,这些标记没有定位到染色体和连锁群上,在进行枸杞品种鉴定及遗传多样性分析时所反映供试材料的信息不够全面。然而,基于枸杞全基因组水平的SSR标记的开发及应用还未见报道。
本研究是基于枸杞全基因组测序结果(未公布)基础上,从600对SSR引物中筛选出多态性高、稳定性好、均匀分布在枸杞12条染色体上的24对引物,以16个枸杞品种为试材,建立枸杞品种SSR分子身份证,以期枸杞品种鉴定及知识产权的保护提供科学依据。
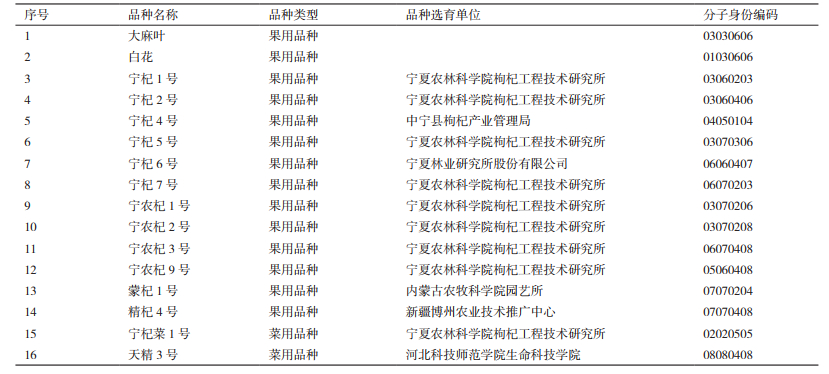
1 材料与方法 1.1 材料所用的16个枸杞品种(表 1),其中包括果用品种14个和菜用品种2个,均采自宁夏农林科学院国家枸杞工程技术研究中心枸杞种质资源圃(38°38'49″N,106°9'10″E)。
采用试剂盒法(天根,DP320)提取枸杞叶片基因组DNA。用1%琼脂糖凝胶电泳检测DNA质量,用BioPhotometer plus核酸蛋白仪(德国,Eppendorf)测定DNA浓度和纯度,并用灭菌的ddH2O将溶液稀释至50 ng/μL,用于后续PCR试验。
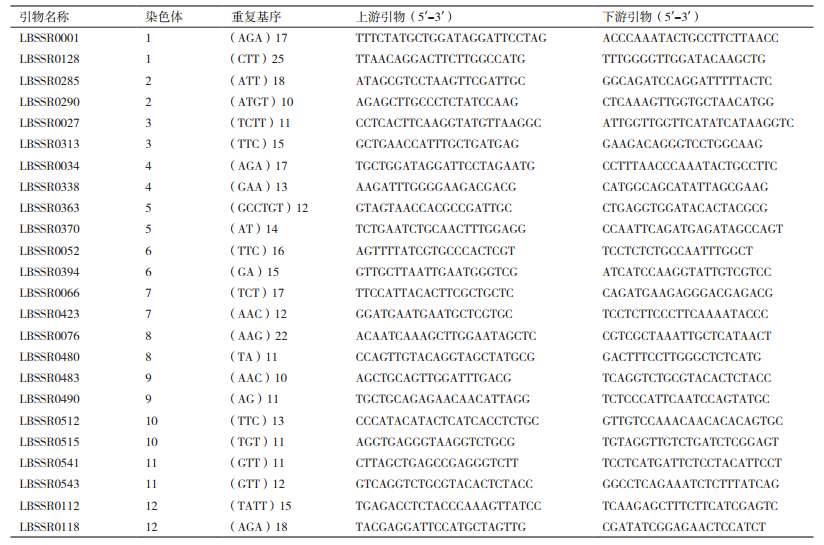
1.2.2 引物来源、设计与合成SSR引物来源于枸杞全基因组测序(数据未公布)序列所开发,利用Primer Premier5.0软件随机设计引物600对,由ABI公司合成,并分别用FAM(蓝色)和HEX(绿色)2种荧光集团修饰上游引物5'端。
1.2.3 PCR扩增及产物检测PCR扩增体系15 μL,含有50 ng/μL DNA模板1 μL、10×Buffer 1.5 μL、10 μmol/L dNTP 1.2 μL、5 μmol/L上游引物1 μL、5 μmol/L下游引物0.2 μL、5 μmol/L M13荧光引物0.8 μL、Taq DNA聚合酶(5 U/μL)0.1 μL和ddH2O 9.2 μL。PCR扩增在GeneAmp PCR System 9600(Perkin Elmer,USA)仪上进行,扩增程序为94℃ 5 min;94℃ 30 s,58℃ 30 s,72℃ 30 s,25个循环;94℃ 30 s,53℃ 30 s,72℃ 30 s,10个循环;最后60℃ 30 min。
在96孔板中每孔加入分子量内标和甲酰胺混合液(0.5:8.5)9 μL,PCR产物1.0 μL,95℃变性3 min,用ABI3730进行自动荧光检测。
1.2.4 数据统计分析利用GeneMapper4.0软件对原始数据进行分析,获得不同样品扩增片段长度。利用SSR数据格式转换软件DataFormater 2.7[18]将扩增片段转换为PowerMarker、Popgene和NTSYS输入文件。利用PowerMarker V 3.25[19]软件计算等位基因数(Number of alleles,NA)、基因型数(Number of genotype,NG)和多态信息含量(Polymorphism Information Content,PIC)。利用Popgene 32软件[20]计算Shannon’s信息指数(Shannon’s information index,I)。利用在线条形码生成器(http://barcode.cnaidc.com/html/BCGcode128.php)构建枸杞品种条形码身份证。
2 结果 2.1 引物筛选通过600对引物对遗传背景和表型性状差异较大2个品种(宁杞1号和宁杞菜1号)进行扩增检测,最终确定多态性高且分别位于12条染色体上的24对SSR引物(表 2)用于分子身份证的构建。
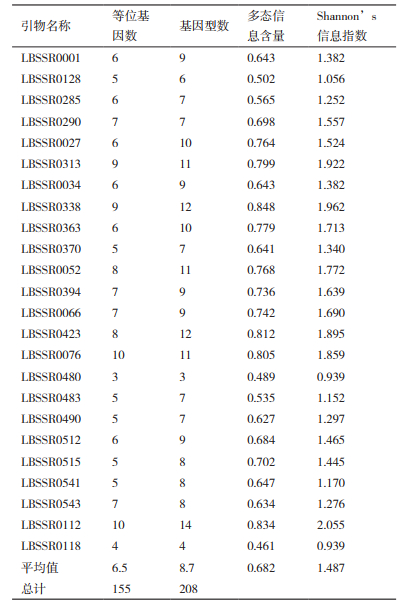
24对引物在16个枸杞品种中共检测到等位基因155个,每对引物检测到的等位基因在3(LBSSR0480)-10(LBSSR0076和LBSSR0112),平均为6.5个(表 3)。24对引物在16个枸杞品种中共检测到基因型数208个,每对引物检测到基因型数在3(LBSSR0480)-14(LBSSR0112),平均为8.7个。多态信息含量(PIC)变幅在0.461(LBSSR01-18)-0.848(LBSSR0338),平均为0.682。Shannon’s信息指数变幅在0.939(LBSSR0480)-2.055(LBSS-R0112),平均为1.487。表明所筛选出的引物具有较高的多态性。
基于最少引物鉴定最多种质的原则。从24对引物扩增结果中筛选出等位基因数、基因型数、PIC值均大于平均值,且Shannon’s信息指数>1.7引物共有6对,即LBSSR0313、LBSSR0338、LBSSR0052、LBSSR0423、LBSSR0076和LBSSR0112。引物两两组合,鉴定其对供试枸杞品种的区分率。引物组合LBSSR0052+LBSSR0423鉴定效率最高,可将16个品种全部区分开来,区分率均为100%,其次是引物组合LBSSR0052+LBSSR0112可以鉴定15个品种,区分率为93.8%(表 4)。利用LBSSR0052和LBSSR0423这2对SSR引物构建供试品种分子身份证。其中,宁杞1号在这2个SSR位点指纹图谱见图 1。

|
| 图 1 宁杞1号在2个SSR位点的SSR指纹图谱 |
将供试品种全部区分开的2对引物LBSSR0052和LBSSR0423获得的等位基因按照小到大的顺序排列,并用阿拉伯数字01开始赋值(表 5)。例如,引物LBSSR0052对16个品种扩增共获得8个等位基因,最小的为114 bp,赋值为01,依次类推,最大的为191 bp赋值08,记为A的位点赋值为09。将每个品种在2个位点获得的等位基因按照赋值数字编码获得每个品种独有的字符串(表 1)。
再利用条形码技术将每个品种的按照相同位点顺序排列的编码字符串转化成每个品种独特的条形码标识即分子身份证(图 2)。

|
| 图 2 宁杞1号身份证条形码 |
随着枸杞全基因组测序开展,构建高通量、准确性高,易操作的覆盖全基因组的枸杞种质资源DNA分子身份证构建将越来越收到重视。一种理想的DNA分子标记具有重现性好、多态性高、易操作、共显性且覆盖整个基因组等特点。DNA分子标记技术发展经历了3个发展阶段:第一阶段以杂交为基础的标记(RFLP);第二个阶段以PCR为基础的标记(RAPD、ISSR和SSR等);第三阶段以单核苷酸为基础的标记(SNP)[21]。近年来,分子标记广泛应用各种物种鉴定,国际植物品种保护联盟在BMT测试指南草案中将构建DNA指纹数据库的标记确定为SSR和SNP[22]。随着高通量测序技术发展,开发了大量的SNP,但由于SNP高额费用限制了应用。相比较,SSR标记成为了最广泛应用的标记。本研究是基于枸杞全基因组测序基础上,设计开发600对SSR引物,从中筛选24对多态性高的SSR引物,构建了16个枸杞品种的分子身份证。
3.2 SSR扩增产物检测方法的确定SSR扩增产物检测方法主要有两种:聚丙烯酰胺凝胶银染技术和毛细管电泳荧光标记技术。郝晨阳等[23]对这两项技术优缺点进行详细的比较,得出荧光标记技术具有高通量、数据收集与处理效率高、数据准确、不同实验室易整合等优点。邵千顺等[16]利用15对引物对17份枸杞种质扩增,采用采用传统的银染技术,15对引物的平均Shannon’s信息指数I为0.311,本研究24对荧光SSR引物在16个枸杞品种检测到平均Shannon’s信息指数I为1.487,显著高于前者。说明本研究采用荧光SSR检测效率高,数据更为精确可靠,为后续建立枸杞分子指纹数据库更科学合理。
3.3 分子身份证的编码方法近年来,基于SSR标记构建分子身份证的编码方法在不断地创新与发展,主要有3类[9, 11-12]:(1)筛选出可以区分所有种质的引物组合,将这些引物扩增到的带型从小到大排序,依次编码,按照固定引物顺序,串联带型编码,组成一串数据,也就是品种分子身份证;(2)筛选出可区分所有种质的核心引物,将核心引物所获得等位基因从小到大排序,依次用数字编码,组成特有字符串构成分子身份证;(3)在第2种方法基础上进行创新,身份证由商品码、指纹码和补充码3部分组成,构建身份证信息更加全面,且能真实反映品种的信息。针对不同的物种及扩增结果采用相对应的编码方法。本研究采用第2种方法,从24对引物中筛选出等位基因数、基因型数和PIC值大于平均值,且Shannon’s信息指数大于1.7,最终确定引物LBSSR0052和LBSSR0423组合,可将供试品种全部区分开。将这2对引物在所有品种扩增获得等位基因由小到大排序,用数字编码赋值,构建17个枸杞品种分子身份证,为后续构建枸杞分子指纹数据库提供合理的科学依据。
4 结论本研究是基于枸杞全基因组测序结果(未公布)基础上,从600对SSR引物中筛选出多态性高、稳定性好、均匀分布在枸杞12条染色体上的24对引物,并对16个枸杞品种进行SSR扩增分析,共检测到155个等位基因,平均为6.5个和208个基因型,平均为8.7个。平均多态信息含量(PIC)和Shannon’s信息指数分别为0.682和1.487。基于最少引物鉴定最多种质的原则,需要同时满足2个条件:(1)检测到等位基因数、基因型数和PIC值均大于平均值,(2)Shannon’s信息指数>1.7。最终确定引物LBSSR0052和LBSSR0423符合要求,而且这2对引物组合可区分全部供试品种。根据这2对引物对所有品种扩增获得等位基因按照由小到大排序,然后将每个品种在这两个SSR位点的赋值依次组合,构建16个枸杞品种的分子身份证。结果表明SSR标记技术可以作为枸杞品种鉴定和分子身份证构建的有效技术手段。
| [1] |
利河伯, 姜丽. 枸杞出口趋势分析[J]. 中国现代中药, 2014, 16(3): 244-246. |
| [2] |
曹有龙. 中国枸杞种质资源[M]. 北京: 中国林业出版社, 2015.
|
| [3] |
钟鉎元, 李健, 樊梅花, 等. 枸杞新品种"宁杞1号"的选育[J]. 宁夏农林科技, 1988, 2: 21-24. |
| [4] |
钟鉎元, 李健, 樊梅花, 等. 枸杞新品种"宁杞2号"的选育[J]. 宁夏农林科技, 1990, 4: 17-20. |
| [5] |
胡忠庆, 周全良, 谢施祎. "宁杞4号"的选育[J]. 宁夏农林科技, 2005, 4: 11-13. |
| [6] |
丛虎滋, 刘富娥, 孙天罡, 等. 枸杞新品系精杞4号特性简介及栽培要点[J]. 黑龙江农业科学, 2013, 12: 165-166. |
| [7] |
滕海涛, 吕波, 赵久然, 等. 利用DNA指纹图谱辅助植物新品种保护的可能性[J]. 生物技术通报, 2009(1): 1-6. |
| [8] |
王慧玲, 闫爱玲, 孙磊, 等. 13个中国葡萄优新品种的分子身份证构建[J]. 生物技术通报, 2016, 32(4): 137-142. |
| [9] |
张靖国, 田瑞, 陈启亮, 等. 基于SSR标记的梨栽培品种分子身份证的构建[J]. 华中农业大学学报, 2014, 33(1): 12-17. DOI:10.3969/j.issn.1000-2421.2014.01.003 |
| [10] |
聂新辉, 尤春源, 李晓方, 等. 新陆早棉花品种DNA指纹图谱的构建及遗传多样性分析[J]. 作物学报, 2014, 40(12): 2104-2117. |
| [11] |
陆徐忠, 倪金龙, 李莉, 等. 利用SSR分子指纹和商品信息构建水稻品种身份证[J]. 作物学报, 2014, 40(5): 823-829. DOI:10.3969/j.issn.1000-2561.2014.05.001 |
| [12] |
高源, 王昆, 王大江, 等. 利用TP-M13-SSR标记构建苹果栽培品种的分子身份证[J]. 园艺学报, 2016, 43(1): 25-37. |
| [13] |
党少飞, 王占林, 张得芳, 等. 枸杞基因组微卫星特征分析[J]. 西北林学院学报, 2016, 31(1): 97-102. DOI:10.3969/j.issn.1001-7461.2016.01.18 |
| [14] |
王瑛.一种鉴定枸杞品种的EST-SSR核心引物组及其鉴定方法和应用: 中国CN201610250639. 4[P]. 2016-4-20.
|
| [15] |
尹跃, 安巍, 赵建华, 等. 枸杞品种SSR荧光指纹图谱构建及遗传关系分析[J]. 西北林学院学报, 2017, 32(1): 137-141. DOI:10.3969/j.issn.1001-7461.2017.01.22 |
| [16] |
邵千顺, 高磊, 南雄雄, 等. 利用SSR技术对十七份枸杞材料进行遗传多样性分析及标准指纹图谱构建[J]. 北方园艺, 2015, 12: 91-95. |
| [17] |
Chen C, Xu M, Wang C, et al. Characterization of the Lycium barbarum fruit transcriptome and development of EST-SSR markers[J]. PLoS One, 2017, 12(11): 1-19. |
| [18] |
樊文强, 盖红梅, 孙鑫, 等. SSR数据格式转换软件DataFormater[J]. 分子植物育种, 2016, 14(1): 265-270. |
| [19] |
Liu K, Muse SV. PowerMarker:an integrated analysis environment for genetic marker analysis[J]. Bioinformatics, 2005, 21(9): 2128-2129. DOI:10.1093/bioinformatics/bti282 |
| [20] |
Yeh FC, Boyle TJB. Population genetic analysis of co-dominant and dominant markers and quantitative traits[J]. Belgian Journal of Botany, 1998, 129: 157. |
| [21] |
Moose SP, Mumm RH. Molecular plant breeding as the foundation for 21st century crop improvement[J]. Plant Physiology, 2008, 147(3): 969-977. DOI:10.1104/pp.108.118232 |
| [22] |
Union for the Protection of New varieties of Plants. Guidelines for Molecular Marker Selection and Database Construction//BMT Guidelines(Proj3.). Geneva: UPOV, 2010: 3-13.
|
| [23] |
郝晨阳, 王兰芬, 贾继增, 等. SSR荧光标记和银染技术的比较分析[J]. 作物学报, 2005, 31(2): 144-149. DOI:10.3321/j.issn:0496-3490.2005.02.002 |