近年来,人们越来越多的通过分析基因层面致病机理来进行疾病治疗[1-4]。在现代医学理论中,特定的致病基因或易感基因均会导致与之对应的疾病发生。而其中最值得注意的当属基因突变中的单碱基突变,包括单个碱基的转换、缺失、插入、颠换四种形式。因其在基因突变中所占的比例极大,所以在研究人类疾病的过程中,单碱基突变的检测至关重要。
检测单碱基错配的方法种类繁多,上世纪就有变性梯度胶电泳法、核糖核酸酶法和等位基因特异性寡聚核苷酸杂交法等各种有效的方法。近年来,随着基因检测领域的不断开拓,又在这些基础上扩展了很多新的内容,其中大多是构建新型高效的分子探针,通过探针与特定靶标配合作用,最终以光、电等信号将作用结果输出,从而进行检测。而实现对单碱基错配的方便、快速、准确的检测对疾病的治疗以及药物的开发等方面都有着极为重要的意义[5-9]。
荧光DNA生物传感器可以将所识别信息转换成可检测的荧光信号,从而实现对物质的定性或定量检测。它有光学传感器所特有的非破坏性和高灵敏性和用量少、选择性好、方法简便等优点[10]。而因氧化石墨烯具有独特的结构、稳定的物理化学性质、较好的水溶性、良好的生物相容性和低毒等性能,在分子水平检测过程中常被应用[11]。近几年研究成果显示,将有优异荧光淬灭能力的氧化石墨烯应用在荧光生物传感技术中,可以快速准确的检测蛋白质、核酸或者其他的生物小分子[12-14]。这种分析方法还有成本较低操作方便等优点,所以在分子水平检测方面有广阔的发展空间和乐观的发展前景。
基于此,为了更好的检测单碱基错配,本研究构建了一个无酶、简单、精确的分析体系。该体系利用氧化石墨烯优异的荧光淬灭能力、对单双链DNA吸附能力的差异和具有稳定的化学性质这些特点,同时结合梳型阳离子共聚物PLL-g-Dex能够促进DNA杂化及链交换反应,并且稳定DNA双链结构的特点,构建了一个快速、灵敏的生物传感体系用于检测DNA单碱基错配。
1 材料与方法 1.1 材料 1.1.1 基本材料聚赖氨酸接枝葡聚糖PLL-g-Dex共聚物和氧化石墨烯均由海南大学热带岛屿资源先进材料教育部重点实验室提供。DNA序列见表 1。
国药集团化学试剂有限公司生产的Na2HPO4·12H2O,EDTA和HCl,西陇化工股份有限公司生产的NaCl,KH2PO4,C2H5OH和NaOH。以上所提及的试剂均为二级试剂。主要仪器及设备见表 2。
在26℃的状态下,将装有干粉DNA的离心管在高转速下离心,约15 s后DNA会全部集中在离心管底,此时停止旋转。按照合成单上浓度为100 μmol/L时的剂量加入无酶水,振荡使管内DNA混匀并充分溶解。再根据紫外检测结果计算出准确浓度,精确的配制成10 μmol/L的溶液装好,在4℃条件下保存备用。
1.2.1.2 GO分散液的制备用超纯水将GO储备液配成氧化石墨稀分散液浓度为60 μg/mL的溶液。超声2 h后在10 000 r/min转速下离心15 min,取其上清液保存备用。
1.2.1.3 PBS缓冲溶液的配制使用KH2PO4、Na2HPO4·12H2O、NaCl、EDTA以及超纯水配制PBS缓冲溶液1 L。使用前的准备:取出适量溶液利用NaOH(0.1 mol/L)、HCl(0.1 mol/L)调节至pH7.2。在120℃下灭菌30 min,冷却后在4℃条件下保存。
1.2.1.4 PLL-g-Dex储备液的配制取2 mg的PLL-g-Dex(由实验室提供),2 mL的超纯水,配制成浓度为1 mg/mL的储备液,在4℃条件下放置以待使用。
1.2.2 荧光检测 1.2.2.1 检测T-DNA(已被TAMRA荧光标记)单独在PBS缓冲液中的荧光值在室温条件下,检测由6 μL的T-DNA(10 μmol/L)和PBS(pH 7.2)缓冲液组成的体系发射出的荧光信号,待信号稳定。最终体系总体积3 mL,T-DNA终浓度20 nmol/L。
1.2.2.2 GO最佳浓度的选择将6 μL的T-DNA(10 μmol/L)分别与100、150、200、250、300、350、400和450 μL的GO溶液(60 μg/mL)混合,待30 min后检测荧光。对比检测结果得到最优的GO猝灭浓度。最终体系总体积3 mL,T-DNA浓度20 nmol/L,GO浓度分别为2、3、4、5、6、7、8、9和10 μg/mL。
1.2.2.3 cDNA最佳浓度的选择分别加6、9、12、15、18和21 μL的cDNA(10 μmol/L)到含有6 μL的T-DNA(10 μmol/L)和450 μL的浓度为GO(60 μg/mL)的PBS(pH 7.2)缓冲液中,均匀混合2 h后,检测体系中显示的荧光值,扫描平衡时间。体系总体积3 mL,T-DNA浓度20 nmol/L,GO浓度9 μg/mL。cDNA的浓度分别为20、30、40、50、60、70、80和90 nmol/L。
1.2.2.4 PLL-g-Dex最佳浓度的选择将完全匹配的20 nmol/L的T-DNA,9 μg/mL的GO,90 nmol/L的cDNA混合后加入到PBS(pH7.2)缓冲液中构成的T-DNA-GO-cDNA体系,再分别加入不同浓度(24、48、72、96、120和144 nmol/L)的PLL-g-Dex,检测体系中显示的荧光值,扫描平衡时间,最终体系总体积3 mL。
1.2.2.5 T-DNA-GO+PLL-g-Dex体系对单碱基错配的选择性将完全匹配的20 nmol/L的T-DNA,9 μg/mL的GO,90 nmol/L的cDNA,96 nmol/L的PLL-g-Dex混合后加入到PBS(pH7.2)缓冲液中构成T-DNA-GO+PLL-g-Dex体系,再向其中分别加入与之互补的cDNA(90 nmol/L),M1、M2、M3检测荧光恢复情况。其中每种错配碱基DNA的终浓度都是90 nmol/L,M1、M2、M3应提前在室温下培育30 min。
若无特殊说明,实验中均采用荧光分光光度计检测荧光,在PBS缓冲液(pH7.2)中进行,其中荧光比色皿装样为3 mL,发射光谱波长范围575-700 nm,激发光谱波长560 nm,曲线所用荧光强度在582 nm的波长。实验数据整合使用origin8.0软件。
2 结果 2.1 方法设计的可行性验证先使T-DNA(20 nmol/L)与不同试剂进行组合,再检测各个体系所发射出的荧光信号,实验结果如图 1所示。图中曲线a表示当在PBS缓冲液中仅加入带有TAMRA标记的T-DNA(20 nmol/L)时,体系发出的荧光值为363,即显示出较强的荧光信号。图中曲线b表示在a组的基础上再加入GO(9 μg/mL)时,体系中绝大部分的荧光被淬灭,被淬灭的荧光占比高达91.6%。图中曲线c表示在b组的基础上再向体系加入cDNA(90 nmol/L)时(所加入的cDNA与T-DNA完全匹配),出现了荧光恢复,数据表明荧光信号约恢复了60%。图中曲线d表示在c组的基础上再向体系加入PLL-g-Dex(96 nmol/L)时,荧光强度恢复率激增,甚至达到a组的荧光强度。因此,我们可以得知实验设计思路是切实可行的。

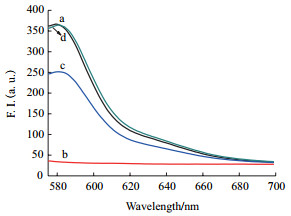
|
| 图 1 不同条件下被TAMRA标记的T-DNA(20 nmol/L)所显示的荧光光谱 a:T-DNA在PBS缓冲液中;b:T-DNA+GO(9 μg/mL);c:T-DNA+GO(9 μg/mL)+cDNA(90 nmol/L);d:T-DNA+GO(9 μg/mL)+cDNA(90 nmol/L)+PLL-g-Dex(96 nmol/L) |
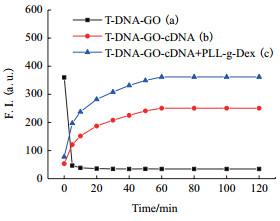
分别检测3组不同条件下被TAMRA标记的T-DNA(20 nmol/L)显示出的荧光光度值和反应时间的关系,结果如图 2所示。
|
|
图 2 T-DNA(20 nmol/L)在不同条件下的荧光光度值和反应时间的关系
(采集荧光信号的时间分别为5、10、20、30、40、50、60、80、100、120 min) (a)单独在GO(9 μg/mL)中;(b)在GO(9 μg/mL)+cDNA(90 nmol/L)中;(c)在GO(9 μg/mL)+cDNA(90 nmol/L)+PLL-g-Dex(96 nmol/L)中 |
由图 2可知,若在T-DNA(20 nmol/L)溶液中加入与之相适应的GO(9 μg/mL)时,可观察到体系中绝大部分荧光在5 min之内被淬灭,之后荧光值以极缓慢的速度继续下降,30 min内荧光值趋于平稳。随后若添加与T-DNA(20 nmol/L)完全匹配的cDNA(90 nmol/L)时,观察到T-DNA-GO-cDNA体系的荧光信号恢复,60 min后荧光信号达到稳定状态。若荧光被淬灭后在T-DNA(20 nmol/L)溶液中同时加入与其相匹配的cDNA(90 nmol/L)和PLL-g-Dex(96 nmol/L)后,可观察到新体系中荧光值恢复更加明显,荧光信号同样是在60 min后达到稳定状态。对比数据可知,此时荧光强度已经恢复到了在PBS缓冲液中仅添加T-DNA时所显示的荧光强度。
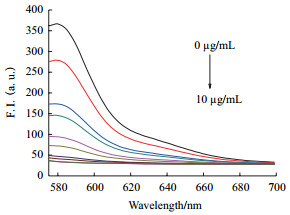
2.3 实验条件的选择 2.3.1 氧化石墨烯最佳浓度的选择随着不同浓度(0、2、3、4、5、6、7、8、9、10 μg/mL)GO的加入,TAMRA标记的T-DNA(20 nmol/L)的荧光强度变化如图 3所示。
|
| 图 3 TAMRA标记的T-DNA(20 nmol/L)在GO不同浓度下(0、2、3、4、5、6、7、8、9和10 μg/mL)的荧光光谱 |
由图 3可知,GO的加入会对被TAMRA标记的T-DNA(20 nmol/L)所显示出的荧光造成影响,随着加入的GO浓度的持续升高,荧光值会不断降低。当所加入的GO浓度为10 μg/mL时,GO过量并且影响了体系检测的灵敏性。但当GO浓度维持在较低水平时,不能有效的淬灭体系显示出的荧光。综合对比认为采用浓度为9 μg/mL的GO为最好,此时体系的荧光被淬灭91.6%。
2.3.2 cDNA最佳浓度的选择在T-DNA(20 nmol/L)和GO(9 μg/mL)的混合液中,分别添加不同浓度的cDNA(20、30、40、50、60、70、80和90 nmol/L),检测T-DNA-GO传感器体系的荧光恢复情况,结果如图 4所示。

|
| 图 4 加入不同cDNA浓度(20、30、40、50、60、70、80和90 nmol/L)的T-DNA-GO的荧光光谱 |
分析图 4可知,cDNA浓度的不同会导致荧光恢复情况有所差异。所加入的cDNA的浓度越高,T-DNA-GO传感器体系的荧光恢复越明显。当cDNA的浓度到达90 nmol/L时,检测到的荧光值最大,达到251.05。此时荧光强度恢复69.2%。
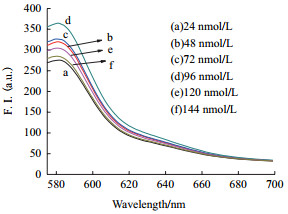
2.3.3 PLL-g-Dex最佳浓度的选择为确定PLL-g-Dex在实验中的最佳浓度,检测在T-DNA(20 nmol/L),GO(9 μg/mL)和cDNA(90 nmol/L)所构成的T-DNA-GO-cDNA体系中分别加入不同浓度(24、48、72、96、120和144 nmol/L)的PLL-g-Dex时体系所显示出的荧光值。结果如图 5所示,当加入96 nmol/L的PLL-g-Dex时,荧光恢复度最高,能达到在PBS缓冲液中仅添加T-DNA时所显示的荧光强度。而其他的浓度效果均不理想,浓度为24 nmol/L时荧光恢复度最低,仅恢复到了275.5。由此,我们选择在使用浓度为96 nmol/L的PLL-g-Dex。
|
| 图 5 T-DNA体系在不同PLL-g-Dex浓度下的荧光光谱 |
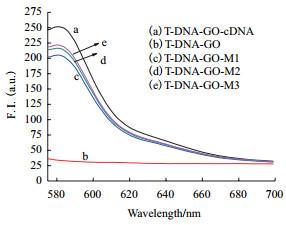
检测T-DNA-GO空白组和在T-DNA-GO中分别加入与之互补的cDNA(90 nmol/L),M1、M2、M3时的荧光恢复情况,结果如图 6所示。当加入cDNA时荧光信号恢复程度最大,达到60.0%。而加入相同浓度的M1、M2、M3时,荧光强度分别恢复到47.2%、50.3%和51.6%。
|
| 图 6 T-DNA-GO(20 nmol/L)体系在不同条件下的荧光光谱 其中GO(9 μg/mL),cDNA,M1,M2,M3(90 nmol/L),PLL-g-Dex(96 nmol/L) |
具体情况如图 7所示,将同等浓度不同类型的碱基突变的目标链加入到T-DNA-GO体系中,发现荧光值的提高程度有所不同,可据此识别双链DNA中的不同类型的碱基错配。明显看出,加入cDNA的体系荧光增强强度最大,之后按顺序依次为M3、M2、M1,即C-G > C-C > C-A > C-T。若再把PLL-g-Dex加进T-DNA-GO体系中(已经含有不同类型的碱基突变的目标链时),体系荧光又会出现变化,如图 7-D所示,增强的信号程度由大到小依次为cDNA、M2、M1、M3,即C-G > C-A > C-T > C-C。其中加入cDNA、M1、M2的体系荧光强度增加明显,分别为31.1%、21.2%和24.6%。而含M3的体系只在原本的基础上多恢复了3.6%,相比于其他体系恢复极小。

|
| 图 7 T-DNA-GO+PLL-g-Dex体系对单碱基错配的选择性情况示意图 A:为T-DNA-GO体系+M1;B:T-DNA-GO体系+M2;C:T-DNA-GO体系+M3;D:加入PLL-g-Dex后各体系中荧光的增加情况的对比图 |
配合氧化石墨烯的生物传感器体系具有高灵敏、低成本、检测快速准确等优势,引起了人们越来越多的研究和关注。近年来,采用氧化石墨烯进行荧光共振能量转移分析以达到实验检测目的的方法种类很多,在检测DNA、离子、生物分子、病原体或细胞方面均具有广阔的应用潜能[15-16]。本研究把PLL-g-Dex加入到基于GO特性的荧光分析体系中,并对含有不同错配碱基的几种体系呈现出来的荧光进行分析,进而确定了我们构建的检测体系对T-DNA序列中出现的C-C碱基错配有较强的选择性。
首先,在验证实验可行性的过程中,当GO加入到带有荧光标记的T-DNA中时,我们观察到荧光被淬灭,这种现象的产生普遍认为是由GO的结构特征所致。在这个过程中,T-DNA以π-π非共价键形式吸附在GO表面,GO吸收荧光标记后又通过荧光共振来传递能量以达到消除荧光的效果[17-21]。之后cDNA的加入使两条单链根据碱基互补配对原则相互之间发生杂交,形成了双链DNA。不同于对单链DNA的高度吸附作用,GO和在核酸骨架中的双链DNA的相互作用明显很弱,即便双链DNA上的磷酸基团和GO上的含氧活性基团可以反应,也只是氢键的结合,并不能稳定存在于常温水溶液中,这种较弱的相互作用不足以分开双链,至此双螺旋结构得以保持,荧光恢复[22-24]。之后我们选择加入PLL-g-Dex,PLL-g-Dex是一种能够稳定DNA双螺旋结构并且可以刺激两条互补的单链DNA之间杂交的功能高分子,因此荧光恢复率明显提高[25-26]。也正是由于PLL-g-Dex和GO特殊的性能使检测体系的稳定性得到提高,再配合低的背景信号,才使所构建的整个检测体系的灵敏度较高。
确定好实验可行以后,我们检测了体系的反应时间,由于GO优异的荧光淬灭能力,荧光值降低很快。随后加入cDNA使荧光恢复的过程包含T-DNA从GO上的脱落和生成双链DNA两个内容,所以恢复荧光耗时比淬灭荧光耗时长。
另外,当在T-DNA-GO中分别加入与之互补的cDNA、M1、M2和M3时,我们检测其荧光值并和T-DNA-GO空白组的荧光值做对照。不同荧光值的出现证明了我们可以用T-DNA-GO体系来检测存在于双链DNA中的单碱基错配。
我们又将PLL-g-Dex加入到体系中,发现对于含完全匹配的cDNA的体系,杂交结果最好,荧光恢复增幅最大,说明PLL-g-Dex对其稳定性最好。对于含有C-A、C-T错配的体系,因为错配影响本身就有部分位置不能恢复荧光,最终的恢复幅度较小,说明PLL-g-Dex对两者也是有稳定性的。但含有C-C碱基错配的体系荧光恢复增幅和其他有明显的不同,并无显著增加,几乎不能说明PLL-g-Dex对这个体系稳定能力的提升有帮助。由此,我们确定了PLL-g-Dex对含有C-C碱基错配的体系有选择性。
总之,本实验以PLL-g-Dex为辅助构建的GO-DNA的荧光生物传感体系可以快速的检测出T-DNA中的C-C碱基错配。这将为关于梳型阳离子PLL-g-Dex或其他物质加入到GO-DNA的荧光生物传感器中所产生的选择能力的进一步研究奠定基础。
4 结论本研究构建了一种以梳型阳离子共聚物PLL-g-Dex辅助的GO-DNA的荧光生物传感体系。研究表明,在T-DNA浓度为20 nmol/L的情况下,最佳的实验点是GO浓度为9 μg/mL,cDNA浓度为90 nmol/L,PLL-g-Dex浓度为96 nmol/L。当体系中含不同类型单碱基错配的寡核苷酸时,含有C-C碱基错配寡核苷酸的体系所呈现出的荧光变化效果与其他几种有明显差异,由此来实现对含有C-C碱基错配链的特异性检测。
| [1] |
Liu JW, Hong T, et al. Recent advance on genome editing for therapy of β-hemoglobinopathies[J]. Hereditas, 2018, 40(2): 95-103. |
| [2] |
Beek NV, Klionsky DJ, Reggiori F. Genetic aberrations in macroautophagy genes leading to diseases[J]. Biochim Biophys Acta, 2018, 1865(5): 803-816. DOI:10.1016/j.bbamcr.2018.03.002 |
| [3] |
Toma C, Shaw AD, Allcock R, et al. An examination of multiple classes of rare variants in extended families with bipolar disorder[J]. Translational Psychiatry, 2018, 8: 65. DOI:10.1038/s41398-018-0113-y |
| [4] |
Singh M, Tyagi SC. Genes and genetics in eye diseases:a genomic medicine approach for investigating hereditary and inflammatory ocular disorders[J]. Int J Ophthalmol, 2018, 11(1): 117-134. |
| [5] |
Lee MH, Lin HY, et al. Detection of DNA Sequences with a Single-Base Mismatch on a Gold-Based and Pyrene-Assisted Platform[J]. Sens Actuators B Chem, 2018, 266: 522-527. DOI:10.1016/j.snb.2018.03.096 |
| [6] |
Lee J, Park G, Min DH. A biosensor for the detection of single base mismatches in microRNA[J]. Chem Commun, 2015, 51(78): 14597-14600. DOI:10.1039/C5CC04706D |
| [7] |
Joda H, Beni V, Katakis I, et al. DNA biosensor based on hybridization refractory mutation system approach for single mismatch detection[J]. Anal Biochem, 2015, 474: 66-68. DOI:10.1016/j.ab.2014.11.021 |
| [8] |
Moradi N, Noori A, Mehrgardi MA, et al. Scanning electrochemical microscopy for electrochemical detection of single-base mismatches by tagging ferrocenecarboxylic acid as a redox probe to DNA[J]. Electroanalysis, 2016, 28(4): 823-832. DOI:10.1002/elan.v28.4 |
| [9] |
Zhu J, Ding Y, Liu X, et al. Toehold-mediated strand displacement reaction triggered isothermal DNA amplification for highly sensitive and selective fluorescent detection of single-base mutation[J]. Biosensors & Bioelectronics, 2014, 59: 276-281. |
| [10] |
Borisov SM, Wolfbeis OS. Optical Biosensors[J]. Chem Rev, 2008, 108(2): 423-461. DOI:10.1021/cr068105t |
| [11] |
Zheng P, Wu N. Fluorescence and sensing applications of graphene oxide and graphene quantum dots:A review[J]. Chem Asian J, 2017, 12(18): 2343-2353. DOI:10.1002/asia.v12.18 |
| [12] |
Huang Y, Wang X, Duan N, et al. Selection and characterization, application of a DNA aptamer targeted to Streptococcus pyogenes, in cooked chicken[J]. Anal Biochem, 2018, 551: 37-42. DOI:10.1016/j.ab.2018.04.015 |
| [13] |
Wang M, Lin Z, et al. DNA-hosted copper nanoclusters/graphene oxide based fluorescent biosensor for protein kinase activity detection[J]. Anal Chim Acta, 2018, 1012: 66-73. DOI:10.1016/j.aca.2018.01.029 |
| [14] |
Wen Y, Xing F, He S, et al. A graphene-based fluorescent nanoprobe for silver (Ⅰ) ions detection by using graphene oxide and a silver-specific oligonucleotide[J]. Chem Commun, 2010, 46(15): 2596-2598. DOI:10.1039/b924832c |
| [15] |
Zhang H, Zhang H, Aldalbahi A, et al. Fluorescent biosensors enabled by graphene and graphene oxide[J]. Biosensors & Bioelectronics, 2017, 89: 96-106. |
| [16] |
Fan K, Guo Z, Geng Z, et al. How graphene oxide quenches fluorescence of rhodamine 6G[J]. Chinese Journal of Chemical Physics, 2013, 26(3): 252-258. DOI:10.1063/1674-0068/26/03/252-258 |
| [17] |
Wang X, Amat M, Wang S, et al. Fluorescence quenching in N-doped graphene derived from graphitic nitrogen[J]. Rsc Advances, 2015, 5(36): 28247-28250. DOI:10.1039/C5RA03721B |
| [18] |
Li R, Liu X, Deng X, et al. Fluorescence determination based on graphene oxide[J]. Materials Letters, 2012, 76(76): 247-249. |
| [19] |
Tan J, Lai Z, Zhang Z, et al. A graphene oxide-based fluorescent aptasensor for the turn-on detection of CCRF-CEM[J]. Nanoscale Research Letters, 2018, 13(1): 66. DOI:10.1186/s11671-017-2403-3 |
| [20] |
Seraj S, Rouhani S. A fluorescence quenching study of naphthalimide dye by graphene:mechanism and thermodynamic properties[J]. Journal of Fluorescence, 2017, 27(5): 1-7. |
| [21] |
Yoo HJ, Baek C, Lee D, et al. Short-Length DNA Adsorption on Graphene Oxide-Coated Microbeads for DNA Target Separation from Clinical Samples[J]. J Nanosci Nanotechnol, 2018, 18(9): 6364-6368. DOI:10.1166/jnn.2018.15656 |
| [22] |
Lee J, Min D H. A simple fluorometric assay for DNA exonuclease activity based on graphene oxide[J]. Analyst, 2012, 137(9): 2024-2026. DOI:10.1039/c2an16214h |
| [23] |
Zhu Q, Xiang D, Zhang C, et al. Multicolour probes for sequence-specific DNA detection based on graphene oxide[J]. Analyst, 2013, 138(18): 5194-5196. DOI:10.1039/c3an00960b |
| [24] |
Yamayoshi A, Miyoshi D, Zouzumi Y, et al. Selective and Robust Stabilization of Triplex DNA Structures Using Cationic Comb-type Copolymers[J]. J Phys Chem B, 2017, 121(16): 4015-4022. DOI:10.1021/acs.jpcb.7b01926 |
| [25] |
Hirano M, Shimada N, Kano A, et al. Analysis of cationic comb-type copolymers/DNA interaction by the single molecular observation and intermolecular force measurement[J]. Nucleic Acids Symposium, 2008, 52(52): 715-716. |






