2. 农业部转基因安全评价重点实验室 中国农业科学院生物技术研究所,北京 100081
2. MOA Key Laboratory on Safety Assessment(Molecular)of Agri-GMO, Chinese Academy of Agricultural Sciences, Beijing 100081
启动子是指DNA分子上被RNA聚合酶、转录调节因子等识别并结合形成转录起始复合物的区域。启动子在基因转录起始和调控中起着非常重要的作用[1]。启动子可以分为组成型、特异型和诱导型。组成型启动子广泛用于基因工程,如花椰菜(Brassica oleracea var. botrytis)花叶病毒(Ca MV)35S启动子[2]、根癌农杆菌Ti质粒T-DNA区域的胭脂碱合成酶基因Nos启动子[3]、章鱼碱合成酶基因Ocs启动子[4]、水稻Actin1基因的Act1启动子[5]和玉米Ubiquitin基因的Ubi启动子[6]等。其中应用最多的35S启动子和Ubi启动子几乎在所有植物组织的全部发育阶段指导靶基因的表达[7]。
然而,外源基因的组成型表达并不总是适用于转基因的研究和应用。用于研究基因功能的组成型表达将掩盖该基因的某些精细功能,特别是与信号转导,能量转化和物质运输相关的功能。外源基因的组成型表达可能在转基因植物中引起额外的代谢负担或能量损耗[8]。此外,在遗传转化中,重复使用相同的启动子可能引起转基因沉默[9-11]。组织特异型启动子又称为器官特异型启动子,它指导基因在植物的特定组织或器官中表达,并表现出发育调节的特性,可以避免植物营养的不必要浪费。组织特异型启动子的特性使其在基因工程中成为一种重要的顺式作用元件,在生物反应器、作物品种改良、抗病、抗虫、抗逆等作物分子育种中广泛应用。
根系在植物生长、发育和适应中起重要作用,同时也负责水分和营养的摄取,并且对整个植物有固定和稳定的作用[12]。普通野生稻是栽培稻的近缘祖先种,宿根繁殖,根系发达,在抗旱[13]等方面进行了广泛研究,但还没有根特异表达基因和启动子的报道。本研究从普通野生稻基因组中克隆了根特异表达启动子并在拟南芥中验证功能,为作物分子育种提供新的调控元件。
1 材料与方法 1.1 材料 1.1.1 植物材料普通野生稻和拟南芥(Columbia-0)均由本实验室保存。
1.1.2 载体与菌株pBinGlyRed3载体、pBinGly-Red3-CaMV 35S-GUS载体(由本实验室保存),pEASY -T1 Simple克隆载体,农杆菌EHA105,大肠杆菌感受态Trans-T1(全式金生物技术有限公司,北京)。
1.2 方法 1.2.1 OrRSG基因表达模式的验证在实验室前期构建的普通野生稻转录组文库中筛选到1个FPKM(Fragments Per Kilobase of transcript per Million mapped reads)值在根中较高,而在地上部分几乎为0的基因,命名为OrRSG(Root Specific Gene)[14]。分别提取普通野生稻不同发育时期地上和地下部分的总RNA,反转录成cDNA。以转录组文库中的OrRSG基因的CDS序列为模板,设计引物(FP:5'-AGAGCGGAAGCGGAGTCAC-3',RP:5'-AACCACCGGACCCATCAAC-3')。利用半定量PCR(RT-PCR)验证OrRSG基因在不同发育阶段的表达模式,选用OsActin(No.AB047313)作为内参基因(FP:TTGTGTTGGACTCTGGTGATG,RP:AAGCTCGTAGCTCTTCTCCAC)。
1.2.2 OrRSG基因启动子克隆及序列分析将OrRSG基因的CDS序列在NCBI数据库进行比对,可以比对到粳稻10号染色体的LOC_Os10g31540基因上,分析普通野生稻基因组中该其因上游序列,克隆LOC_Os10g31540基因ATG上游896 bp序列为OrRSG基因的启动子,扩增引物为(FP:5'-TGGC-AAAGCAAGGATTGCTTA-3';RP:5'-CATGTATTTCAAATCAGAGTGATTATCC-3')。反应程序如下:①预变性:95℃,5 min;②变性:95℃,30 s;③退火:58.5℃,30 s;④延伸:72℃,1min30s;⑤总延伸:72℃,10 min;其中步骤②至④重复36个循环,置于4℃保存。将克隆得到的启动子序列提交到PlantCare(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)网站进行关键的元件预测。
1.2.3 OrRSGp-GUS表达载体的构建OrRSGp启动子克隆引物:FP:5'-ACGCGTAAGGGGATCCTGGCAAAGCAAGGATTGCTTA-3';RP:5'-GATCTACCATGAATTCCATGTATTTCAAATCAGAGTGATTATCC-3'。将克隆自野生稻的OrRSGp序列替换pBinGlyRed3-CaMV 35S-GUS表达载体中的CaMV 35S启动子序列,得到pBinGlyRed3-OrRSGp-GUS表达载体。
1.2.4 拟南芥遗传转化及分子鉴定将pBinGlyR-ed3-CaMV 35S-GUS和pBinGlyRed3-OrRSGp-GUS表达载体转化到农杆菌菌株EHA105中,以含有35S启动子的pBinGlyRed3-CaMV 35S-GUS表达载体作为对照。利用蘸花法转化拟南芥。pBinGlyRed3-CaMV 35S-GUS和pBinGlyRed3-OrRSGp-GUS表达载体的标记基因为DsRed红色荧光报告基因,收获转基因拟南芥的T0代种子,在绿光灯下,用红色3D立体眼镜挑选,挑选显出红光的转基因T0代种子,干燥后种植。当T3代幼苗长至一个月左右,取所有株系叶片提取基因组DNA,在启动子区域和GUS基因编码区设计引物进行PCR验证。其中转pBinGlyRed3-CaMV 35S-GUS表达载体T3代幼苗PCR鉴定引物为(FP:GTAAGGGATGACGCACAATCC,RP:GGTCGTGTAGATTTTCACCGG),转pBinGlyRed3-OrRSGp-GUS表达载体T3代幼苗PCR鉴定引物为(FP:5'- TGGCAAAGCAAGGATTGCTTA-3',RP:5'- CATGTATTTCAAATCAGAGTGATTATCC -3')。
1.2.5 转基因拟南芥的GUS定性分析转基因拟南芥T3代种子点植在1/2MS培养基上,4℃放置2 d后光照培养。发芽后,取1 d、3 d、7 d、15 d和28 d的拟南芥幼苗进行全株GUS染色分析[15];另一部分阳性转基因拟南芥T3代种子在培养基质(蛭石:珍珠岩:营养土体积比为1: 1: 2)中培养,等到植株进入生殖生长后,取其成熟期根、茎、叶、花、荚果进行GUS染色分析。
1.2.6 转基因拟南芥的GUS定量分析选择4个株系的T3代转基因阳性拟南芥种子,在1/2 MS培养基中培养。待拟南芥生长30 d左右分别取其地上部分和地下部分进行GUS定量检测,GUS定量检测方法见Banerjee等[16]。
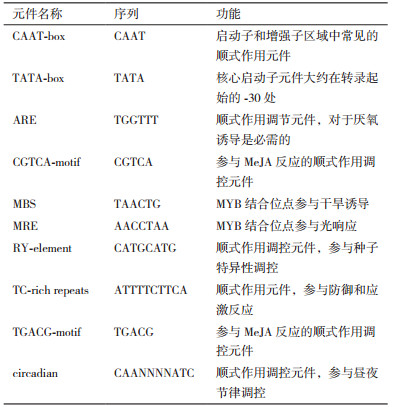
2 结果 2.1 OrRSG基因表达模式与序列分析利用RT-PCR验证OrRSG基因在普通野生稻不同发育时期绿色组织和根部的表达,结果(图 1)表明该基因在幼苗的根中表达,叶中不表达,在成熟期的根中表达,在地上部分不表达。
|
| 图 1 RT-PCR验证OrRSG基因表达模式 1:普通野生稻苗期地上部分;2:普通野生稻苗期根;3:普通野生稻成熟期地上部分;4:普通野生稻成熟期根 |
普通野生稻OrRSG基因序列与栽培稻基因LOC_Os10g31540的相似度为98%(571/582)。该基因位于10号染色体上,基因全长899 bp,CDS长度为582 bp,编码193个氨基酸。该基因在普通野生稻驯化过程中较为保守。
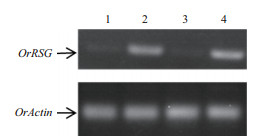
2.2 OrRSG基因启动子克隆及序列分析以LOC_Os10g31540基因上游896 bp片段为启动子区域,设计引物,以普通野生稻全基因组DNA为模板,克隆该片段。凝胶电泳结果(图 2)显示目的条带符合预期大小。
|
| 图 2 OrRSGp克隆片段的凝胶电泳结果 1:Trans 2K DNA Marker;2-3:OrRSGp克隆片段 |
测序结果(表 1)显示,OrRSGp启动子与数据库中栽培稻序列相似度为99%(889/896)。对该启动子序列进行分析,发现其中有CAAT-box、TATA-box等主要功能元件,还有参与茉莉酸甲酯反应的响应元件,以及抗逆等响应元件。转录起始位点位于OrRSG上游44 bp处,为A碱基(图 3)。

|
| 图 3 启动子预测元件及转录起始位点 |
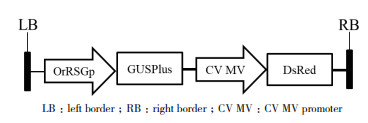
将2.2中克隆得到的OrGSGp启动子片段与pBinGlyRed3-CaMV 35S-GUS表达载体上的CaMV 35S启动子替换,构建根特异表达启动子融合GUS报告基因的载体pBinGlyRed3-OrRSGp-GUS(图 4)。
|
| 图 4 pBinGlyRed3-OrRSGp-GUS表达载体图谱 |
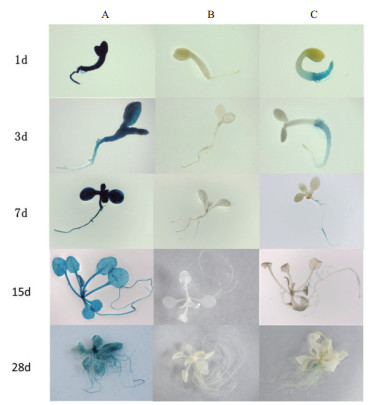
为验证OrRSGp启动子的表达模式,将转OrRSGp-GUS拟南芥幼苗期的全株以及成熟期根、茎、叶、花、荚果进行GUS染色,同时以转CaMV 35S-GUS的阳性株系为阳性对照,以野生型拟南芥为阴性对照。转OrRSGp-GUS拟南芥以及对照拟南芥苗期1 d、3 d、7 d、15 d、28 d的染色结果(图 5)显示,转OrRSGp-GUS拟南芥只在有根部显蓝色,转CaMV 35S-GUS拟南芥全株均蓝色,而野生型拟南芥中没有着色。
|
| 图 5 转OrRSGp-GUS拟南芥幼苗期GUS染色 A:转CaMV 35S-GUS拟南芥苗期GUS染色;B:野生型拟南芥苗期GUS染色;C:转OrRSGp-GUS拟南芥苗期GUS染色 |
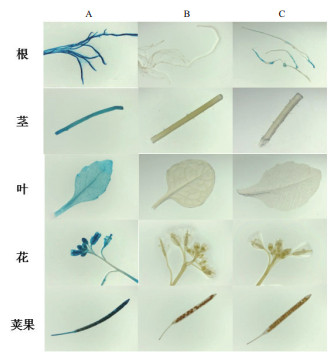
转OrRSGp-GUS拟南芥以及对照在成熟期的根、茎、叶、花以及荚果中的染色结果(图 6)显示,转OrRSGp-GUS拟南芥只在成熟根中显示蓝色,其它部位没有显示蓝色;转CaMV 35S-GUS拟南芥各组织器官中均呈现深蓝色,而野生型各组织器官未见到蓝色。结果表明,OrRSGp启动子在拟南芥中的表达模式为根特异,且在不同时期表达模式一致。
|
| 图 6 转OrRSGp-GUS拟南芥成熟期各组织GUS染色 A:转CaMV 35S-GUS表达载体拟南芥成熟期各组织GUS染色结果;B:野生型拟南芥成熟期各组织GUS染色结果;C:转OrRSGp-GUS表达载体拟南芥成熟期各组织GUS染色结果 |
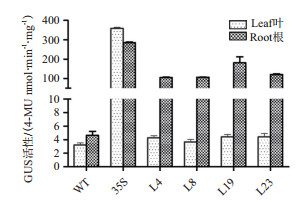
随机挑选4个转GUS基因拟南芥T3代株系,在1/2 MS培养基上培养30 d,对地上和地下部分进行GUS定量检测,以转CaMV 35S-GUS拟南芥,野生型拟南芥作为对照。
GUS定量结果(图 7)显示,GUS蛋白在L4、L8、L19、L23四个转基因株系的叶中含量很低,在根中含量很高,说明OrRSGp启动GUS报告基因在根中特异表达,叶中不表达或表达量很低;野生型拟南芥中GUS蛋白含量在根和叶中都很低;转CaMV 35S-GUS拟南芥在根与叶中均含量很高。
|
| 图 7 转OrRSGp-GUS表达载体拟南芥GUS定量结果 |
根系在植物生长中起着重要的作用,发达的根系有助于吸收深层土壤中的水分,增加作物的抗旱性[17]。利用根特异启动子启动目的基因在根部特异表达,能够促进植物对水分与养分的吸收并且能够提高植物对环境胁迫的耐受力。Jeong等[18-19]在水稻中克隆了Rcc3启动子,发现其在水稻根系中特异表达,将抗旱基因OsNAC10分别连接到组成型启动子GOS2和根特异启动子Rcc3转化水稻,发现在干旱条件下,根特异表达OsNAC10的转基因水稻比组成型表达的产量显著增高。Yu等[20]在水稻中克隆了OsEXPA17启动子,发现其在水稻和拟南芥的根中都特异表达。Chen等[21-22]在大豆中克隆了GmPRP2启动子和GmTIP启动子,分别启动gus基因转化大豆,发现这两个启动子为根特异启动子;Rausch等[23]在马铃薯中克隆了StPT3启动子,启动gus基因转化马铃薯,发现其在转基因马铃薯的根部特异表达。
普通野生稻具有发达的根系,而且具有宿根繁殖特性,已在抗旱方面进行了较为广泛的研究。胡标林等[24]对226份东乡野生稻进行了抗旱性试验,发现东乡野生稻在干旱胁迫下具有比栽培稻发达的根系和高的成活率,即具有更发达水分输送系统。Zhang等[25]构建了栽培稻与普通野生稻的杂交群体,进行QTL分析,定位了12个抗旱相关位点,确定了含有两个来自野生稻片段qSDT2-1和qSDT12-2的导入系IL23具有强大的抗旱性。Zhang等[26]通过模拟干旱,构建了4个小RNA文库进行测序分析,共筛选出231个干旱后差异表达的miRNA。Tian等[14]通过对普通野生稻干旱处理的转录组数据分析,筛选出37个根中特异表达与干旱胁迫相关的基因。
但普通野生稻启动子相关报道还很少,本实验室前期克隆了绿色组织特异表的启动子OrGSP,验证了其在拟南芥绿色组织中表达[27]。目前还未见根特异启动子的报道。本研究从普通野生稻转录组文库中筛选得到了可能的根特异表达基因,经比对和栽培稻中LOC_Os10g31540基因同源,克隆了该基因上游的启动子序列,长度896 bp,包含有CAAT-box、TATA-box等主要功能元件,CGTCA-motif、TGACG-motif等参与茉莉酸甲酯反应的响应元件,以及抗旱、抗逆响应元件MBS,TC-rich repeats等。还有许多其他的调控元件,如circadian参与昼夜节律调控,RY-element参与种子特异性调控等。OrRSGp启动子驱动报告基因主要在根部特异性表达,从GUS染色结果可以看出,启动子强度弱于CaMV 35S组成型启动子,GUS定量结果与该染色结果相符。本研究克隆得到的启动子长度为896 bp,也有可能是由于缺少必要的增强子片段或者抑制子的存在而导致的。下一步将对其进行延长以及缺失分析,以便进一步确定该启动子的关键作用元件。
4 结论本研究克隆了普通野生稻启动子OrRSGp,在拟南芥中验证了其表达模式,证明该启动子为根特异型启动子,为作物分子育种提供了新的调控元件。
| [1] |
Li Y, Sun Y, Yang Q, et al. Cloning and function analysis of an alfalfa(Medicago sativa L.)zinc finger protein promoter MsZPP[J]. Mol Biol Rep, 2012, 39: 855-8569. |
| [2] |
Odell JT, Nagy F, Chua NH, et al. Identification of DNA sequences required for activity of the cauliflower mosaic virus 35S promoter[J]. Nature, 1985, 313: 810-812. DOI:10.1038/313810a0 |
| [3] |
Leisner SM, Gelvin SB. Structure of the octopine synthase upstream activator sequence[J]. Proc Natl Acad Sci USA, 1988, 85: 2552-2557. |
| [4] |
Mol JNM, Antoine RS, Alexander K. Genetic manipulation of floral pigmentation genes[J]. Plant Mol Biol, 1989, 13: 287-294. DOI:10.1007/BF00025316 |
| [5] |
McElroy D, Zhang W, et al. Isolation of an efficient actin promoter for use in rice transformation[J]. Plant Cell, 1990, 2: 163-171. DOI:10.1105/tpc.2.2.163 |
| [6] |
Christensen AH, Sharrock RA, Quail PH. Maize polyubiquitin genes:structure, thermal perturbation of expression and transcript splicing, and promoter activity following transfer to protoplasts by electroporation[J]. Plant Mol Biol, 1992, 18: 675-689. DOI:10.1007/BF00020010 |
| [7] |
Fang RX, Nagy F, et al. Multiple cis-regulatory elements for maximal expression of the Cauliflower Mosaic Virus 35S promoter in transgenic plants[J]. Plant Cell, 1989, 1: 141-150. DOI:10.1105/tpc.1.1.141 |
| [8] |
Shelton AM, Zhao JZ, Roush RT. Economic, ecological, food safety, and social consequences of the deployment of bt transgenic plants[J]. Annu Rev Entomol, 2002, 47: 845-881. DOI:10.1146/annurev.ento.47.091201.145309 |
| [9] |
Bhullar S, et al. Strategies for development of functionally equivalent promoters with minimum sequence homology for transgene expression in plants:cis-elements in a novel DNA context versus domain swapping[J]. Plant Physiol, 2003, 132: 988-998. DOI:10.1104/pp.103.020602 |
| [10] |
Charrier B, Scollan C, Ross S, et al. Co-silencing of homologous transgenes in tobacco[J]. Mol Breed, 2000, 6: 407-419. DOI:10.1023/A:1009672714835 |
| [11] |
Xu L, Ye R, Zheng Y, et al. Isolation of the endosperm specific LPAAT gene promoter from coconut(Cocos nucifera L.)and its functional analysis in transgenic rice plants[J]. Plant Cell Rep, 2010, 29: 1061-1068. DOI:10.1007/s00299-010-0892-y |
| [12] |
Kong X, Zhang M, De Smet, et al. Designer crops:optimal root system architecture for nutrient acquisition[J]. Trends Biotechnol, 2014, 32: 597-598. DOI:10.1016/j.tibtech.2014.09.008 |
| [13] |
周少霞. 江西东乡普通野生稻抗旱渗入系的构建及抗旱基因定位[D]. 北京: 中国农业大学, 2005.
|
| [14] |
Tian X, Long Y, Wang J, et al. De novo transcriptome assembly of common wild rice(O. rufipogon Griff.)and discovery of drought-response genes in root tissue based on transcriptomic data[J]. PLoS One, 2015, 10(7): e0131455. DOI:10.1371/journal.pone.0131455 |
| [15] |
Jefferson RA, Kavanagh TA, Bevan MW. GUS fusions beta-glucuronidase as a sensitive and versatile gene fusion marker in higher plants[J]. EMBO J, 1987, 6(13): 3901-3907. |
| [16] |
Banerjee J, Sahoo D, et al. A region containing an as-1 Element of Dahlia Mosaic Virus(DaMV)suhgenomic transcript promoter plays a key role in green tissue-and root-specific expression in plants[J]. Plant Mol Biol Reporter, 2015, 33(3): 532-556. DOI:10.1007/s11105-014-0766-5 |
| [17] |
Price AH, Young EM, Tomos AD. Quantitative trait loci associated with stomatal conductance, leaf rolling and heading date mapped in upland rice(Oryza sativa)[J]. New Phytol, 1997, 137: 83-91. DOI:10.1046/j.1469-8137.1997.00818.x |
| [18] |
Ohbayashi I, Sugiyama M. Plant nucleolar stress response, a new face in the NAC-dependent cellular stress responses[J]. Front Plant Sci, 2017, 8: 2247. |
| [19] |
Jeong JS, Kim YS, et al. Root-specific expression of OsNAC10 improves drought tolerance and grain yield in rice under field drought conditions[J]. Plant Physiol, 2010, 153(1): 185-197. DOI:10.1104/pp.110.154773 |
| [20] |
Yu Z, Kang B, et al. Root hair-specific expansins modulate root hair elongation in rice[J]. Plant J, 2011, 66(5): 725-734. DOI:10.1111/j.1365-313X.2011.04533.x |
| [21] |
Chen L, Jiang B, Wu C, et al. GmPRP2 promoter drives root-preferential expression in transgenic Arabidopsis and soybean hairy roots[J]. BMC Plant Biology, 2014, 14(1): 245. DOI:10.1186/s12870-014-0245-z |
| [22] |
Chen L, Jiang B, Wu C, et al. The characterization of GmTIP, a root-specific gene from soybean, and the expression analysis of its promoter[J]. Plant Cell, 2015, 121(2): 259-274. |
| [23] |
Rausch C, Daram P, Brunner S, et al. A phosphate transporter expressed in arbuscule-containing cells in potato[J]. Nature, 2001, 414(6862): 462-470. DOI:10.1038/35106601 |
| [24] |
胡标林, 余守武, 万勇, 等. 东乡普通野生稻全生育期抗旱性鉴定[J]. 作物学报, 2007, 33: 425-432. |
| [25] |
Zhang X, Zhou S, Fu Y, et al. Identification of a drought tolerant introgression line derived from Dongxiang common wild rice(O. rufipogon Griff.)[J]. Plant Mol Biol, 2006, 62: 247-59. DOI:10.1007/s11103-006-9018-x |
| [26] |
Zhang JW, Long Y, Xue MD, et al. Identification of microRNAs in Response to Drought in Common Wild Rice(O. rufipogon Griff.)Shoots and Roots[J]. PLoS One, 2017, 12(1): e0170330. DOI:10.1371/journal.pone.0170330 |
| [27] |
赵志强, 薛满德, 等. 普通野生稻绿色组织特异表达启动子的克隆与鉴定[J]. 生物技术通报, 2017, 33(8): 51-57. |