2. 华中农业大学园艺林学学院 园艺植物生物学(教育部)重点实验室,武汉 430070
2. College of Horticultural Sciences and Forestry, Key Lab of Horticultural Plant Biology(MOE), Huazhong Agricultural University, Wuhan 430070
成簇的规律间隔回文重复序列及其相关蛋白系统(Clustered regularly interspaced short palindromic repeats/ Clustered regularly interspaced short palindromic repeats associated protein,CRISPR/CAS)是细菌中广泛存在的应对噬菌体的免疫系统[1-2]。该系统由CRISPR非编码RNA基因及一系列CAS蛋白基因组成。CAS蛋白根据功能可分为3部分,分别作用于CRISPR/CAS介导的免疫反应的3个阶段:在“获得”(Adaptation)阶段,一部分CAS蛋白质可以从入侵噬菌体基因组中捕获DNA片段并整合在CRISPR位点的回文序列中间,这部分CAS基因是所有CRISPR/CAS系统共有的;在“表达和加工”(Expression and processing)阶段,一部分CAS蛋白负责将CRISPR位点转录出的非编码RNA加工成短的引导RNA(gRNA),它们含有~20个来自噬菌体的碱基,并能与噬菌体DNA互补配对;在“干扰”(Interference)阶段,一部分CAS蛋白具有核酸酶活性与gRNA形成复合体,并在其引导下切割噬菌体DNA,以实现抵抗噬菌体入侵的功能[3]。
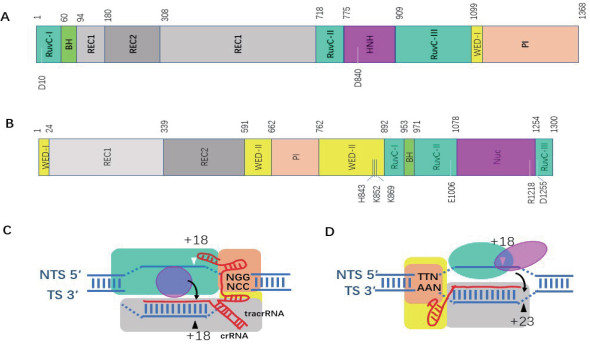
根据CRISPR/CAS系统中构成干扰复合体的蛋白组成,可将该系统分为三大类,第一类干扰复合体包括多个蛋白质分子,第二类干扰复合体仅含有一个蛋白分子,而第三类干扰复合体包含核酸聚合酶和核酸酶等多个蛋白分子[4]。其中第二类因为其干扰复合体较为简单,其机理研究也最为深入,根据其核酸酶的不同可进一步分为3个亚类,分别含有Cas9,Cas12(Cpf1)和Cas13作为其干扰复合体的唯一核酸酶[4-5]。Cas9干扰复合体由1个tracrRNA,1个crRNA和1个Cas9蛋白质组成。tracrRNA的3' 末端包含3个短的发卡结构,其5' 末端与crRNA的3' 末端的重复序列部分互补配对形成双链结构,形成gRNA。gRNA的发卡和双链结构部分与Cas9的REC,WED和PI结构域互作,其crRNA的5' 末端20个与噬菌体互补配对的碱基(称为spacer序列)则主要与REC结构域互作,形成Cas9干扰复合体[6-8]。CRISPR/CAS的靶标DNA除了有与crRNA的spacer序列完全同源的序列外,其下游还有3个保守的序列(Protospacer adjacent motif,PAM)。不同物种的Cas9识别的PAM不同,SpCas9识别的PAM序列为NGG[9]。Cas9干扰复合体与靶标DNA互作时,Cas9的PI结构域结合靶DNA上的PAM序列,crRNA的5' 末端spacer序列与靶标DNA的靶标链形成RNA/DNA双链结构,并与REC结构域和HNH核酸酶结构域互作,后者将靶标链DNA切断,在SpCas9中H840为HNH活性中心的关键残基。Cas9的另一个核酸酶结构域RuvC则与靶标DNA的非模版链互作并切割它,在SpCas9中其活性中心的关键残基包括D10等[6, 10-11](图 1-A和1-C)。Cpf1干扰复合体由一个Cpf1蛋白分子和一个crRNA组成。其crRNA的5' 末端含有一个短的发卡结构形成的“手柄”,而3' 末端的20个碱基则是来自与噬菌体的spacer序列,这种碱基的排列与Cas9干扰复合体中的crRNA是不同的。Cpf1蛋白质分子包含的结构域类型跟Cas9类似,但是排列顺序有很大的不同,主要是Cpf1的PI和WED结构域位于中间,而Cas9的相应结构域位于C端。Cpf1干扰复合体中gRNA与Cpf1结构域互作及复合体与靶标DNA的互作模式跟Cas9干扰复合体有类似之处,如PI结构域结合PAM序列,WED结合gRNA的发卡结构,REC结构域结合gRNA/靶标链DNA双链结构。不同的是Cpf1对两条DNA链的切割依赖于RuvC与Nuc两个结构域形成的单一活性中心,而Cas9则是由RuvC和HNH独立形成两个活性中心分别切割两条链(图 1-B和1-D)[12]。Cas13干扰复合体与Cas9和Cpf1干扰复合体在功能有很大的区别,后两者都靶定切割靶标DNA,而Cas13则靶定单链RNA,它实际上是一个由crRNA与靶标单链RNA形成的双链RNA激活的单链RNA酶,它被激活后不单降解靶标RNA,而是将宿主细胞内的所有RNA降解,导致宿主细胞的死亡[13]。Cas13在结构上也与Cas9和Cpf1有很大的不同,主要由两个helical结构域和两个HEPN结构域组成,后者为RNA酶结构域,Cas13没有DNA酶结构域。Cas13同时具有加工crRNA的功能和非特异性降解RNA的功能,分别由不同的活性中心催化[14-16]。
|
| 图 1 Cas9和Cpf1的结构域和干扰复合体与靶DNA的互作模型 A:SpCas9的序列结构。不同的颜色表示不同的结构域(名称标注在方框中);方框上的数字表示不同结构域的氨基酸在化脓性链球菌(Streptococcus pyogenes)Cas9中的位置;D10和H840分别表示RuvC和HNH两个剪切中心的关键氨基酸[45]。B:FnCpf1的序列结构。不同的颜色表示不同的结构域(名称标注在方框中);方框上的数字表示不同结构域的氨基酸在新凶手弗朗西丝氏菌(Francisella novicida)Cpf1中的位置;H843,K852和K869等表示形成gRNA加工活性中心的关键氨基酸;E1006,R1218和D1255等表示形成DNA剪切中心的关键氨基酸[9]。C:Cas9的作用模型。蓝色线条为靶DNA,NTS表示非靶标链,TS表示靶标链。不同结构域用不同颜色表示并与A中颜色一致。红色线条表示tracrRNA和crRNA形成的gRNA。NGG为靶DNA的PAM序列。白色三角形指示Cas9对靶DNA的切割位置为+18(非靶标链中spacer同源序列5末端为+1)。D:Cpf1的作用模型。与C类似,TTN为PAM序列 |
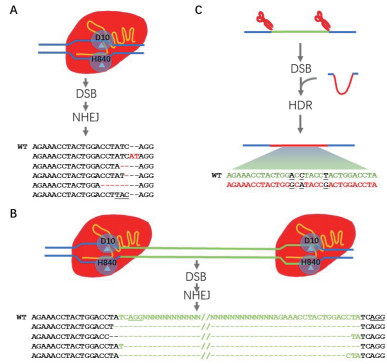
由于CRISPR/CAS系统中的Cas9和Cpf1干扰复合体组成简单,能对DNA进行定点切割,该系统很快被应用到基因的定点突变和编辑领域[17-18]。Cas9在基因定点突变上的应用最先在人类细胞系、小鼠和斑马鱼中报道[19-22],随即被应用到植物的基因定点突变和基于同源重组的基因定点编辑[23-25]。Cas9介导的基因定点突变和编辑可以通过多种方式进行(图 2-A-C)。研究人员通过细胞转染、注射或农杆菌介导转化等方法在细胞内表达核定位的Cas9蛋白质和gRNA序列,从而在活细胞形成Cas9干扰复合体并对靶标DNA单位点进行切割,然后在细胞的非同源末端连接修复通路作用下,被切断的DNA又被连接起来,在这个过程中会发生少量碱基的插入、缺失或变异,进而基因的突变或变异(图 2-A)。如果对同一个基因的两个不同位置设计gRNA,则可以通过上述类似的机制将两个靶位点间的序列删除(图 2-B),或者同时提供一个与切割位点两端有同源的模版,模版的中间含与原序列在特定位置有差异,通过同源重组实现靶基因的定点编辑(图 2-C)。Cpf1稍晚应用于植物基因的编辑[26-28]。目前报道的Cpf1介导的突变机理主要是通过DNA的切割后的非同源末端连接,产生少量碱基的插入或缺失突变。
|
| 图 2 Cas9及其衍生基因介导的基因突变和基因编辑机制 A:原始的Cas9通过诱导双链DNA断裂(DSB)及非同源末端连接(NHEJ)导致的少量碱基的插入或缺失(INDEL)。B:Cas9在相邻的两个位点进行切割,然后切割位点两侧的DNA通过非同源末端连接方式修复,在修复后的DNA链中原来两个切割位点间的序列被删除。A和B中红色图形表示Cas9蛋白,D10及H840表示催化DNA链水解的活性位点,黄色线条代表gRNA,蓝色线条代表靶DNA双链,白色三角指向DNA链的切割位点。C:通过Cas9产生的DSB及同源模版修复(HDR)进行定点编辑。红色剪刀代表Cas9及gRNA复合体,绿色线条代表要编辑的DNA序列,其旁边的蓝色线条代表侧翼DNA序列。红色U形线条表示含有编辑目标序列的同源修复模版,其两侧的蓝色短线条表示与要编辑的DNA的侧翼序列同源的序列 |
鉴于基因编辑在基础研究、医学和农业上的巨大潜力,科研人员对相关的研究倾注了极大的兴趣和努力。在短短的几年里,Cas9和Cpf1介导的各种各样的新功能被开发出来,用于基础研究和应用,例如转录抑制或激活、DNA或是RNA的定位等,同时对现有的系统进行改造升级,提高效率,特异性和扩大靶标范围[9-29]。过去两年中最引人注目的是碱基编辑技术的出现。早期的Cas9介导的突变依赖于目标细胞双链DNA断裂修复过程引入的随机突变,多导致基因的移码或缺失(图 2-A和2-B);而要实现碱基的定点编辑,则需同时靶定编辑位点两侧将待编辑碱基及其侧翼序列删除,同时提供含有编辑过的碱基的模版序列通过同源模版指导修复替换原有序列(图 2-C)。利用这种技术实现碱基的定点编辑的效率很低。
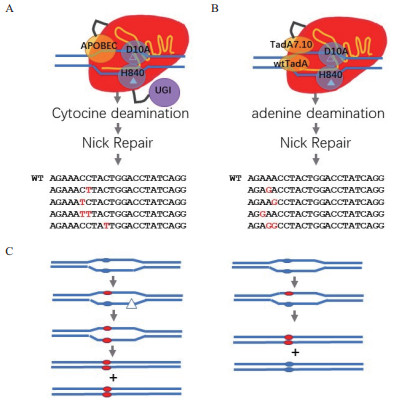
为解决这一问题,研究人员首先将目光投向了胞嘧啶脱氨酶并通过将APOBEC1与不具有DNA酶活性的dCas9融合得到了第一个C->T胞嘧啶编辑器(BE1,APOBEC1-XTEN-dCas9),它将胞嘧啶脱氨基转变为尿嘧啶U,后者在DNA复制过程中与A配对,从而实现C->T的编辑。在细胞中存在着尿嘧啶糖基化酶(UDG),它能将尿嘧啶从DNA链上切除,并触发DNA损伤修复机制将U修正为C,从而降低BE1的编辑效率。于是研究人员通过融合尿嘧啶糖基化酶抑制子(UGI)得到了编辑效率更高的BE2(APOBEC1-XTEN-dCas9-UGI)。为了进一步提高效率,研究人员恢复了dCas9的靶标链切割活性(nCas9)创建了改进型BE3(APOBEC1-XTEN-nCas9-UGI,图 3-A),由于APOBEC1只编辑单链的非靶标链,BE3可以切割靶标链后通过修复可以将碱基编辑引入靶标链中,这样DNA复制后两个子代DNA都是编辑过的(图 3-C),而BE2编辑的DNA其子代DNA只有一个是编辑过的[30]。在人类细胞系中BE3的编辑效率为10%-40%,其伴随的插入缺失突变(INDEL)率为1%左右,INDEL是我们在编辑过程中不希望看到的结果;而与之相对的Cas9切割加同源模版修复(Cas9+HDR)的编辑效率和INDEL概率分别为 < 1%和6%,可见BE3在碱基编辑方面要远远优于Cas9+HDR[30]。在植物中,APOBEC1-XTEN-nCas9首先被应用于C->T碱基编辑,获得了10%左右的编辑效率,但是INDEL概率几乎同样高[31]。密码子优化过的BE3在小麦、水稻和玉米中获得了高达约40%的编辑效率,而INDEL概率几乎为0[32]。BE3介导的胞嘧啶编辑窗口为非靶标链上spacer同源序列5' 末端起第2-9个碱基的范围[30, 32]。通过选择不同的Cas9及Cas9突变体对BE3进行改造,可以扩大靶点的PAM范围,同时通过对APOBEC1进行突变,可以进一步缩小胞嘧啶编辑窗口,从而对更广泛的靶点进行更精准的编辑[33]。
|
| 图 3 基于Cas9的碱基编辑器及其工作原理 A:胞嘧啶编辑器(BE)。D10A表示RuvC结构域中的催化活性中心的突变将Cas9变为缺刻酶(nCas9),黄色圆圈表示APOBEC1的胞嘧啶脱氨基结构域,紫色圆圈表示尿嘧啶糖基化酶抑制子(UGI),黑色曲线代表不同结构域间的连接肽。B:腺嘌呤编辑器(ABE)。nCas9的结构与A中相似,其N末端的两个黄色椭圆分别代表野生型和改造了的tRNA腺嘌呤脱氨基酶结构域。C:左边,基于nCas9的编辑器,能在DNA复制之前将非靶标链上的编辑结果(红色小椭圆)通过缺刻修复过程引入到靶标链,因此DNA复制后两条子链都是编辑过的。右边,基于dCas9的编辑器,只能将非靶标链编辑,DNA复制后只有一条子链是编辑过的 |
研究人员还通过融合海七鳃鳗(Petromyzon marinus)的胞嘧啶脱氨酶(PmCDA1)与Cas9构建了新的胞嘧啶编辑器(nCas9-Linker-PmCDA1),先后在酵母及人类细胞、水稻及番茄转基因植物中实现了定点的C->T和C->G编辑,在PmCDA1后面加上UGI可以大大提高C->T的编辑效率并降低INDEL概率[34-35]。在人类细胞系中nCas9-Linker-PmCDA1-UGI的编辑效率与BE3相当,但是INDEL概率要比BE3高[30, 35]。
基于APOBEC1和CDA1的碱基编辑器实现了C->T和C->G的定点编辑,为实现更多的编辑可能性,通过定向进化和蛋白质工程等手段以大肠杆菌的tRNA腺嘌呤脱氨酶(tRNA Adenine deaminase,TadA)为模版创造了可以对DNA进行腺嘌呤脱氨基反应的突变体TadA*,并与nCas9进行融合形成了腺嘌呤编辑器(Adenine base editor,ABE:TadA*-XTEN-nCas9-NLS)[36]。进一步改进了ABE的结构,采用野生型TadA与TadA*的异源二聚体,并对TadA*进行进一步的点突变,提高了ABE的编辑效率,获得了ABE7.10版本(图 3-B)。ABE将非靶标链上的A脱氨基变成Inosine(I),在复制过程中它与C配对,从而实现A到G的转变。与BE不同的是,抑制Inosine修复途径对提高ABE编辑效率无显著效果[36]。ABE7.10经过密码子优化后,在水稻中实现了对一些蛋白质编码基因的定点编辑[37-38]。
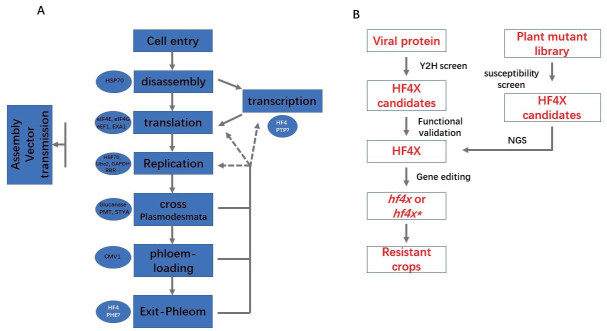
2 植物与病毒互作研究进展植物病毒是严格的胞内寄生病原,它完全依赖宿主细胞的大分子机器完成自身的基因表达和复制。它在寄主上的生活周期包括:进入寄主细胞(Cell entry),卸载基因组(Disassembly),转录(Transcription),翻译(Translation),复制(Replication),跨过胞间连丝(Cross-plasmodesmata),进入韧皮部(Phloem loading),出韧皮部进入系统叶肉细胞(Exit-phloem)等过程(图 4-A)。由于病毒的基因组非常精简,编码的蛋白质数量有限,病毒在完成其生活周期的过程中大量依赖宿主的蛋白质,因此这些过程中都涉及到病毒的蛋白质与宿主的蛋白质相互作用。对于正链RNA病毒来说,宿主蛋白因子在其生活周期中的作用是过去十年中植物病毒学研究的热点,研究人员取得了很多重要的进展[39]。
|
| 图 4 植物病毒互作研究及基因编辑技术在抗病育种中的应用 A:病毒在植物细胞内的生活周期。方框表示病毒生活周期中的不同的阶段,黑色文字标注的椭圆表示已发现的针对不同病毒不同阶段所需的宿主蛋白。HF4PHE和HF4TRX表示尚待发现的帮助病毒出韧皮部进入系统叶和帮助病毒mRNA转录的宿主因子;B:植物病毒互作研究及基因编辑技术相结合在抗病育种中的应用策略 |
植物热激蛋白HSP70在多种病毒侵染过程中大量诱导表达,研究表明HSP70与黄瓜坏死病毒(Cucumber necrosis virus,CNV)的颗粒紧密结合。纯化的CNV病毒颗粒经过HSP70抗体处理后,侵染性降低,相反在植物中过表达HSP70则可以增强CNV的侵染性,暗示HSP70可能在病毒进入宿主细胞后的基因组RNA卸载过程起作用[40-41]。在病毒mRNA的翻译方面,翻译起始和延伸因子(eIF4E,eIF4G和eEF1等)在Potyvirus等多个属的病毒mRNA翻译中的作用已得到证实,并且有多种植物的抗病毒自然变异被定位到这些宿主因子[42]。此外,拟南芥中的基因EXA1最近被证实是Potexvirus病毒属的PIAMV侵染所必需的。该基因在单子叶和双子叶植物中高度保守,含有一个与富含脯氨酸序列互作的GYF结构域和一个与eIF4E互作的保守序列[43]。因此EXA1很可能在病毒的mRNA翻译过程起作用。
在病毒的复制方面,番茄丛矮病毒(Tomato bushy stunt virus,TBSV)的复制酶的激活依赖于植物热激蛋白HSP70和病毒本身的分子伴侣p33[44],而植物的泛素连接酶AtUbc2对p33的泛素化对于其发挥功能有着重要的作用[45]。TBSV的p33很可能通过将胞内脑磷脂(Phosphatidylethanolamine,PE)富集于复制位点促进病毒复制复合体的形成[46]。与此相呼应的是,研究人员在相关的研究中发现了脑磷脂和卵磷脂(Phosphatidylcholine,PC)对病毒的复制有促进作用,而磷脂酸则对病毒复制有抑制作用[47]。此外,糖酵解反应中的甘油醛-3-磷酸脱氢酶(Glyceraldehyde-3-phosphate dehydrogenase,GAPDH)是一个水杨酸(Salicylic acid,SA)结合蛋白,胞质内的GAPDH可以结合TBSV负链RNA促进其复制,SA则通过影响GAPDH的这一功能抑制TBSV的复制[47]。DNA病毒的复制与RNA病毒的复制有很大不同,它依赖于细胞的DNA复制机器和原料,而在完成分化的细胞中RNA的合成还在正常进行,但是DNA复制一般已经停止,因此DNA病毒的复制需重新激活宿主细胞的DNA复制程序。在双生病毒中,Rep和REn蛋白通过抑制植物的RBR蛋白(RETINOBLASTOMA-related),进而解除其对E2F转录因子的抑制,使得E2F激活细胞周期S阶段的基因表达为病毒的复制创造条件[48]。此外Rep和REn还与一系列植物DNA复制叉的组成成分互作,如PCNA、MCM2、RPA32、RFC和DNA聚合酶等,并将其招募到病毒DNA上起始病毒DNA的复制[49]。
胞间连丝(Plasmodesmata)是植物细胞间的连通通道,也是病毒在细胞间运输的必经之路。植物病毒的移动蛋白(Movement protein,MP)通过招募一些改变细胞壁结构的酶,如葡聚糖水解酶(beta-1,3-glucanase)和果胶甲酯酶(Pectin methylesterase)等[50-51],到胞间连丝来增大其孔径以便于病毒的运输。而MP到胞间连丝的运输则是涉及到内质网(ER),细胞膜(PM)和ER-PM连接点的相互作用及一系列MP的互作蛋白的作用,如STYA,ANK和Myosin等[52-55]。韧皮部(Phloem)的筛管(Sieve tube)是植物病毒系统扩散的高速公路,而病毒要从已被侵染的叶肉细胞进入到筛管,需经过束鞘(Boundle sheath),薄壁细胞(Parenchyma cell)和伴细胞(Companion cell)等多层屏障。甜瓜的cmv1突变体使得黄瓜花叶病毒阻隔在束鞘细胞内,无法侵入其它韧皮部细胞[56],这表明病毒在跨越不同类型的韧皮部细胞时需要与不同的宿主因子互作。
3 利用基因编辑创制抗病种质病毒与宿主的互作研究为人们描述了病毒在宿主内复制扩散的分子机理,同时也为植物病害防治提供了着力点。对于同一类型的病毒而言,其依赖的宿主因子往往在不同种的植物宿主中有保守性,这使得改造宿主因子获得隐性抗性的方法具有较大的应用范围。如Potyvirus病毒属的病毒mRNA翻译依赖于宿主翻译延伸因子eIF4E或其同源体,这一机制在多个物种中都是保守的,因此尽管eIF4E自然变异导致的对Potyvirus的抗性只在辣椒番茄和生菜中发现,但研究人员利用CRISPR/Cas9介导的基因编辑很快在拟南芥和黄瓜中创造出eIF4E突变体获得对相应的Potyvirus的抗病种质[57-58]。
病毒与宿主的互作机制的解析以及基因编辑技术的成熟,为创制抗病毒的种质资源提供了一个通用的策略。基于病毒在其生活周期中其自身编码的蛋白必须与宿主蛋白发生直接互作这一原理,我们可以通过酵母双杂交实验,以不同的病毒蛋白为诱饵去筛选宿主的cDNA文库,得到候选的病毒蛋白互作因子(Candidate host factor interacting with viral protein,designated HF4X candidate,图 4-B)。随后通过病毒诱导的基因沉默(VIGS),人工miRNA稳定转化或CRISPR/Cas9等技术将候选基因沉默或敲除,并通过病毒接种实验证实其对病毒复制扩散的必要性,从而找到病毒所依赖的宿主因子(HF4X,图 4-B)。如果HF4X通过CRISPR/Cas9敲除后得到的突变体(hf4x)能正常生长发育,则其隐性纯合体可直接作为抗病种质使用。如果一个候选基因能通过VIGS证实其对病毒的复制扩散的必要性,但转基因过程中有纯合致死的现象,可以找到一些能特异性影响其与病毒蛋白互作的点突变,然后通过碱基编辑器(BE或ABE)将相应的点突变(hf4x*)引入植物中以获得隐性抗性(图 4-B)。事实上,eIF4E在Potyvirus侵染中的作用,最初就是通过其VPg蛋白对拟南芥的cDNA文库进行酵母双杂交筛选发现的[59],后来通过遗传学实验得到了验证[60],并通过基因编辑技术将其应用推广到了黄瓜中[58]。
以上策略是从病毒出发的,此外我们还可以从植物的突变体筛选出发找到不支持病毒侵染的突变体,通过基因克隆找到相应的宿主因子,并通过基因编辑应用到抗病种质的创制上(图 4-B),这部分内容最近的综述有较为详细的阐述,这里不再赘述[61]。迄今为止,这种方法筛选到的可供利用的宿主基因比较有限[61],其原因可能是由于一方面病毒在某一生活阶段依赖的宿主因子具有冗余性,单个基因的突变难以显现其影响,另一方面这些基因可能是宿主正常生长发育所必须的,不能获得纯合突变。因此能通过这种策略筛选到的仅仅为病毒所必需的单基因很可能是个别现象。
4 结语小分子RNA介导的抗病毒基因沉默和抗病基因介导的抗病反应是植物对抗病毒的两种先天免疫机制,在过去的几十年中这两方面的研究为抗病毒育种提供了重要的思路和丰富的抗病种质[62-63]。现在基因编辑技术的飞速发展,使得通过定点突变病毒侵染所需的宿主基因来获得病毒抗性成为一个可以普遍应用的方法,为抗病毒育种提供了新的思路和选择。由于病毒所依赖的宿主蛋白因子大多参与植物本身的生命活动,因此不能简单的通过定点敲除来应用到抗病育种。今后的植物与病毒互作研究中要重视生化方面的研究,注重植物蛋白与病毒蛋白互作的详细机理的研究,找到一些决定互作的关键氨基酸残基,以期通过基因编辑技术实现一些关键氨基酸的改变来特异性的破坏植物蛋白在病毒侵染过程中的功能而不影响其对植物本身生命活动所起的作用。
| [1] |
Ruud J, Embden JDAV, Gasstra W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Molecular Microbiology, 2002, 43(6): 1565-1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [2] |
Amitai G, Sorek R. CRISPR-Cas adaptation: insights into the mechanism of action[J]. Nat Rev Microbiol, 2016, 2: 67-76. |
| [3] |
李铁民. RNA干扰介导原核细胞适应性免疫[J]. 中国细胞生物学学报, 2008, 30(3): 287-290. DOI:10.3969/j.issn.1674-7666.2008.03.002 |
| [4] |
Makarova KS, Haft DH, Barrangou R, et al. Evolution and classification of the CRISPR-Cas systems[J]. Nature Review Microbiology, 2011, 9(6): 467-477. DOI:10.1038/nrmicro2577 |
| [5] |
Shmakov S, Smargon A, Scott D, et al. Diversity and evolution of class 2 CRISPR-Cas systems[J]. Nature Reviews Microbiology, 2017, 15(3): 169-182. DOI:10.1038/nrmicro.2016.184 |
| [6] |
Nishimasu H, Ran FA, Hsu P, et al. Crystal structure of Cas9 in complex with guide RNA and target DNA[J]. Cell, 2014, 156(5): 935-949. DOI:10.1016/j.cell.2014.02.001 |
| [7] |
Jiang F, Doudna JA. The structural biology of CRISPR-Cas systems[J]. Curr Opin Struct Biol, 2015, 30: 100-111. DOI:10.1016/j.sbi.2015.02.002 |
| [8] |
Jiang F, Zhou K, Ma L, et al. A Cas9-guide RNA complex preorganized for target DNA recognition[J]. Science, 2015, 348(6242): 1477-1481. DOI:10.1126/science.aab1452 |
| [9] |
Komor AC, Badran AH, Liu DR. CRISPR-based technologies for the manipulation of eukaryotic genomes[J]. Cell, 2017, 3: 559. |
| [10] |
Jiang F, Taylor DW, Chen JS, et al. Structures of a CRISPR-Cas9 R-loop complex primed for DNA cleavage[J]. Science, 2016, 351(6275): 867-871. DOI:10.1126/science.aad8282 |
| [11] |
Nishimasu H, Nureki O. Structures and mechanisms of CRISPR RNA-guided effector nucleases[J]. Current Opinion in Structural Biology, 2017, 43: 68-78. DOI:10.1016/j.sbi.2016.11.013 |
| [12] |
Swarts DC, Oost JVD, Jinek M. Structural basis for guide RNA processing and seed-dependent DNA targeting by CRISPR-Cas12a[J]. Mol Cell, 2017, 66(2): 221-233. DOI:10.1016/j.molcel.2017.03.016 |
| [13] |
Abudayyeh OO, Gootenberg JS, Konermann S, et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector[J]. Science, 2016, 353(6299): aaf5573. DOI:10.1126/science.aaf5573 |
| [14] |
East-Seletsky A, O'Connell MR, Knight SC, et al. Two distinct RNase activities of CRISPR-C2c2 enable guide-RNA processing and RNA detection[J]. Nature, 2016, 538(7624): 270-273. DOI:10.1038/nature19802 |
| [15] |
Liu L, Li X, Wang J, et al. Two distant catalytic sites are responsible for C2c2 RNase activities[J]. Science Foundation in China, 2017, 168(2): 121-134. |
| [16] |
Liu L, Li X, Ma J, et al. The molecular architecture for RNA-guided RNA cleavage by Cas13a[J]. Cell, 2017, 4: 714-726. |
| [17] |
Wright AV, Nunez JK, Doudna JA. Biology and applications of CRISPR systems: Harnessing nature's toolbox for genome engineering[J]. Cell, 2016, 164(1/2): 29-44. |
| [18] |
Ran YD, Liang Z, Gao CX. Current and future editing reagent delivery systems for plant genome editing[J]. Science China: Life Sciences, 2017, 60(5): 490-505. DOI:10.1007/s11427-017-9022-1 |
| [19] |
Cong L, Ran FA, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 6121: 819-823. |
| [20] |
Hwang WY, Fu Y, Reyon D, et al. Efficient genome editing in zebrafish using a CRISPR-Cas system[J]. Nat Biotechnol, 2013, 31(3): 227-229. DOI:10.1038/nbt.2501 |
| [21] |
Mali P, Yang L, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [22] |
Wang H, Yang H, Shivalila CS, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering[J]. Cell, 2013, 153(4): 910-918. DOI:10.1016/j.cell.2013.04.025 |
| [23] |
Li JF, Norvill JE, Aach J, et al. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9[J]. Nat Biotechnol, 2013, 31(8): 688-691. DOI:10.1038/nbt.2654 |
| [24] |
Nekrasov V, Staskawicz B, Weigel D. Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease[J]. Nat Biotechnol, 2013, 31(8): 691-693. DOI:10.1038/nbt.2655 |
| [25] |
Shan Q, Wang Y, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system[J]. Nat Biotechnol, 2013, 31(8): 686-688. DOI:10.1038/nbt.2650 |
| [26] |
Wang M, Mao Y, et al. Multiplex gene editing in rice using the CRISPR-Cpf1 System[J]. Mol Plant, 2017, 7: 1011-1013. |
| [27] |
Tang X, Lowder LG, Zhang T, et al. A CRISPR-Cpf1 system for efficient genome editing and transcriptional repression in plants[J]. Nat Plants, 2017, 3: 17018. DOI:10.1038/nplants.2017.18 |
| [28] |
Endo A, Masafumi M, Kaya H, et al. Efficient targeted mutagenesis of rice and tobacco genomes using Cpf1 from Francisella novicida[J]. Sci Rep, 2016, 6: 38169. DOI:10.1038/srep38169 |
| [29] |
Li H, Xie K. Recent progresses in CRISPR genome editing in plants[J]. Chinese Journal of Biotechnology, 2017, 33(10): 1700-1711. |
| [30] |
Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533(7603): 420-424. DOI:10.1038/nature17946 |
| [31] |
Lu YM, Zhu JK. Precise editing of a target base in the rice genome using a modified CRISPR/Cas9 system[J]. Molecular Plant, 2017, 10(3): 523-525. DOI:10.1016/j.molp.2016.11.013 |
| [32] |
Zong Y, Wang YP, Li C, et al. Precise base editing in rice, wheat and maize with a Cas9-cytidine deaminase fusion[J]. Nat Biotechnol, 2017, 35(5): 438-440. DOI:10.1038/nbt.3811 |
| [33] |
Kim YB, Komore AC, et al. Increasing the genome-targeting scope and precision of base editing with engineered Cas9-cytidine deaminase fusions[J]. Nat Biotechnol, 2017, 35(4): 371-376. DOI:10.1038/nbt.3803 |
| [34] |
Shimatani Z, Kashojiya S, Takayama M, et al. Targeted base editing in rice and tomato using a CRISPR-Cas9 cytidine deaminase fusion[J]. Nat Biotechnol, 2017, 35(5): 441-443. DOI:10.1038/nbt.3833 |
| [35] |
Nishida K, Arazoe T, Yachie N, et al. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems[J]. Science, 2016, 353(6305): aaf8729. DOI:10.1126/science.aaf8729 |
| [36] |
Gaudelli NM, Komor AC, Rees HA, et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage[J]. Nature, 2017, 551(7681): 464-471. DOI:10.1038/nature24644 |
| [37] |
Hua K, TAO X, Yuan F, et al. Precise A. T to G. C base editing in the rice genome[J]. Molecular Plant, 2018, 11(4): 627-630. DOI:10.1016/j.molp.2018.02.007 |
| [38] |
Yan F, Kuang Y, Ren B, et al. High-efficient A. T to G. C base editing by Cas9n-guided tRNA adenosine deaminase in rice[J]. Molecular Plant, 2018, 11(4): 631-634. DOI:10.1016/j.molp.2018.02.008 |
| [39] |
Wang A. Dissecting the molecular network of virus-plant interactions: the complex roles of host factors[J]. Annual Review of Phytopathology, 2015, 53(1): 45-66. DOI:10.1146/annurev-phyto-080614-120001 |
| [40] |
Alam SB, Rochon DA. Cucumber necrosis virus recruits cellular heat shock protein 70 homologs at several stages of infection[J]. J Virol, 2016, 90(7): 3302-3317. DOI:10.1128/JVI.02833-15 |
| [41] |
Alam SB, Rochon DA. Evidence that Hsc70 is associated with cucumber necrosis virus particles and plays a role in particle disassembly[J]. J Virol, 2017, 91(2): 01555-16. |
| [42] |
Sanfacon H. Plant translation factors and virus resistance[J]. Viruses, 2015, 7(7): 3392-3419. DOI:10.3390/v7072778 |
| [43] |
Hashimoto M, Neriya Y, et al. EXA1, a GYF domain protein, is res-ponsible for loss-of-susceptibility to Plantago asiatica mosaic virus in Arabidopsis thaliana[J]. Plant J, 2016, 88(1): 120-131. DOI:10.1111/tpj.13265 |
| [44] |
Pogany J, Nagy D. Activation of tomato bushy stunt virus RNA-dependent RNA polymerase by cellular heat shock protein 70 is enhanced by phospholipids in vitro[J]. J Virol, 2015, 89(10): 5714-5723. DOI:10.1128/JVI.03711-14 |
| [45] |
Imura Y, Molho M, Chuang C, et al. Cellular Ubc2/Rad6 E2 ubiquitin-conjugating enzyme facilitates tombusvirus replication in yeast and plants[J]. Virology, 2015, 484: 265-275. DOI:10.1016/j.virol.2015.05.022 |
| [46] |
Xu K, Nagy D. RNA virus replication depends on enrichment of phosphatidylethanolamine at replication sites in subcellular membranes[J]. Proc Natl Acad Sci USA, 2015, 112(14): E1782-E1791. DOI:10.1073/pnas.1418971112 |
| [47] |
Tian M, Sasvani Z, Gonzalez PA, et al. Salicylic acid inhibits the replication of tomato bushy stunt virus by directly targeting a host component in the replication complex[J]. Mol Plant Microbe Interact, 2015, 28(4): 379-386. DOI:10.1094/MPMI-09-14-0259-R |
| [48] |
Hanley-Bowdoin L, Bejarano ER, Robertson D, et al. Geminiviruses: masters at redirecting and reprogramming plant processes[J]. Nat Rev Microbiol, 2013, 11(11): 777-788. DOI:10.1038/nrmicro3117 |
| [49] |
Rizvi I, Choudhury NR, Tuteja N. Insights into the functional characteristics of geminivirus rolling-circle replication initiator protein and its interaction with host factors affecting viral DNA replication[J]. Archives of Virology, 2015, 160(2): 375-387. DOI:10.1007/s00705-014-2297-7 |
| [50] |
Fridborg I, Grainger J, Page A, et al. TIP, a novel host factor linking callose degradation with the cell-to-cell movement of potato virus X[J]. Mol Plant Microbe Interact, 2003, 16(2): 132-140. DOI:10.1094/MPMI.2003.16.2.132 |
| [51] |
Bubici G, Carluccio AV, Cillo F, et al. Virus-induced gene silencing of pectin methylesterase protects Nicotiana benthamiana from lethal symptoms caused by Tobacco mosaic virus[J]. European Journal of Plant Pathology, 2015, 141(2): 339-347. DOI:10.1007/s10658-014-0546-y |
| [52] |
Ishikawa K, et al. Dual targeting of a virus movement protein to ER and plasma membrane subdomains is essential for plasmodesmata localization[J]. PLoS Pathogens, 2017, 13(6): e1006463. DOI:10.1371/journal.ppat.1006463 |
| [53] |
Levy A, Zheng JY, Lazarowitzl SG. Synaptotagmin SYTA forms ER-plasma membrane junctions that are recruited to plasmodesmata for plant virus movement[J]. Curr Biol, 2015, 15: 2018-2025. |
| [54] |
Pitzalis N, Heinlein M. The roles of membranes and associated cytoskeleton in plant virus replication and cell-to-cell movement[J]. J Exp Bot, 2017, 69(1): 117-132. |
| [55] |
Ueki S, Spektor R, Natale DM, et al. ANK, a host cytoplasmic receptor for the tobacco mosaic virus cell-to-cell movement protein, facilitates intercellular transport through plasmodesmata[J]. PLoS Pathogens, 2010, 6(11): e1001201. DOI:10.1371/journal.ppat.1001201 |
| [56] |
Guiu-Aragones C, Sánchez-Pina MA, et al. cmv1 is a gate for Cucumber mosaic virus transport from bundle sheath cells to phloem in melon[J]. Mol Plant Pathol, 2016, 17(6): 973-984. DOI:10.1111/mpp.12351 |
| [57] |
Pyott DE, Sheehan E, Molnar A. Engineering of CRISPR/Cas9-mediated potyvirus resistance in transgene-free Arabidopsis plants[J]. Mol Plant Pathol, 2016, 17(8): 1276-1288. DOI:10.1111/mpp.12417 |
| [58] |
Chandrasekaran J, Brumin M, Wolf D, et al. Development of broad virus resistance in non-transgenic cucumber using CRISPR/Cas9 technology[J]. Mol Plant Pathol, 2016, 17(7): 1140-1153. DOI:10.1111/mpp.2016.17.issue-7 |
| [59] |
Wittmann S, Chatel H, et al. Interaction of the viral protein genome linked of turnip mosaic potyvirus with the translational eukaryotic initiation factor(iso)4E of Arabidopsis thaliana using the yeast two-hybrid system[J]. Virology, 1997, 1: 84-92. |
| [60] |
Lellis AD, Kasschau KD, et al. Loss-of-susceptibility mutants of Ar-abidopsis thaliana reveal an essential role for eIF(iso)4E during potyvirus infection[J]. Curr Biol, 2002, 12(12): 1046-1051. DOI:10.1016/S0960-9822(02)00898-9 |
| [61] |
Hashimoto M, Neriya Y, Yamaji Y, et al. Recessive resistance to plant viruses: potential resistance genes beyond translation initiation factors[J]. Front Microbiol, 2016, 7: 1695. |
| [62] |
Deng Y, Liu M, Li X, et al. microRNA-mediated R gene regulation: molecular scabbards for double-edged swords[J]. Science China Life Science, 2018, 61(2): 138-147. DOI:10.1007/s11427-017-9237-4 |
| [63] |
Khalid A, Zhang Q, Yasir M, et al. Small RNA based genetic engineering for plant viral resistance: application in crop protection[J]. Front Microbiol, 2017, 8: 43. |




