2. 广东海洋大学水产学院,湛江 524003;
3. 广东省水生动物健康评估工程技术研究中心,深圳 518116;
4. 中国科学院水生生物研究所,武汉 430072
2. Fisheries College, Guangdong Ocean University, Zhanjiang 524003;
3. Guangdong Provincial Engineering Research Center for Aquatic Animal Health Assessment, Shenzhen 518116;
4. Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072
近年来,我国水产养殖业发展迅速,养殖业面临的诸多问题也日益突出,尤其是病原感染引起的水产病害,给养殖业造成了巨大的经济损失。疾病的发生是由病原、宿主和环境三者相互作用的结果,其中宿主的免疫应答对抵御病原微生物的感染具有重要作用,因此研究鱼类免疫应答机制一直是学术界的热点,特别是在胚胎发育早期发挥关键作用的先天性免疫。肽聚糖识别蛋白(Peptidoglycan recognition proteins,PGRPs)作为一种重要的模式识别受体,在鱼类抵御病原侵染过程中扮演着十分重要的角色。目前有关鱼类PGRPs的研究已经取得了一定的进展,本文综述了鱼类PGRPs的结构、表达及其功能,旨在为鱼类先天性免疫的研究提供理论参考。
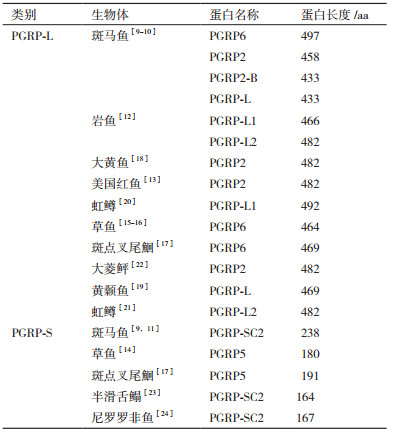
1 PGRPs的发现肽聚糖识别蛋白(Eptidoglycan recognition proteins,PGRPs)是一类可识别肽聚糖(Peptidoglycan,PGN)的模式识别受体家族,最早发现于家蚕血淋巴中[1]。随后,在昆虫[2]、棘皮动物[3]和脊椎动物[4-5]中相继被发现。目前已报道的鱼类PGRP共23种(表 1)。根据氨基酸序列的不同,PGRPs主要分为3类:短型PGRPs(Short-PGRPs,PGRP-S)、中型PGRPs(Intermediate-PGRPs,PGRP-I)和长型PGRPs(Long-PGRPs,PGRP-L)[6]。果蝇PGRPs主要分为两类:一类是较短的转录产物,包括PGRP-SA、-SB1、-SB2、-SC1A、-SC1B、-SC2和-SD;另一类是较长的转录产物,包括PGRP-LAa、-LAb、-LAc、-LB、-LCa、-LCx、-LCy、-LD、-LE和-LF[5, 7]。哺乳动物PGRPs有不同的转录产物:PGRP-S、PGRP-Iα、PGRP-Iβ和PGRP-L,也称为PGLYRP-1、PGLYRP-2、PGLYRP-3和PGLYRP-4[3, 8]。迄今,在包括斑马鱼(Danio rerio)[9-11]、岩鱼(Sebastes schlegeli)[12]、美国红鱼(Sciaenops ocellatus)[13]、草鱼(Ctenopharyngodon idella)[14-16]、斑点叉尾鮰(Ictalurus punctatus)[17]、大黄鱼(Pseudosciaena crocea)[18]、黄颡鱼(Pelteobagrus fulvidraco)[19]、虹鳟(Oncorhynchus mykiss)[20-21]、大菱鲆(Scophthalmus maximus)[22]、半滑舌鳎(Cynoglossus semilaevis)[23]和尼罗罗非鱼(Oreochromis niloticus)[24]等鱼类中发现3类PGRPs,其中PGLYRP-2与哺乳类PGLYRP-2为同源物,而PGLYRP-5(也称为PGRP-SC)和PGLYRP-6则是鱼类所特有的[17]。
在结构上,所有PGRPs的C端通常包含至少一个高度保守的PGRP结构域,与噬菌体T7溶菌酶和细菌的Ⅱ型酰胺酶有一定的相似性,而PGRPs在N端的序列长度差异较大,在不同生物间并不保守[3]。人类PGRPs都含有信号肽结构,而信号肽是蛋白质经典分泌途径的关键结构;然而,斑马鱼PGRP2,PGRP5并不含信号肽结构,但斑马鱼PGRPs也均是分泌蛋白[5, 25-26],其机制尚不明确。尽管鱼类PGRPs在大小、结构域布局及亚细胞定位方面有一定差异,但其C端的PGRP结构域是高度保守的。哺乳动物PGRP结构域的空间构象是由位于中心的几个β片层和外围的3个α螺旋(α1-α3)组成[27]。研究表明,鱼类PGRPs也有类似的结构,而个别剪切体因某个氨基酸位点的缺失,导致结构上稍有不同[9, 16, 24]。
PGRP结构域都含有高度保守的半胱氨酸位点,其形成的二硫键是赋予PGRPs功能活性和维持蛋白结构稳定所必须的重要结构[28]。研究表明,该结构域中的某半胱氨酸位点突变将使其丧失酰胺酶活性[29-31]。晶体结构显示,PGRP结构域构象的正面凹槽含有保守的Zn2+结合位点,由两个组氨酸、一个酪氨酸和一个半胱氨酸组成,是结合肽聚糖的活性中心;其另一面为PGRP特异,且不保守的疏水区域,不同于催化酰胺键水解反应的Ⅱ型酰胺酶结构[31-34]。研究表明,结合PGN的凹槽结构被破坏后,将使其无法识别PGN,从而失去酰胺酶活性[29-30]。通过多重序列比对发现,鱼类PGRPs基本也都含有4个Zn2+结合位点,如图 1所示。说明鱼类PGRPs与哺乳动物可能有类似的功能,但其作用机制是否也依赖于半胱氨酸残基还有待进一步研究。

|
| 图 1 鱼类肽聚糖识别蛋白的多序列比对结果 箭头:保守的Zn2+结合位点 |
鱼类PGRPs在不同器官/组织中广泛表达,但其表达组织和表达量都存在明显差异。与斑马鱼PGRP2类似,美国红鱼、大黄鱼和大菱鲆PGRP2在各组织中普遍存在,尤其在肝脏中表达量最高[9, 13, 18, 22]。岩鱼PGRP-L1在肠和脾脏中表达量较高;其次是鳃和皮肤等,而PGRP-L2仅在肝脏中表达[12]。实验表明,在不同细菌的刺激下,鱼类PGRPs在各免疫组织的表达量均明显改变。因此推测鱼类PGRPs参与了机体的免疫应答过程,且发挥效应的部位各不相同,如草鱼PGRP6a、PGRP6d通过鳃和皮肤发挥作用,而PGRP6b的效应部位是肠,脾脏和肝脏等[16]。鱼类PGRPs的广泛表达将有利于机体及时将体内感染的病原体清除。同时,在非免疫组织中的表达说明PGRPs在生理过程过可能也扮演着重要角色。此外,实验表明,鱼类PGRPs在胚胎发育早期阶段的先天性免疫中发挥着关键作用[18, 26]。
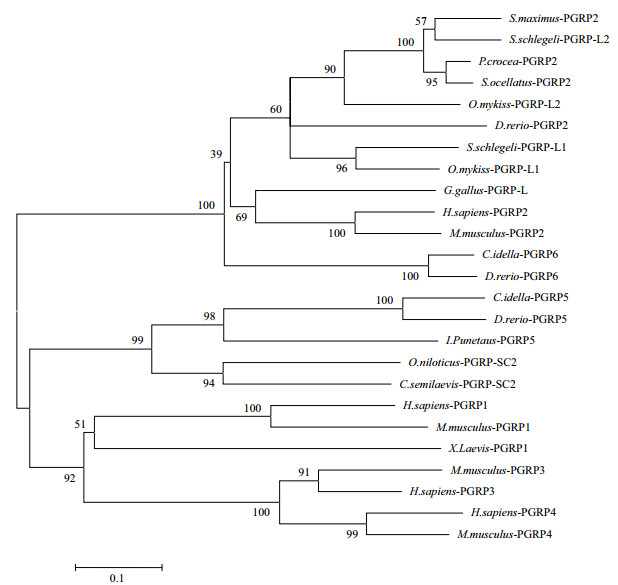
3 PGRPs的进化利用生物信息学软件Clustalx和MEGA5.0构建了N -J(Neighbor-joining)进化树,对来自人类、小鼠、原鸡、非洲爪蟾和斑马鱼等14个物种(表 2)的PGRPs的进化关系进行分析,结果表明,不同物种的PGRP-S和PGRP-L分别独立进化,即同种类型PGRPs聚为一支,而与本物种的其他类型PGRPs同源性相对较低,这与以往的报道高度一致[3, 35](图 2)。
|
| 图 2 PGRPs的系统进化树 |
PGN是几乎所有细菌细胞壁的主要成分,包括L-赖氨酸(Lys型)和二氨基庚二酸(Dap型)两种类型,Lys型PGN主要存在于革兰氏阳性菌中,而Dap型PGN是革兰氏阴性菌的主要组成成分[36]。当细菌或其他含PGN的外源物体侵染机体时,能被宿主PGRPs识别并与之结合,从而激发机体的免疫效应。人PGLYRP3结合PGN后,其PGRP结构域的构象随即发生改变,将目标物固定在凹槽内,提高结合过程的亲和力[34, 36-38]。研究表明,鱼类PGRPs能够结合Lys型和Dap型两种PGN[14-15, 26]。除了PGN以外,人和鼠PGRPs还能够结合脂膦壁酸(LTA),脂多糖(LPS)等聚合物,但对后两者的亲和力明显较低,但猪PGLYRP1对LTA和LPS有很强的亲和力[39-41]。根据文献报道,PGN、脂膦壁酸(Lipoteichoic acid,LTA)和Ploy I:C均能够诱导草鱼PGRP5和PGRP6的高表达[14-16]。说明草鱼PGRP5、PGRP6与哺乳动物PGRPs类似,能够识别不同的病原相关分子模式(Pathogen-associated molecular pattern,PAMPs),至于其相互作用的亲和力是否有差异,还有待进一步的研究。
4.2 酰胺酶活性PGRPs能够水解细菌肽聚糖中N -乙酰胞壁酸与L-丙氨酸之间的酰胺键,将有活性肽聚糖的分子片段化,从而使其失去活性,达到清除细菌的目的[31, 42]。人PGLYRPs中,仅PGLYRP2具有酰胺酶活性[36]。研究表明,保守的Zn2+结合位点对于PGRPs发挥酰胺酶活性是必须的[33-34]。如图 2所示,鱼类PGRPs的C端含有保守的半胱氨酸残基,暗示其和人PGLYRP2一样,都具有酰胺酶活性。文献报道多种鱼类的PGRPs都具有酰胺酶活性,且呈Zn2+依赖性[11-15, 23-24],2012年,孙黎等克隆美国红鱼PGLYRP2和PGLYRP2-AD(酰胺酶结构域),分别进行原核表达,分离纯化重组蛋白,并进行枯草芽孢杆菌肽聚糖的水解实验,结果显示,PGLYRP2和PGLYRP2-AD都具有酰胺酶活性,水解肽聚糖[13]。
4.3 杀菌、抑菌活性当PGRP与微生物细胞壁成分结合后形成复合物,能够激活酚氧化酶,经过一系列反应,将含酚的化合物氧化成醌,转换成黑色素将微生物包裹来限制其感染[2]。哺乳动物PGRPs只有酰胺酶活性或者杀菌活性,与PGLYRP2不同,人PGLYRP-1,PGLYRP-3,PGLYRP-4在对应Cys530位点上是丝氨酸而非半胱氨酸,无法结合Zn2+,因此PGLYRP-1,PGLYRP-3,PGLYRP-4只有杀菌活性[30, 43-44]。后两者形成的异二聚体PGLYRP3:4也同样有高效的杀菌作用[44]。研究表明,鱼类PGRPs同一分子往往兼具酰胺酶和杀菌两种特性[12, 26]。除了酰胺酶活性以外,岩鱼PGRP-L1和PGRP-L2还具有广谱杀菌功能[12]。因此猜测PGRPs的杀菌机制与其自身的酰胺酶活性可能存在某种关联,但目前还没有相关的实验证实这一猜想。斑马鱼PGRPs不仅能有效杀灭革兰氏阳性菌,对革兰氏阴性菌也表现出不同程度的杀菌作用。然而,美国红鱼PGLYRP2只对革兰氏阳性菌有很强的杀灭作用[13]。鱼类PGRPs在抗菌免疫中表现各异,即使同一物种的不同PGRPs分子的作用也不尽相同,至于其作用机制还有待进一步研究。
4.4 免疫调节PGRPs在免疫反应中发挥着重要作用,包括免疫调节过程[31, 42, 45]。除了识别病原体,PGRPs还通过调节机体免疫以便能够及时有效地清除入侵的病原体。研究表明,并非所有的PGRPs都有很强的杀菌作用。例如,半滑舌鳎PGRP-SC2并非是直接杀死细菌的效应物,而是通过提高细胞的吞噬能力来抑制机体内细菌的定植与传播[23]。与此类似,罗非鱼PGRP-SC2并未表现出明显的杀菌作用,却能显著降低靶器官内无乳链球菌的载量,说明其在抵抗无乳链球菌感染过程中扮演着重要角色[24]。
起初认为PGRPs是一个简单的效应分子,随着研究的深入,发现昆虫PGRPs能够激活Toll或IMD信号传导通路或诱导蛋白水解级联反应,从而提高抗菌免疫反应[45]。研究表明,鱼类PGRPs不仅是典型的模式识别受体,还可介导细胞内的Toll、MAPK、JAK/STAT和NF-KB多条信号通路,从而调节下游免疫相关基因的表达。由于昆虫PGRPs的水解肽聚糖的酰胺酶活性,可以防止机体出现过度炎症反应,因此推测哺乳动物PGRPs也有抗炎效应[42, 46-47]。然而,与预期结果相反,实验表明哺乳动物PGLYRP2在PGN感染机体过程中发挥了促炎效应[48]。为探究鱼类PGRPs的功能,Jang等[20]通过构建酰胺酶失活突变体,发现虹鳟PGRP-L1能够抑制IL-1β和TNF-a等下游炎性因子的表达,说明虹鳟PGRP-L1发挥了抗炎功能,且该调节机制与其酰胺酶活性无关。此外,鱼类PGRPs能促进穿孔素、抗菌肽的分泌,从而在机体的抗菌免疫反应中发挥协同效应[16]。
选择性剪切是单个基因剪切合成多功能信使RNA的过程,是真核生物中普遍存在的一种现象,会影响免疫系统中关键蛋白的结构和功能[49]。草鱼PGRP6的不同剪切体虽然都能抑制体内细菌的增长,但其作用机制也各不相同。PGRP6a,PGRP6b和PGRP6c能诱导铁调素和G-溶菌酶的表达,而草鱼PGRP6和PGRP6d却无此功能。另外,4个剪切体中只有PGRP6c能够影响穿孔素的分泌,说明在抑菌活性的基础上,草鱼PGRP6的不同剪切体还有功能特异性[16]。
5 展望PGRPs在机体先天性免疫过程中发挥着重要作用,目前有关鱼类PGRPs的研究已经取得了一定的进展,然而对其作用机制的研究还不够深入。PGRP-L的N端几乎占据整个分子的2/3,该段蛋白的结构差异较大的原因是什么,以及发挥的作用是怎样的;PGRPs的杀菌作用与酰胺酶活性是否存在一定的联系。以往的报道发现,PGRPs能够抵御真菌的感染,且软体动物PGRPs对寄生虫刺激也有一定的防御作用,那么鱼类PGRPs是否也有类似的功能。
免疫信号通路是一个十分复杂的网络。PGRPs作为分泌蛋白,却能显著抑制NF-kB活性,因此,推测细胞内可能存在某种表面受体来协助PGRP完成信号的转导,至于受体是什么又是如何发挥作用的,就不得而知了。研究表明,虹鳟PGRP-L1和核苷酸结合寡聚化结构域(Nucleotide-binding oligomerization domains,NODs)在抗菌免疫应答中存在相互依赖,并能够调节部分炎症因子的表达,但对IL-10的表达却没有任何影响[50]。那么PGRPs与其他免疫信号是否也有一定的关联;又是如何影响下游信号转导,以上问题都需要进一步的研究探索。
| [1] |
Yoshida H, Kinoshita K, Ashida M. Purification of a peptidoglycan recognition protein from hemolymph of the silkworm, Bombyx mori[J]. Journal of Biological Chemistry, 1996, 271(23): 13854-13860. DOI:10.1074/jbc.271.23.13854 |
| [2] |
韦嵩, 宋晓玲, 李赟, 等. 肽聚糖识别蛋白研究进展[J]. 动物医学进展, 2007, 28(1): 61-64. DOI:10.3969/j.issn.1007-5038.2007.01.017 |
| [3] |
Dziarski R, Gupta D. The peptidoglycan recognition proteins(PGRPs)[J]. Genome Biology, 2006, 7(8): 232-232. DOI:10.1186/gb-2006-7-8-232 |
| [4] |
Kang D, Liu G, Lundstrom A, et al. A peptidoglycan recognition protein in innate immunity conserved from insects to mammals[J]. Proceedings of the National Academy of Sciences of USA, 1998, 95(17): 10078-10082. DOI:10.1073/pnas.95.17.10078 |
| [5] |
Liu C, Z Xu, Gupta D, et al. Peptidoglycan recognition proteins:a novel family of four human innate immunity pattern recognition molecules[J]. Journal of Biological Chemistry, 2001, 276(37): 34686-34694. DOI:10.1074/jbc.M105566200 |
| [6] |
Dziarski R, GuptaD. Mammalian PGRPs:novel antibacterial proteins[J]. Cell Microbiology, 2006, 8(7): 1059-1069. DOI:10.1111/cmi.2006.8.issue-7 |
| [7] |
Werner T, Liu G, Kang D, et al. A family of peptidoglycan recognition proteins in the fruit fly Drosophila melanogaster[J]. Proceedings of the National Academy of Sciences of USA, 2000, 97(25): 13772-13777. DOI:10.1073/pnas.97.25.13772 |
| [8] |
Persson C, Oldenvi S, Steiner H. Peptidoglycan recognition protein LF:a negative regulator of Drosophila immunity[J]. Insect Biochemistry Molecular Biology, 2007, 37(12): 1309-1316. DOI:10.1016/j.ibmb.2007.08.003 |
| [9] |
Chang MX, Nie P, Wei Ll. Short and long peptidoglycan recognition proteins(PGRPs)in zebrafish, with findings of multiple PGRP homologs in teleost fish[J]. Molecular Immunology, 2007, 44(11): 3005-3023. DOI:10.1016/j.molimm.2006.12.029 |
| [10] |
Chang MX, Nie P. RNAi suppression of zebrafish peptidoglycan recognition protein 6(zfPGRP6)mediated differently expressed gene involved in Toll-like receptor signalling pathway and caused increased susceptibility to Flavobacterium columnare[J]. Veterinary Immunology and Immunopathology, 2008, 124(3-4): 295-301. DOI:10.1016/j.vetimm.2008.04.003 |
| [11] |
Chang MX, Wang YP, Nie P. Zebrafish peptidoglycan recognition protein SC(zfPGRP-SC)mediates multiple intracellular signaling pathways[J]. Fish and Shellfish Immunology, 2009, 26(2): 264-274. DOI:10.1016/j.fsi.2008.11.007 |
| [12] |
Kim MY, Jang JH, Lee JW, et al. Molecular cloning and characterization of peptidoglycan recognition proteins from the rockfish, Sebastes schlegeli[J]. Fish and Shellfish Immunology, 2010, 28(4): 632-639. DOI:10.1016/j.fsi.2009.12.023 |
| [13] |
Li MF, Zhang M, Wang CL, et al. A peptidoglycan recognition protein from Sciaenops ocellatus is a zinc amidase anda bactericide with a substrate range limited to Gram-positive bacteria[J]. Fish and Shellfish Immunology, 2012, 32(2): 322-330. DOI:10.1016/j.fsi.2011.11.024 |
| [14] |
Li JH, Chang MX, Xue NN, et al. Functional characterization of a short peptidoglycan recognition protein, PGRP5 in grass carp Ctenopharyngodon idella[J]. Fish and Shellfish Immunology, 2013, 35(2): 221-230. DOI:10.1016/j.fsi.2013.04.025 |
| [15] |
Li JH, Yu ZL, Xue NN, Zou PF, et al. Molecular cloning and functional characterization of peptidoglycan recognition protein 6 in grass carp Ctenopharyngodon idella[J]. Developmental and Comparative Immunology, 2014, 42(2): 244-255. DOI:10.1016/j.dci.2013.09.014 |
| [16] |
Yu ZL, Li JH, Xue NN, et al. Expression and functional character-ization of PGRP6 splice variants in grass carp Ctenopharyngodon idella[J]. Developmental and Comparative Immunology, 2014, 47(2): 264-274. DOI:10.1016/j.dci.2014.08.006 |
| [17] |
Sun LY, Liu SK, Wang RJ, et al. Pathogen recognition receptors in channel catfish:Ⅳ Identification, phylogeny and expression analysis of peptidoglycan recognition proteins[J]. Developmental and Comparative Immunology, 2014, 46(2): 291-299. DOI:10.1016/j.dci.2014.04.018 |
| [18] |
Mao Y, Wang J, Zhang ZW, et al. Cloning, mRNA expression, and recombinant expression of peptidoglycan recognition protein Ⅱ gene from large yellow croaker(Pseudosciaena crocea)[J]. Mol Biol Rep, 2010, 37(8): 3897-3908. DOI:10.1007/s11033-010-0046-x |
| [19] |
隗黎丽, 刘毅, 吴华东, 等. 黄颡鱼-基因克隆及其对爱德华氏菌的反应[J]. 水生生物学报, 2016, 40(1): 42-49. |
| [20] |
Jang JH, Kim H, Cho JH. Rainbow trout peptidoglycan recognition protein has an anti-inflammatory function in liver cells[J]. Fish and Shellfish Immunology, 2013, 35(6): 1838-1847. DOI:10.1016/j.fsi.2013.09.015 |
| [21] |
Jang JH, Kim H, Cho JH. Molecular cloning and functional characterization of peptidoglycan recognition protein OmPGRP-L2 from the rainbow trout, Oncorhynchus mykiss[J]. Veterinary Immunology and Immunopathology, 2017, 192(10): 28-32. |
| [22] |
Zhang LN, Gao CB, Liu FQ, et al. Characterization and expression analysis of a peptidoglycan recognition protein gene, SmPGRP2 in mucosal tissues of turbot(Scophthalmus maximus L.)following bacterial challenge[J]. Fish and Shellfish Immunology, 2016, 56(9): 367-373. |
| [23] |
Sun QL, Sun L. A short-type peptidoglycan recognition protein from tongue sole(Cynoglossus semilaevis)promotes phagocytosis and defense against bacterial infection[J]. Fish and Shellfish Immunology, 2015, 47(1): 313-320. DOI:10.1016/j.fsi.2015.09.021 |
| [24] |
Gan Z, Chen SN, Hou J, et al. Molecular and functional characterization of peptidoglycan recognition protein SC2(PGRP-SC2)from Nile tilapia(Oreochromis niloticus)involved in the immune response to Streptococcus agalactiae[J]. Fish and Shellfish Immunology, 2016, 54(7): 1-10. |
| [25] |
Nothwehr SF, Gordon JI. Targeting of proteins into the eukaryotic secretory pathway:Signal peptide structure function relationships[J]. Bioessays, 1990, 12(10): 479-484. DOI:10.1002/(ISSN)1521-1878 |
| [26] |
Li X, Wang S, Qi J, et al. Zebrafish peptidoglycan recognition proteins are bactericidal amidases essential for defense against bacterial infections[J]. Immunity, 2007, 27(3): 518-529. DOI:10.1016/j.immuni.2007.07.020 |
| [27] |
Dziarski R. Peptidoglycan recognition proteins(PGRPs)[J]. Molecular Immunology, 2004, 40(12): 877-886. DOI:10.1016/j.molimm.2003.10.011 |
| [28] |
Guan R, Mariuzza RA. Peptidoglycan recognition proteins of the innate immune system[J]. Trends in Microbiology, 2007, 15(3): 127-134. DOI:10.1016/j.tim.2007.01.006 |
| [29] |
Michel T, Reichhart JM, Hoffmann JA, et al. Drosophila Toll is activated by Gram-positive bacteria through a circulating peptidoglycan recognition protein[J]. Nature, 2001, 414(6865): 756-759. DOI:10.1038/414756a |
| [30] |
Wang ZM, Li X, Cocklin RR, et al. Human peptidoglycan recognition protein-L is an N-acetylmuramoyl-L-alanine amidase[J]. The Journal of Biological Chemistry, 2003, 278(49): 49044-49052. DOI:10.1074/jbc.M307758200 |
| [31] |
Kim MS, Byun M, Oh BH. Crystal structure of peptidoglycan recognition protein LB from Drosophila melanogaster[J]. Nature Immunology, 2003, 4(8): 787-793. DOI:10.1038/ni952 |
| [32] |
Chang CI, Pili-Floury S, Herve M, et al. A Drosophila pattern recognition receptor contains a peptidoglycan docking groove and unusual L, D-carboxypeptidase activity[J]. PLoS Biology, 2004, 2(9): E277. DOI:10.1371/journal.pbio.0020277 |
| [33] |
Reiser JB, Teyton L, Wilson IA, et al. Crystal structure of the Drosophila peptidoglycan recognition protein(PGRP)-SA at 156 A resolution[J]. Journal of Molecular Biology, 2004, 340(4): 909-917. DOI:10.1016/j.jmb.2004.04.077 |
| [34] |
Guan R, Wang Q, Sundberg EJ, et al. Crystal structure of human peptidoglycan recognition protein S(PGRP-S)at 170 A resolution[J]. Journal of Molecular Biology, 2005, 347(4): 683-691. DOI:10.1016/j.jmb.2005.01.070 |
| [35] |
Montano AM, Tsujino F, Takahata N, et al. Evolutionary origin of peptidoglycan recognition proteins in vertebrate innate immune system[J]. BMC Evolutionary Biology, 2011, 11(3): 79-79. |
| [36] |
Royet J, Dziarski R. Peptidoglycan recognition proteins:pleiotropic sensors and effectors of antimicrobial defences[J]. Nature Reviews. Microbiology, 2007, 5(4): 264-277. DOI:10.1038/nrmicro1620 |
| [37] |
Guan R, Roychowdhury A, Ember B, et al. Structural basis for peptidoglycan binding by peptidoglycan recognition proteins[J]. Proceedings of the national academy of sciences of USA, 2004, 101(49): 17168-17173. DOI:10.1073/pnas.0407856101 |
| [38] |
Guan R, Brown PH, Swaminathan CP, et al. Crystal structure of human peptidoglycan recognition protein Ⅰ alpha bound to a muramyl pentapeptide from Gram-positive bacteria[J]. Protein Science, 2006, 15(5): 1199-1206. DOI:10.1110/(ISSN)1469-896X |
| [39] |
Lu X, Wang M, Qi J, et al. Peptidoglycan recognition proteins are a new class of human bactericidal proteins[J]. Journal of Biological Chemistry, 2006, 281(9): 5895-5907. DOI:10.1074/jbc.M511631200 |
| [40] |
Liu C, Gelius E, Liu G, et al. Mammalian peptidoglycan recognition protein binds peptidoglycan with high affinity, is expressed in neutrophils, and inhibits bacterial growth[J]. Journal of Biological Chemistry, 2000, 275(32): 24490-24499. DOI:10.1074/jbc.M001239200 |
| [41] |
Tydell CC, Yuan J, Tran P, et al. Bovine peptidoglycan recognition protein-S:antimicrobial activity, localization, secretion, and binding properties[J]. Journal of Immunology, 2006, 176(2): 1154-1162. DOI:10.4049/jimmunol.176.2.1154 |
| [42] |
Mellroth P, Karlsson J, Steiner H. A scavenger function for a Drosophila peptidoglycan recognition protein[J]. Journal of Biological Chemistry, 2003, 278(9): 7059-7064. DOI:10.1074/jbc.M208900200 |
| [43] |
Gelius E, Persson C, Karlsson J, et al. A mammalian peptidoglycanrecognition protein with N-acetylmuramoyl-L-alanine amidase activity[J]. Biochem Biophys Res Commun, 2003, 306(4): 988-994. DOI:10.1016/S0006-291X(03)01096-9 |
| [44] |
Lu X, Wang M, Qi J, et al. Peptidoglycan recognition proteins are a new class of human bactericidal proteins[J]. Journal of Biological Chemistry, 2006, 281(9): 5895-5907. DOI:10.1074/jbc.M511631200 |
| [45] |
Bischoff V, Vignal C, Duvic B, et al. Down regulation of the Drosophila immune response by peptidoglycan-recognition proteins SC1 and SC2[J]. PLoS Pathogens, 2006, 2(2): 14-14. DOI:10.1371/journal.ppat.0020014 |
| [46] |
Kurata S. Peptidoglycan recognition proteins in Drosophila immunity[J]. Developmental and Comparative Immunology, 2014, 42(1): 36-41. DOI:10.1016/j.dci.2013.06.006 |
| [47] |
Zaidman-Remy A, Herve M, Poidevin M, et al. The Drosophila amidase PGRP-LB modulates the immune response to bacterial infection[J]. Immunity, 2006, 24(4): 463-473. DOI:10.1016/j.immuni.2006.02.012 |
| [48] |
Saha S, Qi J, Wang S, et al. PGLYRP-2 and Nod2 are both required for peptidoglycan-induced arthritis and local inflammation[J]. Cell Host and Microbe, 2009, 5(2): 137-150. DOI:10.1016/j.chom.2008.12.010 |
| [49] |
Nilsen TW, Graveley BR. Expansion of the eukaryotic proteome by alternative splicing[J]. Nature, 2010, 463(7280): 457-463. DOI:10.1038/nature08909 |
| [50] |
Jang JH, Kim H, Jang MJ. PGRP negatively regulates NOD-mediated cytokine production in rainbow trout liver cells[J]. Scientific Reports, 2016, 6: 39344. DOI:10.1038/srep39344 |