2. 河北省科学院生物研究所,石家庄 050000
2. Institute of Biology, Hebei Academy of Sciences, Shijiazhuang 050000
以往,工业生产中主要利用化学催化剂进行工业产品的生产,对条件要求苛刻,且污染环境严重。生物催化剂是具有催化作用的游离或固定化细胞和游离或固定化酶的统称。由于其反应条件温和,绿色、安全,因此被广泛应用于工业、农业、医药和能源等领域。目前,生物催化剂的市场价值约40亿美元,并且酶类的使用范围仍在不断的扩大[1],其中腈水解酶、蛋白酶、脂肪酶[2]、转氨酶和糖基水解酶的研究受到了广泛的关注。随着科技的进步,工业生产中对酶的催化性能及耐受性提出了更高的要求。这就需要我们开发新的生物技术去寻求新型催化剂。
传统的挖掘新型生物催化剂的方法主要基于可培养微生物的功能筛选,然后对阳性菌株进行序列分析,功能验证。人们利用传统的挖掘方法发现了淀粉酶[3]和酯酶[4]等。其中微生物作为催化剂的来源[5]在该方法中起着核心作用。据统计,地球上微生物细胞的总数达1030[6]。但是,大多数微生物存在于其天然微生物群落环境中,与其他微生物相互依赖而生存,很难在实验室条件下进行纯培养[7]。这极大地限制了传统方法挖掘新型酶的效率。宏基因组学通过直接从环境样品中提取全部微生物的DNA,构建宏基因组文库,利用基因组学的研究策略研究环境样品所包含的全部微生物的遗传组成及其群落功能[8],避免了微生物的分离和实验室培养,成为探索生物学和不同自然环境中分子生物多样性的有力工具。而宏基因组学基因挖掘策略中也存在一些限制,可通过合成生物学来弥补。通过这两种方法的结合使用,可以提高挖掘新型生物催化剂的效率,而宏基因组学基因挖掘策略中也存在一些限制,可通过合成生物学来弥补。通过这两种方法的结合使用,可以提高挖掘新型生物催化剂的效率.本文综述了宏基因组学方法挖掘新型生物催化剂的最新研究进展,并对利用合成生物学方法改进宏基因组筛选新型生物催化剂的效率进行了讨论。从而探索提高挖掘新型生物催化剂的方法。
1 宏基因组学挖掘新型生物催化剂宏基因组学挖掘新型生物催化剂的基本流程(图 1):(1)采集样品:微生物的生存环境与其所表达的酶的耐受性及底物特异性具有一致性,可通过所要筛选的生物催化剂的特性寻求合适环境中的微生物群体进行采样。目前微生物样品的来源大致可归结为5大类:陆地环境、水生环境、寄生环境、人工环境及其他特殊环境。(2)提取特定环境中的基因组DNA:一般可分为直接提取法和间接提取法。可根据样品来源微生物的特性及目的催化剂的特性进行优化及选择来提取环境总DNA。(3)构建宏基因组文库:所提取的基因组DNA需要连接到合适的载体中并转化宿主菌。(4)筛选阳性克隆子:根据目的基因的活性或其序列特点进行宏基因组文库的筛选。(5)目的基因的亚克隆和表达:将筛选到的目的基因连入合适的表达载体和宿主菌中表达。(6)目的催化剂的生化特性分析[9]:诱导目的基因表达,纯化目的蛋白,并作生化特性分析。[10]。近年来,人们已经成功利用宏基因组学发现在工业应用中具有潜在利用价值的酶[5, 11]。宏基因组学挖掘新型生物催化剂包括基于活性的筛选和基于序列的筛选。
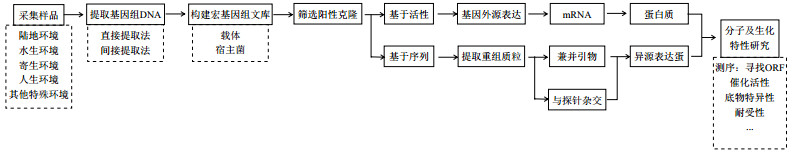
|
| 图 1 宏基因组学挖掘新型生物催化剂流程图 |
基于活性的筛选(Activity-based screening),也称功能驱动筛选(Function-driven screening),是指通过活性检测手段从基因组文库中获得具有特殊活性的克隆方法[9]。基于活性的筛选是在没有任何核苷酸、核糖核苷酸或蛋白质序列分析的情况下,直接对基因潜在功能的筛选。因此,为了有效地从潜在基因(含有潜在功能基因片段的克隆或具有相应表型的菌株)中发现新型催化剂,需要建立合适的功能筛选方法。这些方法最好简单易行,如通过肉眼直接观察或用分光光度计检测来鉴别阳性克隆[12]。通常在固体培养基中添加特殊底物,经过微生物代谢,引起比色变化,透明圈、抑菌圈或生长表型的变化,从而来筛选阳性克隆。本实验室在筛选能将异丁酸合成β-羟基异丁酸菌株时,将菌株培养于以异丁酸为唯一碳源的培养基中,能进行生长的为阳性克隆。在添加特殊底物的液体培养基中,可将细胞或细胞裂解液置于96孔板中,通过分光光度计测量其吸光度的变化来鉴别阳性克隆。最后,利用分子生物学工具和生物体内蛋白调控的知识来挖掘新型酶。近年来有很多利用这种方法成功挖掘具有新型功能的生物催化剂[13-15]的例子。
脂肪酶和酯酶作为生物催化剂,被广泛应用于纺织、食品、洗衣、造纸和纸浆工业、生物柴油生产和精细化学品的合成等领域。Chu等[16]从中国南海取样,构建宏基因组文库。以1%三丁酸甘油酯和1%阿拉伯树胶为底物,筛选出周围带透明圈的阳性菌落。进一步用BamHⅠ内切酶随机消化阳性菌落质粒DNA,构建带有随机片段的克隆子,分离出两种新型酯酶estA和estB。同样,李云娣等[17]在含有1%三丁酸甘油酯的培养平板上,37℃培养3-5 d后观察产生透明圈的阳性克隆子,发现了酯酶ESTYN1[17]。Wierzbicka-Woś等[18]筛选出新型冷活性糖苷水解酶(BglMKg)。在这项研究中,在固体培养基中添加X-gal,阳性克隆将会变为深蓝色,从而筛选出具有新型冷活性糖苷水解酶的菌落。另外作者还用不同的发色底物检验了酶的特殊活性。
如前所述,基于活性筛选的方法已经建立并成功运用,但有些种类的催化剂还没找到合适的功能筛选方法。因此,利用工业相关底物去寻求理想新型(底物特异性、手性、酶活稳定性)催化剂的智能筛选方法仍是我们现在面临的巨大挑战。但这并不是说通用底物在功能筛选中没有作用。解决这一问题的关键是全面重新审视并诠释新型酶的特征,并选择合适的生化检测实验来验证阳性克隆。
1.2 基于序列的筛选基于序列的筛选是以序列相似性为基础,根据已知相关功能基因的保守序列设计探针或PCR引物[19],然后通过杂交或PCR扩增来筛选目标克隆[9],这两种方法都不需要异源宿主细胞中的蛋白表达[20]。当然,其随后的功能表达仍需面临蛋白表达的问题,但是在筛选阶段可以避免这些限制,降低了由异源表达效率低而造成的假阴性。近年来利用此种方法已成功挖掘许多新型催化剂。
梁跃斌等[21]对CAZy数据库中193条细菌源α-L-鼠李糖苷酶进行氨基酸序列分析[22],根据保守氨基酸序列设计简并引物,对健康人体粪便宏基因组DNA[23]进行筛选,将获得的基因片段进行测序,进而通过原核表达获得新型α-L-鼠李糖苷酶基因。Jiang等[24]从北冰洋海水中取样,构建了一个质粒宏基因组文库,并筛选出新型延胡索酸酶基因fumF。另外,Wang等[25]在摩斯拉热液喷口处采样并对构建的宏基因组文库进行随机焦磷酸测序,筛选出糖苷水解酶(GH-57)。Fang等[26]通过保守区域-铜结合位点对中国南海微生物宏基因组文库进行分析,筛选出一长1.32 kb的细菌漆酶基因。
从2009年到2015年宏基因组测序项目从199个增长到607个,同时在GOLD网站上有22 455条非宏基因组研究条目。巨大的测序数据库为探索特定酶活性的基因奠定了基础。尽管如此,这种方法还是具有其局限性,尤其是新颖性。该方法依赖于数据库中已知的序列,仅限于同源酶的分析[27]。迄今为止,新发现的酶的氨基酸序列与数据库中已存在的序列同源性高于50%,一般在70%-90%之间,脂肪类酶除外[28]。因此,同源性低于这个范围的新型酶需要利用其他方法进行筛选,如可采用功能筛选或提高预测软件识别区域结构的能力。
随着注解多样性的增加,利用同源性搜索,我们可以挖掘更具新颖性的基因序列。不管它是否转录为具有新功能的酶还是只具备之前被定义的活性,这种预筛选都是有意义的。虽然目前的筛选方法可以分离新序列,尤其是脂肪酶类序列,但并没有表现出筛选新颖功能酶的用途。基因检测的发展和更复杂的注解程序可以实现这一功能。或者,从宏基因组样品中勘测到的具有新颖性的序列可以作为接下来分子操作的一个基础。
1.3 宏基因组学挖掘新型生物催化剂面临的挑战如Handelsman等[29]所述,宏基因组可定义为“自然界中存在的所有微生物的基因组”,指的是从自然环境中可以直接得到的基因序列。虽然宏基因组学是不依赖于生物培养的获取有价值基因资源的有效方法[16]。但其仍然存在局限性,其具体主要归纳为以下3个方面。首先,挖掘的新型基因需要在异源宿主细胞中进行表达,而各个菌属的菌株在转录、翻译等过程中都具有偏好性(RNA聚合酶对启动子的识别,密码子、核糖体识别等),这就需要提高基因在异源宿主菌中表达目的酶的能力。其次,载体与宿主菌常存在不兼容问题,需要开发新的遗传工具以改善宏基因组文库的构建,使其适合不同宿主的筛选。最后,需要继续完善筛选方法,从而提高挖掘新型催化剂的效率。以自然环境为资源,将迎来更强大有效的探索庞大基因组潜能工具的热潮。
2 用合成生物学来改进宏基因组筛选策略合成生物学在现代生物学基础上融入了工程学思想。它将化学或生物化学合成的DNA生物元件构建为标准化的元件库,并利用这些元件组装成具有全新特征或增强了性能的生物模块、网络、体系乃至生物体(细胞),以满足人类的需求[30]。其中元件指具有特定功能的DNA片段(如基因),模块指多个元件的组合[31]。它主要包括两种技术线路:(1)利用标准化元件设计和建造模块、体系。(2)在天然的代谢网路基础上,利用标准化原件对其进行改造。合成生物学使得基因网路能像电路一样,通过控制各个元件,来调控功能的表达。利用此特点可以很好的解决上面提出的宏基因组筛选策略中遇到的3类问题。
2.1 合成生物学改善新型催化剂的异源表达通过宏基因组学筛选出的阳性克隆子,需要进行目的基因的亚克隆、表达、纯化,才能对目的催化剂的生化特性进行分析。由于目的基因的来源菌株与异源表达宿主菌株系统发育的不同,可能导致异源基因表达失败或低表达,造成宏基因组文库中所需酶回收率低[11, 32]。合成生物学可以解决这一难题。转录初始阶段,由于原核生物中RNA聚合酶对启动子的识别具有很大的偏好性[33],使得宿主体内的RNA聚合酶与宏基因组中的启动子亲和力低成为蛋白异源表达的主要限制。蛋白质异源表达理想的的宿主菌应该具有识别广谱启动子的能力。可以通过插入特定启动子及其σ因子等元件在宿主菌中共表达来实现[34-35]。另外可以利用T7 RNAP表达模块系统实现异源蛋白的高效表达[36],而且这种方法可以使受复杂信号转导机制的基因仅仅通过添加单一诱导物如异丙基-β-D-硫代吡喃半乳糖苷而被诱导表达。同理,在mRNA翻译过程中,核糖体结合位点的差异性也会导致蛋白表达水平的降低[37]。因此同时也需共表达mRNA识别步骤中的蛋白,从而增强宿主菌表达外源基因的能力[38]。然而这些都只是为了提高目的蛋白的表达水平,表达出来蛋白是否有活性是开发新型催化剂的另一个关键因素。表达出来的蛋白需要进行复杂折叠或通过额外的处理步骤(如切割、分泌或肽修饰)形成正确的构象[39]。例如,Ferrer等[40]在大肠杆菌中将目的蛋白与分子伴侣[41]共表达,使得异源蛋白正确的折叠。
在提高异源宿主能力的方法中基因编辑尤其受到了关注[42]。因为在标准的试验条件下目的基因并不是宿主菌(如大肠杆菌)生长所必需的[43],兼容这些基因并大量表达其编码的蛋白意味着巨大的能量消耗和代谢负担[44]。删除非必需基因,减小基因组大小,提高细菌代谢活力,可以提高异源蛋白的表达水平[45]。拥有最小化基因组的细菌,由于其超强的表达能力,成为筛选宏基因组文库最理想的宿主。
2.2 合成生物学改善新型催化剂的载体除了基因工程中最常用的宿主菌大肠杆菌外,在宏基因组筛选中人们还尝试过其他菌株作为宿主菌来提高挖掘新酶的效率[46-47]。而在宿主细胞中的表达,需将目的基因整合入载体并导入宿主菌中,进行稳定遗传及表达,这就需要建立一个通用的、标准化、适合多种宿主菌的广谱工具(载体),以促进各种宿主菌中宏基因组文库的筛选。可以利用合成生物学,重新组装元件,构建新的基因网路[48-49],开发出适合多种宿主细胞的载体。在这些适合于多种宿主的工具中,pSEVA载体因可以广泛应用于100多种不同的细菌种类而受到广泛关注[50]。可以此系统为出发点,设计基因网路,开发新的遗传工具,从而更高效的筛选不同种类细菌的DNA。
2.3 合成生物学改善宏基因组筛选方法目前常用来筛选宏基因组文库中新型催化活性的方法常可以用肉眼观测到。在固体培养基中添加特殊底物,阳性菌落周围底物颜色发生变化[51-52]或底物被降解形成透明光圈[53]。劳动密集型筛选方法如菌落PCR也用于新型催化剂的挖掘[54]。利用合成生物学及生物学知识,通过合理整合调节网路的各个部分[55],设计生物传感器,可以为酶活性的筛选提供更好的方案[56]。生物传感器是将工程基因网路与已报道基因(lacZ、绿色荧光蛋白或萤光素酶等)的整合,通过简单的表观现象筛选阳性克隆,便于生物体内鉴别酶活性的一种工具[57]。这种方法减少了筛选过程中大量的重复劳动,极大的提高了宏基因组文库的筛选效率。合成生物学也可以通过生物合成途径调节酶分子水平。Zelcbuch等[37]通过与核糖体结合位点结合配对基因来调节酶表达的水平,引起蛋白种类几个数量级的变化,这表明工程代谢途径受酶水平的精确调控。这些例子表明可以通过合成生物学方法,关联各调控因子(如启动子和调节子),设计出具有更好性能的酶的基因网路。以基因工程网路中各调控元件为基础重新设计调节系统,并将其应用于挖掘新型催化剂策略中,将会极大提高挖掘新型催化剂的速率。
3 展望新型催化剂在工业中已受到广泛应用,宏基因组筛选策略挖掘新型催化剂将会推动下一代工业流程如生物药物的合成并开创新的机遇。这就要求生物技术的全面综合发展。从基因序列角度来看,随着宏基因组多样性的增加及基因分离、测序技术的发展,宏基因组筛选策略已经开始着手挖掘序列同源性较低的基因,并将推动基于序列的新型催化剂的挖掘。可以将所有原核微生物的宏基因组进行测序[58],为宏基因组学法挖掘新型生物催化剂奠定基础,并为生物技术的发展提供数据参考。
构建工程基因网路可以将合成生物学与编程相结合,它包括设计、建模、实施及调试等环节。例如开发一些电子设备分析生物系统,在这里细胞可以被重新编程,从而以更高的效率执行新的任务[59-60]。设计方面着重规划和构建新的基因网路以便于工业应用[61]。建模包括所提出的基因网路的计算机模拟,评估网路性能与能力并为其选择合适的分子组分做指导[62]。实施部分包括根据组装标准,对适当的组分例如启动子、调节元件、终止子、酶和转运蛋白等进行机械组装[48]。最后,调试步骤需要测试和验证生物体内代谢通络,并且改善生物系统在应激情况中表现出来的对生产不利的特性[63-65]。目前已经有设计并实施成功的生物通路的例子[64, 66-67],并且近年来该领域的发展速度惊人[65, 68]。
将宏基因组筛选策略[69]中各种方法结合起来,并与生物信息学测序能力,信息分析能力[70-73],智能筛选技术和合成技术[73]协同作用将会迎来生物学技术的巨大飞跃。为了实现这个目标并使其效能最大化,需要各领域的科学家们的协调配合。信息学、分子生物学、化学、药物设计、物理学、工程学及系统生物学等学科学术与工业应用的融合发展,将会更好的应对未来的挑战,为生物催化剂及生物产物的发现提供广阔的平台。
| [1] |
Guazzaroni ME, Silva-Rocha R, Ward RJ. Synthetic biology approaches to improve biocatalyst identification in metagenomic library screening[J]. Microbial Biotechnology, 2015, 8(1): 52-64. DOI:10.1111/1751-7915.12146 |
| [2] |
d'Errico C, Borjesson J, Ding H., et al. Improved biomass degradation using fungal glucuronoyl-esterases-hydrolysis of natural corn fiber substrate[J]. Journal of Biotechnology, 2016, 219: 117-123. DOI:10.1016/j.jbiotec.2015.12.024 |
| [3] |
Zhang JW, Zeng RY. Purification and characterization of a cold-adapted alpha-amylase produced by Nocardiopsis sp. 7326 isolated from Prydz Bay, Antarctic[J]. Marine Biotechnology, 2008, 10(1): 75-82. DOI:10.1007/s10126-007-9035-z |
| [4] |
李正阳, 戎振, 王昭, 等. 来源于海洋细菌Altererythrobacterl-uteolus SW109T的新型酯酶E29的克隆表达及其酶学性质[J]. 微生物学通报, 2016(5): 1051-1059. |
| [5] |
Fernandez-Arrojo L, Guazzaroni ME, Lopez-Cortes N, et al. Metagenomic era for biocatalyst identification[J]. Current Opinion in Biotechnology, 2010, 21(6): 725-733. DOI:10.1016/j.copbio.2010.09.006 |
| [6] |
Turnbaugh PJ, Gordon JI. An invitation to the marriage of metagenomics and metabolomics[J]. Cell, 2008, 134(5): 708-713. DOI:10.1016/j.cell.2008.08.025 |
| [7] |
Tedesco P, Pascale DD. Marine metagenomics, a valuable tool for enzymes and bioactive compounds discovery[J]. Frontiers in Marine Science, 2014, 1(1): 38. |
| [8] |
贺纪正, 张丽梅, 沈菊培, 等. 宏基因组学(Metagenomics)的研究现状和发展趋势[J]. 环境科学学报, 2008, 28(2): 209-218. DOI:10.3321/j.issn:0253-2468.2008.02.001 |
| [9] |
王魁, 汪思迪, 黄睿, 等. 宏基因组学挖掘新型生物催化剂的研究进展[J]. 生物工程学报, 2012, 28(4): 420-431. |
| [10] |
张光亚, 方柏山. 宏基因组——生物催化剂的新来源[J]. 生命的化学, 2005, 25(4): 278-281. DOI:10.3969/j.issn.1000-1336.2005.04.002 |
| [11] |
Lorenz P, Eck J. Metagenomics and industrial applications[J]. Nature Reviews Microbiology, 2005, 3(6): 510-516. DOI:10.1038/nrmicro1161 |
| [12] |
Kennedy J, O'Leary ND, Kiran GS, et al. Functional metagenomic strategies for the discovery of novel enzymes and biosurfactants with biotechnological applications from marine ecosystems[J]. Journal of Applied Microbiology, 2011, 111(4): 787-799. DOI:10.1111/jam.2011.111.issue-4 |
| [13] |
Mohamed YM, Ghazy MA, Sayed A, et al. Isolation and characterization of a heavy metal-resistant, thermophilic esterase from a Red Sea brine pool[J]. Scientific Reports, 2013, 3(7477): 3358. |
| [14] |
Fu J, Leiros HK, De PD, et al. Functional and structural studies of a novel cold-adapted esterase from an Arctic intertidal metagenomic library[J]. Applied Microbiology and Biotechnology, 2013, 97(9): 3965-3978. DOI:10.1007/s00253-012-4276-9 |
| [15] |
Fang Z, Li J, Wang Q, et al. A novel esterase from a marine metagenomic library exhibiting salt tolerance ability[J]. Journal of Microbiology and Biotechnology, 2014, 24(6): 771-780. DOI:10.4014/jmb.1311.11071 |
| [16] |
Chu X, He H, Guo C, et al. Identification of two novel esterases from a marine metagenomic library derived from South China Sea[J]. Applied Microbiology and Biotechnology, 2008, 80(4): 615-625. DOI:10.1007/s00253-008-1566-3 |
| [17] |
李云娣, 曹明明, 顾昕琪, 等. 土壤宏基因组文库来源酯酶的鉴定与表征[J]. 微生物学通报, 2017, 44(6): 1255-1262. |
| [18] |
Wierzbicka-Wos A, Bartasun P, Cieslinski H, et al. Cloning and characterization of a novel cold-active glycoside hydrolase family 1 enzyme with beta-glucosidase, beta-fucosidase and beta-galactosidase activities[J]. BMC Biotechnology, 2013, 13(1): 1-12. DOI:10.1186/1472-6750-13-1 |
| [19] |
王海东, 胡忠. 利用宏基因组法筛选新几丁质酶基因[J]. 生物技术进展, 2016, 6(2): 119-124. DOI:10.3969/j.issn.2095-2341.2016.02.07 |
| [20] |
Piel J. Approaches to capturing and designing biologically active small molecules produced by uncultured microbes[J]. Annual Review of Microbiology, 2011, 65(1): 431-453. DOI:10.1146/annurev-micro-090110-102805 |
| [21] |
梁跃斌, 李彬春, 李艳琴. 基于宏基因组学方法挖掘新型α-L-鼠李糖苷酶资源[J]. 中国生物化学与分子生物学报, 2017(1): 66-72. |
| [22] |
McCall C, Xagoraraki I. Comparative study of sequence aligners for detecting antibiotic resistance in bacterial metagenomes[J]. Letters in Applied Microbiology, 2018, 66(3): 162-168. DOI:10.1111/lam.2018.66.issue-3 |
| [23] |
骞宇, 赵欣. 大鼠粪便中细菌基因组DNA提取方法的比较[J]. 食品工业科技, 2014(4): 166-169. |
| [24] |
Jiang C, Wu LL, Zhao GC, et al. Identification and characterization of a novel fumarase gene by metagenome expression cloning from marine microorganisms[J]. Microbial Cell Factories, 2010, 9: 91. DOI:10.1186/1475-2859-9-91 |
| [25] |
Wang H, Gong Y, Xie W, et al. Identification and characterization of a novel thermostable gh-57 gene from metagenomic fosmid library of the Juan de Fuca Ridge hydrothemal vent[J]. Applied Biochemistry and Biotechnology, 2011, 164(8): 1323-1338. DOI:10.1007/s12010-011-9215-1 |
| [26] |
Fang Z, Li T, Wang Q, et al. A bacterial laccase from marine microbial metagenome exhibiting chloride tolerance and dye decol-orization ability[J]. Applied Microbiology and Biotechnology, 2011, 89(4): 1103-1110. DOI:10.1007/s00253-010-2934-3 |
| [27] |
Cheng J, Romantsov T, Engel K, et al. Functional metagenomics reveals novel beta-galactosidases not predictable from gene sequences[J]. PLoS One, 2017, 12(3): e0172545. DOI:10.1371/journal.pone.0172545 |
| [28] |
Bell PJ, Sunna A, Gibbs MD, et al. Prospecting for novel lipase genes using PCR[J]. Microbiology, 2002, 148(8): 2283-2291. DOI:10.1099/00221287-148-8-2283 |
| [29] |
Handelsman J, Rondon MR, Brady SF, et al. Molecular biological access to the chemistry of unknown soil microbes: a new frontier for natural products[J]. Chemistry & Biology, 1998, 5(10): R245-R249. |
| [30] |
吕永坤, 堵国成, 陈坚, 等. 合成生物学技术研究进展[J]. 生物技术通报, 2015, 31(4): 134-148. |
| [31] |
武临专, 洪斌. 微生物药物合成生物学研究进展[J]. 药学学报, 2013, 48(2): 155-160. |
| [32] |
Handelsman J. Metagenomics: application of genomics to uncultured microorganisms[J]. Microbiology and Molecular Biology Reviews, 2004, 68(4): 669-685. DOI:10.1128/MMBR.68.4.669-685.2004 |
| [33] |
Gabor EM, Alkema WB, Janssen DB. Quantifying the accessibility of the metagenome by random expression cloning techniques[J]. Environmental Microbiology, 2004, 6(9): 879-886. DOI:10.1111/emi.2004.6.issue-9 |
| [34] |
Osterberg S, del Peso-Santos T, Shingler V. Regulation of alternative sigma factor use[J]. Annual Review of Microbiology, 2011, 65(1): 37-55. DOI:10.1146/annurev.micro.112408.134219 |
| [35] |
Rhodius VA, Segall-Shapiro TH, Sharon BD, et al. Design of orthogonal genetic switches based on a crosstalk map of sigmas, anti-sigmas, and promoters[J]. Molecular Systems Biology, 2013, 9(1): 702. |
| [36] |
Terron-Gonzalez L, Medina C, Limon-Mortes MC, et al. Heterologous viral expression systems in fosmid vectors increase the functional analysis potential of metagenomic libraries[J]. Scientific Reports, 2013, 3(4): 1107. |
| [37] |
Zelcbuch L, Antonovsky N, Bareven A, et al. Spanning high-dimensional expression space using ribosome-binding site combinatorics[J]. Nucleic Acids Research, 2013, 41(9): e98. DOI:10.1093/nar/gkt151 |
| [38] |
Uchiyama T, Miyazaki K. Functional metagenomics for enzyme discovery: challenges to efficient screening[J]. Current Opinion in Biotechnology, 2009, 20(6): 616-622. DOI:10.1016/j.copbio.2009.09.010 |
| [39] |
Bhat MK. Cellulases and related enzymes in biotechnology[J]. Biotechnology Advances, 2000, 18(5): 355-383. DOI:10.1016/S0734-9750(00)00041-0 |
| [40] |
Ferrer M, Chernikova TN, Timmis KN, et al. Expression of a temperature-sensitive esterase in a novel chaperone-based Escherichia coli strain[J]. Applied and Environmental Microbiology, 2004, 70(8): 4499-4504. DOI:10.1128/AEM.70.8.4499-4504.2004 |
| [41] |
Dobson CM. Protein folding and misfolding[J]. Nature, 2003, 426(6968): 884-890. DOI:10.1038/nature02261 |
| [42] |
Cambray G, Mutalik VK, Arkin AP. Toward rational design of bacterial genomes[J]. Current Opinion in Microbiology, 2011, 14(5): 624-630. DOI:10.1016/j.mib.2011.08.001 |
| [43] |
Medini D, Donati C, Tettelin H, et al. The microbial pan-genome[J]. Current Opinion in Genetics & Development, 2005, 15(6): 589-594. |
| [44] |
Pòsfai G, Plunkett G, Fehér T, et al. Emergent properties of reduced-genome Escherichia coli[J]. Science, 2006, 312(5776): 1044-1046. DOI:10.1126/science.1126439 |
| [45] |
Martinez-Martinez M, Lores I, Pena-Garcia C, et al. Biochemical studies on a versatile esterase that is most catalytically active with polyaromatic esters[J]. Microbial Biotechnology, 2014, 7(2): 184-191. DOI:10.1111/mbt2.2014.7.issue-2 |
| [46] |
Craig JW, Chang FY, Kim JH, et al. Expanding small-molecule functional metagenomics through parallel screening of broad-host-range cosmid environmental DNA libraries in diverse proteobacteria[J]. Applied and Environmental Microbiology, 2010, 76(5): 1633-1641. DOI:10.1128/AEM.02169-09 |
| [47] |
Guazzaroni ME, Morgante V, Mirete S, et al. Novel acid resistance genes from the metagenome of the Tinto River, an extremely acidic environment[J]. Environmental Microbiology, 2013, 15(4): 1088-1102. DOI:10.1111/emi.2013.15.issue-4 |
| [48] |
Arkin A. Setting the standard in synthetic biology[J]. Nat Biotechnol, 2008, 26(7): 771-774. DOI:10.1038/nbt0708-771 |
| [49] |
Shetty RP, Endy D, Knight TFJR, et al. Engineering BioBrick vectors from BioBrick parts[J]. J Biol Eng, 2008, 2: 5. DOI:10.1186/1754-1611-2-5 |
| [50] |
Silva-Rocha R, Martinez-Garcia E, Calles B, et al. The Standard European Vector Architecture(SEVA): a coherent platform for the analysis and deployment of complex prokaryotic phenotypes[J]. Nucleic Acids Research, 2013, 41(Database issue): 666-675. |
| [51] |
Hu XP, Heath C, Taylor MP, et al. A novel, extremely alkaliphilic and cold-active esterase from Antarctic desert soil[J]. Extremophiles: Life under Extreme Conditions, 2012, 16(1): 79-86. DOI:10.1007/s00792-011-0407-y |
| [52] |
Ko KC, Han Y, Cheong DE, et al. Strategy for screening metagenomic resources for exocellulase activity using a robotic, high-throughput screening system[J]. Journal of Microbiological Methods, 2013, 94(3): 311-316. DOI:10.1016/j.mimet.2013.07.010 |
| [53] |
Wong DW, Chan VJ, McCormack AA. Functional cloning and expression of a novel Endo-alpha-1, 5-L-arabinanase from a metagenomic library[J]. Protein and Peptide Letters, 2009, 16(12): 1435-1441. DOI:10.2174/092986609789839313 |
| [54] |
Hrvatin S, Piel J. Rapid isolation of rare clones from highly complex DNA libraries by PCR analysis of liquid gel pools[J]. Journal of Microbiological Methods, 2007, 68(2): 434-436. DOI:10.1016/j.mimet.2006.09.009 |
| [55] |
Voigt CA. Genetic parts to program bacteria[J]. Current Opinion in Biotechnology, 2006, 17(5): 548-557. DOI:10.1016/j.copbio.2006.09.001 |
| [56] |
Nasuno E, Kimura N, Fujita MJ, et al. Phylogenetically novel LuxI/LuxR-type quorum sensing systems isolated using a metagenomic approach[J]. Applied and Environmental Microbiology, 2012, 78(22): 8067-8074. DOI:10.1128/AEM.01442-12 |
| [57] |
Uchiyama T, Miyazaki K. Product-induced gene expression, a product-responsive reporter assay used to screen metagenomic libraries for enzyme-encoding genes[J]. Applied and Environmental Microbiology, 2010, 76(21): 7029-7035. DOI:10.1128/AEM.00464-10 |
| [58] |
Cowan DA, Arslanoglu A, Burton SG, et al. Metagenomics, gene discovery and the ideal biocatalyst[J]. Biochemical Society Transactions, 2004, 32(2): 298-302. DOI:10.1042/bst0320298 |
| [59] |
Purnick PE, Weiss R. The second wave of synthetic biology: from modules to systems[J]. Nature Reviews Molecular Cell Biology, 2009, 10(6): 410-422. DOI:10.1038/nrm2698 |
| [60] |
Weber W, Fussenegger M. Synthetic gene networks in mammalian cells[J]. Current Opinion in Biotechnology, 2010, 21(5): 690-696. DOI:10.1016/j.copbio.2010.07.006 |
| [61] |
Canton B, Labno A, Endy D. Refinement and standardization of synthetic biological parts and devices[J]. Nat Biotechnol, 2008, 26(7): 787-793. DOI:10.1038/nbt1413 |
| [62] |
Koide T, Pang WL, Baliga NS. The role of predictive modelling in rationally re-engineering biological systems[J]. Nature Reviews Microbiology, 2009, 7(4): 297-305. DOI:10.1038/nrmicro2107 |
| [63] |
Gardner TS, Cantor CR, Collins JJ. Construction of a genetic toggle switch in Escherichia coli[J]. Nature, 2000, 403(6767): 339-342. DOI:10.1038/35002131 |
| [64] |
Moon TS, Clarke EJ, Groban ES, et al. Construction of a genetic multiplexer to toggle between chemosensory pathways in Escherichia coli[J]. Journal of Molecular Biology, 2011, 406(2): 215-227. DOI:10.1016/j.jmb.2010.12.019 |
| [65] |
Siuti P, Yazbek J, Lu TK. Synthetic circuits integrating logic and memory in living cells[J]. Nat Biotechnol, 2013, 31(5): 448-452. DOI:10.1038/nbt.2510 |
| [66] |
3Rd CR, Dunlop MJ, Elowitz MB. A synthetic three-color scaffold for monitoring genetic regulation and noise[J]. J Biol Eng, 2010, 4(1): 10. DOI:10.1186/1754-1611-4-10 |
| [67] |
Silva-Rocha R, de Lorenzo V. Implementing an OR-NOT(ORN)logic gate with components of the SOS regulatory network of Escherichia coli[J]. Molecular BioSystems, 2011, 7(8): 2389-2396. DOI:10.1039/c1mb05094j |
| [68] |
Regot S, Macia J, Conde N, et al. Distributed biological computation with multicellular engineered networks[J]. Nature, 2011, 469(7329): 207-211. DOI:10.1038/nature09679 |
| [69] |
Porter TM, Hajibabaei M. Scaling up: A guide to high throughput genomic approaches for biodiversity analysis[J]. Molecular Ecology, 2018, 27(2): 313-338. DOI:10.1111/mec.2018.27.issue-2 |
| [70] |
Jiang Y, Wang J, Xia D, et al. EnSVMB: Metagenomics fragments classification using ensemble SVM and BLAST[J]. Scientific Reports, 2017, 7(1): 9440. DOI:10.1038/s41598-017-09947-y |
| [71] |
Siegwald L, Touzet H, Lemoine Y, et al. Assessment of common and emerging bioinformatics pipelines for targeted metagenomics[J]. PLoS One, 2017, 12(1): e0169563. DOI:10.1371/journal.pone.0169563 |
| [72] |
Zhai P, Yang L, Guo X, et al. MetaComp: comprehensive analysis software for comparative meta-omics including comparative metagenomics[J]. BMC Bioinformatics, 2017, 18(1): 434. DOI:10.1186/s12859-017-1849-8 |
| [73] |
Ofaim S, Ofek-Lalzar M, Sela N, et al. Analysis of microbial functions in the rhizosphere using a metabolic-network based framework for metagenomics interpretation[J]. Front Microbiol, 2017, 8: 1606. DOI:10.3389/fmicb.2017.01606 |




