2. 上海海洋大学水产动物遗传育种中心上海市协同创新中心,上海 201306;
3. 上海海洋大学水产科学国家级实验教示范中心,上海 201306
2. Shanghai Collaborative Innovation for Aquatic Animal Genetics and Breeding, Shanghai Ocean University, Shanghai 201306;
3. National Demonstration Center for Experimental Fisheries Science Education, Shanghai Ocean University, Shanghai 201306
鳜(Siniperca chuatsi)属硬骨鱼纲、鲈形目(Perciformes)、鮨科(Serranidae)、鳜属(Siniperca),俗称季花鱼、桂花鱼,其肉质细嫩、营养丰富、没有小刺,是我国传统淡水名贵经济鱼类。自20世纪80年代以来,鳜人工繁殖技术的突破推动了其养殖业的发展。目前鳜产业年产值超200亿元,但在养殖过程中也存在品种退化、病害频发等问题,鳜良种选育仍然是提高其产量和品质的首要途径。
成熟的microRNA(miRNA)是一类20-24 nt的非编码小分子RNA,其在进化和功能上均具有保守性[1]。通常,miRNA与靶基因mRNA的3'端非编码区(3'-untranslated region,3'-UTR)不完全互补配对结合,引起靶基因mRNA的降解,从而负调控靶基因的表达,即在转录后水平上影响表型[2]。miRNA的发现和技术的成熟,丰富了人们对蛋白质合成控制的认识,补充了在RNA水平对靶mRNA进行更有效调节的机制,展现了细胞内基因表达调控全方位、多层次的信号网络系统。
miRNA的研究对象遍及动物、植物和微生物,且miRNA几乎参与从生物胚胎发育到死亡的生命阶段所有的生物过程[3]。miRNA在水产动物(鱼、虾、蟹、贝)中也有研究报道,如miR-430在斑马鱼胚胎发育阶段的表达和功能[4]。有关鳜miRNA的研究起步较晚,数量也较少,可分为miRNA转录组和单个miRNA两类研究。迄今为止,通过高通量测序研究鳜miRNA转录组的报道有3篇:第一篇见于2013年,该研究以翘嘴鳜红肌(慢肌)与白肌(快肌)为研究对象,发现了186个保守和3个新miRNAs,在两种肌肉纤维中存在60个显著差异表达miRNA[5];以饲养6个月后的慢长和快长翘嘴鳜为研究对象,混合大脑、脑垂体、肝脏和肌肉样本,发现252个已知miRNAs和12个新miRNAs,其中36个miRNAs在组间表达差异显著[6];以孵化后第30天的慢长和快长翘嘴鳜肌肉为材料,鉴定出433个保守miRNAs,组间8个差异miRNAs[7]。就单个miRNA而言,miRNA-181a通过靶定小清蛋白(Parvalbumins,PVALBs)而影响鳜肌肉松弛率[8];miR-143可以通过调控靶基因肌分化因子(MyoD)而促进成肌细胞增殖和肌肉增生[9];对翘嘴鳜进行饥饿后再投喂,miR-10c、miR-107a、miR-133a-3p、miR-140-3p、miR-181a-5p、miR-206、miR-214[10]和miR-222[11]的相对表达会显著上调,上述miRNAs可能与鳜鱼骨骼肌快速生长相关。此外,还发现温度升高会降低miR-146a在鳜胚胎发育各个时期的表达[12]。,
综上所述,目前关于鳜miRNA转录组的研究多以肌肉等不同组织为研究对象,鱼龄最低为孵化后30 d,研究的单个miRNA大部分与鳜鱼骨骼肌发育相关。为了深入理解鳜的发育机制,本研究以鳜孵化后第3天、第17天和第28天的全鱼为材料,通过高通量测序建立上述各阶段miRNA组,并选取miR-199-5p和miR-203b进一步分析其可能的mRNA靶基因及相关的调控通路。本研究首次了解鳜孵化后前30 d内miRNAs表达特征、可以为深入了解miRNA对鳜胚胎发育过程的调控作用奠定基础、丰富有关miRNAs和靶基因间作用的认知,也可为鳜分子标记辅助育种提供候选基因。
1 材料与方法 1.1 材料实验用鳜鱼苗取自上海市浦东新区孙农水产养殖场。亲鱼经激素催产、人工授精。受精卵在直径70 cm的孵化桶中孵化,水温为23-25℃。孵化当天记为第0天(D0),分别在孵化后第3天(D3)、第17天(D17)、第28天(D28)、第60天(D60),第90天(D90)取样,每批随机取20-50尾(日龄越小取样越多)。取其中15尾D3,10尾D17、5尾D28全鱼分别混合后用于小RNA测序文库制备(图 1)。其余样品用于荧光定量分析,荧光定量分析前需做如下处理:D17、D28样本去除头、尾,D60、D90样本采集白肌(去除鳞片后背鳍起点下背侧第一肌节区域)。另取3尾体重500 g左右的健康鳜,用MS-222麻醉,于冰上解剖,取肝、脑、肠、白肌样本用于组织表达谱分析。所有样品分装于冻存管,迅速置于液氮中,并长期存放于-80℃冰箱备用。

|
| 图 1 孵化后D3、D17和D28鳜形态 |
采用Trizol法提取样品的总RNA,使用紫外分光光度计(Eppendorf BioPhotometer)和琼脂糖/EB凝胶电泳法分别确定总RNA的纯度和完整性。取质量较好的RNA样品用于相关分析。采用TruSeqSmall RNA Sample Prep Kits(Illumina,San Diego,USA)试剂盒制备小RNA测序文库。流程如下:用T4 RNA连接酶2将腺苷化单链DNA 3'接头和5'接头连接到小RNA上,进行反转录反应,对反转录产生的cDNA序列进行PCR扩增,将140-160 bp长度范围的PCR产物用6% polyacrylamide Tris-borate-EDTA胶回收,完成文库的制备,对构建好的文库用Illumina Hiseq2000/2500进行测序,测序读长为单端1X50 bp。
1.2.2 miRNA组数据分析使用联川生物公司开发的软件处理数据。首先,清理由于样本制备、测序接头、非典型miRNA特征序列以及测序仪器光学数码处理而产生的非纯序列,保留碱基长度在18-26 nt序列。然后,将剩余序列比对RFam数据库和重复序列数据库(Repbase)以去除rRNA、tRNA、snRNA和snoRNA等。经清理、过滤后的数据称之为Valid数据并用于后续分许。鳜尚无基因组数据,故以斑马鱼基因组为参考。使用Bowtie软件与miRbase21.0斑马鱼及其他脊椎动物的己知的miRNA进行保守性分析比对,并鉴定出鳜3个不同发育时期的miRNAs。在比对分析中允许5'和3'末端长度变化以及序列内部存在一个错配的情况。用Expdiff方法两两比较并判断不同发育阶段样品中miRNA的表达量是否存在显著差异。利用维恩图(Venn diagrams)直观地显示出样品两两间共同检出的miRNA个数以及差异miRNA的个数。
1.2.3 引物设计及合成根据鳜MyoD(Gene ID:JN561167.1)、斑马鱼Wnt5b(Gene ID NM_130937)https://www.ncbi.nlm.nih.gov/nuccore/NM_130937。用Primer Premier 6.0软件设计并由上海生工生物工程有限公司合成相关引物,序列见表 1。
利用定量RT-PCR检测miR-199-5p / miR-203b在肝、脑、肠、白肌及在D3、D17、D28、D60和D90的相对表达量。
1.2.5 miR-199-5p和miR-203b的靶基因验证设计并合成靶基因3'UTR区域包含miR -203b作用位点及突变的引物序列(表 1),上游引物插入XhoⅠ酶切位点CTCGAG,下游引物插入NotⅠ酶切位点GCGGCCGC(表 1序列中下画线部分)。PCR产物经过纯化后与psiCHECKTM-2载体相连后进行测序,测序正确的质粒命名为psiCHECKTM2- MyoD-3'-UTR(wt)和psiCHECKTM2- MyoD-3'-UTR(mutant)。培养293T细胞系,将细胞接种到96孔板中,待细胞密度达到70%时,将miR-203b mimic分别和psiCHECKTM2-MyoD-3'-UTR(wt)和psiCHECKTM2- MyoD-3'-UTR(mutant)进行共转染,以psiCHECKTM-2为对照,按Promega公司Dual-Luciferase Reporter Assay System说明书步骤检测细胞荧光素酶表达量,重复3次。另构建miR-199-5p靶基因质粒psiCHECKTM2-Wnt-3'-UTR(wt)和psiCHECKTM2- Wnt-3'-UTR(mutant),实验操作同上。本实验所用293T细胞系、psiCHECK -2空载体购自ATCC生物公司(美国)。
1.2.6 Myod/Wnt在孵化后90 d内的表达变化利用定量RT-PCR检测Myod / Wnt mRNA在D3、D17、D28、D60和D90的相对表达量。
1.2.7 数据分析应用荧光定量比较Ct值法(2-△△Ct法)分析各基因相对表达量。细胞试验重复处理3次,数据均以平均值±标准差表示。采用SPSS软件进行单因素方差分析(one-way ANOVA),Duncan法进行多组样本间差异显著性分析,以P < 0.05作为统计意义上的显著水平。
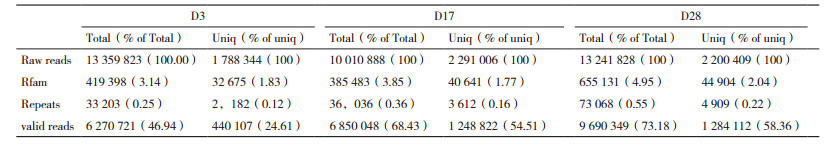
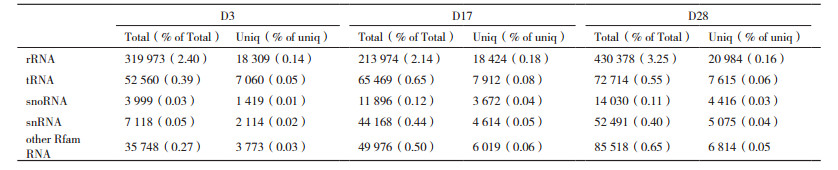
2 结果 2.1 孵化后30 d内3个时期鳜miRNA转录组测序与分析以孵化后不同发育时期(D3、D17和D28)的鳜全鱼为研究对象分别构建小RNA文库。利用高通量测序获得原始测序数据总数(Total)和原始测序种数(Unique),3个时期的测序总数分别为13 359 823、10 010 888和13 241 828,测序种数分别为1 788 344、2 291 006和2 200 409(表 2)。经清理过滤得到的valid数据总数和种数分别为6 270 721,46.94%;6 850 048,68.43%;9 690 349,73.18%和440 107,24.61%;1 248 822,54.51%;1 284 112,58.36%(表 2)。Valid数据大部分分布在20-24 nt,符合Dicer酶切割的典型特征。和Rfam数据库比对发现的rRNA、scRNA、snoRNA、snRNA和tRNA等非miRNA序列组成,见表 3。
共鉴定出1 084个miRNAs,其中已知的(known,reads既有miRbase支撑也有基因组位置支撑)miRNAs 432个,保守的(conservative,reads有miRbase支撑但pre-miRNA没有基因组位置支撑)miRNAs 624个,新的(novel,reads无miRbase支撑但有基因组位置支撑)miRNA 28个。D17时期鉴定出的miRNA个数最多为926个,在D3和D28时期分别有763和862个。在3个发育时期都表达的有628个miRNAs,只在D3、D17和D30中表达的分别有71、104和70个miRNAs。
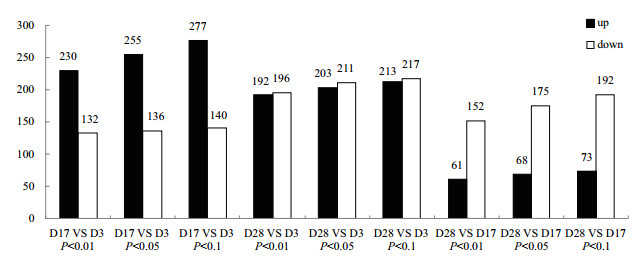
分别比较D17与D3,D28与D3,D28与D17两两之间的差异表达miRNAs(P < 0.1、P < 0.05或P < 0.01)。P < 0.1时,两两之间差异miRNAs个数分别为417,430,265(包含上调和下调);P < 0.05时,差异miRNAs个数分别为391、414和243;P < 0.01时,差异miRNAs个数分别为362、388和213。不同显著性p值的阈值下,D17和D3间差异miRNAs数均多于D28和D17间差异miRNAs数。D17和D3相比,上调miRNAs明显多于下调;D28和D17相比,下调miRNAs明显多于上调(图 2)。
|
| 图 2 不同时期两两之间的差异表达miRNAs |
在众多差异表达的miRNAs中筛选统计学上差异最显著的(P value = 0)进一步分析发现,在D3到D28阶段持续下调的miRNAs有dre-miR-10d-5p,dre-miR-9-7-3p,dre-miR-10c-5p,dre-miR-222a-3p,dre-miR-26a-5p和dre-miR-199-5p。持续上调的miRNAs有dre-miR-200c-3p,aca-miR-194-5p,dre-miR-192,dre-let-7a,dre-let-7h,dre-let-7e,dre-miR-1388-5p,dre-miR-1,dre-miR-199-3-3p,dre-let-7g,dre-miR-122,dre-miR-203a和dre-miR-203b。
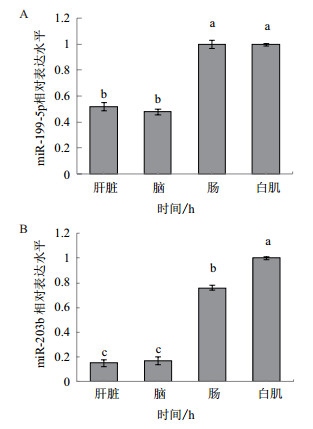
2.2 miR-199-5p / miR-203b组织表达谱及其在孵化后90 d内表达量的变化miR-199-5p / miR-203b在检测的组织中均有一定的表达,但表达丰度存在一定差异,miR-199-5p在白肌和肠道组织中的表达较高(图 3-A),miR-203b在白肌中的表达最高(图 3-B)。
|
| 图 3 miR-199-5p / miR-203b组织表达谱 |
进一步验证miR-199-5p / miR-203b与肌肉发育是否相关,利用实时荧光定量检测miR-199-5p / miR-203b在D3(全鱼),D17(躯干)、D28(躯干),D60(白肌)、D90(白肌)的相对表达情况。miR-199-5p随发育时期延续而表达逐渐减弱(图 4-A)。miR-203b随发育时期延续而表达逐渐增强(图 4-B)。
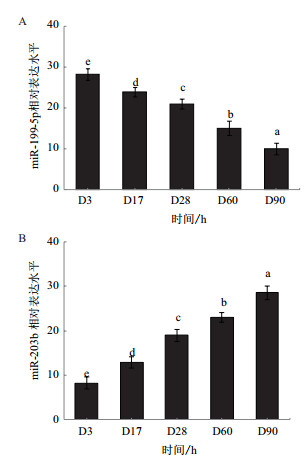
|
| 图 4 miR-199-5p / miR-203b在孵化后90 d内表达变化量 |
利用TargetScan预测miR-199-5p和miR-203b的靶基因分别为Wnt和Myod。
与转染psiCHECKTM-2空载体或psiCHECKTM2- Wnt-3'-UTR(mutant)相比,miR-199-5p mimic和psiCHECKTM2- Wnt -3'-UTR(wt)共转染时荧光素酶活性显著下降(P < 0.05)(图 5-A)与转染psiCHECKTM-2空载体或psiCHECKTM2- Myod-3'-UTR(mutant)相比,miR-203b mimic和psiCHECKTM2-MyoD-3'-UTR(wt)共转染时荧光素酶活性显著下降(P < 0.05)(图 5-B)。
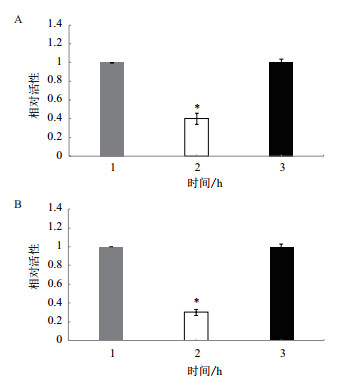
|
| 图 5 miR-199-5p和miR-203b的靶基因验证 A:1.转染psiCHECKTM-2空载体,2.共转染miR-199-5p mimic和psiCHECKTM2-Wnt-3'-UTR(wt),3.共转染miR-199-5p mimic和psiCHECKTM2- Wnt-3'-UTR(mutant);B:1.转染psiCHECKTM-2空载体,2.共转染miR- 203b mimic和psiCHECKTM2-MyoD-3'-UTR(wt),3.共转染miR-203b mimic和psiCHECKTM2-MyoD-3'-UTR(mutant)。*表示差异显著(P<0.05) |
Wnt mRNA随发育时期延续而表达逐渐增强(图 6-A),MyoD mRNA在D17表达量较高,之后表达量逐渐减弱(图 6-B)。
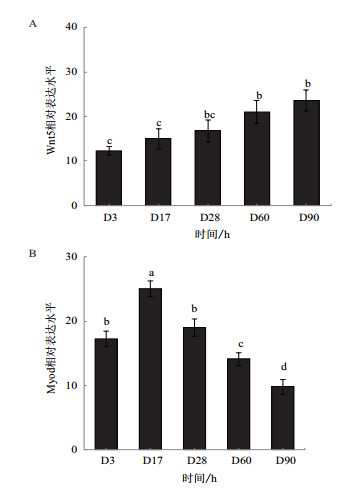
|
| 图 6 Wnt/ Myod在孵化后90 d内表达变化 |
本研究利用高通量测序技术获得孵化后D3、D17和D28时期鳜全鱼miRNA转录组,经清理过滤后得到的valid数据总数分别为6 270 721、6 850 048和9 690 349,样品间存在一定的差异,D28时期获得的数据最多,推测发育早期个体较小对文库的建立可能有一定影响。本研究中共鉴定出已知的miRNA known 432个,不同发育时期中鉴定出的miRNA个数存在明显的差异,有在3个发育时期都表达的也有只在某一时期表达的miRNAs,D17时期鉴定出的miRNA个数最多,且D17和D3间差异miRNAs数多于D28和D17间差异miRNAs数,这些充分说明生物体发育过程中miRNA对基因表达调控的时效性,也暗示了在D17时期miRNA更广泛的参与调控,相应得个体本身也可能经历着更多的发育变化。
对差异表达最显著(P value = 0)且在3个时期都表达的miRNAs进行归类,有在上述发育阶段持续下调的miRNAs(dre-miR-10d-5p,dre-miR-9-7-3p,dre-miR-10c-5p,dre-miR-222a-3p,dre-miR-26a-5p和dre-miR-199-5p),也有持续上调的miRNAs(dre-miR-200c-3p,aca-miR-194-5p,dre-miR-192,dre-let-7a,dre-let-7h,dre-let-7e,dre-let-7g,dre-miR-1388-5p,dre-miR-1,dre-miR-199-3-3p,dre-miR-122,dre-miR-203a-3p和dre-miR-203b-3p)。
已有的研究表明这些miRNAs与生物个体发育相关,如miR-10家族会和调控生物形体发育的Hox基因共表达,并对Hox的转录起到调控作用[13];miR-26调节血管平滑肌细胞的分化,并通过抑制ctdsp2(Carboxy-terminal domain RNA polymerase Ⅱ polypeptide A small phosphatase 2)而参与神经形成[14-15];miR-221可以通过靶定干细胞标志物CD117而阻止内皮细胞迁移和增生[16],此外也和血管形成相关[17];mir-200家族在上皮细胞-间充间质转化中起到重要作用,并可以维持细胞的上皮表型[18];MiR-194只在脊椎动物中被发现,其在小鼠中的靶基因是RhoB(Ras homolog gene family,member B),该基因可以调控微丝骨架重组,进而影响内耳螺旋神经元形成[19];miR-192是由p53诱导的miRNA,可以广泛的参与细胞周期调控[20],在羊骨骼肌发育过程中,miR-192可以调控羊肌肉卫星细胞sheep satellite cells(SCs)增殖和成肌分化,其靶基因是眼癌(Retinoblastoma 1,RB1)[21];miR-1388可以抑制GATA1(Erythroid transcription factor)和ALAS2(Erythroid-specific delta-aminolaevulinate synthase)的表达,进而影响红细胞分化[22];miR-1属于肌肉特异性miRNA,参与调控心肌在内的多种肌肉组织的发育和生理[23];let-7是被大家所熟知的可以控制干细胞分裂和分化时期的保守miRNA[24];miR-122在人体肝脏中随个体发育会不断增加直至成年,占到肝脏总miRNA的70%以上,是在各种组织表达最多的miRNA之一[25];MiR-199a-3p对成肌细胞分化起着非常重要的作用。抑制miR-199a-3p,肌细胞生成素(Myogenin,MyoG)、肌球蛋白重链(MyHC)表达均显著升高,肌萎缩标记基因MuRF1表达降低,同时肌管形成增加,融合指数和肌管直径升高[26]。本研究中,上述miRNAs在鳜早期发育过程中表现活跃(差异表达显著),且存在持续上调或下调的规律性表达,表明上述miRNAs参与了鳜早期发育,且在功能上可能是保守的。
在持续下调的基因中选择dre-miR-199-5p做深入分析。miR-199是一类家族,只在脊椎动物中被发现且参与调控多种发育机制,在肌肉发育调控方面,miR-199a是重要的心肌细胞尺寸调控基因[27]。miR-199a-5p通过靶向SIRT1促进心肌纤维化相关基因表达[28];miR-199a-5p参与调控平滑肌肥大以及器官重构。抑制miR-199a-5p的表达,可以上调包括WNT2在内的诸多靶基因,进而促进膀胱平滑肌细胞增殖,同时减小细胞体积[29],Wnt信号通路参与调控肌肉的形成与分化[30]。本实验中,鳜孵化后到D90,Wnt mRNA表达随发育推移而持续增强。miR-199-5p在白肌和肠道组织中的表达较高,miR-199-5p随发育时期延续而表达逐渐减弱,miR-199-5p和Wnt mRNA的表达表现为负相关,双荧光报告实验验证了Wnt是miR-199-5p的靶基因。综上,说明miR-199-5p可能和鳜肌肉生长相关。
在持续上调的基因中选择dre-miR-203做深入分析。通常miR-203在皮肤发育和生理过程中发挥重要调控作用。有研究表明该miRNA也和骨骼肌相关,如在鸡骨骼肌中,miR-203可以抑制细胞增殖、成肌细胞的增殖和分化,上述过程中miR-203靶基因为c-JUN和MEF2C,c-JUN在细胞增殖中起重要作用,而MEF2C是肌肉发育的关键转录因子[31]。同时,miR-203可以通过调控和肌纤维肥大相关的靶基因EIF4进而影响鸡肌肉质量[32]。在罗非鱼骨骼肌中,稚鱼时期miR-203b表达低,成鱼时期高。静默miR-203b在罗非鱼体内的表达,可以导致MyoD上调,并激活下游基因。MyoD与成肌细胞增殖和肌肉增生相关[33]。本实验中,MyoD mRNA在D17表达量较高,之后表达量逐渐减弱,可能由于D17时期肌肉增生较快,而D28之后肌肉增生下降,转向肥大。miR-203b在鳜白肌中的表达最高,随发育时期延续miR-203b表达逐渐增强,D28之后,miR-203b和MyoD表达呈负相关,双荧光报告实验验证MyoD是miR-203b的靶基因,综上说明miR -203b可能和鳜肌肉生长相关,且在发育后期miR-203b对MyoD抑制作用更加明显。
本研究中发现的miRNAs涉及个体发育的各个方面,其中和肌肉发育相关的miRNAs较多。以往只以肌肉为材料研究鳜骨骼肌发育相关miRNA,本研究以鳜发育早期全鱼为材料建立转录组,筛选显著差异的miRNA,也能发现和鳜肌肉生长相关的候选miRNA,可能是因为在早期胚胎阶段肌肉本身所占的比例较大,取样时肌肉生长相关的候选miRNA会富集。
4 结论本研究利用高通量测序技术获得鳜孵化后3个发育时期(D3、D17和D28)全鱼miRNA转录组,共鉴定出432个已知的miRNAs,28个新的miRNA。鳜早期发育过程中miRNA的调控作用具有时效性,表现为上述不同发育时期中miRNA个数存在明显差异,其中D17时期miRNA个数最多。一些和发育相关的miRNAs在上述时期的表达呈现持续上调或下调的特点。实时荧光定量表明miR-199-5p和miR-203b在鳜白肌中均有较高的表达,miR-199-5p / miR-203和WnT / MyoD在一定发育时期分别表现负相关,双荧光素酶报告验证了WnT和MyoD分别是miR-199-5p和miR-203的靶基因。综上,miR-199-5p、miR-203可能与鳜骨骼肌肉发育相关。
| [1] |
Carthew RW, Sontheimer EJ. Origins and Mechanisms of miRNA and siRNA[J]. Cell, 2009, 1364: 642-655. |
| [2] |
徐艳珺. miRNA-375在胃癌中的功能及作用机制[D]. 杭州: 浙江大学, 2010.
|
| [3] |
范文涛, 钟杨生, 陈芳艳, 等. miRNA在动物胚胎形成、发育中的研究进展[J]. 生物学杂志, 2016, 332: 91-94. |
| [4] |
李法君, 李明爽, 付春鹏, 等. microRNA在水产动物中的研究进展[J]. 水产学报, 2016, 406: 976-992. |
| [5] |
Chu WY, Liu LS, Li YL, et al. Systematic identification and differential expression profiling of MicroRNAs from white and red muscles of Siniperca chuatsi[J]. Current Molecular Medicine, 2013, 138: 1397-1407. |
| [6] |
Tu J, Tian C, Zhao P, et al. Identification and profiling of growth-related microRNAs in Chinese perchSiniperca chuatsi[J]. BMC Genomics, 2017, 18489: 1-9. |
| [7] |
王博, 田园园, 孙成飞, 等. 翘嘴鳜microRNA转录组分析及生长相关miRNA鉴定[J]. 基因组学与应用生物学, 2017, 362: 603-613. |
| [8] |
Chu WY, Liu LS, Chen L, et al. Rapid muscle relaxation in Siniperca chuatsi is coordinated by parvalbuminPVALBand MiR-181a[J]. Current Molecular Medicine, 2015, 158: 772-779. |
| [9] |
Chen L, Wu P, Guo XH, et al. miR-143:a novel regulator of MyoD expression in fast and slow muscles of Siniperca chuatsi[J]. Current Molecular Medicine, 2014, 143: 370-375. |
| [10] |
Zhu X, Chen D, Hu Y, et al. The microRNA signature in response to nutrient restriction and refeeding in skeletal muscle of chinese perchSiniperca chuatsi[J]. Marine Biotechnology, 2015, 172: 180-189. |
| [11] |
朱鑫, 胡毅, 王开卓, 等. 翘嘴鳜miR-222的表达特征[J]. 水生生物学报, 2015, 392: 315-320. DOI:10.7541/2015.42 |
| [12] |
朱鑫, 易潭, 陈琳, 等. 翘嘴鳜miR-146a在胚胎发育过程中的表达及温度对其表达的影响[J]. 湖南师范大学自然科学学报, 2015, 385: 21-26. |
| [13] |
Lund AH. miR-10 in development and cancer[J]. Cell Death & Differentiation, 2010, 172: 209-214. |
| [14] |
Leeper NJ, Raiesdana A, Kojima Y, et al. MicroRNA-26a is a novel regulator of vascular smooth muscle cell function[J]. Journal of Cellular Physiology, 2011, 2264: 1035-1043. |
| [15] |
Dill H, Linder B, Fehr A, et al. Intronic miR-26b controls neuronal differentiation by repressing its host transcript, ctdsp2[J]. Genes & Development, 2012, 261: 25-30. |
| [16] |
Vasilatou D, Papageorgiou S, Pappa V, et al. The role of microRNAs in normal and malignant hematopoiesis[J]. European Journal of Haematology, 2010, 841: 1-16. |
| [17] |
Nicoli S, Knyphausen CP, Zhu LJ, et al. miR-221 is required for endothelial tip cell behaviors during vascular development[J]. Developmental Cell, 2012, 222: 418-429. |
| [18] |
周欣亮, 张雪, 刘巍. miR-200家族在上皮-间质转化中的研究进展[J]. 肿瘤防治研究, 2013, 4010: 993-997. DOI:10.3971/j.issn.1000-8578.2013.10.019 |
| [19] |
Du J, Zhang X, Cao H, et al. MiR-194 is involved in morphogenesis of spiral ganglion neurons in inner ear by rearranging actin cytoskeleton via targeting RhoB[J]. International Journal of Developmental Neuroscience, 2017, 63: 16-26. DOI:10.1016/j.ijdevneu.2017.09.004 |
| [20] |
Georges SA, Biery MC, Kim SY, et al. Coordinated regulation of cell cycle transcripts by p53-Inducible microRNAs, miR-192 and miR-215[J]. Cancer Research, 2008, 6824: 10105-10112. |
| [21] |
Qian Z, Ye K, Wang HY, et al. Expression profiling and functional characterization of miR-192 throughout sheep skeletal muscle development[J]. Scientific Reports, 2016, 630281: 1-12. |
| [22] |
Xu Q, Cai C, Hu X, et al. Evolutionary suppression of erythropoiesis via the modulation of TGF-β signalling in an Antarctic icefish[J]. Molecular Ecology, 2015, 2418: 4664-4678. |
| [23] |
Mishima Y, Stahlhut C, Giraldez AJ. miR-1-2 gets to the heart of the matter[J]. Cell, 2007, 1292: 247-249. |
| [24] |
Roush S, Slack FJ. The let-7 family of microRNAs[J]. Trends in Cell Biology, 2008, 1810: 505-516. |
| [25] |
Chang J, Nicolas E, Marks D, et al. miR-122, a mammalian liver-specific microRNA, is processed from hcr mRNA and may downregulate the high affinity cationic amino acid transporter CAT-1[J]. RNA Biology, 2004, 12: 106-113. |
| [26] |
贾龙. MiR-199a-3p在C2C12成肌细胞分化过程中的作用及机制研究[D]. 杨凌: 西北农林科技大学, 2014.
|
| [27] |
Shen G, Chan WY. Flexible and versatile as a chameleon-sophisticated functions of microRNA-199a[J]. Int J Mol Sci, 2012, 137: 8449-8466. |
| [28] |
张灼, 朱杰宁, 肖珍, 等. 微小RNA-199a-5p通过靶向SIRT1促进心肌纤维化相关基因表达[J]. 中国病理生理杂志, 2017, 3310: 1781-1787. DOI:10.3969/j.issn.1000-4718.2017.10.008 |
| [29] |
Hashemi GA, Burkhard FC, Rehrauer H, et al. MicroRNA miR-199a-5p regulates smooth muscle cell proliferation and morphology by targeting Wnt2 signaling pathway[J]. Journal of Biological Chemistry, 2015, 19011: 7067-7086. |
| [30] |
Cisternas P, Henriquez JP, Brandan E, et al. Wnt signaling in skeletal muscle dynamics:myogenesis, neuromuscular synapse and fibrosis[J]. Molecular Neurobiology, 2014, 491: 574-589. |
| [31] |
罗文, 聂庆华, 张细权. miR-203的短暂表达及其对鸡骨骼肌细胞增殖和分化的抑制作用[J]. 中国家禽, 2014, 3617: 1. |
| [32] |
罗文, 聂庆华, 张细权. miR-203在鸡成肌细胞中靶向抑制EIF4E基因表达的研究[J]. 中国家禽, 2015, 378: 6-11. |
| [33] |
Yan B, Guo JT, Zhu CD, et al. miR-203b:a novel regulator of MyoD expression in tilapia skeletal muscle[J]. Journal of Experimental Biology, 2013, 2163: 447-451. |