2. 中国农业科学院饲料研究所,北京 100081
2. Feed Research Institute, Chinese Academy of Agricultural Sciences, Beijing 100081
乳酸氧化酶能够催化乳酸氧化为丙酮酸,是重要的制备丙酮酸原料[1]。丙酮酸作为一种新型医药、农药和日化中间体,许多氨基酸的生物合成都需要以丙酮酸为起始物质,国内外市场需求增长极其迅速,价格居高不下。如L-二羟基-苯丙氨酸(L-DOPA)作为一种有效的抗癌药物,就是以丙酮酸为原料,在酚基裂解酶的作用下转化而来[2]。同时乳酸氧化酶也广泛应用于传感器的制备,以检测血清样品的乳酸含量[3-4]。乳酸氧化酶作为一种黄素蛋白酶,以FMN作为辅基,其电子转移的形式不同于经典的电子传递链,而是直接以氧作底物,FMN和酶蛋白结合非常牢固,整个反应过程不需要游离的外源辅酶参与,没有辅酶再生的问题[5]。
但是目前研究发现的乳酸氧化酶大多热稳定性较差,只能在30-50℃下保持稳定[6-7],在化工合成过程中极易失去大部分酶活性,导致生产效率低下、资源浪费等问题,因此提高乳酸氧化酶的热稳定性具有重要意义。这不仅可以拓宽它的工业应用范围,而且可以研究酶蛋白结构与热稳定性之间的关系。国外学者已利用各种方法来尝试提高乳酸氧化酶的热稳定性,Lillis等[8]发现将乳酸氧化酶固定化可以大大提高乳酸氧化酶的稳定性,Hamamatsu等[9]通过易错PCR结合定向突变的方式,也得到了有效提高热稳定性的突变体。但我国关于乳酸氧化酶的研究则主要集中于产酶菌株的分离、酶基因克隆以及工业应用领域的拓宽[1, 10-11],对于乳酸氧化酶热稳定性改造方面的研究报道较少。
本研究通过同源建模、序列比对、分子对接技术以及酶热稳定性系统(Enzyme thermal stability system,ETSS)软件等生物信息学手段对乳酸氧化酶的蛋白结构进行理性分析,尝试在不损伤酶活力的前提下基于蛋白表面电荷分布优化对其进行热稳定性改良,以期进一步提高乳酸氧化酶的热稳定性,拓宽其应用领域,同时也为以后相关乳酸氧化酶热稳定性的分子改造提供一定的理论指导。
1 材料与方法 1.1 材料 1.1.1 菌种和载体实验所用重组载体pET30a-EgLOD为本实验室克隆、构建并保存,其中EgLOD基因来源于鹑鸡肠球菌(Enterococcus gallinarum strain),GenBank号为MG547691。大肠杆菌感受态Escherichia coli DMT、BL21购自北京全式金生物技术有限公司。
1.1.2 试剂FastPfu DNA聚合酶购自北京全式金生物技术有限公司;DNA琼脂糖凝胶回收试剂盒购自OMEGA公司;质粒小提中量试剂盒购自北京TianGen公司;蛋白定量试剂盒购自Bio-Rad公司;蛋白Marker购买于北京GeneStar生物公司;IPTG购自北京Solarbio科技有限公司;其他化学试剂为进口或国产分析纯。引物由北京梓熙生物技术有限公司合成。
1.1.3 仪器PCR,凝胶成像系统(美国Bio-Rad),高速冷冻离心机(日本HIMAC),酶标仪(美国Thermo),紫外分光光度计(日本JASO),移液枪(德国Eppendorf),His-tag蛋白磁珠(Beaver),其他实验仪器均为国产。
1.2 方法 1.2.1 EgLOD同源模建采用在线BLASTp(https://blast.ncbi.nlm.nih.gov/Blast.cgi)在pdb数据库(http://www.rcsb.org/pdb/home/home.do)中搜索同源模板,并进行E值和序列一致性等参数的比较分析。利用Discovery Studio(DS)软件进行单模板同源模建,采用DS内置的拉氏构象图(Ramachandra plot)评价结构合理性,内置的Minimization工具和Verify 3D优化程序进行结构优化,并进行三维立体结构的可视化分析。
1.2.2 分子对接及结构分析采用DS软件对配体和受体进行半柔性CDOCKER模拟对接。配体分子为优化后的EgLOD pdb蛋白结构,受体分子为FMN和乳酸的pdb结构(PDB库中下载),根据文献报道的活性位点选定对接区域[12-13],将RMSD阈值设为0.5Å以确保对接构象尽可能具有多样性。根据能量打分和构象评估选择最优对接结果进行后续分析。再利用DS对对接后的受体配体复合物进行相互作用力的分析。
1.2.3 多序列比对在对蛋白结构进行分析的基础上,为了进一步筛选可能影响乳酸氧化酶热稳定性的结合位点,通过查阅文献,筛选不同来源但结构相似的其他乳酸氧化酶序列,利用ClustalX 2.0进行多序列比对,ESPript 3.0(http://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi)进行序列催化位点、溶剂可及性、保守性和序列二级结构的分析。
1.2.4 蛋白表面电荷分析本文将蛋白结构看成一个球体,每一个带电荷氨基酸则被看成一个点电荷分布在球体上。利用酶热稳定性系统软件[14-15]计算蛋白结构中每个点电荷的总静电势能Eij。负值的Eij表示所给定的氨基酸残基对整个蛋白的稳定性起到积极的相互作用。相反,正值的Eij表示所给定的氨基酸残基对整个蛋白的稳定性起到不利的相互作用。因此,从理论上讲,改变这些Eij为正值的氨基酸残基就有可能提高蛋白的热稳定性。但ETSS软件所计算得到的符合条件的氨基酸数量依然十分庞大,在实际操作中仍需要经过其他手段辅助筛选,得到效果最优突变体。根据以下4个原则,对蛋白进行再设计:优先选择Eij为较高正值的残基进行突变试验;为了保证突变不会使酶活力损失,尽可能选择一些远离活性中心的氨基酸残基;保留EgLOD乳酸氧化酶中相对保守的氨基酸;通过多序列比对来选择突变点,当正值Eij残基所在的位置出现带相反电荷的氨基酸残基时,则将该正值Eij残基突变成不带电荷或者带相反电荷的残基;否则,将该正值Eij残基突变成Ala。
1.2.5 EgLOD突变体的构建以实验室构建并保存的重组质粒pET30a-EgLOD为模板,利用FastPfu DNA聚合酶通过上游引物和下游引物对质粒进行扩增,直接用PCR的方法一步构建定向单点突变。提取经DMT感受态克隆后的质粒,转化表达菌株BL21(DE3),最终得到构建好的突变体表达菌株命名为:BL21-pET30a-EgLOD-Mutant。设计突变引物见表 1。
PCR产物直接用DMT酶在37℃处理2 h后转化DMT感受态细胞,在固体LB培养基平板(含50 μg/mL Kana)上筛选获得阳性转化子。挑单克隆至600 μL液体LB培养基(含50 μg/mL Kana)上过夜培养,提取质粒并进行测序,将测序结果与pET30a-EgLOD比对以确定是否突变成功。
1.2.6 EgLOD及其突变体表达及纯化诱导表达:将构建好的表达菌株接种至30 mL LB培养液中过夜活化,后按1%的接种量转接至300 mL的大瓶LB(含50 μg/mL Kana)中,37℃,220 r/min培养至OD 600值约为0.6-0.8(约2-3 h)时加入终浓度为0.6 mmol/L IPTG诱导表达,并转至20℃,190 r/min诱导培养约10-12 h。12 000 r/min,10 min离心收集菌体。
蛋白纯化:将离心收集的菌体称湿重,以每克湿重加入3 mL磷酸钠缓冲液的比例加入缓冲液,将菌体吹打重悬。冰浴超声破碎菌体,200 W功率,4 s破碎,3 s暂停,共运行30 min。离心收集上清,上清即为EgLOD及各突变体表达的粗酶液。粗酶液采用His-tag蛋白磁珠纯化,将预处理的磁珠与粗酶液样品混合,4℃慢速旋转混合孵育1 h,使两者充分结合,依次加入5 mL不同咪唑浓度的清洗液各3次进行杂蛋白的洗脱,最后加入洗脱液收集洗脱峰,经酶活测定和SDS-PAGE验证,收集得到纯蛋白洗脱峰。
1.2.7 EgLOD及其突变体的比活测定乳酸氧化酶比活力单位定义:每毫克乳酸氧化酶蛋白中所含有的乳酸氧化酶活力单位(U/mg)。
在酶标条中加入5 μL不同浓度的蛋白BSA标品(0.125、0.25、0.50、0.75、1.0、1.5、2.0 mg/mL)和待测样品乳酸氧化酶(WT/D250N/ D281N),然后加入250 µL考马斯亮蓝显色剂,混匀。室温反应10 min后,测定OD595波长处吸光值。根据所测得的标品吸光值算出标准曲线,将样品(野生型与各突变体)的吸光值代入公式计算出样品的蛋白质含量(mg/mL)。
1.2.8 EgLOD及其突变体的热稳定性测定乳酸氧化酶酶活力测定采用DNP法[16],通过检测体系中丙酮酸产生量来确定酶活。具体方法如下:500 μL缓冲液+500 μL乳酸钠(5 mmol/L)+100 μL酶液混匀,水浴保温20 min;加入500 μL 0.1% DNP,水浴保温10 min;加入500 μL 4 mol/L氢氧化钠,水浴保温10 min;510 nm比色读值,并通过标准曲线计算酶活。
酶活单位规定:1 min催化乳酸生成1 nmol丙酮酸的酶量为1个酶活力单位(U)。
乳酸氧化酶热稳定性测定方法:将纯化后的酶液分别放置在水浴55℃、60℃中,分别处理5、10、20、30、60和90 min,最适条件下进行活性检测,以未经热处理的酶液作参照,计算剩余酶活百分比。
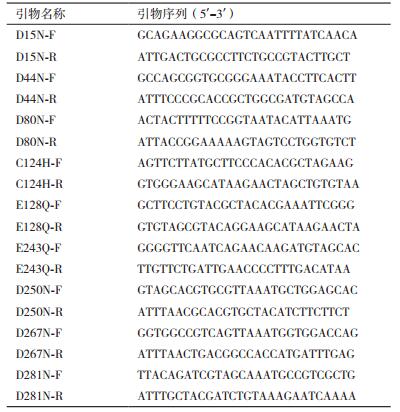
2 结果 2.1 EgLOD同源模建EgLOD与绿色气球菌(Aerococcus viridans)来源的乳酸氧化酶(PDB ID:4RJE)序列一致性达48.7%,相似性为53.2%,以4RJE为模板进行单模板同源模建并经能量最小化和结构优化,最后得到的蛋白结构其拉氏构象图及Verify 3D验证均达到可接受范围。EgLOD是典型的黄素酶,蛋白结构为(β/α)8桶状结构(图 1),辅酶FMN和底物乳酸钠的结合位点在β折叠片C末端[12]。
|
| 图 1 乳酸氧化酶EgLOD的蛋白结构及分子对接 |
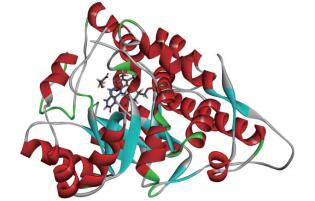
对接结果显示辅酶FMN紧紧结合于EgLOD酶蛋白β折叠片C末端,底物乳酸同时与FMN和乳酸氧化酶蛋白作用。2D平面图显示受体氨基酸与配体FMN辅酶、乳酸钠的相互作用力如图 2。FMN辅酶与乳酸氧化酶蛋白之间有多个相互作用的氨基酸残基,形成了非常丰富的作用键网络。对比乳酸钠和FMN与蛋白分子形成作用的氨基酸残基,发现有3个氨基酸残基同时与两个配体均有作用力,分别为Y37,H261,R264。而在观察蛋白结构时发现有8个氨基酸位于EgLOD酶蛋白表面,分别为Y37、R179、Y211、K237、H261、H264、D292和R296。这些都是与催化密切相关的氨基酸,在ETSS计算后应注意这些氨基酸的选择。
|
| 图 2 FMN、乳酸钠与EgLOD间的相互作用力 A、B:FMN与EgLOD之间的相互作用力;C、D:乳酸钠与EgLOD之间的相互作用力 |
利用ETSS软件计算EgLOD蛋白表面可电离的氨基酸残基的总静电势能Eij,结果如图 3所示。带电荷氨基酸残基共有97个,其中42个氨基酸的总电势能为正值,55个氨基酸的总电势能为负值。
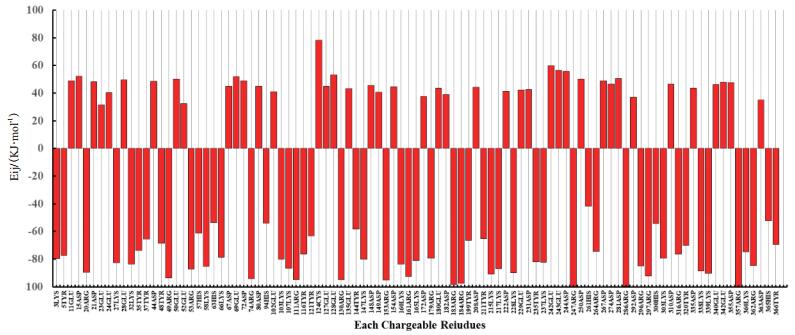
|
| 图 3 ETSS软件对EgLOD总静电势能Eij的计算结果 |
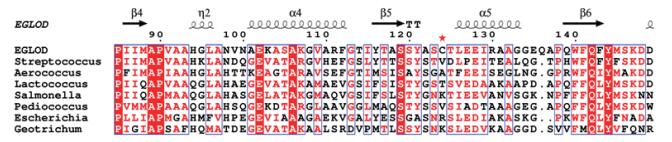
结合DS分子间作用力分析以及多序列比对结果(图 4),根据再设计4条原则进行筛选,我们选择了9个位点进行改造,分别为D15、D44、D80、C124、E128、E242、D250、D267、D281。突变方向为将Asp/Glu突变为结构相似但不带电荷的Asn/Gln,将不带电荷的Cys突变为带正电荷的His。
|
| 图 4 EgLOD与不同来源的乳酸氧化酶多序列比对 |
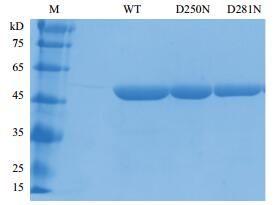
测定酶活后发现,E242Q、D267N突变后失活,D15N、D44N、D80N、C124H、E128Q突变后酶活损失较大,因此着重对D250N、D281N进行了详细研究。EgLOD野生型及其突变体构建成功后进行表达,纯化后经SDS-PAGE电泳验证,结果如图 5所示。EgLOD野生型及其突变体纯化后蛋白均呈单一条带,大小在49 kD左右,与理论分子量一致。测定比活后发现,D250N、D281N的比活分别为36.7 U/mg、37.3 U/mg,与野生型38.0 U/mg相近,比活损失不大。
|
| 图 5 EgLOD野生型及其突变体的SDS-PAGE分析 |
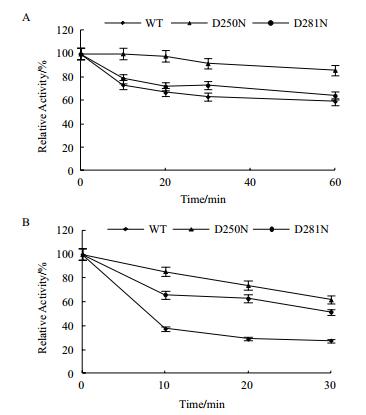
将纯化后的EgLOD野生型及突变体置于55℃与60℃下,每隔一定时间测量其残留活性,并规定未经热处理的酶活为100%,计算热处理后剩余酶活的百分比,得到EgLOD野生型及突变体酶失活曲线,如图 6所示。突变体D250N温度稳定性最好,在60℃下放置30 min后,仍保持有62%的剩余酶活;D281N在60℃下放置30 min后,也可以保持有52%的剩余酶活;与之相比,野生型EgLOD保持约28%的残留活性。
|
| 图 6 乳酸氧化酶EgLOD野生型及突变体的温度稳定性 A:酶在55℃下的温度稳定性测定;B:酶在60℃下的温度稳定性测定 |
目前国内对于乳酸氧化酶热稳定性改造的研究非常少,只有少数研究在乳酸氧化酶的固定化时,发现可以同时提高其温度稳定性[17]。而国外通常使用易错PCR[9]或DNA改组[18]等方式进行随机突变以及大量筛选来获得有效的突变体,但是其中绝大多数突变体都是以损失酶催化活力为代价来提高蛋白质稳定性的[9]。我们通过Discovery Studio软件为乳酸氧化酶建模,并以此分析蛋白与辅酶以及底物的作用键网络。我们发现由于乳酸氧化酶的蛋白结构较为复杂,蛋白必须在辅酶FMN存在的条件下才能与底物发生催化反应,而辅酶FMN与蛋白之间存在着复杂的作用键网络,因此在对乳酸氧化酶进行热稳定性分子改造时,极易造成作用键网络的破坏而导致蛋白酶活降低甚至失活。因此,探究一种既能提高蛋白质稳定,又不损失酶活的蛋白质改造策略是非常有意义的。
电荷相互作用的优化是一种成功应用于提高蛋白质稳定性的理性设计思路[19-20],但国内应用该思路进行蛋白改造的研究较少仍不成熟。本研究利用ETSS软件,对EgLOD进行热稳定性模拟改良,以期能提高EgLOD的稳定性并扩展其应用范围。结合ETSS软件的计算结果和再设计的4项原则,一共设计了9个突变体。经过试验验证,D250和D281两个氨基酸位点对提高EgLOD热稳定性起着非常重要的作用。在60℃下处理30 min后野生型EgLOD酶活仅剩20%左右,而D250N和D281N这两个突变体在同等条件处理后仍然能够保持50%以上的酶活,明显提高了EgLOD的热稳定性。实验结果表明降低蛋白表面的静电势能,能够有效稳定蛋白,确实证明蛋白表面带电情况与蛋白稳定性相关。
在酶工程中,对酶蛋白热稳定性改良的同时,保持酶活不损失是非常有必要的。本研究的主要目的是保证EgLOD酶活不损失的前提下进行热稳定性改良。在比对突变体与野生型EgLOD的比活时发现,这两个突变体的比活降低幅度很小,基本与野生型酶活相当。这可能是因为我们在选取目标位点时,在ETSS计算结果的基础上,综合考虑了这些氨基酸残基在蛋白结构中的作用和它们的序列保守程度,尽量挑选远离活性位点和保守序列的氨基酸进行突变。同时,本研究中我们将Asp突变为Asn,这两个氨基酸残基的结构相似,对于蛋白结构的影响较小,因此突变体的比活没有大幅降低。通过本实验证实了通过表面电荷分布的优化改造蛋白质热稳定性是一种行之有效的策略。
4 结论将ETSS计算与DS分子间作用力分析以及多序列比对相结合,对EgLOD进行蛋白热稳定性改造,获得了两个有效突变体D250N和D281N,将60℃下放置30 min后的剩余酶活由野生型的28%分别提高至62%和52%,且其比活没有损失。
| [1] |
谷劲松, 曲音波. 丙酮酸的酶法转化及乳酸氧化酶的研究进展[J]. 微生物学通报, 2003, 30(1): 86-90. DOI:10.3969/j.issn.0253-2654.2003.01.022 |
| [2] |
Park HS, Lee JY, Kim HS. Production of L-DOPA(3, 4-dihydroxyphenyl-L-alanine)from benzene by using a hybrid pathway[J]. Biotechnology & Bioengineering, 1998, 58(2-3): 339. |
| [3] |
Gavalas VG, Chaniotakis NA. Lactate biosensor based on the adsorption of polyelectrolyte stabilized lactate oxidase into porous conductive carbon[J]. Microchimica Acta, 2001, 136(3-4): 211-215. DOI:10.1007/s006040170056 |
| [4] |
Xu JJ, Zhao W, Luo XL, et al. A sensitive biosensor for lactate based on layer-by-layer assembling MnO2 nanoparticles and lactate oxidase on ion-sensitive field-effect transistors[J]. Chemical Communications, 2005, 6(6): 792-794. |
| [5] |
Unterweger B, Stoisser T, Leitgeb S, et al. Engineering of Aerococcus viridans L-lactate oxidase for site-specific PEGylation:characterization and selective bioorthogonal modification of a S218C mutant[J]. Bioconjugate Chemistry, 2012, 23(7): 1406. DOI:10.1021/bc2006847 |
| [6] |
Toda A, Nishiya Y. Gene cloning, purification, and characterization of a lactate oxidase from Lactococcus lactis, subsp. cremoris, IFO3427[J]. Journal of Fermentation & Bioengineering, 1998, 85(5): 507-510. |
| [7] |
Sztajer H, Wang W, Lünsdorf H, et al. Purification and some properties of a novel microbial lactate oxidase[J]. Applied Microbiology & Biotechnology, 1996, 45(5): 600. |
| [8] |
Lillis B, Grogan C, Berney H, et al. Investigation into immobilisation of lactate oxidase to improve stability[J]. Sensors & Actuators B Chemical, 2000, 68(1-3): 109-114. |
| [9] |
Hamamatsu N, Nomiya Y, Aita T, et al. Directed evolution by accumulating tailored mutations:thermostabilization of lactate oxidase with less trade-off with catalytic activity[J]. Protein Engineering Design & Selection, 2006, 19(11): 483-489. |
| [10] |
刘真, 常雁红, 刘仁霖, 等. 磁性壳聚糖微球的制备及对乳酸氧化酶的固定化研究[J]. 现代化工, 2007(s2): 310-313. |
| [11] |
王艳, 周林, 姚丽丽. 耻垢分枝杆菌产乳酸氧化酶条件的研究[J]. 食品与发酵工业, 2006, 32(8): 28-31. DOI:10.3321/j.issn:0253-990X.2006.08.007 |
| [12] |
Umena Y, Yorita K, Matsuoka T, et al. The crystal structure of L-lactate oxidase from Aerococcus viridans at 2. 1A resolution reveals the mechanism of strict substrate recognition[J]. Biochemical & Biophysical Research Communications, 2006, 350(2): 249. |
| [13] |
Demirjian A. Crystallographic study on the interaction of L-lactate oxidase with pyruvate at 1. 9 Angstrom resolution[J]. Biochemical & Biophysical Research Communications, 2007, 358(4): 1002-1007. |
| [14] |
Zhang L, Tang X, Cui D, et al. A method to rationally increase protein stability based on the charge-charge interaction, with application to lipase LipK107[J]. Protein Science A Publication of the Protein Society, 2014, 23(1): 110-116. DOI:10.1002/pro.2388 |
| [15] |
Tu T, Luo H, Meng K, et al. Improvement in Thermostability of an Achaetomium sp. strain Xz8 endopolygalacturonase via the optimization of charge-charge interactions[J]. Applied & Environmental Microbiology, 2015, 81(19): 6938-6944. |
| [16] |
谷劲松. 乳酸氧化酶转化DL-乳酸生成丙酮酸[D]. 济南: 山东大学, 2002. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y480151
|
| [17] |
周林. 乳酸氧化酶的制备及其固定化催化DL-乳酸生产丙酮酸研究[D]. 长沙: 中南大学, 2007. http://cdmd.cnki.com.cn/Article/CDMD-10533-2007172696.htm
|
| [18] |
Minagawa H, Shimada J, Kaneko H. Effect of mutations at Glu160 and Val198 on the thermostability of lactate oxidase[J]. Febs Journal, 2003, 270(17): 3628-3633. |
| [19] |
Strickler SS, Gribenko AV, Gribenko AV, et al. Protein stability and surface electrostatics:a charged relationship[J]. Biochemistry, 2006, 45(9): 2761. DOI:10.1021/bi0600143 |
| [20] |
Schweiker KL, Zarrine-Afsar A, Davidson AR, et al. Computational design of the Fyn SH3 domain with increased stability through optimization of surface charge charge interactions[J]. Protein Science, 2007, 16(12): 2694-2702. DOI:10.1110/(ISSN)1469-896X |