2. 吉林省人参基因资源开发与利用工程研究中心,长春 130118
2. Research Center for Ginseng Genetic Resources Development and Utilization, Changchun 130118
在生物进化过程中,植物在生物和非生物胁迫下产生了很多新的转录因子基因家族,转录因子可以通过调控靶基因的表达从而提高植物体抵御这些胁迫的能力。其中,AP2/ERF转录因子超家族是植物最大转录因子家族之一,不仅参与调控植物的生长发育,在响应逆境胁迫方面也起到非常重要的作用。第一个AP2/ERF转录因子基因于1994年在模式植物拟南芥中被分离出来,且被证实参与了花发育过程的调控[1]。最初认为AP2/ERF转录因子只存在于植物中,后来在原核生物和蓝藻中也发现该类转录因子[2]。在植物中,AP2/ERF转录因子编码的蛋白质具有与DNA结合的能力[3]。但是在非植物体中,有报道发现AP2结构域能够与富含组氨酸和天冬酰胺的HNH核酸酶结构域结合[2]。有报道称植物与非植物体之间可能发生基因交换,那么,植物体中的AP2/ERF转录因子可能来源于携带该转录因子的细菌或病毒。通过细菌或病毒侵染不同植物体,从而实现了AP2/ERF转录因子跨物种转移。木本植物染色体上出现串联重复序列的情况可能是由于部分AP2/ERF转录因子出现了转座[4]。本文综述了近年来研究人员对AP2/ERF转录因子家族在植物生长发育、次生代谢、胁迫应答及信号传导等方面的研究进展,旨为后续进一步探究该家族基因的功能及推广应用奠定理论基础。
1 AP2/ERF蛋白的分类和结构特征乙烯应答元件结合因子(Ethylene responsive element binding factor,ERF)转录因子的命名是由于其能响应乙烯的调节,然而,该功能并不是AP2/ERF家族普遍具有的功能,其保守结构域也不受乙烯信号的直接影响,但该转录因子的命名一直被沿用至今。AP2/ERF转录因子至少含有一个名为AP2结构域的DNA结合结构域,是由约60个氨基酸残基按照3个β折叠和一个α螺旋的方式所构成一个典型的三维结构[5]。2002年,Sakuma等[6]依据AP2/ERF转录因子的AP2结构域相似性将拟南芥中145条AP2/ERF转录因子基因分为5大类,即AP2、ERF、DREB、RAV和Soloist。AP2亚家族包含两个相似度很高并且串联重复出现的AP2结构域。含有一个AP2结构域和一个B3结构域的AP2/ERF转录因子被命名为RAV亚家族。Soloist类AP2/ERF转录因子也包含一个AP2结构域,但是它的氨基酸基序和基因结构与其他类AP2/ERF转录因子存在很大的不同。DREB(脱水应答元件结合蛋白)亚家族和ERF(乙烯应答转录因子)亚家族都只含有一个AP2结构域,但是它们的主要区别在于AP2结构域的第14位和第19位氨基酸残基的不同。DREB亚家族的第14位氨基酸是缬氨酸,第19位是谷氨酸;ERF亚家族的第14位氨基酸是丙氨酸,第19位是天冬氨酸。而DREB亚家族和ERF亚家族又进一步细分为6个亚组(A1-A6;B1-B6)。由于氨基酸序列的不同,两个亚家族对DNA亲和力和特异性区别较大。例如,DREB蛋白可以特异性结合与ABA、干旱和低温响应基因相关联的A/GCCGAC元件[7];而ERF亚家族的成员可以特异性结合基因组上游的AGCCGCC元件(GCC-box),从而参与调控乙烯应答、抗病以及非生物胁迫[8]。然而,Welsch等[9]报道称一些AP2/ERF转录因子成员可以同时与A/GCCGAC元件和GCC-box,甚至能和其他DNA元件结合。2006年,Nakano等[10]利用新增加的拟南芥的基因组注释信息,并考虑到AP2/ERF基因的内含子-外显子数量和结构将DREB与ERF基因合并成一个整体,分为4类12个组(Groups I to IV;Groups V to X;Groups VI-L;Groups Xb-L)。该分类与Sakuma等[6]的分类结果基本一致。如今,Sakuma和Nakano这两种分类方式在文献中均有使用。然而,相比Nakano分类方式,Sakuma等对AP2/ERF转录因子基因家族分类方式更加清晰。因此,本文采用Sakuma等的分类方式对AP2/ERF转录因子进行分类和功能分析。
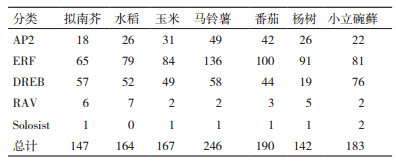
2 不同植物AP2/ERF转录因子的分布及数量目前人类已经完成了对拟南芥、水稻和番茄等物种的全基因组测序,在不同植物中,AP2/ERF转录因子及其各个亚家族成员的数量不同。分析发现,在这些植物中,AP2/ERF转录因子数量较多,其中,马铃薯中AP2/ERF转录因子达到246条。现有报道的植物中,ERF和DREB类的AP2/ERF转录因子所占比例均超过该家族的50%以上。然而,Solosist类的AP2/ERF转录因子数量很少,大部分植物中只有1条,而在水稻中甚至没有该亚家族的AP2/ERF转录因子。Nakano等报道了拟南芥中存在147条AP2/ERF转录因子,其中18条属于AP2亚家族,65条属于ERF亚家族,57条属于DREB亚家族,6条属于RAV亚家族,1条属于Solosist。Song等[11]报道番茄中AP2亚家族有42个成员,ERF亚家族有100个成员,DREB亚家族有44个成员,RAV亚家族有3个成员,有一个成员属于Solosist(表 1)。
首先,AP2/ERF转录因子被报道参与了种子发育过程。植物种子萌发过程受许多因素影响,其中包括湿度、温度、光照及种子的成熟程度。此外,植物激素如ABA在种子的萌发过程中也起到重要作用。研究显示在种子发育过程中ABA浓度较高,种子处于休眠状态;当种子萌发时,ABA含量则显著下降[12]。Wang等[13]发现在高浓度ABA处理时,转大豆AP2/ERF转录因子GmSGR基因的拟南芥种子发芽率明显高于非转基因植株。因此,作者认为AP2/ERF转录因子GmSGR基因是ABA响应的负调控基因,从而参与植物的种子发育过程。Ohto等[14]发现缺失AP2基因(ap2-6、ap2-7和ap2-11)的拟南芥突变体出现了发育异常现象,主要表现为种子膨大,且重量明显增加,从而证明AP2基因能够影响拟南芥的种子发育过程。Lasserre等[15]对转AtERF38基因的拟南芥植株成熟种子进行GUS染色,通过显微镜观察其木质部的木栓质积累异常、外珠被次生壁增厚,利用RT-PCR技术检测到AtERF38基因的表达量与次生壁增厚现象成正相关,由此证明AtERF38基因能正向调节种子次生壁的形成。
除了种子发育,AP2/ERF转录因子还被报道异常表达时会影响植物多个器官的形态。例如,籼稻(Oryza sativa subsp. indica)9311中的一个AP2/ERF家族基因OsEATB可以降低水稻节间的伸长,其机制是通过下调赤霉素生物合成基因贝壳烯酶A的表达水平,限制了赤霉素响应乙烯的诱导,最终导致成熟的水稻植株株高下降[16]。Zhao等[17]研究发现,水稻中ERF3基因是一个具有促进根冠形成的AP2/ERF转录因子,通过抑制ERF3基因的表达,得到相比野生型产生根冠的数量与长度明显减少的植株,利用甲苯胺蓝染色观察发现其根冠发育被推迟,由此推测ERF3基因在根冠形成和发育初期行使重要的功能。而水稻中另一AP2/ERF家族成员HL6具有增加水稻叶片与茎表明绒毛数目的功能,Sun等[18]将超表达HL6基因植株与对照组相比绒毛数量显著增加而HL6突变体茎、叶表面绒毛数量明显减少,但是绒毛长度并没有发生改变,推测虽然HL6基因调控水稻茎、叶表面绒毛数目但绒毛长度由其他基因调控。Krizek等[19]发现在拟南芥中过表达ant-4 ail6-2(AP2亚家族)基因时,严重影响了拟南芥花的发育,具体表现为花丝和萼片的膨大,作者推测该现象可能是由于ant-4 ail6-2(AP2亚家族)基因能够参与生长素的运输所导致。腾飞[20]的研究证明了拟南芥ERF055基因会影响合子胚的发育,导致胚胎发育滞后,不能形成正常的胚,过表达的ERF055基因导致拟南芥幼苗真叶显著变小,而ERF055-RNAi转基因植株的真叶相对野生型变大。有研究发现,乙烯对植物果实形状有着重要影响[21]。Wang等[22]通过检测弯曲角度与黄瓜基因表达量的相关关系发现,弯曲果实凸面的基因ERF025(csa3g042390)比直果中表达量高60多倍,包括生长素相关基因IAA4在内,其他基因如csa3g207390、csa3g042390和csa5g167120也都存在这一现象,这些基因全部参与乙烯介导的信号通路。由此推测,乙烯影响果实形状是由于乙烯的处理引起AP2/ERF转录因子表达发生改变,从而引起植物性状发生改变。
ERF转录因子最初的命名正是由于其能响应乙烯调节,如Yang等[23]在2005年就发现经过ERF转录因子调节乙烯转录,ERF亚族成员积极或消极地调节乙烯诱导EIN3下游基因的表达。此外,AP2/ERF还能响应其他植物激素如细胞分裂素和生长素的调节。Rashotte等[24]发现一些ERF-Ⅵ蛋白介导细胞分裂素的转录,将其命名为细胞分裂素响应因子Cytokinin Responsive Factors(CRFs)。2011年,Iwase等[25]发现拟南芥AP2转录因子中WOUND INDUCED DEDIFFERENTIATION 1(WIND1)基因能够刺激受伤植物伤口位置的细胞分泌细胞分裂素,促进细胞增殖分化,抑制WIND1基因的表达时,植物愈伤组织形成能力显著下降。在拟南芥中,另一个AP2/ERF转录因子RAP2.6被认为是植物分生组织再生过程的调节基因,在细胞损伤的修复再生过程中是必不可少的,该基因活性与植物生长素IAA浓度成反比,证明RAP2.6基因是IAA合成抑制基因[26]。总之,AP2/ERF转录因子通过多种途径,直接或间接参与了植物发育的多个进程。
4 AP2/ERF转录因子参与植物次生代谢调控作为一类广泛参与植物生命活动过程的转录因子,AP2/ERF基因家族不仅参与了植物的初生代谢,还在次生代谢方面发挥重要作用。目前,由于AP2/ERF转录因子在调控药用植物主要药用活性成分合成方面效果显著,多数研究主要集中于其参与药用植物次生代谢过程。1971年,中国药学家屠呦呦发现了青蒿素,它是从黄花蒿的茎叶中提取的新型抗疟药。Lu等[27]通过超表达青蒿中AaERF1和AaERF2基因,增强了青蒿素生物合成途径中的关键酶基因CYP71AV1的表达,青蒿素的积累量也有所增加;而在青蒿中沉默AaERF1和AaERF2基因后,青蒿素的合成明显被抑制。因此,青蒿中AaERF1和AaERF2基因与青蒿素合成呈正相关。同时,ABA和JA诱导处理后,转录因子AaERF3的转录水平有所提高,且青蒿素的产量也显著提高,由此作者推测AaERF3基因与青蒿素合成也存在一定相关性。长春花中长春碱是一种临床上被应用于治疗白血病和肿瘤的药物。1999年,Menke等[28]利用酵母单杂从长春花中筛选出两条AP2/ERF转录因子(ORCA2和ORCA3),这两条基因能够增加长春碱合成途径关键酶基因——异胡豆苷合成酶基因的的表达量,从而能够促进长春碱前体物质异胡豆苷的合成,进而增加长春碱的积累量。作为一种二萜生物碱类化合物,紫杉醇是目前已发现的最优秀的天然抗癌药物,其半化学合成的前体物质紫杉烯是由牻牛儿基牻牛儿基焦磷酸(Geranyl geranyl pyrophosphate,GGPP)在紫杉烯合酶(Taxadiene synthase,TASY)催化下生成的。张蒙[29]从红豆杉转录组中筛选了94条AP2/ERF转录因子,并克隆了其中两条基因:TcERF12和TcERF15,通过酵母双杂交、红豆杉细胞体内共表达及超表达等多个实验,最终证明TcERF12和TcERF15能够分别抑制和激活红豆杉中TASY的表达。除了药用植物,AP2/ERF转录因子还被报道能够调控烟碱的合成。烟碱又叫尼古丁,是烟草中主要的活性成分。2005年,De Sutter等[30]发现经过茉莉酸甲酯处理过的烟草中AP2/ERF转录因子NtJAP1和NtORC1基因的表达水平都有所提高,且显著上调了烟草烟碱生物合成途径下游的关键酶N-甲基转移酶(PMT)合成基因启动子的表达,烟碱的含量也同时增加,推测AP2/ERF转录因子NtJAP1和NtORC1基因正向调节烟草烟碱的合成。此外,De Boer等[31]也通过利用茉莉酸甲酯对烟草进行处理发现,NtPMT1a基因的表达量上升,同时,NtERF32的表达也显著增强,在烟草中过表达NtERF32后,NtPMT1a转录水平增加,且检测到烟草烟碱和总生物碱含量都有所增加。因此,作者认为AP2/ERF转录因子能够调控烟草烟碱的生物合成。木质素类化合物已被认定为板蓝根中主要抗病毒有效成分,而AP2/ERF转录因子可能与板蓝根中木脂素/木质素的生物合成有关[32]。Ma等[33]通过荧光定量PCR分析,敲除AP2/ERF成员li049基因会导致木脂素/木质素的生物合成途径内的基因转录水平下降,证明li049基因在木脂素/木质素的合成途径中扮演重要角色。
5 AP2/ERF转录因子参与生物与非生物胁迫应答AP2/ERF转录因子在参与植物应对生物、非生物胁迫方面也具有重要功能。有研究表明,一些AP2/ERF基因异位表达可以提高植物体应对胁迫的能力,包括抵抗高盐、干旱、缺氧及低温胁迫等。例如,拟南芥中部分AP2/ERF转录因子诱导编码脯氨酸合成关键酶的表达,促进脯氨酸的合成,游离的脯氨酸含量增加能够增强植物的耐寒性[34]。与之不同的是,拟南芥DREB/CBF2基因通过减缓拟南芥的生长和推迟叶片和花的衰老抵抗低温胁迫[35]。AtERF6基因的超表达可以抑制拟南芥中活性氧应答基因的表达,减轻由于高温产生的活性氧对植物的损伤,从而提高植物对高温胁迫的抗性[36]。Dossa等[37]对芝麻中132条AP2/ERF转录因子进行了分析发现,其中有41条DREB基因,绝大多数在根部表达水平更高,功能集中于应对干旱胁迫。通过转ERF转录因子SNORKEL1(SK1)和SNORKEL2(SK2)基因水稻与野生型水稻对比发现,转基因水稻植株更高,激素表达水平也不同,证明SK1与SK2具有促进水稻节间分生组织生长、调节植物体内乙烯和脱落酸等激素合成的功能,以此帮助植物抵抗淹水胁迫[38]。Park等[39]发现了几株拟南芥AtERF71/HRE2基因缺失突变体,突变体植株表现为不耐盐性。将AtERF71/HRE2基因转入突变体拟南芥中,植株表现出耐盐性,成功抵抗渗透胁迫,且植物体内超氧化物的含量增加,应对低氧胁迫的能力也有所增强。拟南芥ERF转录因子RELATED TO AP2 2(RAP2.2)与35S启动子结合后在植物体内超表达,可以提高植物对灰霉病的抗性。相反,对RAP2.2基因进行敲除,植物体患灰霉病的死亡率极高,说明RAP2.2基因具有正向调节植物抗灰霉病的功能[40]。
6 AP2/ERF转录因子参与非生物信号传导AP2/ERF转录因子作为乙烯信号传导途径的最终目的基因,可以反馈调节激素的生物合成,如乙烯、细胞分裂素、赤霉素和脱落酸,同时也能响应生长素、茉莉酸甲酯等生物信号,因此视该家族为植物激素信号连接的关键调节器[41]。Mishra等[42]通过实时定量PCR分析发现,在茉莉酸甲酯诱导处理3 h条件下PsAP2基因的表达量相比正常条件下高出2.8倍,而经乙烯诱导处理1 h的PsAP2基因表达量为正常条件下的3.7倍,脱落酸处理5 h后PsAP2基因的表达量为正常条件下的3倍,以此证明该家族基因确实具有响应激素调节的功能。Xiao等[43]通过乙烯处理香蕉果实,确定AP2/ERF家族成员MaERF9与MaERF11表达量变化显著,又监控香蕉果实软硬程度及乙烯释放量与MaERF9与MaERF11及香蕉中乙烯合成关键基因MaACS1和MaACO1表达量之间的关系,结果发现,经少量外源乙烯诱导可以促进这些基因表达,并促进香蕉果实合成乙烯并释放,当处理25 d后,乙烯会转而抑制基因的转录水平,同时减少乙烯的释放,证明AP2/ERF类转录因子参与乙烯的生物合成及信号传导。
7 展望从1994年第一条AP2/ERF转录因子在拟南芥中被分离出来至今,科学家们对AP2/ERF转录因子的研究已有20余年,随着研究的逐渐深入,研究人员们已经明确AP2/ERF转录因子在植物生理过程中的重要地位[44]。虽然AP2/ERF转录因子在分子育种方面具有很重要的功能,但是目前的研究都还只停留在基因功能的初步验证程度,虽然经过了大量的转基因实验证实了该类转录因子对于实际应用具有重要意义,但是关于已经投入实际应用的转基因材料却鲜有报道,这或许是该类转录因子在实验阶段向应用阶段过渡中存在瓶颈,需要研究人员进一步探索突破。具有转录抑制活性的转录因子存在提高代谢产物的现象,推测虽然转录因子行使的是转录抑制活性,但其作用的下游靶基因是产物合成的抑制基因,因此我们需要着重关注对于转录因子调控特异靶基因的机制和乙烯效应的下游分子网络、信号通路的精细调节以及乙烯信号与其他植物激素信号之间的交叉反应,利用荧光基因或同位素标记或许能够成为直观的检测手段。
由于分子技术的日益进步和蛋白质组分析灵敏度的提高,转录因子的活性和数量的增多与减少不需要进行非常复杂的操作就能确定,这为研究转录因子的功能提供了极大的便利。利用基因工程技术手段,通过进行同源物种基因超表达或基因编辑的方法验证基因的功能也成为目前基因功能研究领域的主要方式。根据AP2/ERF转录因子应对生物、非生物胁迫及调控植物次生代谢产物合成等一系列特性,使创造兼具抗病、高产及营养型转基因农作物品种成为可能。
| [1] |
Jofuku KD, Montagu MV, Okamuro JK. Control of Arabidopsis flower and seed development by the homeotic gene APETALA2[J]. Plant Cell, 1994, 6(9): 1211-1225. DOI:10.1105/tpc.6.9.1211 |
| [2] |
Wessler SR. Homing into the origin of the AP2 DNA binding domain[J]. Trends in Plant Science, 2005, 10(2): 54-56. DOI:10.1016/j.tplants.2004.12.007 |
| [3] |
Magnani E, Sjölander K, Hake S. From endonucleases to transcription factors: evolution of the AP2 DNA binding domain in plants[J]. Plant Cell, 2004, 16(9): 2265. DOI:10.1105/tpc.104.023135 |
| [4] |
Licausi F, Giorgi FM, Zenoni S, et al. Genomic and transcriptomic analysis of the AP2/ERF superfamily in vitis vinifera[J]. BMC Genomics, 2010, 11(1): 719-734. DOI:10.1186/1471-2164-11-719 |
| [5] |
Allen MD, Yamasaki K, OhmeTakagi M, et al. A novel mode of DNA recognition by a beta-sheet revealed by the solution structure of the GCC-box binding domain in complex with DNA[J]. EMBO J, 1998, 17(18): 5484-5496. DOI:10.1093/emboj/17.18.5484 |
| [6] |
Sakuma Y, Liu Q, Dubouzet JG, et al. DNA-binding specificity of the ERF/AP2 domain of Arabidopsis DREBs, transcription factors involved in dehydration- and cold-inducible gene expression[J]. Biochem Biophys Res Commun, 2002, 290(3): 998-1009. DOI:10.1006/bbrc.2001.6299 |
| [7] |
Stockinger EJ, Gilmour SJ, Thomashow MF. Arabidopsis thaliana CBF1 encodes an AP2 domain-containing transcriptional activator that binds to the C-repeat/DRE, a cis-acting DNA regulatory element that stimulates transcription in response to low temperature and water deficit[J]. Proc Nati Acad Sci USA, 1997, 94(3): 1035-1040. DOI:10.1073/pnas.94.3.1035 |
| [8] |
Ohme-Takagi M, Shinshi H. Ethylene-inducible DNA binding proteins that interact with an ethylene-responsive element[J]. Plant Cell, 1995, 7(2): 173-182. DOI:10.1105/tpc.7.2.173 |
| [9] |
Welsch R, Maass D, Voegel T, et al. Transcription factor RAP2. 2 and its interacting partner SINAT2: stable elements in the carotenogenesis of Arabidopsis leaves[J]. Plant Physiology, 2007, 145(3): 1073-1085. DOI:10.1104/pp.107.104828 |
| [10] |
Nakano T, Suzuki K, Fujimura T, et al. Genome-wide analysis of the ERF gene family in Arabidopsis and rice[J]. Plant Physiology, 2006, 140(2): 411-432. DOI:10.1104/pp.105.073783 |
| [11] |
Song XM, Li Y, Hou XL. Genome-wide analysis of the AP2/ERF transcription factor superfamily in Chinese cabbage(Brassica rapa ssp. pekinensis)[J]. BMC Genomics, 2013, 14(1): 573-586. DOI:10.1186/1471-2164-14-573 |
| [12] |
Agarwal P, Agarwal PK, Nair S, et al. Stress-inducible DREB2A transcription factor from Pennisetum glaucum is a phosphoprotein and its phosphorylation negatively regulates its DNA-binding activity[J]. Mol Genet Genomics, 2007, 277(2): 189-198. DOI:10.1007/s00438-006-0183-z |
| [13] |
Wang C, Wang H, Zhang J, et al. A seed-specific AP2-domain transcription factor from soybean plays a certain role in regulation of seed germination[J]. 中国科学:生命科学, 2008, 51(4): 336-345. |
| [14] |
Ohto MA, Fischer RL, Goldberg RB, et al. Control of seed mass by APETALA2[J]. Proc Nati Acad Sci USA, 2005, 102(8): 3123-3128. DOI:10.1073/pnas.0409858102 |
| [15] |
Lasserre E, Jobet E, Llauro C, et al. AtERF38(At2g35700), an AP2/ERF family transcription factor gene from Arabidopsis thaliana, is expressed in specific cell types of roots, stems and seeds that undergo suberization[J]. Plant Physiology & Biochemistry, 2008, 46(12): 1051-1061. |
| [16] |
Qi WW, Sun F, Wang QJ, et al. Rice ethylene-response AP2/ERF factor OsEATB restricts internode elongation by down-regulating a gibberellin biosynthetic gene[J]. Plant Physiology, 2011, 157(1): 216-228. DOI:10.1104/pp.111.179945 |
| [17] |
Zhao Y, Cheng SF, Song YL, et al. The Interaction between Rice ERF3 and WOX11 promotes crown root development byregulating gene expression involved in cytokinin signaling[J]. Plant Cell, 2015, 27(9): 2469-2483. DOI:10.1105/tpc.15.00227 |
| [18] |
Sun W, Gao D, Xiong Y, et al. Hairy Leaf 6, an AP2/ERF transcription factor, interacts with OsWOX3B and regulates trichome formation in rice[J]. Mol Plant, 2017, 10(11): 1417-1433. DOI:10.1016/j.molp.2017.09.015 |
| [19] |
Krizek BA. AINTEGUMENTA and AINTEGUMENTA-LIKE6 act redundantly to regulate Arabidopsis floral growth and patterning[J]. Plant Physiology, 2009, 150(4): 1916-1929. DOI:10.1104/pp.109.141119 |
| [20] |
滕飞. 拟南芥AP3/ERF基因ERF055调控茎端分生组织发育的功能研究[D]. 泰安: 山东农业大学, 2013.
|
| [21] |
Colle M, Weng YQ, Kang YY, et al. Variation in cucumber(Cucumis sativus L.)fruit size and shape results from multiple components acting pre-anthesis and post-pollination[J]. Planta, 2017, 246(4): 641-658. DOI:10.1007/s00425-017-2721-9 |
| [22] |
Wang CH, Xin M, Zhou XY, et al. The novel ethylene-responsive factor CsERF025 affects the development of fruit bending in cucumber[J]. Plant Mol Biol, 2017, 95(4-5): 1-13. |
| [23] |
Yang Z, Tian L, Latoszek GM, et al. Arabidopsis, ERF4 is a transcriptional repressor capable of modulating ethylene and abscisic acid responses[J]. Plant Mol Biol, 2005, 4: 585-596. |
| [24] |
Rashotte AM, Mason MG, Hutchison CE, et al. A subset of Arabidopsis AP2 transcription factors mediates cytokinin responses in concert with a two-component pathway[J]. Proc Nati Acad Sci USA, 2006, 103(29): 11081-11085. DOI:10.1073/pnas.0602038103 |
| [25] |
Iwase A, Mitsuda N, Koyama T, et al. The AP2/ERF transcription factor WIND1 controls cell dedifferentiation in Arabidopsis[J]. Current Biology Cb, 2011, 21(6): 508-514. DOI:10.1016/j.cub.2011.02.020 |
| [26] |
Asahina M, Satoh S. Spatially selective hormonal control of RAP2. 6L and ANAC071 transcription factors involved in tissue reunion in Arabidopsis[J]. Proc Nati Acad Sci USA, 2011, 108(38): 16128-16132. DOI:10.1073/pnas.1110443108 |
| [27] |
Lu X, Zhang L, Zhang F, et al. AaORA, a trichome-specific AP2/ERF transcription factor of Artemisia annua, is a positive regulator in the artemisinin biosynthetic pathway and in disease resistance to Botrytis cinerea[J]. New Phytol, 2013, 198(4): 1191-1202. DOI:10.1111/nph.12207 |
| [28] |
Menke FL, Champion A, Kijne JW, et al. A novel jasmonate- and elicitor-responsive element in the periwinkle secondary metabolite biosynthetic gene strinteracts with a jasmonate- and elicitor-inducible AP2-domain transcription factor, ORCA2[J]. EMBO Journal, 1999, 18(16): 4455-4463. DOI:10.1093/emboj/18.16.4455 |
| [29] |
张蒙. 中国红豆杉中JA信号调控紫杉醇生物合成模式研究[D]. 武汉: 华中科技大学, 2016.
|
| [30] |
De Sutter V, Vanderhaeghen R, Tilleman S, et al. Exploration of jasmonate signalling via automated and standardized transient expression assays in tobacco cells[J]. Plant Journal for Cell & Molecular Biology, 2005, 44(6): 1065-1076. |
| [31] |
De Boer K, Tilleman S, Pauwels L, et al. APETALA2/ETHYLENE RESPONSE FACTOR and basic helix-loop-helix tobacco transcription factors cooperatively mediate jasmonate-elicited nicotine biosynthesis[J]. Plant J, 2011, 66(6): 1053-1065. DOI:10.1111/tpj.2011.66.issue-6 |
| [32] |
Zeng JK, Li X, Xu Q, et al. EjAP2-1, an AP2/ERF gene, is a novel regulator of fruit lignification induced by chilling injury, via interaction with EjMYB transcription factors[J]. Plant Biotechnology Journal, 2015, 13(9): 1325-1334. DOI:10.1111/pbi.12351 |
| [33] |
Ma RF, Xiao Y, Lv ZY, et al. AP2/ERF transcription factor, Ii049, positively regulates lignan biosynthesis in isatis indigotica through activating salicylic acid signaling and lignan/lignin pathway genes[J]. Frontiers in Plant Science, 2017, 8: 1361-1377. DOI:10.3389/fpls.2017.01361 |
| [34] |
Gilmour SJ, Sebolt AM, Salazar MP, et al. Overexpression of the Arabidopsis CBF3 transcriptional activator mimics multiple biochemical changes associated with cold acclimation[J]. Plant Physiology, 2000, 124(4): 1854-1865. DOI:10.1104/pp.124.4.1854 |
| [35] |
Sharabi SM, Samach A, Porat R, et al. Overexpression of the CBF2 transcriptional activator in Arabidopsis suppresses the responsiveness of leaf tissue to the stress hormone ethylene[J]. Plant Biology, 2010, 12(4): 630-638. |
| [36] |
张弘. 中国樱桃PpcERF基因克隆与功能研究[D]. 金华: 浙江师范大学, 2015.
|
| [37] |
Dossa K, Xin W, Li D, et al. Insight into the AP2/ERF transcription factor superfamily in sesame and expression profiling of DREB subfamily under drought stress[J]. BMC Plant Biology, 2016, 16(1): 171-186. DOI:10.1186/s12870-016-0859-4 |
| [38] |
Hattori Y, Nagai K, Furukawa S, et al. The ethylene response factors SNORKEL1 and SNORKEL2 allow rice to adapt to deep water[J]. Nature, 2009, 460(7258): 1026-1030. DOI:10.1038/nature08258 |
| [39] |
Park HY, Seok HY, Woo DH, et al. AtERF71/HRE2 transcription factor mediates osmotic stress response as well as hypoxia response in Arabidopsis[J]. Biochem Biophys Res Commun, 2011, 414(1): 135-141. DOI:10.1016/j.bbrc.2011.09.039 |
| [40] |
Zhao Y, Wei T, Yin KQ, et al. Arabidopsis RAP2. 2 plays an important role in plant resistance to Botrytis cinerea and ethylene responses[J]. New Phytol, 2012, 195(2): 450-460. DOI:10.1111/j.1469-8137.2012.04160.x |
| [41] |
Gu C, Guo ZH, Hao PP, et al. Multiple regulatory roles of AP2/ERF transcription factor in angiosperm[J]. Botanical Studies, 2017, 58(1): 6-13. DOI:10.1186/s40529-016-0159-1 |
| [42] |
Mishra S, Phukan UJ, Tripathi V, et al. PsAP2 an AP2/ERF family transcription factor from Papaver somniferum enhances abiotic and biotic stress tolerance in transgenic tobacco[J]. Plant Mol Biol, 2015, 89(1-2): 173-186. DOI:10.1007/s11103-015-0361-7 |
| [43] |
Xiao YY, Chen JY, Kuang JF, et al. Banana ethylene response factors are involved in fruit ripening through their interactions with ethylene biosynthesis genes[J]. J Exp Bot, 2013, 64(8): 2499-2510. DOI:10.1093/jxb/ert108 |
| [44] |
Smaczniak C, Li N, Boeren S, et al. Proteomics-based identification of low-abundance signaling and regulatory protein complexes in native plant tissues[J]. Nat Protoc, 2012, 7(12): 2144-2158. DOI:10.1038/nprot.2012.129 |