2. 浙江大学生命科学学院,杭州 310027
2. College of Life Sciences, Zhejiang University, Hangzhou 310027
随着温室效应的加剧,近些年全球气候逐渐变暖,20世纪全球平均气温约提高0.6℃[1],经预测,到2050年平均温度将可能提高2-5℃[2]。高温会对植物的生长发育产生直接影响,在较高环境温度下植物会出现叶片偏下、开花提前及下胚轴伸长等表型,与光形态建成对应,植物在环境温度升高条件下的一些形态上的改变被称为热形态建成(thermomorphogenesis)。现有研究表明,调控这些表型的信号途径与植物遮荫下的分子信号途径多有重叠,表型也十分类似[3]。
光和环境温度作为最重要的环境信号,极大地影响了植物的生长发育。光敏色素互作因子(Phytochrome interacting factors,PIFs),是一类可以与光受体光敏色素蛋白相互作用的bHLH家族转录因子。PIFs在光调控的植物生长发育过程中十分重要,能够影响植物的下胚轴伸长、幼苗的暗形态建成、促进避荫反应等[4]。29℃的温和高温(mild high temperature)能诱导拟南芥野生型幼苗下胚轴伸长,而PIF4缺失突变体pif4对温和高温不敏感,下胚轴几乎不伸长[4]。隐花素(cryptochrome,CRY)是光裂解酶类似的蓝光受体,在拟南芥苗期主要调控其下胚轴伸长。CRY1对温度响应十分重要,而温和高温下CRY1通过与PIF4以蓝光依赖的形式相结合,进而调控PIF4在温和高温下的转录活性[5]。最近,有报道指出光敏色素B(phyB)不仅可作为光的受体,同时能感应环境温度的变化[6],但温和高温下phyB到PIF4的信号传递目前不是很清楚。
另外一条独立于PIF4的调控植物响应光和温度信号,控制下胚轴伸长的信号通路包含COP1(CONSTITUTIVE PHOTOMORPHOGENIC 1)和HY5(ELONGATED HYPOCOTYL 5)。HY5是bZIP家族转录因子,不但在白光下抑制拟南芥下胚轴伸长,同时在常温下也能通过与PIF4竞争下游靶标基因启动子来抑制下胚轴伸长[7]。在黑暗条件下或环境温度升高后,HY5被泛素连接酶COP1降解,从而解除了HY5对下胚轴伸长的抑制效果[7-10]。COP1在细胞核中发挥作用,但温和高温下COP1如何进核的分子机理目前不清楚。
温度的升高会使生长素表达水平上升,进而调节下胚轴伸长等植物生长活动[11]。有研究表明当温度升高时,拟南芥中生长素响应基因表达水平也有所变化[12]。拟南芥在响应温和高温时,PIF4能够直接结合生长素合成基因,如YUC8,TAA1,CYP79B2等基因的启动子。PIF4通过激活TAA1和CYP79B2基因的表达,进而介导吲哚乙酸(IAA)的合成[13]。而IAA的积累能够促进AUX/IAA蛋白的降解,进而消除对ARF蛋白的抑制作用。最后ARF通过激活SAUR19等基因的表达促进胚轴伸长[14]。其它植物激素,例如,油菜素内脂BR和赤霉素GA在植物热形态建成中也发挥着重要的作用[12, 15-17]。
本实验利用拟南芥PIF4缺失突变体和PIF4过表达等材料开展研究。遗传分析表明,PIF4、PIF5和PIF7中,PIF4在热形态建成中的作用最重要。通过对RNA-Seq结果和结合CHIP-Seq分析找到了拟南芥热形态建成中PIF4调控的下游基因以及结合靶标基因。
1 材料与方法 1.1 材料实验所使用的植物材料有拟南芥(Arabidopsis thaliana)Columbia生态型(Col-0),拟南芥Columbia背景突变体pif4-101,拟南芥野生型背景下PIF4: PIF4-myc转基因植株,拟南芥pif4-101背景下PIF4: PIF4,PIF4: PIF5,PIF4: PIF7转基因植株。所使用的菌株有大肠杆菌(Escherichia coli)TOP10,农杆菌菌株(Agrobacterium tumefaciens)GV3101。这些材料均为本实验室获得和保存。
1.2 方法 1.2.1 拟南芥的平皿培养将同批收获的种子放于1.5 mL离心管中,用1 mL体积比为50%的酒精迅速清洗1次,用体积比0.3%的NaClO消毒15 min后,于超净台内用无菌水清洗3次,播种在含质量浓度1.2%蔗糖的1/2MS固体培养基上。4℃春化2-3 d后置于22℃光照培养箱,光16 h/暗8 h。
1.2.2 下胚轴长度测量和分析将相应材料于22℃培养4 d后,在第5天光照开始时移至29℃培养4 d,以22℃培养的植物作为对照。用相机分别拍下对应天数的不同材料的拟南芥幼苗生长照片,利用ImageJ软件进行数据测量分析。
1.2.3 RNA-Seq拟南芥Columbia野生型(wild-type,WT)和pif4-101突变体4℃春化2 d后于22℃培养7 d。在光照开始时将幼苗移至29℃处理1 d,以22℃培养的植物作为对照。于黑暗周期结束后收集拟南芥幼苗用于RNA-Seq。根据Illumina标准步骤构建cDNA文库,Illumian Hiseq3000测序(晶能公司)。GO分析采用AgriGO(v2.0)进行分析[18]。
1.2.4 染色质免疫共沉淀-定量PCR(ChIP-qPCR)分别将拟南芥Columbia野生型及PIF4: PIF4-myc转基因植株于22℃培养,第12天29℃处理24 h,以22℃培养的植物作为对照。用甲醛固定液固定,后用2.5 mol/L甘氨酸解交联,MilliQ水清洗材料3次。去除水分,冻于-80℃保存。将材料用液氮研磨成粉末,加入30 mL EB1,用2层Miracloth过滤。离心后用1 mL EB2重悬,离心并用300 μL EB3重悬沉淀,并覆盖于600 μL EB3上。离心并用300 μL NLB重悬沉淀,使用biorupt进行超声,超30 s停30 s。离心取部分上清做Input。其余每个重复加入50 μL protein A beads去除与beads的非特异性结合。取上清加入2 μg myc抗体,于4℃旋转过夜。次日加protein A beads 110 μL,4℃ 2 h后低速离心留beads。分别用低盐、高盐和LiCl和TE缓冲液清洗beads。分两次加入300 μL洗脱缓冲液65℃煮15 min,合并。用洗脱缓冲液将Input补齐到500 μL,与免疫沉淀后的DNA-蛋白复合物一起于65℃解交联约6 h。用蛋白酶K去除蛋白,RnaseI去除RNA。沉淀DNA,并用无水乙醇及70%乙醇清洗沉淀,晾干后用无菌水溶解。
以MYC抗体沉淀的DNA为模板,进行qPCR扩增,以actin启动子序列作为阴性对照。qPCR实验使用SYBR Premix Ex TaqTM Ⅱ(TaKaRa)试剂,在Bio-Rad CFX96TM仪器上完成。反应体系按照TaKaRa及Bio-Rad CFX96TM仪器配制。得到数据后分析处理并作图。
1.2.5 实时荧光定量PCR实验(qRT-PCR)RNA提取使用植物总RNA提取试剂盒(TIANGEN),总RNA保存于-80℃。cDNA使用M-MLV(TaKaRa)反转录得到,保存在-20℃。
qRT-PCR实验使用SYBR Premix Ex TaqTM Ⅱ(TaKaRa)试剂,在Bio-Rad CFX96TM仪器上完成。反应体系按照TaKaRa及Bio-Rad CFX96TM仪器配制。得到数据后分析处理并作图。
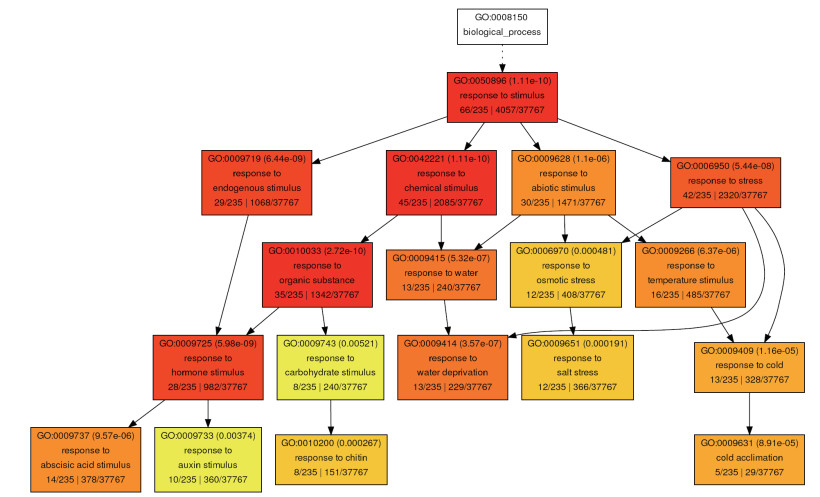
2 结果 2.1 PIF4在拟南芥热形态建成中的主要生物学功能本实验在拟南芥PIF4突变体pif4-101背景下采用PIF4自身启动子分别回补PIF4、PIF5和PIF7,利用这3种互补遗传材料来研究PIF4及同源蛋白在拟南芥热形态建成中的生物学功能的区别。野生型拟南芥和pif4-101、PIF4: PIF5、PIF4: PIF7在22℃生长8 d后,4种材料的幼苗下胚轴长度基本一致(图 1-A),而PIF4: PIF4的下胚轴则明显长于其他材料。而在22℃生长4 d、29℃生长4 d的条件下,PIF4: PIF4的下胚轴伸长更长,野生型和PIF4: PIF5的下胚轴比22℃时伸长的幅度接近,而pif4-101、PIF4: PIF7则几乎不伸长(图 1-B)。

|
| 图 1 PIF4突变体的表型遗传互补分析 以PIF4突变体pif4-101为背景,转入PIF4自身启动子驱动的PIF4(PIF4: PIF4)、PIF5(PIF4: PIF5)和PIF7(PIF4: PIF7)。野生型WT和PIF4突变体pif4-101植物为对照。A:发芽8 d后的照片。B:代表性株系下胚轴长度分析,误差线为8颗幼苗的统计数据;C:PIF4、PIF5和PIF7在各幼苗中的表达量。相对表达量为选定基因与内参基因Actin的表达量的比值。野生型中22℃的表达值归一化为1,误差线为3个样品的统计数据。*:P < 0.05;**:P < 0.01;***:P < 0.001。B图采用t-test统计分析,C图采用ANOVA统计分析 |
基因表表达分析(图 1-C)表明,PIF4: PIF4植物中正常条件下PIF4表达量比野生型中高,可能是PIF4: PIF4植物正常温度条件下下胚轴比较长的原因。这些结果表明,拟南芥热形态建成中PIF7的功能较小,PIF5也具有PIF4类似的功能,但PIF4的功能明显比PIF5的功能强。因此,PIF4在拟南芥热形态建成中具有重要的功能。
2.2 热形态建成中PIF4调控的下游基因为了在全基因组水平上理解热形态建成过程中PIF4调控的下游基因表达,将拟南芥野生型和突变体pif4-101在22℃培养7 d后,于光周期中白光开始时用29℃处理24 h,在黑暗结束后取样,以不处理的植物作对照,收集幼苗全苗进行转录组测序(RNA-Seq)。对转录组数据进行统计分析后显示,在拟南芥野生型与pif4-101中分别有602个基因和512个基因受温和高温处理上调,并有529个基因和484个基因受温和高温处理下调。其中有248个基因只在野生型中特异上调,198个基因只在野生型中特异下调(图 2,附表)。

|
| 图 2 PIF4突变体与野生型热处理的RNA-SEQ分析 野生型WT和PIF4突变体pif4-101温和高温(29℃)的处理后进行RNA-Seq,维恩图分析基因表达差异(29℃与22℃条件下基因表达值之比FC > 2,q值< 0.05) |
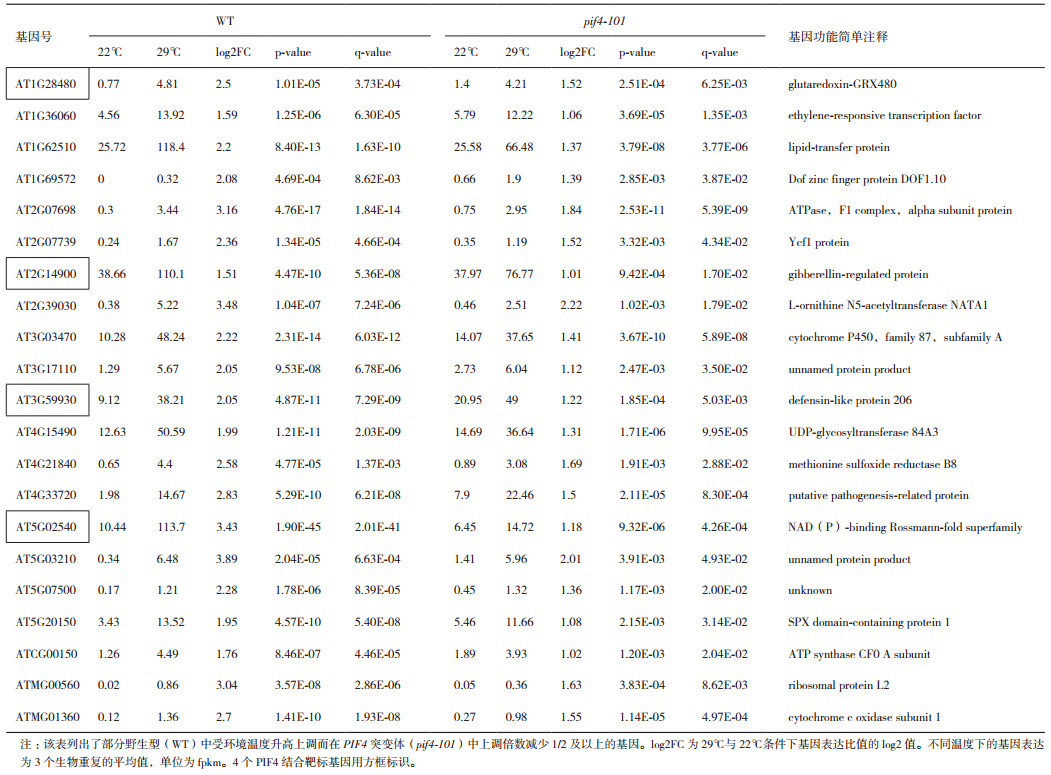
对于这些只在野生型中上调的248基因,我们认为它们是PIF4依赖的热响应基因。GO分析(图 3)显示,PIF4依赖的热响应基因中主要参与脱落酸响应、生长素响应、几丁质响应、脱水响应、盐胁迫、冷驯化等过程。综上所述,这些结果表明,PIF4依赖的热响应基因主要参与植物激素信号通路和逆境应答。
|
| 图 3 PIF4依赖的热响应基因GO分析 将PIF4依赖的热响应基因(248)采用AgriGO(v2.0)进行分析,参数为默认参数 |
对于354个在野生型和突变体pif4-101中上调的下游基因,对其上调幅度也进行了比较。结果(表 1)显示,有21个基因在pif4-101突变体中的上调倍数是野生型(WT)中上调倍数的1/2及以下,我们把这些基因视作是部分依赖PIF4调控的热形态建成基因。
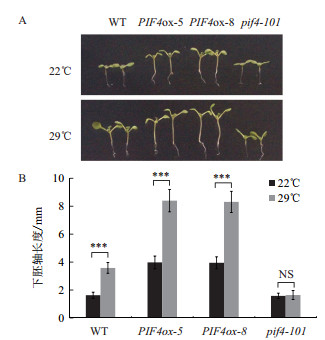
为了检测上述PIF4依赖和部分依赖的基因是否是PIF4的直接靶标基因,我们在拟南芥野生型背景下过表达myc标签融合的PIF4-myc材料(PIFox)。野生型WT和PIF4ox-5、PIF4ox-8转基因材料在22℃正常生长8 d后,PIF4过表达转基因材料表现出下胚轴变长的表型。在22℃生长4 d后移入29℃培养4 d,野生型的下胚轴出现一定程度的伸长,PIF4过表达转基因材料下胚轴伸长现象更加明显(图 4)。这表明PIF4过表达材料正常温度下可能已经激活部分下游基因,并且能响应环境温度升高的变化,这些材料可以用于后续结合靶标基因的实验。
|
| 图 4 PIF4过表达的表型分析 将myc标签与PIF4融合,在PIF4启动子的驱动下,在野生型背景下筛选PIF4-myc过表达转基因材料(PIFox)后进行表型分析。PIF4突变体pif4-101植物为对照。误差线为24个幼苗的统计数据。***:P < 0.001;NS:P > 0.05不显著(t-test) |
在研究植物激素BR调控下胚轴伸长过程时,王志勇课题组采用pifq四突变体背景下自身启动子驱动的PIF4-myc过表达材料,正常生长条件下进行染色质共免疫沉淀深度测序(ChIP-Seq)分析,得到4 583个候选的PIF4结合靶标基因[15-16]。
为了研究热形态建成中PIF4的结合靶标基因,我们比较了上述248个PIF4依赖的基因以及PIF4候选的结合靶标基因,得到70个热形态建成过程中被PIF4结合的候选靶标基因。
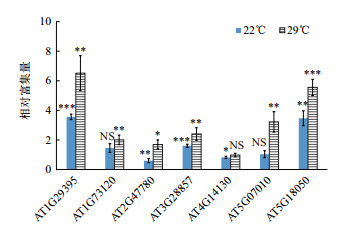
我们随机选择了7个靶标基因,包括AT1G29395(COR413IM1)、AT1G73120(F-box蛋白)、AT2G47780(LDAP2)、AT3G28857(PRE5)、AT4G14130(XTR7)、AT5G07010(ATST2A)、AT5G18050(SAUR22),利用PIF4-myc过表达材料(PIFox-5)进行ChIP-qPCR实验。结果(图 5)表明,AT1G29395、AT3G28857、AT5G18050这3个基因的启动子区域在正常温度下就可以被PIF4-myc富集(≥1.5倍,P < 0.05),在环境温度升高后7个候选靶标基因中,6个基因(AT1G29395、AT1G73120、AT2G47780、AT3G28857、AT5G07010、AT5G18050)的启动子区域被PIF4-myc富集(≥1.5倍,P < 0.05),说明通过生物信息分析得到的70个PIF4结合靶标基因中绝大部分为实际PIF4结合靶标基因(附表)。
|
| 图 5 PIF4体内结合下游基因启动子的分析 将PIF4-myc过表达转基因材料在常温(22℃)和温和高温(29℃)下处理,采用myc抗体进行染色质共免疫沉淀(ChIP),针对7个候选结合靶标基因启动子区域设计引物,采用定量PCR(qPCR)进行比较。相对富集量为PIF4-myc在目标基因启动子和内参基因(Actin)启动子上富集量之间的比值。数据为3次ChIP的平均值。***:P < 0.001;**:P < 0.01;*:P < 0.05;NS:P > 0.05不显著(t-test) |
我们也比较了上述21个PIF4部分依赖的基因以及PIF4候选的结合靶标基因,得到4个PIF4热形态建成过程中被PIF4结合的靶标基因(表 1)。我们把这74个PIF4结合靶标基因视作热形态建成中PIF4结合的靶标基因,这些靶标基因是否被PIF4直接结合可以采用凝胶迁移EMSA实验进行证明。
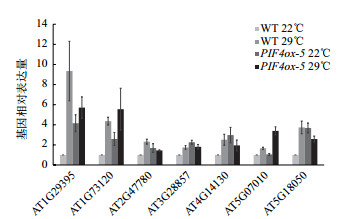
2.4 PIF4过表达对下游基因的影响为了研究PIF4过表达对下游基因表达的影响,将野生型(WT)和PIF4-myc过表达材料(PIF4ox-5)进行不同温度处理,检测以上提到的7个基因的表达情况。结果(图 6)表明,6个基因(AT1G29395、AT1G73120、AT2G47780、AT3G28857、AT4G14130、AT5G18050)正常温度下PIF4ox-5中的表达量高于野生型中的表达量,只有1个基因(AT5G07010)在温和高温处理后PIF4ox-5中表达量才高于野生型中的表达量。
|
| 图 6 PIF4过表达对下游基因表达的影响 将野生型(WT)和PIF4-myc过表达转基因材料(PIF4ox-5)在常温(22℃)和温和高温(29℃)下处理1 d,针对7个候选直接靶标基因编码区域设计引物,采用定量RT-PCR(qRT-PCR)对这些基因的表达量进行比较。基因相对表达量为目标基因表达量和内参基因(Actin)表达量之间的比值。数据为3次生物重复的平均值 |
bHLH家族转录因子PIF4与关敏色素phyB互作,参与植物光形态建成,后续更多的研究表明,PIF4是植物热形态建成中的重要调控节点[19-23]。我们的实验结果也表明,在几个PIFs中,PIF4在植物热形态建成中功能最重要。PIF4基因的表达受时钟的调控,同时也受温和高温的诱导[20, 24]。PIF4蛋白也受到翻译后修饰调控。例如,在白光下phyB和BIN2可以磷酸化PIF4,导致PIF4的降解[25-26]。环境温度升高后磷酸化的PIF4积累[27],这主要与DET1稳定PIF4有关[7]。此外,PAR1和CRY1也可以与PIF4互作,温和高温下抑制PIF4的转录活性[5, 28-29]。
PIF4的靶标基因中有很多是植物激素合成和响应基因[12, 15],因此一般认为PIF4在热形态建成中主要通过植物激素,如生长素途径发挥作用[11, 23]。事实上,生长素合成基因比如YUC8和生长素响应基因比如IAA4和IAA29,受温和高温的诱导上调并且依赖于PIF4[13, 20]。但是,PIF4在全基因组水平的热形态建成中的下游基因并未见报道。我们采用RNA-Seq的方法,得到了温和高温处理后受PIF4调控的269个下游基因(包括21个PIF4部分依赖的下游基因)。这些基因中生长素响应途径的基因(IAA6、IAA19、IAA34、SAUR22、SAUR57、SAUR59)和脱落酸响应途径的基因得到显著富集。此外,一些细胞壁伸长相关基因和逆境胁迫响应基因也得到富集。我们的结果为理解植物热形态建成的基因调控提供了帮助。
这些PIF4调控的热形态建成下游基因中,据估算大约有25%为热形态建成中PIF4调控的结合靶标基因,其他为PIF4间接调控的下游基因。例如,编码膜蛋白COR413IM1的基因AT1G29395被证明是PIF4的直接调控基因。温和高温处理下的该基因的诱导表达依赖于PIF4,野生型中组成性表达PIF4正常温度下该基因上调表达。而编码细胞壁成分、改变有关的木葡聚糖内糖基转移酶XTR7的基因AT4G14130并不被PIF4直接结合,是PIF4间接调控的下游基因。温和高温处理下该基因的诱导表达也依赖于PIF4,野生型中组成性表达PIF4正常温度下该基因也上调表达。
此外,PIF4对下游靶标基因的结合能力也有差别,当PIF4组成性表达后,有的靶标基因在正常温度下就可以被PIF4结合,而有些靶标基因只有在温和温度处理后才被PIF4结合。例如,编码磺基转移酶ATST2A的AT5G07010启动子只有在温和高温处理下才显著被PIF4结合。温和高温处理下该基因的诱导表达依赖于PIF4,野生型中组成性表达PIF4,正常温度下该基因没有上调表达,但温和高温处理后该基因明显诱导表达。这些结果暗示其他的温和高温处理依赖的转录调控因子可能与PIF4一起调控了该基因的表达。PIF4过表达植物正常条件下下胚轴伸长,温和高温下有进一步的伸长,而在检测的7个基因中,只有1个基因(ATST2A)的表达量在PIF4过表达植物中温和高温下有进一步上调表达,说明一部分PIF4调控基因在温和高温下对下胚轴的伸长非常重要。
4 结论获得拟南芥热形态建成中248个PIF4完全依赖和21个PIF4部分依赖的下游基因,这些下游基因主要参与植物激素和非生物逆境响应。这些PIF4调控的热形态建成下游基因有74个基因是PIF4结合靶标候选基因。选择7个PIF4候选靶标基因,有6个基因被PIF4直接结合。
注:本论文中附表见电子版(http://biotech.caas.cn)
| [1] |
IP CC. Climate Change 2001:The Scientific Basis[M]. The Third Assessment Report of Working Group. Cambridge: Cambridge Univ Press, 2001.
|
| [2] |
Meinshausen M, Meinshausen N, Hare W, et al. Greenhouse-gas emission targets for limiting global warming to 2 degrees C[J]. Nature, 2009, 458(7242): 1158-U96. DOI:10.1038/nature08017 |
| [3] |
Proveniers MCG, Van Zanten M. High temperature acclimation through PIF4 signaling[J]. Trends in Plant Science, 2013, 18(2): 59-64. DOI:10.1016/j.tplants.2012.09.002 |
| [4] |
Castillon A, Shen H, Huq E. Phytochrome interacting factors:central players in phytochrome-mediated light signaling networks[J]. Trends in Plant Science, 2007, 12(11): 514-521. DOI:10.1016/j.tplants.2007.10.001 |
| [5] |
Ma D, Li X, Guo Y, et al. Cryptochrome 1 interacts with PIF4 to regulate high temperature-mediated hypocotyl elongation in response to blue light[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(1): 224-229. DOI:10.1073/pnas.1511437113 |
| [6] |
Jung J H, Domijan M, Klose C, et al. Phytochromes function as thermosensors in Arabidopsis[J]. Science, 2016, 354(6314): 886-889. DOI:10.1126/science.aaf6005 |
| [7] |
Gangappa SN, Kumar SV. DET1 and HY5 control PIF4-mediated thermosensory elongation growth through distinct mechanisms[J]. Cell Reports, 2017, 18(2): 344-351. DOI:10.1016/j.celrep.2016.12.046 |
| [8] |
Park YJ, Lee HJ, Ha JH, et al. COP1 conveys warm temperature information to hypocotyl thermomorphogenesis[J]. New Phytologist, 2017, 215(1): 269-280. DOI:10.1111/nph.14581 |
| [9] |
Delker C, Sonntag L, James GV, et al. The DET1-COP1-HY5 pathway constitutes a multipurpose signaling module regulating plant photomorphogenesis and thermomorphogenesis[J]. Cell Reports, 2014, 9(6): 1983-1989. DOI:10.1016/j.celrep.2014.11.043 |
| [10] |
Osterlund MT, Hardtke CS, Wei N, et al. Targeted destabilization of HY5 during light-regulated development of Arabidopsis[J]. Nature, 2000, 405(6785): 462-466. DOI:10.1038/35013076 |
| [11] |
Gray WM, Ostin A, Sandberg G, et al. High temperature promotes auxin-mediated hypocotyl elongation in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(12): 7197-7202. DOI:10.1073/pnas.95.12.7197 |
| [12] |
Stavang JA, Gallego-Bartolome J, Gomez MD, et al. Hormonal regulation of temperature-induced growth in Arabidopsis[J]. Plant J, 2009, 60(4): 589-601. DOI:10.1111/j.1365-313X.2009.03983.x |
| [13] |
Sun J, Qi L, Li Y, et al. PIF4-Mediated activation of YUCCA8 expression integrates temperature into the auxin pathway in regulating Arabidopsis hypocotyl growth[J]. PLoS Genetics, 2012, 8(3): e1002594. DOI:10.1371/journal.pgen.1002594 |
| [14] |
Franklin KA, Lee SH, Patel D, et al. PHYTOCHROME-INTERACTING FACTOR 4(PIF4)regulates auxin biosynthesis at high temperature[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(50): 20231-20235. DOI:10.1073/pnas.1110682108 |
| [15] |
Oh E, Zhu JY, Wang ZY. Interaction between BZR1 and PIF4 integrates brassinosteroid and environmental responses[J]. Nature Cell Biology, 2012, 14(8): 802-U64. DOI:10.1038/ncb2545 |
| [16] |
Oh E, Zhu JY, Bai MY, et al. Cell elongation is regulated through a central circuit of interacting transcription factors in the Arabidopsis hypocotyl[J]. Elife, 2014, 3. |
| [17] |
Bai MY, Shang JX, Oh E, et al. Brassinosteroid, gibberellin and phytochrome impinge on a common transcription module in Arabidopsis[J]. Nature Cell Biology, 2012, 14(8): 810-U78. DOI:10.1038/ncb2546 |
| [18] |
Tian T, Liu Y, Yan H, et al. agriGO v2. 0:a GO analysis toolkit for the agricultural community, 2017 update[J]. Nucleic Acids Research, 2017, 45(W1): W122-W129. DOI:10.1093/nar/gkx382 |
| [19] |
Huq E, Quail PH. PIF4, a phytochrome-interacting bHLH factor, functions as a negative regulator of phytochrome B signaling in Arabidopsis[J]. Embo Journal, 2002, 21(10): 2441-2450. DOI:10.1093/emboj/21.10.2441 |
| [20] |
Koini M A, Alvey L, Allen T, et al. High temperature-medated adaptations in plant architecture require the bHLH transcription factor PIF4[J]. Current Biology, 2009, 19(5): 408-413. DOI:10.1016/j.cub.2009.01.046 |
| [21] |
Leivar P, Quail PH. PIFs:pivotal components in a cellular signaling hub[J]. Trends in Plant Science, 2011, 16(1): 19-28. DOI:10.1016/j.tplants.2010.08.003 |
| [22] |
Bae G, Choi G. Decoding of light signals by plant phytochromes and their interacting proteins[J]. Annual Review of Plant Biology, 2008, 59: 281-311. DOI:10.1146/annurev.arplant.59.032607.092859 |
| [23] |
Quint M, Delker C, Franklin KA, et al. Molecular and genetic control of plant thermomorphogenesis[J]. Nature Plants, 2016, 15190. |
| [24] |
Nusinow DA, Helfer A, Hamilton EE, et al. The ELF4-ELF3-LUX complex links the circadian clock to diurnal control of hypocotyl growth[J]. Nature, 2011, 475(7356): 398-U161. DOI:10.1038/nature10182 |
| [25] |
Lorrain S, Allen T, Duek PD, et al. Phytochrome-mediated inhibition of shade avoidance involves degradation of growth-promoting bHLH transcription factors[J]. Plant J, 2008, 53(2): 312-323. |
| [26] |
Bernardo-Garcia S, De Lucas M, Martinez C, et al. BR-dependent phosphorylation modulates PIF4 transcriptional activity and shapes diurnal hypocotyl growth[J]. Genes & Development, 2014, 28(15): 1681-1694. |
| [27] |
Foreman J, Johansson H, Hornitschek P, et al. Light receptor action is critical for maintaining plant biomass at warm ambient temperatures[J]. Plant J, 2011, 65(3): 441-452. DOI:10.1111/tpj.2011.65.issue-3 |
| [28] |
Roig-Villanova I, Bou-Torrent J, Galstyan A, et al. Interaction of shade avoidance and auxin responses:a role for two novel atypical bHLH proteins[J]. Embo Journal, 2007, 26(22): 4756-4767. DOI:10.1038/sj.emboj.7601890 |
| [29] |
Hao Y, Oh E, Choi G, et al. Interactions between HLH and bHLH factors modulate light-regulated plant development[J]. Molecular Plant, 2012, 5(3): 688-697. DOI:10.1093/mp/sss011 |