2. 山东农业大学植物保护学院 山东省农业微生物重点实验室,泰安 271018
2. Department of Plant Pathology, College of Plant Protection, Shandong Agricultural University/Shandong Province Key Laboratory of Agricultural Microbiology, Tai'an, 271018
植物的生长发育以及对外界环境的适应都离不开植物激素的调节,这种调节依赖特异的转录因子对下游基因的表达调控。基因的特异性转录需要将调控信号恰当地传递到负责转录的通用RNA聚合酶Ⅱ装置上。然而,绝大多数转录因子并不能够直接与RNA聚合酶Ⅱ结合,两者之间还需要一个桥梁来共同形成转录预起始复合物(Pre-initiation-complex,PIC)[1]。近年来的研究发现中介体能够招募转录因子形成转录复合物,使转录因子与RNA聚合酶Ⅱ连接起来,并对转录调控的进行发挥重要作用。Mediator25(MED25)是中介体的一个亚基,它可以与激素途径中的特定转录因子互作并调控下游基因的转录,从而影响植物的各种生理过程。
1 植物中介体的发现和研究中介体首次在酵母中发现,其含有25个亚基,而哺乳动物和植物中则分别由31和35个亚基组成[2-4]。中介体复合物一般由4个模块组装而成,分别是头部(head)、中部(middle)、尾部(tail)以及细胞周期素依赖性激酶(Cyclin-dependent kinase 8,CDK8)[1]。其中头部模块主要与RNA聚合酶Ⅱ相结合,由MED6、MED8、MED11、MED17、MED18、MED19、MED20和MED22等亚基组成;中部模块同样可与RNA聚合酶Ⅱ结合,同时对中介体结构稳定性十分重要,由MED1、MED4、MED5、MED7、MED9、MED10、MED21和MED31等亚基组成;尾部模块提供了多种激活子的结合位点,由MED2、MED3、MED14、MED15和MED16等亚基组成[5];激酶域主要包括:CDK8、Cyclinc、MED12和MED13,它们可以与以上3个模块组成的中介体中心结构可逆的结合,通过磷酸化转录因子、破坏尾部或与转录激活子的结合,以及影响中介体与RNA聚合酶Ⅱ的结合来负向调控转录[5]。对中介体复合物的结构分析,进一步明确了中介体复合物与RNA聚合酶Ⅱ的结合方式对稳定转录体系的重要意义。
植物中介体的研究相对比较滞后,直到2007年才被发现并分离出来[6]。研究人员首先通过序列比对的方法寻找酵母中介体亚基在拟南芥中的同源基因,却仅仅找到MED6、MED7和MED10等少数亚基,另外多数亚基的序列相似性很低,因此难以判定植物中是否也存在中介体复合物[2]。于是研究人员用拟南芥MED6和MED7蛋白制备抗体,以拟南芥悬浮细胞总蛋白为材料进行免疫沉淀,并结合质谱分析最终分离到了拟南芥中的中介体复合物[6]。研究发现拟南芥中介体共有21个保守的亚基和6个植物特有的亚基。与中介体复合物同时洗脱的还有RNA聚合酶Ⅱ的一些肽段,证实得到的是一个中介体-RNA聚合酶Ⅱ的复合物。尽管没有同时将中介体的激酶域洗脱下来,研究人员通过同源比对发现拟南芥中也存在同源的MED12、MED13、CDK8和CycC(Cyclin C)。有意思的是,在分离到的中介体亚基中,有一些是之前报道过的调控特定途径的蛋白,如MED25/PFT1(PHYTO-CHROME AND FLOWERING TIME)参与开花调控途径[7],MED14/SWP(STRUWWELPETER)参与细胞的增殖[8],CDK8/HEN3(HUA ENHANCER3)参与花器官的发育等[9]。拟南芥中介体复合物的分离纯化为人们研究植物体内的转录调控提供了一个新的切入点。
2 植物MED25的研究进展 2.1 植物MED25的发现MED25在植物中的发现存在一定的偶然性。首先,在筛选拟南芥诱导突变群体的过程中鉴定出一个隐性pft1突变体,其在亚适光条件下显示晚开花表型。这表明突变基因调控避荫性(shade-avoidance)[7]。随后的分析发现PFT1受上游的光敏色素B(phyB)控制,进而调控下游控制开花时间基因(FLOWERING TIME,FT)的表达来正向调节开花过程[7]。虽然当时的研究已经揭示PFT1是一种与动物的转录激活子相似的核蛋白,然而直到后来,在纯化植物中介体复合物的研究中才发现PFT1编码的其实就是中介体亚基MED25[6]。自此,med25突变体被应用在不同的功能筛选中,并鉴定出各种由PFT1/MED25调控的植物生理功能[10-11]。除已知MED25参与了开花时间的调控外,还有报道MED25亚基参与了生物和非生物胁迫响应,并与转录因子DREB2A(DRE-binding protein 2A)、ZFHD1(Zinc-finger homeo domain1)以及MYB-LIKE相互作用[12-13]。MED8和MED25一样参与植物的开花调控和对尖孢镰刀菌Fusarium oxysporum的抗性[13],以及植物器官大小的调控[14-15]。最新证据表明MED25介导的某些功能在双子叶植物和单子叶植物之间是保守的[16]。随着拟南芥中介体复合物的分离,对MED25功能的研究近几年成为了热点。
2.2 MED25的功能结构域酵母不含有MED25的同源基因,在植物中也仅发现单基因编码的MED25[17]。目前还不清楚MED25在中介体复合物中的位置。然而,从MED25与许多转录因子以及MED16(一种尾部模块亚基)相互作用推断MED25最可能与中介体复合物的尾部模块相关[17-18]。
MED25含有执行特定功能的多个蛋白域,分别是von Willebrand Factor Type A(vWF-A)结构域、MD结构域、ACID结构域、以及Q-rich结构域(图 1)[16]。其中位于MED25蛋白N末端的vWF-A结构域是其与中介体复合物尾部亚基MED16相互作用所必需的[18];中间域(Middle Domain)目前未发现执行具体功能;激活子相互作用结构域(Activator interacting domain,ACID)是MED25与转录激活因子(如AP2/ERF和MYC)以及抑制因子(如JAZ蛋白)相互作用所必需的[19-21]。位于MED25蛋白C-末端保守的富含谷氨酰胺的(Q-rich)结构域,它可能参与到转录激活功能[6-7, 12]。Q-rich区的存在或缺失以及长度都会影响MED25调控植物开花的功能。MED25在Q-rich区域的两种不同的选择性剪切在进化上是保守,并且与该蛋白执行的不同功能有关[22]。MED25不直接与RNA聚合酶Ⅱ相互作用,而是通过招募转录因子形成转录复合物,然后将转录因子与RNA聚合酶Ⅱ连接起来,从而激活或抑制下游基因的转录[12, 23-24]。
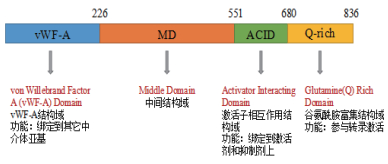
|
| 图 1 拟南芥MED25(AtMED25)的蛋白功能结构域 |
近年来的研究结果表明MED25在多种激素相关途径中发挥着重要的作用,它既可以单一调控某一种激素信号途径,又可以在植物激素信号途径crosstalk中起重要调节作用[16]。
3.1 MED25与茉莉酸(JA)信号途径在茉莉酸信号途径中,中介体亚基MED25作为一个正向调节因子,参与拟南芥对生物和非生物胁迫的反应[25]。MYC2是内源激素茉莉酸信号通路的核心转录因子,调控茉莉酸反应的多个方面,包括根生长、机械受伤反应和抗病反应等[26]。在低茉莉酸水平下,JAZ蛋白复合物通过直接与MYC2互作、招募组蛋白脱乙酰酶(HDAC)或抑制RNA聚合酶Ⅱ介导复合物的活性等作用来抑制MYC2的转录活性,从而调节茉莉酸介导的基因表达的不同方面[27-30];在高茉莉酸水平下,JAs被COI1-JAZ共受体复合物感知,这导致JAZ抑制子作用的破坏和MYC2转录因子的释放,从而调控多种茉莉酸介导的抗胁迫反应[26]。在发现MED25之前,MYC和其它调节JA反应途径的转录因子招募中介体复合物的机制尚不清楚[13]。近年来研究发现,MED25突变后影响了MYC2的功能及茉莉酸诱导的基因表达[21]。一系列生化证据表明,在茉莉酸诱导的基因转录过程中,MED25的一个重要作用是把RNA聚合酶Ⅱ及相关的转录辅助因子招募到MYC2靶标基因的启动子区域,进而精细调节MYC2的转录调控活性[31]。另外,研究发现MED25与各种茉莉酸相关的转录因子之间的相互作用进一步证明了MED25参与茉莉酸信号传导的作用[32]。首先,使用MED25的保守ACID结构域作为诱饵,对来自62个家族的1589个拟南芥属转录因子进行高通量酵母双杂交筛选[33-34],鉴定了8个与MED25相互作用的转录因子,其中有6个属于AP2-ERF家族中,一个属于GARP-G2样家族中,另一个属于B-Box家族中[32, 35]。研究发现AP2-ERF家族的3个转录因子(At3g23220、At3g23240和At4g18450)作用于茉莉酸信号传导途径的下游[32]。它们与目的基因启动子的GCC-box结合,然后通过AP2-ERF家族蛋白的C末端保守EDLL基序与MED25的ACID域结合,并将转录信息传递给RNA聚合酶Ⅱ,介导了一系列基因的表达[23]。另外,包括ORA59和ERF1(Ethylene response factor 1)以及MYC家族的转录因子包括MYC2、MYC3和MYC4等与JA响应相关的转录因子也显示与MED25有直接相互作用[19-20]。相关研究显示,MED25作用于ERF1、ORA59和MYC2的下游,并且对于JA介导的病原体防御基因如PDF1.2的激活是必需的[13, 19, 36]。另外有证据显示,MED25对于MYC2介导的昆虫防卫基因VSP的激活也是必须的[13, 19]。
与MYC转录因子类似,JAZ家族的抑制子如JAZ9通过其保守的JAS(JA Associated)域与MED25直接互作,而这种互作会干扰MED25与MYC2的互作,证明抑制子和激活子通过与MED25的竞争结合来调控茉莉酸信号途径[21, 37]。
此外,MED25还可与中介体复合物的CDK8互作,正向调控JA响应的基因如PDF1.2的表达[38]。另外CDK8与MED25都可与调控蜡质合成的AP2/ERF家族转录因子WIN1(WAX Inducer 1)互作,共同调控表皮角质层的形成和植物的防卫反应[39]。
3.2 MED25与生长素(Auxin)信号途径研究发现MED25还参与调控生长素介导的根系发育过程。在拟南芥中MED25的功能丧失会促进主根和侧根的生长以及侧根和不定根形成[40-41]。而MED25过表达可以减少这些反应[41]。已知生长素(吲哚-3-乙酸,IAA)在侧根发育的各个阶段发挥重要作用[42-43]。IAA或合成植物生长素物质如萘乙酸(NAA)的施用可刺激侧根的形成,而用极性生长素转运抑制剂处理能抑制侧根发生[44-45]。有研究表明MED25在侧根原基形成期通过负向调节植物生长素运输和相关基因表达来调节侧根的形成[41]。
最新研究表明,中介体复合物CDK8结构域的一部分:MED13/MAB2/GCT(MACCHI-BOU2/GRAND CENTRAL)通过与生长素信号途径的ARF7和ARF19以及Aux/IAA14的结合来调节生长素信号传导[46]。在低生长素水平下,Aux/IAA14通过与共阻遏蛋白TPL以及中介体亚基MED13形成复合物来抑制核心中介体复合物与RNA聚合酶Ⅱ之间的相互作用,从而抑制ARF7和ARF9的作用。在高生长素水平下,Aux/IAA14降解并且导致TPL和MED13从复合物解离[46]。ARF7和ARF9通过与LBD16(LOBDOMAIN16)启动子中发现的生长素应答元件(AuxE)结合,并与MED25相互作用以募集核心介体复合物和RNA聚合酶Ⅱ,从而调控基因的表达[46-47]。
3.3 MED25与乙烯(Ethylene)信号途径MED25在乙烯信号途径中既可以作为负调控元件调节植物的抗病反应,又可以作为正调控元件调节植物响应缺素症。
小麦白粉病作为一种世界性病害,在很长的一段时间里,科学家对防御其的转录调控系统缺乏深入的了解[48-49]。最近研究发现,小麦中介体亚基编码基因TaMED25的敲除可以降低小麦对白粉病的易感性,同时乙烯信号途径的转录因子TaEIL1也可以负调控小麦对白粉病的抗性[50]。将TaMED25的ACID结构域和TaEIL1进行酵母双杂交实验,发现两者能够互作,并共同调节乙烯信号下游靶基因TaERF1的表达,进而激活乙烯对病原菌信号的响应,负向调控对白粉病的抗性[51]。
营养缺素方面,MED25通过乙烯信号途径正向调控缺铁反应。med25突变体显示出对铁元素缺乏的敏感性增加,表明MED25是这些反应的正调节剂[52-53]。MED25通过与乙烯信号传导组分EIN3和EIL1以及调控铁元素缺乏反应的另一介体复合物亚基MED16相互作用[54],将铁缺乏信号传递给RNA聚合Ⅱ以激活参与铁稳态的基因IRT1(铁调节的转运蛋白1)和FRO2(铁还原氧化酶2)的表达[55-56],以此来调控应对铁元素缺乏的胁迫反应。
3.4 MED25与脱落酸(ABA)信号途径MED25在蛋白水平上可以与ABI5相互作用,并负向调控脱落酸(ABA)信号途径[31]。研究发现,拟南芥med25突变体对ABA抑制种子萌发和幼苗生长更加敏感,显示MED25也参与调控ABA信号途径[31, 57-58]。拟南芥MED25在脱落酸信号转导途径中主要是与ABI5(ABA-INSENSITIVE 5)相互作用,而ABI5是一个bZIP家族转录因子,调控种子成熟和萌发过程中的ABA信号[59]。有趣的是,酵母双杂交实验发现MED25并不与ABI5的转录激活区互作,因此这种调控还存在某种未知的机制[31]。与茉莉酸信号转导途径中所起的正调控作用相反,MED25负调控依赖于ABI5的ABA信号途径。此外med25突变体内的ABI5蛋白水平显著升高,是导致突变体对ABA超敏感的原因[31]。进一步的实验表明MED25与ABI5转录因子相互作用并负向调控ABI5蛋白的降解过程,实现对早期幼苗发育的ABA应答基因(EM1、EM6和RAB18)的表达来影响ABA信号通路[31, 60]。
3.5 MED25共调节多种激素途径截至目前,对中介体亚基MED25调控激素方面的报道主要集中在茉莉酸、生长素、乙烯和脱落酸信号途径中,而MED25又极有可能是多个激素途径crosstalk的共享元件。前文提到,MYC2是拟南芥中茉莉酸(JA)信号传导途径主要调节剂[28],有趣的是MYC2还是JA信号与其它激素如脱落酸(ABA)、水杨酸(SA)、赤霉素(GAs)和生长素(IAA)信号传导途径之间的连接纽带[26]。MYC2通过其N末端的转录激活结构域(TAD)特异性地与中介体亚基MED25的ACID结构域和MD中间结构域的10个氨基酸部分互作,来募集转录起始所需的中介体复合物进而对激素信号作出响应[19-20]。可以看出MED25作为多个激素信号通路调节机制的共享元件,既可以和转录激活子(如MYCs和ERFs)也可以和抑制子(JAZs、ABI5)相互作用,也可以和中介体复合物的其它亚基或组成部分(MED16和CDK8等)相互作用,改变中介体亚基与转录因子和RNA聚合酶Ⅱ之间的相互作用,以实现多种激素信号的精细调控。最新的研究表明,MED25参与JA和乙烯共同调节的植物的抗病途径[61]。胍丁胺香豆酰基转移酶(AtACT)是防御坏死病原体的次生代谢物质-羟基肉桂酸酰胺(HCAAs)生物合成反应的关键酶[62-63],而ORA59是茉莉酸和乙烯介导的植物防御反应的关键正调控因子[64]。研究发现ORA59作用于AtACT的启动子区,MED25可以促进ORA59同源二聚体对AtACT启动子的活性从而合成HCAAs来共同调节茉莉酸和乙烯的响应。
4 总结与展望中介体是研究真核生物基因转录调控的重要切入点,也是目前研究的热点。中介体亚基MED25广泛参与了茉莉酸、生长素、乙烯、脱落酸等激素途径的信号转导、调控和转录相关的多个生物学过程。可以看出,MED25是一个多功能的中介体亚基,如同一个信号处理器,接收来自不同激素受体的信号,并输出合适的信号给转录机器以启动激素介导的特异性生理反应,使得特定的信号途径得以精细而准确的调控。MED25可以通过与激素途径中特异的转录因子以及中介体复合物相互作用来正向或负向调控激素信号转导,但MED25如何实现多激素途径信号的准确传递还不清楚,其分子机制还需要进一步的探明。在这种多维信号转导的过程中,中介体的构象是否发生变化,以及哪些转录因子会使其构象发生变化进而调控植物生长发育的机理尚不清楚。
目前MED25参与其它植物激素途径如水杨酸,赤霉素和油菜素内酯等方面的报道还未出现,但已有报道其它中介体亚基参与了这些激素途径,如MED14、MED15、MED16和MED19a调节水杨酸引发的对活体和半活体病原菌的免疫性[64-66];番茄中介体亚基SlMED18正调节赤霉素生物合成和信号转导以及生长素的极性运输等[67]。相信不远的未来,MED25在这些激素途径中的作用也会被一一揭示。
同样因为中介体对于植物转录调控和胁迫响应的重要作用,其亚基或复合物也成为病原菌效应分子攻击的对象。有报道拟南芥霜霉菌(Hyaloperono-spora arabidopsidis)分泌的RXLR家族效应子HaRxL44能与MED19a互作,导致MED19a通过蛋白酶体途径降解,使水杨酸信号通路的相关基因表达降低,茉莉素/乙烯信号途径得到加强,改变了植物激素的平衡,最终成功突破植物的防卫反应[66]。MED25作为多个激素信号通路调节机制的共享元件,也极有可能参与相关的病原菌和植物的互作反应中。
综上所述,MED25如何与其它亚基共同调节激素介导的信号途径,以及MED25如何调控多种激素拮抗和交叉的分子机理,都将是研究者的关注热点。目前对于MED25在植物生长发育、抗病、抗虫、抗逆境胁迫等方面的作用研究也在蓬勃开展,希望本文能够为进一步研究以MED25为代表的中介体功能提供参考。
| [1] |
Flanagan PM, Rd KR, Sayre MH, et al. A mediator required for activation of RNA polymerase Ⅱ transcription in vitro[J]. Nature, 1991, 350(6317): 436-438. DOI:10.1038/350436a0 |
| [2] |
Mathur S, Vyas S, Kapoor S, et al. The mediator complex in plants: structure, phylogeny, and expression profiling of representative genes in a dicot(Arabidopsis)and a Monocot(Rice)during reproduction and abiotic stress[J]. Plant Physiology, 2011, 157(4): 1609-27. DOI:10.1104/pp.111.188300 |
| [3] |
Allen BL, Taatjes DJ. The mediator complex: a central integrator of transcription[J]. Nature Reviews Molecular Cell Biology, 2015, 16(3): 155-166. DOI:10.1038/nrm3951 |
| [4] |
Samanta S, Thakur JK. Importance of mediator complex in the regulation and integration of diverse signaling pathways in plants[J]. Front Plant Sci, 2015, 6(7): 388-394. |
| [5] |
Poss Z C, Ebmeier CC, Taatjes DJ. The mediator complex and transcription regulation[J]. Critical Reviews in Biochemistry & Molecular Biology, 2013, 48(6): 575-608. |
| [6] |
Bäckström S, Elfving N, Nilsson R, et al. Purification of a plant mediator from Arabidopsis thaliana identifies PFT1 as the Med25 subunit[J]. Molecular Cell, 2007, 26(5): 717. DOI:10.1016/j.molcel.2007.05.007 |
| [7] |
Cerdán PD, Chory J. Regulation of flowering time by light quality[J]. Nature, 2003, 423(6942): 881. |
| [8] |
Autran D, Jonak C, Belcram K, et al. Cell numbers and leaf development in Arabidopsis: a functional analysis of the STRUWWELPETER gene[J]. Embo Journal, 2002, 21(22): 6036. DOI:10.1093/emboj/cdf614 |
| [9] |
Wang W, Chen X. HUA ENHANCER3 reveals a role for a cyclin-dependent protein kinase in thespecification of floral organ identity in Arabidopsis[J]. Development, 2004, 131(13): 3147. DOI:10.1242/dev.01187 |
| [10] |
Zheng W, Zhai Q, Sun J, et al. Bestatin, an inhibitor of aminopeptidases, provides a chemical genetics approach to dissect jasmonate signaling in Arabidopsis[J]. Plant Physiology, 2006, 141(4): 1400-1413. DOI:10.1104/pp.106.080390 |
| [11] |
Seguelaarnaud M, Smith C, Uribe MC, et al. The Mediator complex subunits MED25/PFT1 and MED8 are required for transcriptional responses to changes in cell wall arabinose composition and glucose treatment in Arabidopsis thaliana[J]. BMC Plant Biology, 2015, 15(1): 1-13. DOI:10.1186/s12870-014-0410-4 |
| [12] |
Elfving N, Davoine C, Benlloch R, et al. The Arabidopsis thaliana Med25 mediator subunit integrates environmental cues to control plant development[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(20): 8245. DOI:10.1073/pnas.1002981108 |
| [13] |
Kidd BN, Edgar CI, Kumar KK, et al. The mediator complex subunit PFT1 is a key regulator of jasmonate-dependent defense in Arabidopsis.[J]. Plant Cell, 2009, 21(8): 2237-2252. DOI:10.1105/tpc.109.066910 |
| [14] |
Xu, Li. Control of final organ size by Mediator complex subunit 25 in Arabidopsis thaliana[J]. Development, 2011, 138(20): 4545-4554. DOI:10.1242/dev.071423 |
| [15] |
Xu R, Li Y. The Mediator complex subunit 8 regulates organ size in Arabidopsis thaliana[J]. Plant Signaling & Behavior, 2012, 7(2): 182-183. |
| [16] |
Kazan K. The Multitalented MEDIATOR25[J]. Front Plant Sci, 2017, 8. |
| [17] |
Kidd B N, Cahill D M, Manners J M, et al. Diverse roles of the Mediator complex in plants.[J]. Seminars in Cell & Developmental Biology, 2011, 22(7): 741-748. |
| [18] |
Larsson M, Uvell H, Sandström J, et al. Functional studies of the yeast med5, med15 and med16 mediator tail subunits.[J]. PLoS One, 2013, 8(8): e73137. DOI:10.1371/journal.pone.0073137 |
| [19] |
ÇevikV, KiddB N, ZhangP, 等. MEDIATOR25 acts as an integrative hub for the regulation of jasmonate-responsive gene expression in Arabidopsis[J]. Plant Physiology, 2012, 160(1): 541-555. |
| [20] |
Fernández-Calvo P, Solano R. The Arabidopsis bHLH transcription factors MYC3 and MYC4 are targets of JAZ repressors and act additively with MYC2 in the activation of jasmonate responses[J]. Plant Cell, 2011, 23(2): 701-15. DOI:10.1105/tpc.110.080788 |
| [21] |
Zhang F, Yao J, Ke J, et al. Structural basis of JAZ repression of MYC transcription factors in jasmonate signalling[J]. Nature, 2015, 525(7568): 269-73. DOI:10.1038/nature14661 |
| [22] |
Rival P, Press M O, Bale J, et al. The conserved PFT1 tandem repeat is crucial for proper flowering in Arabidopsis thaliana[J]. Genetics, 2014, 198(2): 747-754. DOI:10.1534/genetics.114.167866 |
| [23] |
Yang Y, Li L, Qu L J. Plant Mediator complex and its critical functions in transcription regulation[J]. Journal of Integrative Plant Biology, 2016, 58(2): 106-118. DOI:10.1111/jipb.v58.2 |
| [24] |
Dolan W L, Chapple C. Conservation and divergence of mediator structure and function: insights from plants[J]. Plant & Cell Physiology, 2016, 58(1): pcw176. |
| [25] |
Kazan, Kemal. Diverse roles of jasmonates and ethylene in abiotic stress tolerance[J]. Trends in Plant Science, 2015, 20(4): 219-229. DOI:10.1016/j.tplants.2015.02.001 |
| [26] |
Kemal Kazan, John M. Manners. MYC2: the master in action[J]. Molecular Plant, 2013, 6(3): 686-703. DOI:10.1093/mp/sss128 |
| [27] |
Kazan, Kemal, John M. JAZ repressors and the orchestration of phytohormone crosstalk[J]. Trends in Plant Science, 2012, 17(1): 22-31. DOI:10.1016/j.tplants.2011.10.006 |
| [28] |
Boter M, Ruízrivero O, Abdeen A, et al. Conserved MYC transcription factors play a key role in jasmonate signaling both in tomato and Arabidopsis[J]. Genes Dev, 2004, 18(13): 1577-1591. |
| [29] |
Lorenzo O, Chico JM, Sánchez-Serrano JJ, et al. JASMONATE-INSENSITIVE1 encodes a MYC transcription factor essential to discriminate between different jasmonate-regulated defense responses in Arabidopsis[J]. Plant Cell, 2004, 16(7): 1938. DOI:10.1105/tpc.022319 |
| [30] |
Dombrecht B, Xue GP, Sprague SJ, et al. MYC2 differentially modulates diverse jasmonate-dependent functions in Arabidopsis[J]. Plant Cell, 2007, 19(7): 2225-2245. DOI:10.1105/tpc.106.048017 |
| [31] |
Chen R, Jiang H, Li L, et al. The Arabidopsis mediator subunit MED25 differentially regulates jasmonate and abscisic acid signaling through interacting with the MYC2 and ABI5 transcription factors[J]. Plant Cell, 2012, 24(7): 2898. DOI:10.1105/tpc.112.098277 |
| [32] |
O u, B in, Y in, et al. A high-throughput screening system for Arabidopsis transcription factors and its application to Med25-dependent transcriptional regulation[J]. Molecular Plant, 2011, 4(3): 546-555. DOI:10.1093/mp/ssr002 |
| [33] |
Riechmann JL, Heard J, Martin G, et al. Arabidopsis transcription factors: genome-wide comparative analysis among eukaryotes[J]. Science, 2000, 290(5499): 2105. DOI:10.1126/science.290.5499.2105 |
| [34] |
Gong W, Shen YP, Ma LG, et al. Genome-wide ORFeome cloning and analysis of Arabidopsis transcription factor genes[J]. Plant Physiology, 2004, 135(2): 773-82. DOI:10.1104/pp.104.042176 |
| [35] |
Nakano T, Suzuki K, Fujimura T, et al. Genome-wide analysis of the ERF gene family in Arabidopsis and rice[J]. Plant Physiology, 2006, 140(2): 411. DOI:10.1104/pp.105.073783 |
| [36] |
Kidd BN, Aitken EA, Schenk PM, et al. Plant mediator: mediating the jasmonate response[J]. Plant Signal Behav, 2010, 5(5): 718-720. |
| [37] |
Zhang F, Ke J, Zhang L, et al. Structural insights into alternative splicing-mediated desensitization of jasmonate signaling[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(7): 1720. DOI:10.1073/pnas.1616938114 |
| [38] |
Zhu Y, Mengiste T. CYCLIN-DEPENDENT KINASE8 differentially regulates plant immunity to fungal pathogens through kinase-dependent and -independent functions in Arabidopsis[J]. Plant Cell, 2014, 26(10): 4149-4170. DOI:10.1105/tpc.114.128611 |
| [39] |
Sela D, Buxdorf K, Shi J X, et al. Overexpression of AtSHN1/WIN1 provokes unique defense responses[J]. PLoS One, 2013, 8(7): e70146. DOI:10.1371/journal.pone.0070146 |
| [40] |
黄维娜, 康玉凡. 乙烯在幼苗根生长发育中调控作用的研究进展[J]. 中国农学通报, 2013(12): 6-12. |
| [41] |
Rayagonzález J, Ortizcastro R, Ruízherrera L F, et al. PHYTOCHROME AND FLOWERING TIME1/MEDIATOR25 regulates lateral root formation via auxin signaling in Arabidopsis[J]. Plant Physiology, 2014, 165(2): 880. DOI:10.1104/pp.114.239806 |
| [42] |
Casimiro I, Beeckman T, Graham N, et al. Dissecting Arabidopsis, lateral root development[J]. Trends in Plant Science, 2003, 8(4): 165-71. DOI:10.1016/S1360-1385(03)00051-7 |
| [43] |
Hirota A, Kato T, Fukaki H, et al. The auxin-regulated AP2/EREBP Gene PUCHI is required for morphogenesis in the early lateral root primordium of Arabidopsis[J]. Plant Cell, 2007, 19(7): 2156. DOI:10.1105/tpc.107.050674 |
| [44] |
Casimiro I, Marchant A, Bhalerao RP, et al. Auxin transport promotes Arabidopsis lateral root initiation.[J]. Plant Cell, 2001, 13(4): 843. DOI:10.1105/tpc.13.4.843 |
| [45] |
Himanen K, Boucheron E, Vanneste S, et al. Auxin-mediated cell cycle activation during early lateral root initiation[J]. Plant Cell, 2002, 14(10): 2339-2351. DOI:10.1105/tpc.004960 |
| [46] |
Ito J, Fukaki H, Onoda M, et al. Auxin-dependent compositional change in mediator in ARF7- and ARF19-mediated transcription.[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(23): 6562. DOI:10.1073/pnas.1600739113 |
| [47] |
Muto H, Watahiki MK, Nakamoto D, et al. Specificity and similarity of functions of the Aux/IAA genes in auxin signaling of Arabidopsis revealed by promoter-exchange experiments among MSG2/IAA19, AXR2/IAA7, and SLR/IAA14[J]. Plant Physiology, 2007, 144(1): 187-96. DOI:10.1104/pp.107.096628 |
| [48] |
Bourras S, Mcnally KE, Ben-David R, et al. Multiple avirulence loci and allele-specific effector recognition control the Pm3 race-specific resistance of wheat to powdery mildew[J]. Plant Cell, 2015, 27(10): 2991. |
| [49] |
Parlange F, Roffler S, Menardo F, et al. Genetic and molecular characterization of a locus involved in avirulence of Blumeria graminis, f. sp. tritici, on wheat Pm3, resistance alleles[J]. Fungal Genetics & Biology, 2015, 82: 181-192. |
| [50] |
Duan X, Wang X, Fu Y, et al. TaEIL1, a wheat homologue of AtEIN3, acts as a negative regulator in the wheat-stripe rust fungus interaction[J]. Molecular Plant Pathology, 2013, 14(7): 728. DOI:10.1111/mpp.2013.14.issue-7 |
| [51] |
Liu J, Zhang T, Jia J, et al. The wheat mediator subunit TaMED25 interacts with the transcription factor TaEIL1 to negatively regulate disease resistance against powdery mildew[J]. Plant Physiology, 2016, 170(3): 1799. |
| [52] |
Kazan K, Manners JM. Linking development to defense: auxin in plant-pathogen interactions[J]. Trends in Plant Science, 2009, 14(7): 373-382. DOI:10.1016/j.tplants.2009.04.005 |
| [53] |
Carlos L, Romera FJ, García MJ, et al. Ethylene participates in the regulation of Fe deficiency responses in strategy Ⅰ plants and in rice[J]. Front Plant Sci, 2015, 6(155): 1056. |
| [54] |
Brumbarova, Tzvetina, Bauer, et al. Molecular mechanisms governing Arabidopsis iron uptake[J]. Trends in Plant Science, 2015, 20(2): 124-133. DOI:10.1016/j.tplants.2014.11.004 |
| [55] |
Yang Y, Ou B, Zhang J, et al. The Arabidopsis Mediator subunit MED16 regulates iron homeostasis by associating with EIN3/EIL1 through subunit MED25[J]. Plant Journal, 2014, 77(6): 838-51. DOI:10.1111/tpj.2014.77.issue-6 |
| [56] |
Zhang Y, Wu H, Wang N, et al. Mediator subunit 16 functions in the regulation of iron uptake gene expression in Arabidopsis[J]. New Phytologist, 2014, 203(3): 770-83. DOI:10.1111/nph.12860 |
| [57] |
Bu QY, Li HM, Zhao QZ, et al. The Arabidopsis RING finger E3 ligase RHA2a is a novel positive regulator of abscisic acid signaling during seed germination and early seedling development[J]. Plant Physiology, 2009, 150(1): 463-481. DOI:10.1104/pp.109.135269 |
| [58] |
Li H, Jiang H, Bu Q, et al. The Arabidopsis RING finger E3 ligase RHA2b acts additively with RHA2a in regulating abscisic acid signaling and drought response[J]. Plant Physiology, 2011, 156(2): 550-563. DOI:10.1104/pp.111.176214 |
| [59] |
Lopez-Molina L, Mongrand S, Chua NH. A postgermination developmental arrest checkpoint is mediated by abscisic acid and requires the ABI5 transcription factor in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(8): 4782-4787. DOI:10.1073/pnas.081594298 |
| [60] |
Lopezmolina L, Mongrand S, Mclachlin DT, et al. ABI5 acts downstream of ABI3 to execute an ABA-dependent growth arrest during germination[J]. Plant Journal, 2002, 32(3): 317-328. DOI:10.1046/j.1365-313X.2002.01430.x |
| [61] |
Li J, Zhang K, Meng Y, et al. Jasmonic acid/Ethylene signaling coordinates hydroxycinnamic acid amides biosynthesis through ORA59 transcription factor[J]. Plant Journal for Cell & Molecular Biology, 2018, 95(3): 444-457. |
| [62] |
Campos L, Lisón P, López-Gresa MP, et al. Transgenic tomato plants overexpressing tyramine N-hydroxycinnamoyltransferase exhibit elevated hydroxycinnamic acid amide levels and enhanced resistance to Pseudomonas syringae[J]. Mol Plant Microbe Interact, 2014, 27(10): 1159-1169. DOI:10.1094/MPMI-04-14-0104-R |
| [63] |
Atsushi M, Kenji M, Takeshi S, et al. Acquired immunity of transgenic torenia plants overexpressing agmatine coumaroyltransferase to pathogens and herbivore pests[J]. Scientific Reports, 2012, 2(689): 689. |
| [64] |
Canet J V, DobãN A, Tornero P. Non-recognition-of-BTH4, an Arabidopsis mediator subunit homolog, is necessary for development and response to salicylic acid[J]. Plant Cell, 2012, 24(10): 4220-4235. DOI:10.1105/tpc.112.103028 |
| [65] |
Zhang X, Wang C, Zhang Y, et al. The Arabidopsis mediator complex subunit16 positively regulates salicylate-mediated systemic acquired resistance and jasmonate/ethylene-induced defense pathways[J]. Plant Cell, 2012, 24(10): 4294. DOI:10.1105/tpc.112.103317 |
| [66] |
Caillaud M, Asai S, Rallapalli G, et al. A downy mildew effector attenuates salicylic acid-triggered immunity in arabidopsis by interacting with the host mediator complex[J]. PLoS Biology, 2013, 11(12): e1001732. DOI:10.1371/journal.pbio.1001732 |
| [67] |
Wang Y, Hu Z, Zhang J, et al. Silencing SlMED18, tomato mediator subunit 18 gene, restricts internode elongation and leaf expansion[J]. Scientific Reports, 2018, 8(1): 3285. DOI:10.1038/s41598-018-21679-1 |




