植物自身是一个复杂的微生态系统,栖息着大量微生物,其中尤以细菌数量最多,包括定殖于植物根际的根际细菌、附生于植物组织(茎、叶、花及果实等)表面的附生细菌、以及分布于各器官或组织内部的内生细菌[1-2],它们与宿主协同进化,在促进植物生长、抵御病虫害、增强寄主对环境胁迫的抗性方面具有重要作用。植物附生和内生细菌现已作为新型的微生物资源,研究者们在水稻、玉米、番茄、辣椒、白菜等多种作物中分离到具有固氮、促生和生防作用的菌株[3-5]。研究土壤以及植物附生和内生细菌的种类、分布、定殖规律,并比较它们相互间的异同,对筛选植物促生菌具有重要意义。
研究植物附生和内生细菌群落主要采用传统的分离培养方法,依靠形态特征、生理生化特性等进行分类鉴定和计数。但是,可培养细菌仅占自然界细菌总数的1%-10%,传统方法对细菌群落结构及多样性的认识是不全面和有选择性的[6]。近年来,以16S rRNA分析为基础的分子生物学手段的应用为细菌群落结构的解析提供了更高效的研究技术,如末端限制性片段长度多样性(Terminal restriction fragment length polymorphism,T-RFLP)、变性梯度凝胶电泳(Denaturing gel gradient electrophoresis,DGGE)等。这些不依赖于培养的分子生态学方法,通过对条带图谱和序列的分析,更加快速、完整地反映植物附生和内生细菌的群落结构和多样性[7-8]。
本实验以番茄(Solanum lycopersicum cv.)为研究对象,采用传统平板培养法结合DGGE技术对番茄根区和根际土壤以及各组织附生和内生细菌的数量、丰富度、多样性等进行定量和比较分析,明确土壤细菌以及组织附生、内生细菌间的分布规律及相关性,为筛选植物促生菌,提高植物的健康水平提理论依据。
1 材料与方法 1.1 材料供试番茄选用中杂302(S. lycopersicum cv. Zho-ngza No.302)由中国农业科学院蔬菜花卉研究所选育,番茄于2016年2月播种,4月定植于该所日光温室中,常规栽培管理,植株长势良好,至7月当番茄第四穗位果成熟时进行取样。
1.2 方法 1.2.1 番茄组织样品采集随机选取20株健康番茄植株,依次收集根系、成熟叶片、位于第2至第4叶片间的茎段、以及第2和第3穗成熟果实,各组织样品均无明显的损伤。根区土壤的收集采用抖落法[9],将松散与根系结合的土壤抖落后,过2 mm筛,去除根组织及大块土壤、石头块等。将各土壤样品和植物组织样品分别装入无菌自封袋中,4℃冰箱保存待用。3次生物学重复。
1.2.2 根区土壤中细菌的分离培养取番茄根区土壤(0.3 g),置于无菌三角瓶中,加入无菌PBS溶液(NaCl 8 g/L、KCl 0.2 g/L、Na2HPO4 1.44 g/L、KH2PO4 0.24 g/L,pH 7.4),于25℃、150 r/min振荡1 h后,静置10 min,取上清液用无菌水按10倍梯度稀释(10-1-10-6),稀释后的悬液分别取100 μL涂布于TSA培养基(胰蛋白胨15 g/L、大豆胨5 g/L、NaCl 5 g/L、琼脂15 g/L,pH 7.0),每个梯度悬液涂布3个平行,将培养皿置于37℃培养箱中倒置培养,12 h后观察细菌生长情况并计数。
1.2.3 根际和番茄组织附生细菌的分离培养取抖落根区土壤的番茄根以及茎、叶片等组织,置于无菌三角瓶中,加入含有0.01% Triton X-100的PBS溶液中,25℃、150 r/min振荡3 h,将振荡后的液体经5 000 r/min离心20 min,用无菌PBS溶液重悬沉淀,分别得到根际、茎和叶片的附生细菌悬液。将菌悬液用无菌水按10倍梯度稀释后,分别涂布于TSA培养基上,37℃倒置培养,并观察、计数。
1.2.4 番茄组织内生细菌的分离培养对上述经PBS溶液清洗的番茄根、茎和叶片,参照文献[10]方法进行表面灭菌(80%乙醇,2 min;5% NaClO溶液,10 min),用无菌水清洗3遍,并将清洗液涂布于TSA培养基,以确认表面灭菌是否彻底。参照文献[11-13]分离番茄组织内生细菌,用无菌刀片将各灭菌后的组织切成约0.5 cm × 0.5 cm的小块,置于无菌研钵中研磨,研磨物中加入PBS溶液于150 r/min振荡1 h后,经4层无菌纱布过滤,滤液5 000 r/min离心20 min,用无菌PBS溶液重悬沉淀,得各组织的内生细菌悬液。将细菌悬液用无菌水梯度稀释后涂布于TSA培养基上,37℃倒置培养后观察、计数。
对于果实中内生细菌的分离,将番茄果实经表面灭菌后(80%乙醇,2 min;5% NaClO溶液,10 min),在中部横切,分离其中果皮、胎座、种子及其表面胶状物等4个部分。将各部分研磨后,过滤、离心,分离各部分的内生细菌,并涂布TSA平板,观察、计数,方法同上。
1.2.5 番茄组织附生和内生细菌的16S rRNA拷贝数分析利用实时荧光定量PCR法计算各样品中细菌的16S rRNA拷贝数[14]。
16S rRNA拷贝数标准曲线的绘制:采用Easy-Pure Bacteria Genome DNA Kit(全式金,北京)提取大肠杆菌DH5α(Escherichia coli DH5α)的基因组DNA。采用细菌通用引物27F(5'-AGAGTTTGAT-CCTGGCTCAG-3')和1492R(5'-GGTTACCTTGTTA-CGACTT-3')扩增16S rRNA序列,采用Zymoclean Gel DNA Recovery Kit(ZYMO,USA)对PCR产物进行切胶回收,采用NanoDrop 2000测定其浓度。使用ddH2O稀释DNA至浓度为1、10-1、10-2、10-3、10-4、10-5和10-6 ng/μL(1 ng模板对应的16S rRNA拷贝数为6.14 × 108),并以此为模板,采用细菌通用引物SRV3-1(5'-CGGTCCAGACTCCTACGGG-3')和SRV3-2(5'-TTACCGCGGCTGCTGGCAC-3')对16S rRNA的V3区进行PCR扩增。Real-time PCR反应体系(20 μL):2 × Top Green qPCR SuperMix(全式金,北京)10 μL、引物(10 μmol/L)各0.5 μL、模板(10 ng/μL)1.0 μL,用ddH2O补足20 μL;反应程序为95℃ 5 min;95℃ 10 s,55℃ 10 s,72℃ 10 s,45个循环。以模板中16S rRNA拷贝数的lg值为横坐标,以Real-time PCR所得的Ct值为纵坐标,绘制标准曲线。
附生和内生细菌基因组DNA提取:将分离得到的番茄根区土壤、根际、组织附生和内生细菌悬液,利用FastDNATM Spin Kit for Soil试剂盒(MPbio,USA)进行基因组DNA提取,并采用DNA产物纯化试剂盒(天根,北京)对提取的DNA进行纯化。采用NanoDrop 2000(Thermo Scientific,USA)测定DNA浓度,用0.8%的琼脂糖凝胶电泳检测DNA的完整性。
细菌16S rRNA拷贝数分析:以纯化后的细菌基因组DNA为模板,利用细菌通用引物SRV3-1和SRV3-2进行PCR扩增,PCR反应体系和程序同1.2.5。根据Real-time PCR反应的Ct值,结合标准曲线计算出番茄各组织附生和内生细菌的16S rRNA拷贝数。
1.2.6 番茄组织附生和内生细菌的DGGE分析以番茄根区土壤、根际、组织附生和内生细菌基因组DNA为模板,采用通用引物341F-GC(5'-CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGCCTACGGGAGGCAGCAG-3')和518R(5'-ATTACCGCGGCTTGCTGG-3')扩增细菌16S rRNA的V3区片段。PCR反应体系(50 μL):2× Fast Pfu SuperMix(全式金,北京)25 μL、引物(10 μmol/L)各2 μL、模板(10 ng/μL)1 μL,用ddH2O补足至50 μL。PCR反应条件:95℃ 5 min;93℃ 30 s,65℃ 40 s,72℃ 1 min,10个循环;93℃ 30 s,60℃ 40 s,72℃ 1 min,10个循环;93℃ 30 s,55℃ 40 s,72℃ 1 min,9个循环;93℃ 30 s,55℃ 40 s,72℃ 5 min。PCR产物经0.8%琼脂糖凝胶电泳检测后,-20℃保存备用。
DGGE试验参照房嫚嫚等[15]方法并进行适当修改。PCR产物采用DcodeTM基因突变检测系统(Bio-Rad,USA)进行DGGE电泳,聚丙烯酰胺梯度为6%-12%,变性剂梯度为20%-55%(以尿素7 mol/L、甲酰胺40%时的变性剂浓度为100%)。电泳结束后,凝胶经SYBR Green I(Invitrogen,USA)染色后在成像系统中观察并拍照。使用Quantity One(v4.6.2,Bio-Rad)对DGGE图谱进行分析,2个样本的相似性通过戴斯系数(Cs)条带模式进行估测(公式1),用非加权平均法(UPGMA)检验各番茄样品的相似性。
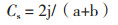 (1)
(1)
其中,j是两条DGGE泳道中共有的条带数,a和b分别为两条DGGE泳道各自的条带数。
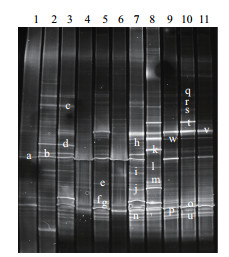
采用多样性指数(H)、丰富度指数(S)和均匀度指数(EH)等指标比较各样品的细菌多样性。H的计算公式如下:
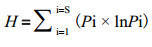 (2)
(2)
其中,S为样品中所有条带的数目总和,Pi为样品中某一条带(i)的强度在该样品所有条带总强度中所占的比率(%)。
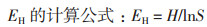 (3)
(3)
试验结果采用3次独立生物学重复的平均值±标准差(x±s)表示,采用Microsoft Excel 2010进行数据处理和作图,用DPS 2000软件对数据进行方差分析,运用邓肯氏新复极差法进行差异显著性(P < 0.05)检验。
2 结果 2.1 番茄组织附生和内生可培养细菌的分离与计数对番茄根区和根际土壤中的细菌进行分离培养并计数(图 1-A),根区和根际土壤中可培养细菌数量分别为8.19×107和1.81×109 CFU/g土壤,根际土壤中的细菌数量显著高于根区土壤,是根区土壤的约22倍。对番茄根、茎和叶片附生的可培养细菌进行计数(图 1-B),根附生的细菌数量最多(3.05×107 CFU/g FW),茎和叶片附生的细菌数量次之,分别为8.53×106和1.12×107 CFU/g FW。
|
| 图 1 番茄根区和根际土壤(A)、组织附生(B)和内生(C)可培养细菌数量 不同小写字母表示在P < 0.05水平差异显著,下同 |
图 1-C表示番茄各组织内生的可培养细菌数量,内生细菌的数量远低于附生细菌。各组织内生细菌数量依次为:根 > 果皮 > 叶片 > 胎座≈种子 > 茎 > 种子表面胶状物。根内生细菌数量最多,为1.16×105 CFU/g FW,而种子表面胶状物中细菌数量最少,只有6.62×102 CFU/g FW。
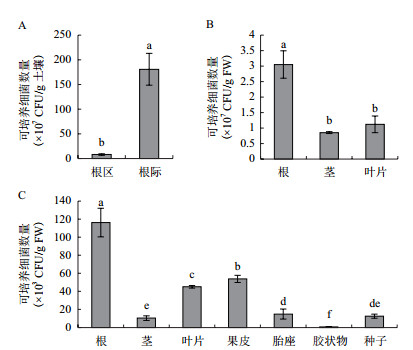
2.2 番茄组织附生和内生细菌的16S rRNA拷贝数分析采用Real-time PCR检测番茄土壤及各组织附生和内生的细菌16S rRNA拷贝数(图 2)。根区和根际土壤中16S rRNA拷贝数分别为1.96×109/g和5.13×109/g(图 2-A);根、茎和叶片的附生细菌16S rRNA拷贝数分别为3.05×108/g、2.22×108/g和1.60×108/g(图 2-B),与传统细菌培养法计数结果相比,变化趋势一致,但数量上较传统培养法多1-2个数量级。对于内生细菌16S rRNA拷贝数,较传统培养法,在数量上多2-4个数量级。其中,根中拷贝数最多,叶片与茎内生数量相当。果实的内生细菌16S rRNA拷贝数依次为果皮 > 种子 > 胎座 > 种子表面胶状物(图 2-C)。
|
| 图 2 番茄根区和根际土壤(A)、组织附生(B)和内生(C)细菌16S rRNA拷贝数 |
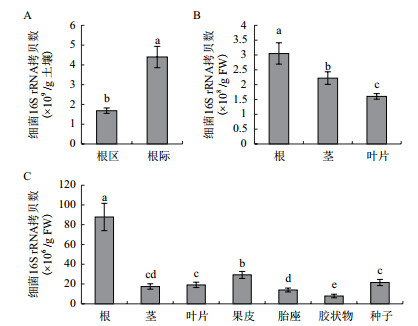
采用DGGE技术对番茄根区和根际土壤及各组织附生和内生细菌进行多样性分析,各样品的条带几乎布满整个泳道(图 3),表明DGGE的变性剂梯度范围合理,且各样品均具有较高的细菌多样性。各样品所含条带数目不等,其中番茄根际土壤条带数最多,说明根际微生物最丰富。
|
| 图 3 番茄组织附生和内生细菌DGGE图谱 1:根区土壤;2:根际;3:根内生;4:茎附生;5:茎内生;6:叶片附生;7:叶片内生;8:果皮内生;9:胎座内生;10:胶状物内生;11:种子内生 |
在DGGE图谱中,条带a、e、f、n、o、p、u、v出现在所有样品中,其中条带a、e、o在各样品中位置相同,亮度也相近,条带f、n、p、u、v在内生菌样品中条带亮度较高,而在土壤和附生菌样品中较弱,说明这些条带代表的细菌种类在番茄组织内生菌中占有优势地位。在7个番茄组织(根、茎、叶片、果实)内生细菌样品中,检测到共有条带10个,即a、e、f、l、n、o、p、u、v、w,其中条带l、w仅在内生菌样品中出现,在土壤和附生菌中均未检测到。在4种果实(果皮、胎座、种子及其表面胶状物)的内生细菌样品中共检测到12个共有条带,即条带a、e、f、k、l、n、o、p、t、u、v、w,其中k、t为仅在果实样品中出现的特征条带。
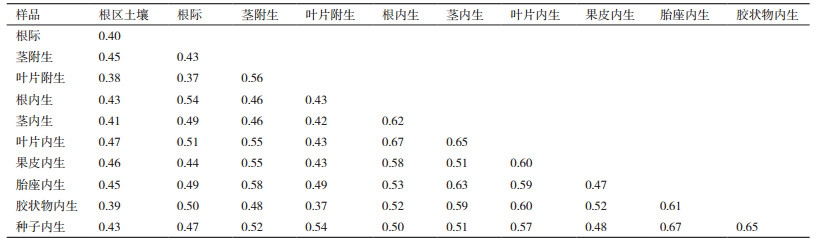
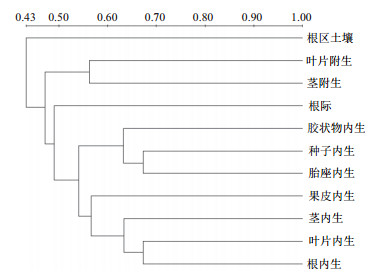
根据DGGE图谱分析各样品细菌群落间的相似度(表 1),并进行聚类分析,结果(图 4)表明,进化树被分为3个分支,分别为根区土壤细菌分支、附生细菌分支和内生细菌分支。在附生菌分支中,叶片附生和茎附生细菌群落间相似度为0.56,高于二者与根际细菌间的相似度。而在内生菌分支中,茎内生、叶片内生和根内生样品间的细菌群落相似度达到0.62以上,三者聚类在一起。此外,果实中胎座内生、种子内生和种子表面胶状物内生的细菌群落间的相似度分别为0.61、0.67和0.65,三者聚为一类。
|
| 图 4 番茄组织附生和内生细菌DGGE图谱的聚类分析 |
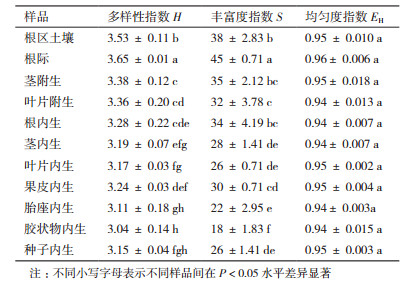
根据DGGE图谱计算各样品中细菌多样性指数H、丰富度指数S和均匀度指数EH(表 2)。在11个番茄样品中,根际细菌的H指数和S指数最高;其次为根区土壤;茎和叶片附生细菌的H指数和S指数低于土壤样品中;内生细菌样品中,除根和果皮内生细菌的H指数和S指数与附生菌相当,其余样品的H指数和S指数显著低于附生细菌,其中尤以种子表面胶状物内生细菌的H指数和S指数最低。番茄各样品的均匀度指数均约0.95,差异不显著。
植物组织器官表面和内部着生的各类细菌,是植物生态系统的重要组成,与植物密切互作,消长相关。植物的种类、生长发育情况等影响着这些细菌的群落结构,而这些细菌又通过自身的生命活动影响植物的生长发育[16]。本实验中,采用传统平板培养方法结合16S rRNA拷贝数分析发现,对于日光温室中栽培的番茄,根际细菌数量最多,远高于根区土壤,根系分泌物中丰富的糖、蛋白、脂肪等有机物质为微生物生存提供有机碳源,使得根际微生物数量比根际外的土壤微生物数量高几倍到几十倍[17]。对于番茄各组织的附生细菌数量,以根部最多,茎和叶片次之。这是由于在根周围的土壤中,细菌可以存活几年甚至几十年,而茎和叶片等组织的生活周期相对较短[18],且相较根部,茎、叶表面细菌生存环境相对严苛,可被利用的营养成分较少,温湿度波动较大,且紫外线辐射对细菌的生存也会有较大影响[19],从而导致根系附生的细菌数量显著大于茎、叶片表面附生的细菌数量。
研究还发现,番茄内生细菌的数量显著低于附生细菌,且在不同组织中有显著不同,以根内生细菌数量最多,这与之前在辣椒、番茄、茄子等植物中的报道结果一致[20-22],主要是由于根所在的土壤环境含有比空气中更为丰富的细菌资源,且根的表面积显著大于其他组织或器官,使根成为细菌进入植物的主要入口,导致根内生细菌数量最多,种类最丰富。果皮内生细菌的数量和多样性仅次于根,推测果皮中富含葡萄糖、果糖、蔗糖、半乳糖等糖类物质,以及苹果酸、柠檬酸、琥珀酸、丁酸、脯氨酸等有机酸和氨基酸[23],可为细菌的生长繁殖提供良好的营养条件,引起细菌在果皮中富集。
本实验进一步采用DGGE技术对土壤细菌、以及番茄各组织附生和内生细菌的多样性进行了比较分析。DGGE图谱显示番茄各样品均具有较高的细菌多样性,但以根际细菌的多样性指数和丰富度指数最高,根区土壤次之,附生和内生样品最低,其中尤以种子表面胶状物中内生细菌的多样性指数和丰富度指数最低。有8条条带存在于番茄所有样品中,其中3条条带在各样品中位置相同,亮度相近,说明这些条带代表的细菌在土壤和番茄各组织表面和内部均占有优势地位;有5条条带在番茄内生组织中亮度较高,而在其他样品中较弱,说明这些条带代表的细菌种类在番茄组织内生菌中占有优势地位。对DGGE图谱的聚类分析发现,根际细菌与组织附生和内生细菌的群落相似度显著高于根区土壤,根际与茎、叶片附生细菌聚为一个分支,7个组织内生样品聚为一个内生分支,暗示细菌在番茄植株表面和内部的定殖具有选择性。番茄植株不同组织内生细菌间的相似度最高,根、茎和叶片内生细菌群落的相似度约为0.65,果实胎座、种子表面胶状物以及种子内生细菌群落的相似度也达到0.6以上。这些结果表明,番茄植株各组织,尤其是相邻组织的内生细菌间可能具有流动性[24]。相似度分析还发现,相较于附生细菌,根际细菌与组织内生细菌群落间的相似度更高,推测番茄内生细菌可能主要来源于根际,通过根部吸收,并在进入植物内部后在其体内迁移,最终在各个组织间选择性地定殖[25-26]。
本实验通过平板培养计数结合分子生物学手段,明确了番茄根区、根际土壤以及根、茎、叶片、果实、种子等组织的附生和内生细菌的数量、多样性及各样品间的相似度。但上述方法也存在一定的局限性,未能在种属水平上鉴定各样品的细菌群落组成。故采用高通量测序技术鉴定番茄各样品的细菌群落结构,全面解析土壤细菌以及组织附生和内生细菌间的相互关系,将是下一步开展的工作重点。
4 结论番茄根区、根际土壤以及组织附生和内生细菌数量依次为:根际土壤 > 根区土壤 > 附生 > 内生,组织内生中以根和果皮内生细菌数量最多,叶片、茎和种子次之,种子表面胶状物内生细菌数量最少。DGGE分析表明细菌在番茄植株表面和内部的定殖具有选择性,且组织内生细菌与根际细菌群落密切相关,相邻组织的内生细菌群落结构相似,表明内生细菌在组织间具有流动性。
| [1] |
Brader G, Compant S, Vescio K, et al. Ecology and genomic insights into plant-pathogenic and plant-nonpathogenic endophytes[J]. Annual Reviews of Phytopathology, 2017, 55: 61-83. DOI:10.1146/annurev-phyto-080516-035641 |
| [2] |
Kutschera U, Khanna R. Plant gnotobiology:Epiphytic microbes and sustainable agriculture[J]. Plant Signaling and Behavior, 2016, 11(12): e1256529-1-e1256529-4. |
| [3] |
Xu MS, Sheng JP, Chen L, et al. Bacterial community compositions of tomato(Lycopersicon esculentum MilL.)seeds and plant growth promoting activity of ACC deaminase producing Bacillus subtilis(HYT-12-1)on tomato seedlings[J]. World Journal of Microbiology and Biotechnology, 2014, 30: 835-845. DOI:10.1007/s11274-013-1486-y |
| [4] |
孙建光, 罗琼, 等. 小麦、水稻、玉米、白菜、芹菜内生固氮菌及其系统发育[J]. 中国农业科学, 2012(7): 1303-1317. |
| [5] |
谈泰猛, 黎继烈, 申爱荣, 等. 辣椒疫病拮抗菌的分离、鉴定及其生防效果[J]. 生态学杂志, 2017, 36(4): 988-994. |
| [6] |
刘开朗, 王加启, 卜登攀, 等. 环境微生物群落结构与功能多样性研究方法[J]. 生态学报, 2010, 30(4): 1074-1080. |
| [7] |
Smalla K, Oros-Sichler M, Milling A, et al. Bacterial diversity of soils assessed by DGGE, T-RFLP and SSCP fingerprints of PCR-amplified 16S rRNA gene fragments:Do the different methods provide similar results?[J]. J Microbiol Methods, 2010, 69(3): 470-479. |
| [8] |
李德斌, 杨洪一, 卢磊. 变性梯度凝胶电泳在微生物生态学中的应用[J]. 生物技术通报, 2010(12): 88-92. |
| [9] |
杨瑞娟, 王腾飞, 等. 禾本科作物伴生对番茄根区土壤酶活性、微生物及根结线虫的影响[J]. 中国蔬菜, 2017(3): 38-42. |
| [10] |
邹媛媛, 刘琳, 刘洋, 等. 不同水稻品种种子固有细菌群落的多样性[J]. 植物生态学报, 2012, 36(8): 880-890. |
| [11] |
黄晓辉, 杨友才, 谭周进, 等. 四个品种烟草内生微生物的分布特征[J]. 生态学报, 2009, 29(12): 6827-6833. DOI:10.3321/j.issn:1000-0933.2009.12.062 |
| [12] |
杨清香, 王育水, 张晶, 等. 小麦土霉素抗性内生细菌分离、鉴定及分布[J]. 生态学报, 2009, 29(8): 4580-4587. |
| [13] |
Midha S, Bansal K, et al. Genomic resource of rice seed associated bacteria[J]. Front Microbiol, 2016, 6: 1551-1558. |
| [14] |
Links MG, Demeke T, et al. Simultaneous profiling of seed-associa-ted bacteria and fungi reveals antagonistic interactions between microorganisms within a shared epiphytic microbiome on Triticum and Brassica seeds[J]. New Phytologist, 2014, 202: 542-553. DOI:10.1111/nph.12693 |
| [15] |
房嫚嫚, 张志刚, 董春娟, 等. 蔬菜育苗用草炭细菌多样性分析[J]. 中国蔬菜, 2012(20): 33-38. |
| [16] |
国辉, 毛志泉, 刘训理. 植物与微生物互作的研究进展[J]. 中国农学通报, 2011, 27(9): 28-33. |
| [17] |
李春俭, 马玮, 张福锁. 根际对话及其对植物生长的影响[J]. 植物营养与肥料学报, 2008, 14(1): 178-183. |
| [18] |
Vorholt JA. Microbial life in the phyllosphere[J]. Nature Review Microbiology, 2012, 10: 828-840. DOI:10.1038/nrmicro2910 |
| [19] |
潘建刚, 呼庆, 齐鸿雁, 等. 叶际微生物研究进展[J]. 生态学报, 2011, 31(2): 583-592. |
| [20] |
王美琴, 刘慧平, 韩巨才, 等. 番茄内生细菌种群动态分析及拮抗菌株的筛选[J]. 中国农学通报, 2010(9): 277-282. |
| [21] |
何红, 蔡学清, 洪永聪, 等. 辣椒内生细菌的分离及拮抗菌的筛选[J]. 中国生物防治, 2002, 18(4): 171-175. |
| [22] |
洪鹏翔, 邱思鑫, 等. 4种茄科作物内生细菌的分离及拮抗菌的筛选[J]. 福建农林大学学报, 2007(36): 347-351. |
| [23] |
齐红岩, 李天来, 邹琳娜, 等. 番茄果实不同发育阶段糖分组成和含量变化的研究初报[J]. 沈阳农业大学学报, 2001, 32(5): 346-348. |
| [24] |
Compant S, Kaplan H, et al. Endophytic colonization of Vitis vinifera L. by Burkholderia phytofiimans strain PsJN:from the rhizosphere to inflorescence tissues[J]. FEMS Microbiol Ecol, 2008, 1: 84-93. |
| [25] |
Compant S, Reiter B, et al. Endophytic colonization of Vitis vinifera L. by plant growth-promoting bacterium Burkholderia sp. strain PsJN[J]. Appl Environ Microbiol, 2005, 71: 1685-1693. DOI:10.1128/AEM.71.4.1685-1693.2005 |
| [26] |
Compant S, Clement C, Sessitsch A. Plant growth-promoting bacteria in the rhizo- and endosphere of plants:their role, colonization, mechanisms involved and prospects for utilization[J]. Soil Biol Biochem, 2010, 42(5): 669-678. DOI:10.1016/j.soilbio.2009.11.024 |