2. 清华大学生命科学学院,北京 100084
2. School of Life Sciences, Tsinghua University, Beijing 100084
茉莉素(Jasmonate)是一类新型植物激素,包括茉莉酸(JA)及其环戊烷酮类衍生物。茉莉素在植物界中广泛存在,对植物的生存至关重要[1]。茉莉素既是植物生长发育的调节物,也是植物防御反应的信号分子。茉莉素调控植物生长发育的诸多方面,包括根的生长、雄蕊发育、叶片衰老、表皮毛形成等[1, 2];同时,茉莉素也介导植物对病原侵染、昆虫噬咬等多种生物胁迫和低温、干旱、臭氧、紫外线等多种非生物胁迫的抗性反应[3-7]。近年来,茉莉素的生物合成、信号转导和生理功能等方面均得到了广泛研究,取得了许多重要进展。本文将主要从茉莉素对植物生物胁迫反应的调控和病原生物对植物茉莉素途径的操控两方面进行综述。
1 茉莉素生物合成的调控机理茉莉素的生物合成经历了依次发生于叶绿体、过氧化物酶体和细胞质中的多步催化过程(图 1-A)。首先,叶绿体内膜上释放出来的α-亚麻酸(α-LeA)在脂肪氧合酶(LOX)、丙二烯氧化物合成酶(AOS)、丙二烯氧化物环化酶(AOC)等酶的催化作用下形成12-氧-植物二烯酸(OPDA);随后,OPDA被转运至过氧化物酶体中,在OPDA还原酶3(OPR3)的作用下形成3-氧代-2(顺2’-戊烯基)-环戊烷-1-辛酸(OPC-8),然后经过三轮β-氧化形成茉莉酸[8]。近期有研究表明,除了经典的OPR3途径外,OPDA也可先经过三轮β-氧化形成4,5-双脱氢茉莉酸(ddh-JA),然后经OPR2催化形成茉莉酸[9]。茉莉酸在过氧化物酶体中合成后会被释放到细胞质进行进一步的修饰,形成多种茉莉酸的衍生物,如茉莉酸甲酯、茉莉酸氨基酸偶联物等。其中,经JAR1催化形成的(+)-7-iso-JA-L-Ile(JA-Ile的一种构象)是植物体内最早被发现的具有活性的茉莉素形式[10-11]。最近的研究表明,(+)-7-iso-JA-Leu、(+)-7-iso-JA-Val、(+)-7-iso-JA-Met和(+)-7-iso-JA-Ala也是具有活性的植物内源茉莉素形式[12]。值得注意的是,JA-Ile具有四种不同的构象,但只有(+)-7-iso-JA-L-Ile这一种构象是具有生物活性的;同时,(+)-7-iso-JA-L-Ile不稳定,在环境因素(如pH等)的影响下很容易转化为没有生物活性的构象形式[10-12]。
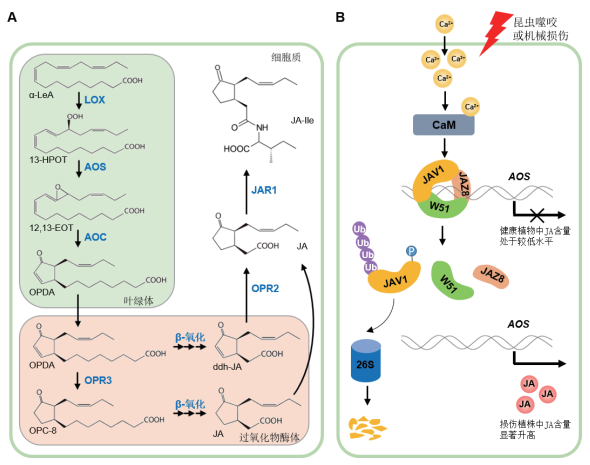
|
| 图 1 茉莉素的生物合成与调控 A:茉莉素的生物合成途径。叶绿体中的α-亚麻酸(α-LeA)在LOX、AOS、AOC的催化下依次形成13-氢过氧化亚油酸(13-HPOT)、12,13-十八碳三烯酸(12,13-EOT)和OPDA。随后,OPDA被转运至过氧化物酶体中,先经OPR3催化形成OPC-8,再经三轮β-氧化形成茉莉酸(JA);此外,OPDA也可先经三轮β-氧化形成ddh-JA,而后ddh-JA转移至细胞质中并经OPR2催化形成茉莉酸。茉莉酸可在细胞质中被JAR1进一步催化形成JA-Ile。B:损伤对茉莉素生物合成的诱导。在健康植物中,JAV1-JAZ8-WRKY51(JJW)蛋白复合体结合并抑制AOS等茉莉素合成基因的表达,使植物体内茉莉素含量维持在较低水平;在当植物受到昆虫噬咬或机械损伤时,细胞内钙离子浓度升高,激发了钙调蛋白(CaM)依赖的JAV1磷酸化,导致JAV1不能与JAZ8互作、失去细胞核定位能力、被泛素化并经26S蛋白酶体降解,从而使JJW复合体解体,解除了JJW复合体对AOS等茉莉素合成基因表达的抑制作用,使得茉莉素能够迅速合成。 |
茉莉素的合成受到严格的调控。在正常生长状态下,植物体内的茉莉素处于较低水平,以降低茉莉素对植物生长的抑制作用;而当植物受到损伤等外界刺激或接收到特定生长发育信号时,植物体内的茉莉素含量迅速升高,从而激活茉莉素响应基因的表达[1, 13]。
损伤如何诱导茉莉素的合成是茉莉素领域一个重要的科学问题。曾有研究表明,机械损伤能够诱导植物细胞的膜电位变化,进而诱导茉莉素的快速合成和茉莉素响应基因的表达;而与离子通道调控相关的谷氨酸受体基因家族中部分基因(GLR3.2,GLR3.3和GLR3.6)的突变会抑制损伤诱导的膜电位变化和茉莉素响应基因表达,表明植物细胞离子通道状态改变引起的膜电位变化在损伤诱导茉莉素合成过程中发挥重要作用[14]。最近的研究表明,VQ家族蛋白JAV1可能是连接离子通道状态变化/细胞膜电位变化和细胞内茉莉素合成的关键分子[15]。当植物处于正常生长状态时,JAV1蛋白能够与JAZ8、WRKY51互作并形成JAV1-JAZ8-WRKY51蛋白复合体(简称JJW复合体),结合并抑制茉莉素合成基因的表达,维持植物体内茉莉素含量处于较低水平;而当植物受到昆虫咬噬等损伤后,细胞内钙离子浓度迅速升高,从而激活了钙调蛋白依赖的JAV1磷酸化,而磷酸化的JAV1则不能与JAZ8互作、其亚细胞定位也发生改变(由细胞核定位转变为细胞质定位)、并被26S蛋白酶体降解,从而使JJW复合体解体、解除了JJW复合体对茉莉素合成的抑制作用,最终导致茉莉素能够在损伤后迅速大量合成[15-16](图 1-B)。
2 茉莉素的信号转导机制1998年,COI1基因的克隆[17]开启了茉莉素信号转导研究的序幕,此后茉莉素信号通路中的关键因子陆续被分离出来,信号转导过程逐渐清晰。目前认为,茉莉素信号转导过程大致如下:当植物处于静息状态时,体内的茉莉素含量较低,大量积累的负调控因子JAZ蛋白与下游一系列转录因子结合并抑制其活性,从而抑制了茉莉素反应;而当植物受到伤害等外界刺激或接收到特定生长发育信号时,体内的茉莉素含量迅速升高,SCFCOI1复合体中的COI1蛋白与活性茉莉素分子结合,进而与底物JAZ蛋白相互作用并将其泛素化降解,解除了JAZ蛋白对下游转录因子的抑制作用,从而激活了下游茉莉素反应[5, 8, 18]。
茉莉素受体COI1是一个F-box蛋白[17],可与SKP、CUL1和RBX1等蛋白形成SCFCOI1复合体[19],作为一种泛素连接酶特异性识别底物JAZ蛋白[20-22]。JAZ蛋白可以在茉莉素诱导下与COI1蛋白发生直接相互作用并被SCFCO1泛素化,进而被26S蛋白酶体所降解[21-22]。生化及晶体学实验结果表明,COI1能直接与活性茉莉素分子JA-Ile或COR结合[23],而JAZ蛋白可进一步稳定COI1与JA-Ile的结合[23-25]。此外,SCFCOI1复合体的完整性对维持COI1蛋白自身的稳定性也至关重要,SCFCOI1复合体组分SKP、CUL1的突变会导致COI1蛋白的稳定性大大降低[26]。
作为茉莉素途径的负调控因子,JAZ蛋白通过抑制下游一系列转录因子的活性、从而抑制各种茉莉素反应。已报道的可与JAZ蛋白直接互作的下游转录因子有20余个,包括MYC2、MYC3、MYC4、MYC5、bHLH3、bHLH13、bHLH14、bHLH17、MYB21、MYB24、FIL、ICE1、ICE2、EIN3、EIL1、TOE1、TOE2、RSS3、WRKY57、GL3、EGL3、TT8、MYB75、GL1等[8, 18]。JAZ蛋白通过作用于不同的下游转录因子,介导茉莉素对植物生长发育及抗性不同方面的调控,例如,JAZ通过与MYC2、MYC3、MYC4互作介导茉莉素对植物抗性、根的生长和叶片衰老等方面的调控[27-31],通过与MYB21、MYB24互作介导茉莉素对植物育性的调控[32]。目前认为,JAZ蛋白至少可通过下列三种方式抑制下游转录因子的活性:(1)通过与下游转录因子互作,干扰下游转录因子与MED等转录共激活因子的结合[33-34];(2)通过直接招募HDA6,抑制组蛋白乙酰化[35];(3)通过自身EAR结构域(如JAZ5/6/7/8)直接招募或通过NINJA间接招募转录共抑制因子TPL[36-37]。
3 茉莉素对植物生物胁迫反应的调控茉莉素在调控植物对昆虫和腐生型真菌的抗性反应中发挥重要作用。茉莉素合成突变体(如fad3 fad7 fad8、aos、opr3等)和受体突变体(如coi1-1)对昆虫和腐生型真菌的抗性都显著低于野生型植物[1]。目前,已经鉴定到了一些调控植物对昆虫和腐生型真菌抗性的茉莉素途径基因,它们大多为转录因子,通过诱导植物表面物理屏障(如表皮毛等)的形成、调控次生代谢产物的合成、诱导防御基因表达等多种方式,介导茉莉素对植物生物胁迫反应的调控。
MYC2、MYC3和MYC4在调控茉莉素介导的植物抗性反应中发挥重要作用。MYC2、MYC3和MYC4功能冗余地调控植物对昆虫的抗性,myc2 myc3 myc4三突变体对灰翅夜蛾(Spodoptera littoralis)和甜菜夜蛾(Spodoptera exigua)的抗性显著低于野生型植物[28-29, 38]。进一步研究发现,MYC2、MYC3和MYC4可通过促进芥子油苷的合成增强植物对昆虫的抗性:MYC2、MYC3和MYC4能够与调控芥子油苷合成的一些MYB家族转录因子直接互作,形成转录因子复合体调控芥子油苷合成基因的表达;同时,MYC2、MYC3和MYC4也能够结合到许多芥子油苷合成基因的启动子序列上,直接调控这些基因的表达[28]。此外,MYC2还能够直接与萜类化合物合成基因TPS10、TPS11和TPS21的启动子结合并激活它们的表达,促进植物萜类化合物的合成[39],从而增强萜类化合物介导的植物对病虫害的直接和间接抗性[6]。值得注意的是,不同于对害虫抗性的调控作用,MYC2在植物对病原菌的抗性中起负调控作用,myc2突变体对灰霉菌(Botrytis cinerea)等病原菌的抗性强于野生型,这可能是由于MYC2与EIN3互作并抑制EIN3的功能,从而抑制了乙烯介导的植物对病原菌的抗性[38]。
茉莉素可显著诱导植物花色素苷的积累和表皮毛的产生。花色素苷和表皮毛分别作为植物的一种化学和物理屏障,能有效提高植物对昆虫、微生物等病虫害的抗性,降低紫外线等非生物胁迫对植物造成的伤害[40-41]。拟南芥花色素苷的合成和表皮毛产生均受WD-repeat/bHLH/MYB复合体的调控[40, 42]。JAZ蛋白能够与WD-repeat/bHLH/MYB复合体的bHLH组分(GL3、EGL3和TT8)和R2R3 MYB组分(MYB75、GL1)直接互作,抑制WD-repeat/bHLH/MYB复合体的活性,从而抑制植物花色素苷的积累和表皮毛的产生;茉莉素通过诱导JAZ蛋白降解,解除JAZ蛋白对WD-repeat/bHLH/MYB复合体的抑制作用,从而促进色素苷的积累和表皮毛的产生,增强植物对病虫害和紫外等非生物胁迫的抗性[43]。此外,JAZ蛋白也可与YAB家族转录因子FIL互作调控色素的合成:FIL能够结合到MYB75的启动子上并激活MYB75的表达,从而促进色素合成;而JAZ蛋白通过与FIL互作,抑制FIL对MYB75的转录激活,从而抑制色素合成[44]。
IIId亚家族bHLH家族转录因子bHLH3、bHLH13、bHLH14和bHLH17作为转录抑制因子,抑制茉莉素响应基因的表达[45-48]。bhlh3 bhlh13 bhlh14 bhlh17四突变体表现出对茉莉素超敏的表型,且对昆虫(甜菜夜蛾)和腐生真菌(灰霉菌)的抗性增强[46]。bHLH3、bHLH13、bHLH14和bHLH17虽不具有转录激活活性,但具有DNA结合能力,能够通过拮抗MYC2、MYC3、MYC4等转录因子发挥转录抑制功能[45-48]。
JAV1是茉莉素途径的一个负调控因子[15-16]。如前文所述,JAV1能够与JAZ8和WRKY51形成JAV1-JAZ8-WRKY51复合体,抑制茉莉素合成基因的表达[15]。JAV1的突变体对灰霉菌和甜菜夜蛾的抗性强于野生型,而其超表达植株则对灰霉菌和甜菜夜蛾更敏感[16]。同样,JAV1与WRKY51的双突变体在伤诱导后会比野生型植物积累更多的茉莉素,其对甜菜夜蛾的抗性也明显强于野生型植物[15]。植物抗性上的增强往往伴随着生长上的抑制[49]。然而有趣的是,JAV1虽然在植物抗性中发挥重要作用,却对植物生长发育没有明显影响[16]。
近期有研究表明,植物对茉莉素的响应水平以及对昆虫的抗性受植物年龄影响,与幼嫩植物相比,成株植物对茉莉素的响应水平较低,但对昆虫的抗性更强[50]。进一步研究发现,不同年龄植物对茉莉素的响应水平受miRNA156的靶标SPL9调控:SPL9能够直接与JAZ蛋白互作,并抑制COI1介导的JAZ蛋白降解;随着植物逐渐生长成熟,miR156水平逐渐降低,SPL9逐渐积累,使得JAZ蛋白含量逐渐升高,最终导致茉莉素响应水平逐渐降低[50]。值得注意的是,虽然成株植物的茉莉素响应水平低于幼嫩植物,但由于成株植物中积累了更多的芥子油苷等抗性物质,因此成株植物对昆虫的抗性强于幼嫩植物[50]。
4 病原生物对植物茉莉素途径的操控茉莉素在调控植物对昆虫和病原微生物的抗性反应中发挥重要作用。相应地,昆虫、细菌、真菌、病毒及植原体等诸多植物病原生物都进化出了多种策略来操控茉莉素途径,通过激活或抑制茉莉素途径,促进自身的侵染或传播(图 2)。
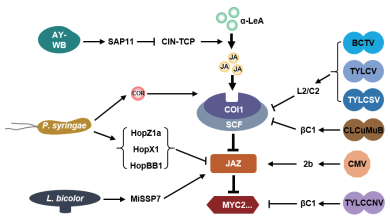
|
| 图 2 微生物对茉莉素途径的操控策略 AY-WB利用其效应蛋白SAP11与CIN-TCP互作并抑制CIN-TCP的活性,从而抑制茉莉素的合成;P. syringae通过分泌茉莉素类似物冠菌素(COR)或利用其效应蛋白HopZ1a、HopX1和HopBB1与JAZ蛋白互作并降低JAZ的稳定性或功能,激活茉莉素途径;BCTV、TYLCV、TYLCSV和CLCuMuB分别利用其效应蛋白L2/C2和βC1影响SCFCOI1复合体的功能,抑制茉莉素途径;L. bicolor和CMV分别利用其效应蛋白MiSS7和2b与JAZ蛋白互作并促进JAZ的积累,从而抑制茉莉素途径;TYLCCNV利用其效应蛋白βC1与MYC2互作并抑制MYC2的功能,抑制MYC2调控的茉莉素响应基因表达。 |
一些昆虫通过激活水杨酸(SA)途径间接抑制茉莉素途径。甜菜夜蛾分泌的唾液含有葡萄糖氧化酶,能够催化葡萄糖形成过氧化物,激活水杨酸途径,从而间接抑制茉莉素途径[51]。马铃薯甲虫(Leptinotarsa decemlineata)通过其口器内的共生微生物激活水杨酸途径,间接抑制茉莉素途径,其对茉莉素途径的抑制作用在经抗生素处理后消失,而经移植共生菌群后又得到恢复,表明了共生微生物在马铃薯甲虫抑制茉莉素途径中的关键作用[52]。
一些昆虫利用茉莉素途径下游ERF和MYC分支间的拮抗作用操控茉莉素途径。菜青虫(Pieris rapae)通过口器分泌物激活ERF分支,抑制MYC分支介导的植物对昆虫的抗性,同时增强了ERF分支介导的植物对腐生菌的抗性,减轻其它病原生物对宿主植物侵害,以更多地从植物宿主摄取营养[53]。
4.2 细菌对茉莉素途径的操控一些细菌通过合成茉莉素类似物激活植物茉莉素途径,以利于侵染。丁香假单胞菌(Pseudomonas syringae,简称P.syringae)能够合成冠菌素(茉莉素类似物),冠菌素的结构与(+)-7-iso-JA-L-Ile较为相似,能够被茉莉素受体COI1识别、诱导COI1与JAZ蛋白结合、激活茉莉素途径[23-25](图 2)。冠菌素具有较强的茉莉素活性,与(+)-7-iso-JA-L-Ile的生物活性相当,比商业化生产的JA-Ile(4种构象的混合物)的生物活性高约100倍[10]。丁香假单胞菌利用冠菌素激活茉莉素途径,从而促进植物气孔打开,利于丁香假单胞菌从植物气孔入侵;同时,由于茉莉素途径与水杨酸途径具有拮抗作用,冠菌素激活的茉莉素途径干扰了水杨酸途径,从而抑制了水杨酸途径介导的植物抗性反应,利于丁香假单胞菌侵染[54-56]。
一些细菌利用自身分泌的效应蛋白激活植物茉莉素途径,以利于侵染。丁香假单胞菌效应蛋白AvrB可通过与MPK4及分子伴侣HSP90/RAR1互作,促进MPK4的磷酸化,从而激活茉莉素途径[57];此外,AvrB还可通过与RIN4互作激活AHA1,促进COI1与JAZ蛋白的互作,从而激活茉莉素途径[58]。但AvrB通过RIN4/AHA1或MPK4/HSP90/RAR1激活茉莉素途径的具体分子机制还不清楚。分别来源于丁香假单胞菌Psy株系和Pta株系的效应蛋白HopZ1a和HopX1都通过作用于植物JAZ蛋白来激活茉莉素途径[59-60],但它们的作用机制有所不同。HopZ1a具有乙酰转移酶活性,通过诱导JAZ蛋白的乙酰化,从而促进COI1依赖的JAZ蛋白的降解[59];而HopX1则利用其自身具有的半胱氨酸蛋白酶活性降解JAZ蛋白,此降解过程不依赖于COI1[60](图 2)。最近的研究表明,效应蛋白HopBB1也参与了丁香假单胞菌对植物茉莉素途径的激活[61]。TCP14能够抑制茉莉素响应基因的表达,而HopBB1通过与TCP14和JAZ3互作,促使TCP14被SCFCOI1泛素化降解,解除了TCP14对茉莉素响应基因的抑制作用;此外,HopBB1与JAZ3的互作也阻碍了JAZ3对MYC2的抑制作用,这些机制都使得HopBB1能够激活茉莉素途径[61](图 2)。
4.3 真菌对茉莉素途径的操控腐生型病原真菌或共生真菌往往通过抑制茉莉素途径、降低茉莉素介导的植物抗性,利于腐生型病原真菌的侵染或共生真菌与植物的共生。灰霉菌产生的一种胞外多糖β-(1,3)(1,6)-D-glucan能够诱导植物体内水杨酸的合成,利用水杨酸-茉莉素途径间的拮抗,抑制茉莉素介导的植物抗性[62]。核盘菌(Sclerotinia sclerotiorum)编码的效应蛋白SSITL通过抑制茉莉素和乙烯途径促进侵染,但SSITL抑制茉莉素和乙烯途径的具体分子机制还不清楚[63]。杨树共生真菌双色蜡蘑(Laccaria bicolor,简称L.bicolor)编码的效应蛋白MiSSP7能够与杨树的JAZ6蛋白互作并抑制JAZ6蛋白的降解,从而抑制杨树茉莉素信号通路以促进双色蜡蘑与杨树的共生[64](图 2)。
也有一些真菌通过激活茉莉素途径促进侵染。尖孢镰刀菌(Fusarium oxysporum)、漆蜡蘑(Laccaria laccata)、黑曲霉(Aspergillus niger)等真菌都已被发现能够合成JA、JA-Ile等茉莉素类物质[65]。尖孢镰刀菌Fo5176株系还可通过其效应蛋白SIX4激活植物茉莉素途径进而促进侵染[66]。值得一提的是,这些激活茉莉素途径的真菌并不都是通过间接抑制SA途径促进侵染的。例如,coi1突变体对尖孢镰刀菌的抗性强于野生型植物,说明茉莉素途径促进尖孢镰刀菌的侵染,但尖孢镰刀菌在coi1单突变体和coi1/NahG(过量表达SA羟基酶而不能积累SA)双突变体上的侵染能力相近,说明尖孢镰刀菌并不是利用JA-SA之间的拮抗关系来促进侵染的[67]。此外,SIX4并不显著影响植物SA途径,进一步映证了尖孢镰刀菌并不是通过操控JA途径间接影响SA途径来促进侵染的[66]。这些研究暗示,茉莉素介导的植物与真菌间的互作可能比我们预想的更加复杂。
4.4 病毒和植原体对茉莉素途径的操控一些以昆虫为介体进行传播的病毒和植原体,通过抑制植物茉莉素途径,抑制植物对介体昆虫的抗性或提高植物对介体昆虫的吸引性,从而促进自身的传播。翠菊黄化植原体AY-WB株系(Aster Yellows phytoplasma strain Witches' Broom,AY-WB)分泌的效应蛋白SAP11通过与CIN-TCP转录因子互作并促进CIN-TCP的降解,抑制CIN-TCP对茉莉素合成基因(LOX2等)的转录激活,从而抑制茉莉素的合成,进而抑制茉莉素介导的植物对其传播介体翠菊叶蝉的抗性,以促进自身的传播[68](图 2)。黄瓜花叶病毒(Cucumber mosaic virus,CMV)能够显著干扰植物茉莉素途径,进而影响宿主植物与介体昆虫间的互作[69-70]。最近的研究表明,CMV编码的2b蛋白能够与植物JAZ蛋白直接互作,抑制SCFCOI1介导的JAZ蛋白降解,从而抑制茉莉素途径,增强植物对病毒传播媒介蚜虫的吸引力,以利于病毒传播[71](图 2)。番茄黄曲叶病毒(Tomato yellow leaf curl China virus,TYLCCNV)编码的βC1蛋白通过与植物AS1和MYC2蛋白互作,抑制植物茉莉素途径,降低植物对其介体昆虫烟粉虱的抗性[72-74](图 2)。
一些植物病毒通过抑制茉莉素途径促进侵染。水稻齿叶矮缩病毒(Rice ragged stunt virus,RRSV)的侵染能够诱导miR319的积累,降低miR319靶标基因TCP21的表达,削弱了TCP21对茉莉素合成基因的激活作用,抑制了茉莉素的合成和茉莉素响应基因的表达,从而抑制了茉莉素介导的植物对RRSV的抗性,利于RRSV的侵染[75]。甜菜曲顶病毒(Beet curly top virus,BCTV)编码的L2蛋白及其在番茄黄化曲叶病毒(Tomato yellow leaf curl virus,TYLCV)和番茄黄叶卷撒丁病毒(Tomato yellow leaf curl Sardinia virus,TYLCSV)中的同源蛋白C2,通过与植物COP9复合体中的CSN5亚基互作,影响SCF复合体组分CUL1的去RUB化修饰,从而影响了包括SCFCOI1在内的E3泛素连接酶的活性,进而抑制了茉莉素途径介导的植物病毒抗性,促进了病毒侵染[76]。木尔坦棉花曲叶病毒(Cotton leaf curl Multan virus,CLCuMuB)编码的βC1蛋白通过与烟草SKP1蛋白互作,抑制SCFCOI1复合体的形成,从而抑制茉莉素途径,促进病毒侵染[77](图 2)。
值得注意的是,许多操控植物茉莉素途径的病毒效应蛋白(包括CMV编码的2b蛋白、TYLCCNV编码的βC1蛋白、BCTV编码的L2、CLCuMuB编码的βC1蛋白等)同时也是RNA沉默抑制子(Viral suppressor of RNA silencing,VSR)[78-81],暗示病毒VSR蛋白功能的多重性,即既通过抑制植物RNA沉默促进病毒在植物内的增殖,又通过抑制茉莉素途径促进病毒在植物内的增殖或在植物间的传播。
5 展望植物固定生长不能移动,在长期的进化过程中,植物形成了独特而复杂的防御机制来抵抗昆虫和微生物的入侵;与此同时,昆虫和微生物也进化出了各种机制来逃避、克服或利用植物的防御系统,以实现对宿主植物的寄生。茉莉素在调控植物对昆虫和微生物的抗性反应中发挥重要作用。相应地,包括昆虫、真菌、细菌、病毒在内的许多生物都通过多种策略操控茉莉素途径,直接或间接影响植物抗性,从而促进侵染或传播。近年来,茉莉素信号转导机制的逐渐明晰和部分重要植物病原全基因组测序的完成,为系统和深入研究茉莉素介导的植物与病原生物互作提供了极大便利,相关研究领域也取得了很大进展。
已有的研究表明,茉莉素在调控植物对病毒的抗性方面发挥一定作用,但对于不同的植物病毒,茉莉素的调控作用却不尽相同。例如,茉莉素正调控植物对RRSV(双链RNA病毒)、CLCuMuB(DNA病毒)和BCTV(DNA病毒)的抗性[75-77],却负调控植物对烟草花叶病毒(单链RNA病毒)的抗性[82]。茉莉素究竟如何调控植物对病毒的抗性还有待进一步研究。此外,茉莉素在调控植物对腐生型真菌的抗性反应中发挥关键作用,然而与细菌、病毒等病原微生物相比,腐生型真菌操控茉莉素途径的研究还很少见,作用于茉莉素途径的腐生型真菌效应蛋白还有待更加广泛地鉴定和研究。对这些重要科学问题的探索,将使人们对茉莉素介导的植物与病原生物的互作有更加全面和深入的认识,从而为抗病虫害植物分子育种奠定理论基础。
| [1] |
Wasternack C, Hause B. Jasmonates: biosynthesis, perception, signal transduction and action in plant stress response, growth and development. An update to the 2007 review in Annals of Botany[J]. Ann Bot, 2013, 111(6): 1021-1058. DOI:10.1093/aob/mct067 |
| [2] |
Huang H, Liu B, Liu L, et al. Jasmonate action in plant growth and development[J]. J Exp Bot, 2017, 68(6): 1349-1359. DOI:10.1093/jxb/erw495 |
| [3] |
Zhang L, Zhang F, et al. Jasmonate signaling and manipulation by pathogens and insects[J]. J Exp Bot, 2017, 68(6): 1371-1385. |
| [4] |
Yan C, Xie D. Jasmonate in plant defence: sentinel or double agent?[J]. Plant Biotechnol J, 2015, 13(9): 1233-1240. DOI:10.1111/pbi.12417 |
| [5] |
Browse J. Jasmonate passes muster: A receptor and targets for the defense hormone[J]. Annu Rev Plant Biol, 2009, 60: 183-205. DOI:10.1146/annurev.arplant.043008.092007 |
| [6] |
Howe GA, Jander G. Plant immunity to insect herbivores[J]. Annu Rev Plant Biol, 2008, 59: 41-66. DOI:10.1146/annurev.arplant.59.032607.092825 |
| [7] |
Liechti R, Farmer EE. The jasmonate pathway[J]. Science, 2002, 296(5573): 1649-1650. DOI:10.1126/science.1071547 |
| [8] |
Wasternack C, Song S. Jasmonates: biosynthesis, metabolism, and signaling by proteins activating and repressing transcription[J]. J Exp Bot, 2017, 68(6): 1303-1321. |
| [9] |
Chini A, Monte I, Zamarreno AM, et al. An OPR3-independent pathway uses 4, 5-didehydrojasmonate for jasmonate synthesis[J]. Nat Chem Biol, 2018, 14(2): 171-178. DOI:10.1038/nchembio.2540 |
| [10] |
Fonseca S, Chini A, Hamberg M, et al. (+)-7-iso-Jasmonoyl-L-isoleucine is the endogenous bioactive jasmonate[J]. Nat Chem Biol, 2009, 5(5): 344-350. DOI:10.1038/nchembio.161 |
| [11] |
Staswick PE, Tiryaki I. The oxylipin signal jasmonic acid is activated by an enzyme that conjugates it to isoleucine in Arabidopsis[J]. Plant Cell, 2004, 16(8): 2117-2127. DOI:10.1105/tpc.104.023549 |
| [12] |
Yan J, Li S, Gu M, et al. Endogenous bioactive jasmonate is composed of a set of(+)-7-iso-JA-amino acid conjugates[J]. Plant Physiol, 2016, 172(4): 2154-2164. |
| [13] |
Browse J, Howe GA. New weapons and a rapid response against insect attack[J]. Plant Physiol, 2008, 146(3): 832-838. DOI:10.1104/pp.107.115683 |
| [14] |
Mousavi SAR, Chauvin A, Pascaud F, et al. GLUTAMATE RECEPTOR-LIKE genes mediate leaf-to-leaf wound signalling[J]. Nature, 2013, 500(7463): 422-426. DOI:10.1038/nature12478 |
| [15] |
Yan C, Fan M, Yang M, et al. Injury activates Ca2+/calmodulin-dependent phosphorylation of JAV1-JAZ8-WRKY51 complex for jasmonate biosynthesis[J]. Mol Cell, 2018, 70(1): 136-149. DOI:10.1016/j.molcel.2018.03.013 |
| [16] |
Hu P, Zhou W, Cheng ZW, et al. JAV1 Controls jasmonate-regulated plant defense[J]. Mol Cell, 2013, 50(4): 504-515. DOI:10.1016/j.molcel.2013.04.027 |
| [17] |
Xie DX, Feys BF, James S, et al. COI1: An Arabidopsis gene required for jasmonate-regulated defense and fertility[J]. Science, 1998, 280(5366): 1091-1094. DOI:10.1126/science.280.5366.1091 |
| [18] |
Song S, Qi T, Wasternack C, et al. Jasmonate signaling and crosstalk with gibberellin and ethylene[J]. Curr Opin Plant Biol, 2014, 21: 112-119. DOI:10.1016/j.pbi.2014.07.005 |
| [19] |
Xu L, Liu F, Lechner E, et al. The SCFCOI1 ubiquitin-ligase complexes are required for jasmonate response in Arabidopsis[J]. Plant Cell, 2002, 14(8): 1919-1935. DOI:10.1105/tpc.003368 |
| [20] |
Yan Y, Stolz S, Chételat A, et al. A downstream mediator in the growth repression limb of the jasmonate pathway[J]. Plant Cell, 2007, 19(8): 2470-2483. DOI:10.1105/tpc.107.050708 |
| [21] |
Thines B, Katsir L, Melotto M, et al. JAZ repressor proteins are targets of the SCFCOI1 complex during jasmonate signalling[J]. Nature, 2007, 448(7154): 661-665. DOI:10.1038/nature05960 |
| [22] |
Chini A, Fonseca S, Fernández G, et al. The JAZ family of repressors is the missing link in jasmonate signalling[J]. Nature, 2007, 448(7154): 666-671. DOI:10.1038/nature06006 |
| [23] |
Yan JB, Zhang C, Gu M, et al. The Arabidopsis CORONATINE INSENSITIVE1 protein is a jasmonate receptor[J]. Plant Cell, 2009, 21(8): 2220-2236. DOI:10.1105/tpc.109.065730 |
| [24] |
Sheard LB, Tan X, Mao HB, et al. Jasmonate perception by inositol-phosphate-potentiated COI1-JAZ co-receptor[J]. Nature, 2010, 468(7322): 400-405. DOI:10.1038/nature09430 |
| [25] |
Katsir L, Schilmiller AL, Staswick PE, et al. COI1 is a critical component of a receptor for jasmonate and the bacterial virulence factor coronatine[J]. Proc Natl Acad Sci USA, 2008, 105(19): 7100-7105. DOI:10.1073/pnas.0802332105 |
| [26] |
Yan J, Li H, Li S, et al. The Arabidopsis F-box protein CORONATINE INSENSITIVE1 is stabilized by SCFCOI1 and degraded via the 26S proteasome pathway[J]. Plant Cell, 2013, 25(2): 486-498. DOI:10.1105/tpc.112.105486 |
| [27] |
Qi T, Wang J, et al. Regulation of jasmonate-induced leaf senesc-ence by antagonism between bHLH subgroup Ⅲe and Ⅲd factors in Arabidopsis[J]. Plant Cell, 2015, 27(6): 1634-1649. DOI:10.1105/tpc.15.00110 |
| [28] |
Schweizer F, Fernández-Calvo P, Zander M, et al. Arabidopsis basic helix-loop-helix transcription factors MYC2, MYC3, andMYC4 regulate glucosinolate biosynthesis, insect performance, and feeding behavior[J]. Plant Cell, 2013, 25(8): 3117-3132. DOI:10.1105/tpc.113.115139 |
| [29] |
Fernandez-Calvo P, Chini A, Fernandez-Barbero G, et al. The Arabidopsis bHLH transcription factors MYC3 and MYC4 are targets of JAZ repressors and act additively with MYC2 in the activation of jasmonate responses[J]. Plant Cell, 2011, 23(2): 701-715. DOI:10.1105/tpc.110.080788 |
| [30] |
Cheng Z, Sun L, Qi T, et al. The bHLH transcription factor MYC3 interacts with the Jasmonate ZIM-domain proteins to mediate jasmonate response in Arabidopsis[J]. Mol Plant, 2011, 4(2): 279-288. DOI:10.1093/mp/ssq073 |
| [31] |
Chen Q, Sun J, Zhai Q, et al. The basic helix-loop-helix transcription factor myc2 directly represses plethora expression during jasmonate-mediated modulation of the root stem cell niche in Arabidopsis[J]. Plant Cell, 2011, 23(9): 3335-3352. DOI:10.1105/tpc.111.089870 |
| [32] |
Song S, Qi T, Huang H, et al. The jasmonate-ZIM domain proteins interact with the R2R3-MYB transcription factors MYB21 and MYB24 to affect jasmonate-regulated stamen development in Arabidopsis[J]. Plant Cell, 2011, 23(3): 1000-1013. DOI:10.1105/tpc.111.083089 |
| [33] |
Zhang F, Yao J, Ke J, et al. Structural basis of JAZ repression of MYC transcription factors in jasmonate signalling[J]. Nature, 2015, 525(7568): 269-273. DOI:10.1038/nature14661 |
| [34] |
Chen R, Jiang H, Li L, et al. The Arabidopsis mediator subunit MED25 differentially regulates jasmonate and abscisic acid signaling through interacting with the MYC2 and ABI5 transcription factors[J]. Plant Cell, 2012, 24(7): 2898-2916. DOI:10.1105/tpc.112.098277 |
| [35] |
Zhu Z, An F, Feng Y, et al. Derepression of ethylene-stabilized transcription factors(EIN3/EIL1)mediates jasmonate and ethylene signaling synergy in Arabidopsis[J]. Proc Natl Acad Sci USA, 2011, 108(30): 12539-12544. DOI:10.1073/pnas.1103959108 |
| [36] |
Shyu C, Figueroa P, Depew CL, et al. JAZ8 lacks a canonical degron and has an EAR motif that mediates transcriptional repression of jasmonate responses in Arabidopsis[J]. Plant Cell, 2012, 24(2): 536-550. DOI:10.1105/tpc.111.093005 |
| [37] |
Pauwels L, Barbero GF, Geerinck J, et al. NINJA connects the co-repressor TOPLESS to jasmonate signalling[J]. Nature, 2010, 464(7289): 788-791. DOI:10.1038/nature08854 |
| [38] |
Song S, Huang H, Gao H, et al. Interaction between MYC2 and ETHYLENE INSENSITIVE3 modulates antagonism between jasmonate and ethylene signaling in Arabidopsis[J]. Plant Cell, 2014, 26(1): 263-279. DOI:10.1105/tpc.113.120394 |
| [39] |
Hong GJ, Xue XY, Mao YB, et al. Arabidopsis MYC2 interacts with DELLA proteins in regulating sesquiterpene synthase gene expression[J]. Plant Cell, 2012, 24(6): 2635-2648. DOI:10.1105/tpc.112.098749 |
| [40] |
Ishida T, Kurata T, Okada K, et al. A genetic regulatory network in the development of trichomes and root hairs[J]. Annu Rev Plant Biol, 2008, 59: 365-386. DOI:10.1146/annurev.arplant.59.032607.092949 |
| [41] |
Winkel-Shirley B. Biosynthesis of flavonoids and effects of stress[J]. Curr Opin Plant Biol, 2002, 5(3): 218-223. DOI:10.1016/S1369-5266(02)00256-X |
| [42] |
Broun P. Transcriptional control of flavonoid biosynthesis: a complex network of conserved regulators involved in multiple aspects of differentiation in Arabidopsis[J]. Curr Opin Plant Biol, 2005, 8(3): 272-279. DOI:10.1016/j.pbi.2005.03.006 |
| [43] |
Qi T, Song S, Ren Q, et al. The Jasmonate-ZIM-Domain proteins interact with the WD-Repeat/bHLH/MYB Complexes to regulate jasmonate-mediated anthocyanin accumulation and trichome initiation in Arabidopsis thaliana[J]. Plant Cell, 2011, 23(5): 1795-1814. DOI:10.1105/tpc.111.083261 |
| [44] |
Boter M, Golz JF, Gimenez-Ibanez S, et al. FILAMENTOUS FLOWER is a direct target of JAZ3 and modulates responses to jasmonate[J]. Plant Cell, 2015, 27(11): 3160-3174. DOI:10.1105/tpc.15.00220 |
| [45] |
Fonseca S, Fernandez-Calvo P, et al. bHLH003, bHLH013 and bHLH017 are new targets of JAZ repressors negatively regulating JA responses[J]. PLoS One, 2014, 9(1): e86182. DOI:10.1371/journal.pone.0086182 |
| [46] |
Song SS, Qi TC, Fan M, et al. The bHLH subgroup Ⅲd factors negatively regulate jasmonate-mediated plant defense and development[J]. PLoS Genetics, 2013, 9(7): e1003653. DOI:10.1371/journal.pgen.1003653 |
| [47] |
Sasaki-Sekimoto Y, Jikumaru Y, Obayashi T, et al. Basic helix-loop-helix transcription factors JASMONATE-ASSOCIATED MYC2-LIKE1(JAM1), JAM2, and JAM3 are negative regulators of jasmonate responses in Arabidopsis[J]. Plant Physiol, 2013, 163(1): 291-304. DOI:10.1104/pp.113.220129 |
| [48] |
Nakata M, Mitsuda N, Herde M, et al. A bHLH-type transcription factor, ABA-INDUCIBLE BHLH-TYPE TRANSCRIPTION FACTOR/JA-ASSOCIATED MYC2-LIKE1, acts as a repressor to negatively regulate jasmonate signaling in Arabidopsis[J]. Plant Cell, 2013, 25(5): 1641-1656. DOI:10.1105/tpc.113.111112 |
| [49] |
Ning Y, Liu W, Wang GL. Balancing immunity and yield in crop plants[J]. Trends Plant Sci, 2017, 22(12): 1069-1079. DOI:10.1016/j.tplants.2017.09.010 |
| [50] |
Mao YB, Liu YQ, et al. Jasmonate response decay and defense metabolite accumulation contributes to age-regulated dynamics of plant insect resistance[J]. Nat Commun, 2017, 8: 13925. DOI:10.1038/ncomms13925 |
| [51] |
Musser RO, Hum-Musser SM, Eichenseer H, et al. Herbivory: caterpillar saliva beats plant defences[J]. Nature, 2002, 416(6881): 599-600. DOI:10.1038/416599a |
| [52] |
Chung SH, Rosa C, Scully ED, et al. Herbivore exploits orally secreted bacteria to suppress plant defenses[J]. Proc Natl Acad Sci USA, 2013, 110(39): 15728-15733. DOI:10.1073/pnas.1308867110 |
| [53] |
Verhage A, Vlaardingerbroek I, Raaymakers C, et al. Rewiring of the jasmonate signaling pathway in Arabidopsis during insect herbivory[J]. Front Plant Sci, 2011, 2: 47. |
| [54] |
Geng X, Cheng J, Gangadharan A, et al. The coronatine toxin of Pseudomonas syringae is a multifunctional suppressor of Arabidopsis defense[J]. Plant Cell, 2012, 24(11): 4763-4774. DOI:10.1105/tpc.112.105312 |
| [55] |
Zheng XY, Spivey NW, Zeng W, et al. Coronatine promotes Pseudomonas syringae virulence in plants by activating a signaling cascade that inhibits salicylic acid accumulation[J]. Cell Host Microbe, 2012, 11(6): 587-596. DOI:10.1016/j.chom.2012.04.014 |
| [56] |
Melotto M, Underwood W, Koczan J, et al. Plant stomata function in innate immunity against bacterial invasion[J]. Cell, 2006, 126(5): 969-980. DOI:10.1016/j.cell.2006.06.054 |
| [57] |
Cui H, Wang Y, et al. Pseudomonas syringae effector protein AvrB perturbs Arabidopsis hormone signaling by activating MAP kinase 4[J]. Cell Host Microbe, 2010, 7(2): 164-175. DOI:10.1016/j.chom.2010.01.009 |
| [58] |
Zhou Z, Wu Y, Yang Y, et al. An Arabidopsis plasma membrane proton ATPase modulates JA signaling and is exploited by the Pseudomonas syringae effector protein AvrB for stomatal invasion[J]. Plant Cell, 2015, 27(7): 2032-2041. DOI:10.1105/tpc.15.00466 |
| [59] |
Jiang S, Yao J, Ma KW, et al. Bacterial effector activates jasmonate signaling by directly targeting JAZ transcriptional repressors[J]. PLoS Pathog, 2013, 9(10): e1003715. DOI:10.1371/journal.ppat.1003715 |
| [60] |
Gimenez-Ibanez S, Boter M, Fernandez-Barbero G, et al. The bacterial effector HopX1 targets JAZ transcriptional repressors to activate jasmonate signaling and promote infection in Arabidopsis[J]. PLoS Biology, 2014, 12(2): e1001792. DOI:10.1371/journal.pbio.1001792 |
| [61] |
Yang L, Teixeira PJ, Biswas S, et al. Pseudomonas syringae type Ⅲ effector HopBB1 promotes host transcriptional repressor degradation to regulate phytohormone responses and virulence[J]. Cell Host Microbe, 2017, 21(2): 156-168. DOI:10.1016/j.chom.2017.01.003 |
| [62] |
El Oirdi M, El Rahman TA, Rigano L, et al. Botrytis cinerea manipulates the antagonistic effects between immune pathways to promote disease development in tomato[J]. Plant Cell, 2011, 23(6): 2405-2421. DOI:10.1105/tpc.111.083394 |
| [63] |
Zhu W, Wei W, Fu Y, et al. A secretory protein of necrotrophic fungus Sclerotinia sclerotiorum that suppresses host resistance[J]. PLoS One, 2013, 8(1): e53901. DOI:10.1371/journal.pone.0053901 |
| [64] |
Plett JM, Daguerre Y, Wittulsky S, et al. Effector MiSSP7 of the mutualistic fungus Laccaria bicolor stabilizes the Populus JAZ6 protein and represses jasmonic acid(JA)responsive genes[J]. Proc Natl Acad Sci USA, 2014, 111(22): 8299-8304. DOI:10.1073/pnas.1322671111 |
| [65] |
Gimenez-Ibanez S, Chini A, Solano R. How microbes twist jasmonate signaling around their little fingers[J]. Plants(Basel), 2016, 5(1): 9. |
| [66] |
Thatcher LF, Gardiner DM, Kazan K, et al. A highly conserved effector in Fusarium oxysporum is required for full virulence on Arabidopsis[J]. Mol Plant Microbe In, 2012, 25(2): 180-190. DOI:10.1094/MPMI-08-11-0212 |
| [67] |
Thatcher LF, Manners JM, Kazan K. Fusarium oxysporum hijacks COI1-mediated jasmonate signaling to promote disease development in Arabidopsis[J]. Plant J, 2009, 58(6): 927-939. DOI:10.1111/tpj.2009.58.issue-6 |
| [68] |
Sugio A, Kingdom HN, MacLean AM, et al. Phytoplasma protein effector SAP11 enhances insect vector reproduction by manipulating plant development and defense hormone biosynthesis[J]. Proc Natl Acad Sci USA, 2011, 108(48): E1254-E1263. DOI:10.1073/pnas.1105664108 |
| [69] |
Ziebell H, Murphy AM, Groen SC, et al. Cucumber mosaic virus and its 2b RNA silencing suppressor modify plant-aphid interactions in tobacco[J]. Sci Rep, 2011, 1: 187. DOI:10.1038/srep00187 |
| [70] |
Lewsey MG, Murphy AM, MacLean D, et al. Disruption of two defensive signaling pathways by a viral RNA silencing suppressor[J]. Mol Plant Microbe Intrant, 2010, 23(7): 835-845. DOI:10.1094/MPMI-23-7-0835 |
| [71] |
Wu D, Qi T, Li WX, et al. Viral effector protein manipulates host hormone signaling to attract insect vectors[J]. Cell Res, 2017, 27(3): 402-415. |
| [72] |
Li R, Weldegergis BT, Li J, et al. Virulence factors of geminivirus interact with MYC2 to subvert plant resistance and promote vector performance[J]. Plant Cell, 2014, 26(12): 4991-5008. DOI:10.1105/tpc.114.133181 |
| [73] |
Yang JY, Iwasaki M, Machida C, et al. betaC1, the pathogenicity factor of TYLCCNV, interacts with AS1 to alter leaf development and suppress selective jasmonic acid responses[J]. Gene Dev, 2008, 22(18): 2564-2577. DOI:10.1101/gad.1682208 |
| [74] |
Zhang T, Luan JB, Qi JF, et al. Begomovirus-whitefly mutualism is achieved through repression of plant defences by a virus pathogenicity factor[J]. Mol Ecol, 2012, 21(5): 1294-1304. DOI:10.1111/j.1365-294X.2012.05457.x |
| [75] |
Zhang C, Ding Z, Wu K, et al. Suppression of jasmonic acid-mediated defense by viral-inducible microRNA319 facilitates virus infection in rice[J]. Mol Plant, 2016, 9(9): 1302-1314. DOI:10.1016/j.molp.2016.06.014 |
| [76] |
Lozano-Duran R, Rosas-Diaz T, Gusmaroli G, et al. Geminiviruses subvert ubiquitination by altering CSN-mediated derubylation of SCF E3 ligase complexes and inhibit jasmonate signaling in Arabidopsis thaliana[J]. Plant Cell, 2011, 23(3): 1014-1032. DOI:10.1105/tpc.110.080267 |
| [77] |
Jia Q, Liu N, et al. CLCuMuB betaC1 subverts ubiquitination by interacting with NbSKP1s to enhance geminivirus infection in Nicotiana benthamiana[J]. PLoS Pathog, 2016, 12(6): e1005668. DOI:10.1371/journal.ppat.1005668 |
| [78] |
Westwood JH, Lewsey MG, Murphy AM, et al. Interference with jasmonic acid-regulated gene expression is a general property of viral suppressors of RNA silencing but only partly explains virus-induced changes in plant-aphid interactions[J]. J Gen Virol, 2014, 95: 733-739. DOI:10.1099/vir.0.060624-0 |
| [79] |
Rojas MR, Hagen C, Lucas WJ, et al. Exploiting chinks in the plant's armor: evolution and emergence of geminiviruses[J]. Annu Rev Phytopathol, 2005, 43: 361-394. DOI:10.1146/annurev.phyto.43.040204.135939 |
| [80] |
Cui X, Li G, et al. A begomovirus DNAbeta-encoded protein binds DNA, functions as a suppressor of RNA silencing, and targets the cell nucleus[J]. J Virol, 2005, 79(16): 10764-10775. DOI:10.1128/JVI.79.16.10764-10775.2005 |
| [81] |
Li HW, Lucy AP, Guo HS, et al. Strong host resistance targeted against a viral suppressor of the plant gene silencing defence mechanism[J]. EMBO J, 1999, 18(10): 2683-2691. DOI:10.1093/emboj/18.10.2683 |
| [82] |
Oka K, Kobayashi M, Mitsuhara I, et al. Jasmonic acid negatively regulates resistance to tobacco mosaic virus in tobacco[J]. Plant Cell Physiol, 2013, 54(12): 1999-2010. DOI:10.1093/pcp/pct137 |




