棉花作为我国重要的经济作物,在推动国民经济的发展与人民生活水平的提高方面起着重要作用。随着棉花产业的持续发展,新品种审定速度快、数量多,而骨干亲本数量有限且反复利用导致棉花品种的遗传差异越来越小。另外,随着转基因技术在棉花育种中的应用,在原品种基础上改良少数或单个性状的衍生品种增多,完全依据形态性状进行棉花品种的辨别越来越困难[1],且传统的棉花品种真实性和纯度是通过田间种植的方法鉴定的,耗时长、成本高、时效性差,易受环境因素的影响[2]。目前,生产上应用的棉花类型有常规品种和杂交品种,大多数优势杂交棉花品种是通过人工去雄制种,制种过程中,母本因去雄不彻底或漏去雄易形成自交铃,严重影响杂交种纯度,而常规品种的遗传纯度(位点纯合)和非遗传混杂(生产或加工时的异源种子的掺入、异源花粉的异花授粉等)也会给棉花品种纯度和遗传多样性的鉴别带来很大问题。如果不能及时、准确地进行鉴定,则会给棉花生产带来严重的损失。因此,对棉花品种快速、准确而高效的纯度检测,对于棉花品种的推广应用意义重大。
棉花品种纯度的检测方法有种子形态学、幼苗鉴定、蛋白质电泳以及田间小区种植鉴定等。近年来,随着生物技术的飞速发展与在育种途径的多样化,分子标记技术因其准确可靠、批量操作及不受环境因素制约等优点已被广泛应用于农作物品种真实性与纯度鉴定中[3]。其中,由于SSR多态性丰富,而且覆盖整个基因组的编码区和非编码区,成为一种新兴分子标记[4]。棉花基因组中存在着丰富的SSR标记,SSR标记以其独特的优势,已被广泛用于棉花的遗传图谱构建、目标基因定位、遗传多样性分析、种质鉴定及分子标记辅助选择等方面的研究。马轩等[5]利用SSR标记构建了彩色棉的指纹图谱,为彩色棉品种的真伪鉴定和纯度检测提供了方法。武耀廷等[6]针对SSR引物的多态性进行了筛选,认为筛选的SSR标记可对棉花杂交种进行纯度鉴定。刘勤红等[4]以转基因抗虫杂交棉鲁棉研15号母本、父本及其F1代为材料,通过筛选217对棉花异源四倍体的SSR引物,获得了十几个区分鲁棉研15号亲本及其F1代的标记位点,为鲁棉研15号杂交种的纯度鉴定提供了方法。郭宝生等[7]利用9对SSR引物对46份陆地棉与海岛棉品种(系)进行了遗传多样性分析,结果表明,渐渗系主要遗传背景为陆地棉,由于渐渗位点和基因不同,从而造成一定的差异,被聚类到不同小组。滕中华等[8]通过对4个棉花栽培种的29份棉花品种/品系SSR遗传多样性分析表明,棉花栽培种陆地棉、海岛棉、中棉和草棉种内遗传差异小,认为种间杂交渐渗系的培育是增加陆地棉遗传多样性的有效途径。李武等[9]利用SRAP标记对国内外的56份海岛棉品种进行了遗传多样性分析,结果表明,我国现在育成的品种相对于早期品种遗传多样性在逐渐降低。吴大鹏等[10]利用SSR技术研究了不同海岛棉生产国的海岛棉品种资源的遗传亲缘关系和遗传多样性,结果表明SSR标记能较好地揭示供试棉花品种之间的遗传差异和亲缘关系。李成奇等[11]对来自前苏联、中国、美国和埃及的20份海岛棉种质资源进行SSR分析,较好地揭示了供试棉花品种之间的遗传差异和亲缘关系。
目前,采用田间调查品种性状的方法因耗时长、成本高、效果差而特别容易受到环境条件的影响,SSR标记因其操作简单、重复性好、对DNA质量要求不高、属共显性标记而被认为是品种鉴定较为理想的分子标记之一,成为品种鉴定技术的研究热点[12]。基于SSR分子标记已构建了水稻[13-14]、大豆[15]、小麦[16-17]、玉米[18-19]、马铃薯[20]等主要农作物的指纹图谱。SSR标记技术在棉花中也逐步得到了广泛的应用[21-22]。本研究采用SSR引物首先对现有的58份棉花种质资源进行纯度鉴定,确保对种质资源的有效利用,并通过遗传多样性分析,进一步了解种质间的亲缘关系与遗传组成,旨为杂交亲本的选择利用、合理组配提供参考依据。
1 材料与方法 1.1 材料供试材料为中国农业科学院棉花研究所提供的棉花种质资源,包括国家审定品种及国外引进品种共58份(表 1)。所用26对核心SSR引物由国家区试站付小琼老师提供,由上海生物工程公司合成。
主要仪器包括细胞破碎仪(Retsch MM400)、高速低温离心机(Bio-Rad)、PCR仪(Bio-Rad)、水浴锅、DYY-6C型电泳仪和微量移液枪(Eppendorf)等。
主要试剂包括N,N-甲叉双丙烯酰胺(Bis)、丙烯酰胺(Acr)、三羟甲基氨基甲烷(Tris)、四甲基乙二胺(TEMED)、过硫酸铵(APS)、硼酸、三氯甲烷、异丙醇、异戊醇、CTAB、无水乙醇和硝酸银等,以上试剂均为分析纯,购自上海生物工程公司。10×buffer、dNTPs、Taq酶等购自天根生物公司。
1.2 方法 1.2.1 棉花叶片DNA的提取在棉花幼苗期(5-6片真叶)进行取样,取植株顶部完全展开的幼嫩叶片,每个棉花材料连续选取10株,取完后植株挂牌,叶片采集后立即放入冰盒内带回-80℃保存。采用CTAB法提取棉花叶片DNA[23],并且参照付小琼等[4]方法进行改进,用核酸和蛋白测定仪测定棉花基因组DNA在230、260和280 nm处的OD值,读取稀释后样品DNA浓度,根据样品OD260/OD280和OD260/OD230的比值判断DNA样品的纯度,并用1%琼脂糖凝胶电泳检测基因组DNA的质量及完整性。
1.2.2 SSR-PCR扩增与聚丙烯酰胺凝胶电泳SSR-PCR反应体系为20 μL,包含10×PCR Buffer 2.0 μL、dNTPs(10 mmol/L)0.8 μL、Taq酶(2.5 U)0.3 μL、上游引物(10 μmol/L)1 μL、下游引物(10 μmol/L)1 μL、DNA模版1.0 μL和dd H2O 13.9 μL。PCR反应程序为95℃ 5 min;94℃ 30 s,45 s,72℃ 90 s,30个循环;72℃ 10 min;4℃保存。
取2 μL PCR产物加Loading buffer,于8%聚丙烯酰胺凝胶上进行电泳,恒压200 V,时间1.5 h,电泳结束后拆板,并用蒸馏水漂洗2次,将胶加入染色液(0.2%的AgNO3、1%的冰醋酸、10%的无水乙醇)进行银染,然后用蒸馏水漂洗2次,再加入显影液(1.5%的无水NaOH,每200 mL溶液加1 mL甲醛)进行显色,最后用数码相机对凝胶进行照相,并统计条带。
1.2.3 条带统计与数据处理依据SSR-PCR产物在凝胶上的相对位置,对各引物扩增的基因带型进行统计,按条带有无分别赋值,有带记为1,无带记为0,统计总位点数和多态性位点数。根据各引物扩增谱带结果计算SSR标记的变异的多态性信息含量(Polymorphism information content,PIC),PIC=1-∑(Pi)2,其中,Pi表示第i条谱带出现的频率[24]。各类群内多样性指数H0 =-∑PilnPi,n个不同群体的平均多样性指数Hgroup =ΣH0/n,总的多样性指数Hsp= ΣPlnP是把所有的供试材料群体作为一个整体时,以表型频率P计算而得到的多样性[25]。利用NTsys-pc 2.10软件进行遗传多样性分析,计算各样本间的遗传相似系数(Genetic similarity,GS);聚类分析按基于Nei-Li遗传相似系数按不加权成对数算术平均法(Unweighted pair group method with arithmetic,UPGMA)进行。
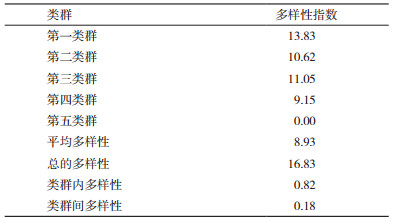
2 结果 2.1 棉花基因组DNA质量检测棉花种质材料经CTAB方法提取DNA后,于1%琼脂糖凝胶电泳检测其完整性(图 1),并经核酸测定仪测定其质量,结果表明,提取的DNA质量较好,OD260/OD280在1.73-2.00,可以作为进行SSR-PCR扩增的模板。
|
| 图 1 DNA琼脂糖检测 1-16:部分种质材料基因组DNA;M:D2000 DNA Marker |
利用26对核心引物对供试材料进行SSR-PCR电泳检测,分析纯度。读取条带时,同一材料中若每株植株有2对及以上的引物扩增谱带与其他植株不同,则定义为杂株,未达到2对引物扩增谱带不同的植株定义为正常株。通过电泳检测发现,各种质资源材料中所含杂株数情况不一(图 2),在同一引物扩增条件下,M1和M2材料中的所有植株谱带一致,表明在该引物条件下,M1和M2材料纯度高,无杂株;M3材料中的1和7植株与其他植株谱带不同,M4材料中的4植株与其他植株的谱带不同,在该引物检测条件下,将M3材料的植株1和7、M4材料的植株4视为杂株,表明M3和M4材料的品种纯度降低。

|
| 图 2 同一SSR引物进行的不同棉花种质资源电泳检测 M1-M4:不同棉花材料;1-10:同一材料的不同植株 |
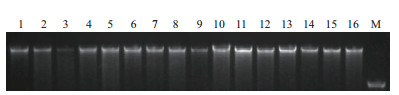
不同棉花种质资源的SSR标记纯度检测结果如图 3所示,纯度概率在20%-90%,其中AL5503的纯度最低,为20%,占供试材料的1.7%;冀2053、海兴耐盐10、冀省331、国丰棉5号、中远9120、华中远92-217、豫棉11号、Ari3697、L142-9、PD3249、SC-1(GP72)等11份种质材料的纯度较高,为90%,占供试材料的19.0%;纯度为70%-80%的种质材料最多,共有31份,占供试材料的53.4%。
|
| 图 3 不同棉花种质资源SSR标记纯度检测 |
分子标记检测可以与田间性状观察相互验证,在引物2和引物5扩增条件下,供试材料“冀省03H17”的植株中出现了谱带不一致的情况(图 4-A,图 4-B),此时将具有特异谱带的植株定义为杂株。通过田间定株观察表型性状,发现杂株的叶形与叶色与正常株存在一定的差异(图 4-C和图 4-D),与正常植株叶片相比,杂株叶形表现为典型“鸡脚叶”,裂口较深,且叶色较浅有斑纹。分子标记鉴定与田间性状观察结果一致,说明SSR纯度检测的准确度较高,可作为快速检测棉花种质资源纯度的方法。

|
| 图 4 棉花种质材料SSR检测与表型鉴定对比 A:引物2扩增谱带;B:引物5扩增谱带;(1和3:“冀省03H17”中杂株模板;2和4:“冀省03H17”中正常株模板;C:“冀省03H17”中杂株叶片;D:“冀省03H17”中正常株叶片 |
由表 2可知,26对SSR核心引物在58份材料中共扩增出85种多态性基因型,每个标记检测到的基因型数为2-5,最多的为NAU1167、DPL0528、DPL0917(图 5)和TMB1638引物,最少的为NAU3419、HAU1300、NAU1362、MON168、BNL3033、BNL1026、NAU1102、CGR6410、BNL3173和CIR170引物,平均每对引物扩增出3.27个基因型,基因型数大于平均值的标记12个,占比46.2%;标记间的多态性信息含量介于0.073 7-0.928 1,平均为0.363 9,其中多态性信息含量大于平均数的标记14个,占比53.8%,基因型数和多态性信息均大于平均值的标记5个,占比19.2%。
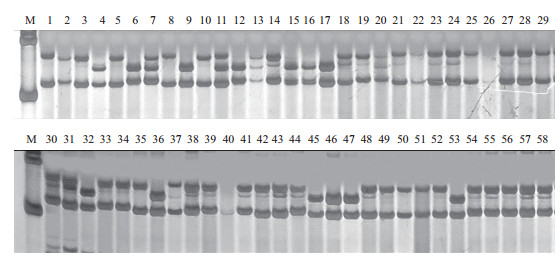
|
| 图 5 引物DPL0917对58份棉花种质资源的扩增电泳图 |
采用Nei-Li相似系数(GS)计算供试材料的电泳谱带结果,得到供试材料相似系数矩阵。58份棉花种质资源材料的GS变化范围为0.305 1-0.898 3,变幅为0.593 2。由相似系数矩阵可以看出,“AL5503”与“SC-1(GP72)的遗传相似系数最小,为0.305 1,其次是与“G51-1”、“冀省病检17”、“西南远917”等材料的相似系数最小,其遗传距离较大,表明这些材料之间的亲缘关系最远。而“川109-1”和“中远9120”的遗传相似系数最大,为0.898 3,即两者遗传距离最小,表明它们的亲缘关系最近,该结果与其遗传背景接近有关。
2.5 聚类分析对供试棉花种质资源遗传差异按UPGMA法进行聚类分析。聚类结果(图 6)显示,58份种质材料在遗传相似系数0.63水平上分为5个类别。第一类包含24份供试材料,占41.4%,其遗传相似系数为0.491 5-0.864 4,变化幅度为0.372 9,多样性指数为13.83。该组内又可分为3个亚类,其中“冀1972”与组内其他材料的遗传相似系数较小,表明在遗传背景上,与其他材料有较大差异;第二类包含7份供试材料,分别为“日辉棉6号”、“F112”、“苏远04-129”、“泗阳6821”、“皖2067-657”、“LineF(HY330)”和“PD3249”,占12.1%,其遗传相似系数为0.542 4-0.847 5,变化幅度为0.305 1,多样性指数为10.62。其中“LineF(HY330)”和“PD3249”为国外引进种质资源;第三类包含18份供试材料,占31.0%,包括“中远”系列材料3份,“冀”系列材料6份,另外还包括“豫棉11号”、“鲁7219”等材料,表明该组内主要包含黄河流域的棉花种质资源。组内遗传相似系数为0.559 3-0.898 3,变化幅度为0.339 0,多样性指数为11.05;第四类包含8份供试材料,占13.8%,分别为“鲁E1138”、“百棉985”、“苏棉20”、“丰优棉809”、“徐棉266”、“中棉所8号”、“苏研118”和“源棉2号”,遗传相似系数为0.576 2-0.796 6,变化幅度为0.220 4,多样性指数为9.15;第五类包含1份供试材料,为“AL5503”,该材料与“SC-1(GP72)”的遗传相似系数最小,为0.305 1,与“冀省资长”的遗传相似系数最大,为0.661 0,表明在所有供试材料中,“AL5503”与“SC-1(GP72)”的遗传背景差异较大,而与“冀省资长”的遗传背景最为接近(图 7)。
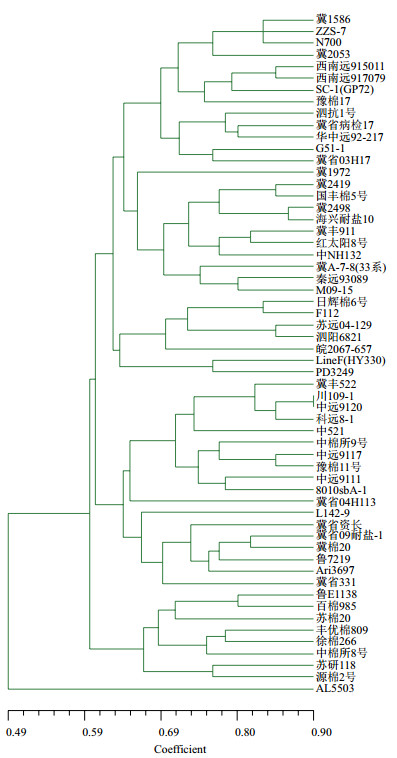
|
| 图 6 棉花种质资源聚类图 |

|
| 图 7 棉花种质资源聚类分析二维分布图 |
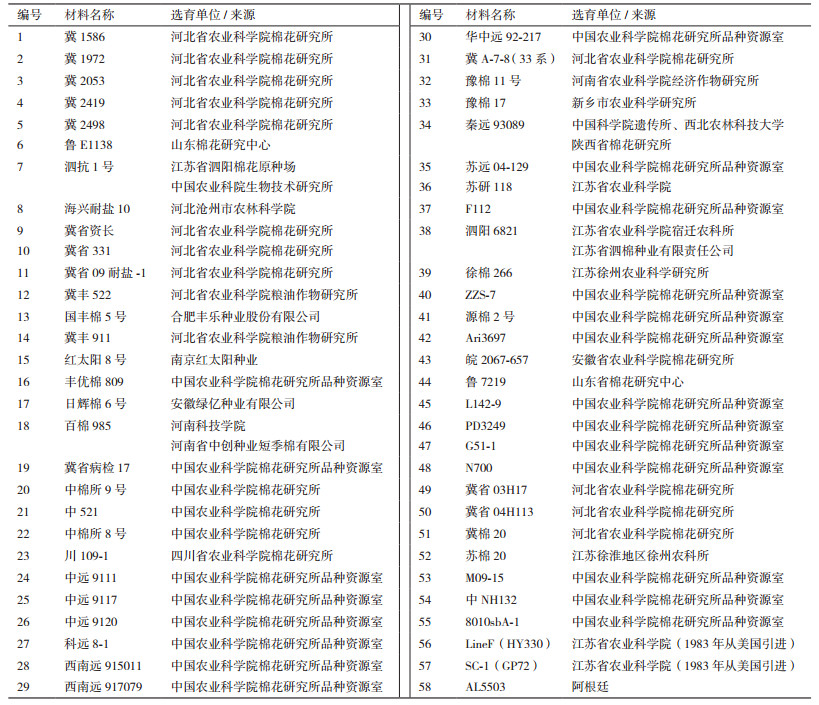
基于聚类分析结果,分别统计每个位点谱带出现的频率,按公式计算各类群内的多样性指数、平均多样性指数、类群内多样性指数及类群间多样性指数(表 3)。从表 3可以看出,除了第五类群只有一个材料外,其他各群体多样性指数在9.15-13.83,平均多样性指数为8.93,类群内多样性指数为0.82,类群间多样性指数为0.18,表明棉花种质资源的遗传变异更多的是来群体内,各类群间差异不大。
棉花的性状大多属数量性状,由多基因控制,易受环境影响。现有的鉴定方法中,同工酶和蛋白质电泳可检测的位点少,蛋白质类型不多,多态性水平低,对于不同品种不能有效区分。分子标记则是在分子水平上揭示不同材料之间存在的遗传差异,如棉花产量、纤维品质均表现为数量性状的遗传方式,易受外界环境条件影响,分子标记技术可在棉株生育早期检测后期性状,准确鉴定棉花品种间的差异,对于提高种子纯度检测效率有重要意义。一种理想的分子标记应具有多态性高、重复性和稳定性好、简便宜操作、带型清晰易统计、在染色体上分布均匀及开发使用成本低等特点[26]。SSR标记具有按孟德尔遗传方式分离、共显性遗传等特点,且多态性较高,程序简单,成本较低,目前已广泛应用于许多作物的基因定位、QTL分析、品种真实性鉴定、种子纯度检测及遗传多样性研究当中[27]。本研究结果表明,53.4%的种质材料纯度在70%-80%,来源于阿根廷的“AL5503”材料纯度最低,这可能是由引进过程中繁种方式及选种时的机械混杂造成的。通过田间表型观察发现,“冀省03H17”材料植株中存在叶色与叶形区别于其他植株的杂株,结合SSR谱带类型发现,该杂株扩增出了特异谱带类型,表明SSR纯度检测的准确性较高,是一种快速进行棉花品种纯度检测的方法。
3.2 棉花种质资源聚类结果分析58份棉花种质资源材料的相似系数矩阵表明,来源于阿根廷的“AL5503”与其他材料的遗传相似系数较小,同为远缘后代的“川109-1”和“中远9120”的遗传相似系数最大,说明种质间的遗传相似性与遗传背景有关。在遗传相似系数0.63水平上将58份资源分为5大类,与系谱分析结果[28]部分吻合。例如,“豫棉11号”是以(中棉所12号×(河南69×(完紫×科遗)))组合选育而成的,而“中棉所12号”又是经“乌干达4号”与“冀棉1号”杂交获得的,所以“豫棉11号”既带有“乌干达棉”的血统,又带有“冀棉1号”的血统。“冀棉20”则是从((海陆野×海陆野)×冀棉10号)组合后代中选育而成的,“冀棉10号”则是从(冀棉5号×冀棉1号)的组合中选育得到的,因此,“冀棉20”也带有“冀棉1号”的血统。“中棉所9号”则是从((中棉所4号×乌干达3号)×陕3778)的组合中得到的,带有乌干达棉血统。所以,“豫棉11号”、“冀棉20”和“中棉所9号”被归在了第三类。“徐棉266”是由((苏棉20×渝棉1号)×徐99-2)复合组合中选育而成的,与“苏棉20”归在第四类。这表明,这些品种资源的DNA聚类与其地理生态来源无关,而是与品种的亲缘关系相关性较高,亲缘关系近,则较早聚为一类,反之,则较晚才聚一类。但也有不吻合的情况,如“豫棉17号”是以“朝阳1号”后代((中383-1×373-3)×中6331)组合后代在病圃中选抗病株育成,追溯其系谱来源,“豫棉17号”带有“中棉所8号”的血统,但两材料却分别归属于第一类和第四类,追溯其系谱发现,尽管“豫棉17号”是从“中棉所8号”的复交后代群体中选育而成,但由于经过多代复交,其复交花粉来源于杂交后代,所以其遗传基础比较复杂,不能单从系谱来源来推断其聚类结果,利用分子标记分析的是总DNA水平的差异,其聚类结果能更真实地反映品种间的遗传差异。对58份种质材料的遗传相似系数聚类分析结果基本上反映了品种之间的亲缘关系。
品种选育过程是复杂的,育成品种的遗传基础不仅与选择方法、目标性状及生态环境因素有一定的关系,还与选用的亲本有直接关系[29]。通过遗传多样性分析,可明确各材料间亲缘关系的远近,结合种质材料自身品质和产量性状的优缺点进行优势互补,为针对性的选择杂交亲本提供参考依据,对拓展棉花种质基础、发掘和利用优异等位基因进行新品种培育具有重要意义。
4 结论利用SSR分子标记方法,对58份棉花种质资源的纯度与遗传多样性进行了检测与分析,所测材料的纯度概率介于20%-90%,且分子标记检测结果与田间表型相吻合。SSR-PCR共扩增出85种多态性基因型,每个标记检测到的基因型数在2-5,平均为3.27个,引物多态性信息量(PIC)介于0.073 7-0.928 1,平均为0.363 9,在GS=0.63水平上,将供试材料分为5大类,系谱分析表明,这些品种资源的DNA聚类与其地理生态来源无关,而是与品种的亲缘关系相关性较高,表明棉花种质资源遗传的多样性与复杂性。
| [1] |
张玉翠, 杨伟华, 匡猛, 等. 32个棉花主栽品种DNA指纹图谱构建及遗传多样性分析[J]. 棉花学报, 2012(2): 120-126. |
| [2] |
匡猛, 杨伟华, 许红霞, 等. 中国棉花主栽品种DNA指纹图谱构建及SSR标记遗传多样性分析[J]. 中国农业科学, 2011, 44(1): 20-27. |
| [3] |
付小琼, 杨付新, 刘逢举, 等. 棉花总DNA快速提取技术及其在国家棉花区试中的应用[C]. 安阳: 中国棉花学会2011年年会论文汇编. 2011: 130-133.
|
| [4] |
刘勤红, 王芙蓉, 张军, 等. 利用SSR标记鉴定鲁棉研15号杂交种纯度的研究[J]. 山东农业科学, 2003(2): 7-9, 16. |
| [5] |
马轩, 杜雄明, 孙君灵. 18个彩色棉品系的SSR指纹分析[J]. 植物遗传资源学报, 2003, 4(4): 305-310. |
| [6] |
武耀廷, 张天真, 郭旺珍, 等. 陆地棉品种SSR标记的多态性及用于杂交种纯度检测的研究[J]. 棉花学报, 2001, 13(3): 131-133. |
| [7] |
郭宝生, 张建宏, 刘素恩, 等. 棉花品种分子标记遗传多样性检测[J]. 华北农学报, 2010, 25(S1): 47-49. DOI:10.7668/hbnxb.20102.S1.012 |
| [8] |
滕中华, 王晓雯, 张建, 等. 棉花栽培品种/品系的SSR标记遗传多样性分析[J]. 西南大学学报:自然科学版, 2012, 34(12): 20-26. |
| [9] |
李武, 倪薇, 林忠旭, 等. 海岛棉遗传多样性的SRAP标记分析[J]. 作物学报, 2008, 34(5): 893-898. |
| [10] |
吴大鹏, 房嫌嫌, 马梦楠, 等. 四个国家海岛棉品种资源的亲缘关系和遗传多态性研究[J]. 棉花学报, 2010, 22(2): 104-109. |
| [11] |
李成奇, 王晓芸, 张晓飞, 等. 百棉系列棉花自交系品种(系)SSR指纹图谱构建[J]. 棉花学报, 2011, 23(3): 228-234. |
| [12] |
Zhang HY, Wang H, Guo S, et al. Identification and validation of a core set of microsatellite markers for genetic diversity analysis in watermelon[J]. Citrullus lanatus Thunb Matsum & Nakai Euphytica, 2012, 186: 324-342. |
| [13] |
庄杰云, 施勇烽, 应杰政, 等. 中国主栽水稻品种微卫星标记数据库的初步构建[J]. 中国水稻科学, 2006(5): 460-468. |
| [14] |
程保山, 万志兵, 洪德林. 35个粳稻品种SSR指纹图谱的构建及遗传相似性分析[J]. 南京农业大学学报, 2007(3): 1-8. |
| [15] |
张军, 赵团结, 盖钧镒. 大豆育成品种农艺性状QTL与SSR标记的关联分析[J]. 作物学报, 2008, 34(12): 2059-2069. |
| [16] |
李根英, DreisigackerS, WarburtonML, 等. 小麦指纹图谱数据库的建立及SSR分子标记试剂盒的研发[J]. 作物学报, 2006, 32(12): 1771-1778. DOI:10.3321/j.issn:0496-3490.2006.12.001 |
| [17] |
王立新, 李云伏, 常利芳, 等. 建立小麦品种DNA指纹的方法研究[J]. 作物学报, 2007, 33(10): 1738-1740. DOI:10.3321/j.issn:0496-3490.2007.10.027 |
| [18] |
王凤格, 赵久然, 郭景伦, 等. 比较三种DNA指纹分析方法在玉米品种纯度及真伪鉴定中的应用[J]. 分子植物育种, 2003, 1(5/6): 655-661. |
| [19] |
赵久然, 王凤格, 郭景伦, 等. 中国玉米新品种DNA指纹库建立系列研究[J]. 玉米科学, 2003, 11(2): 3-5. |
| [20] |
段艳凤, 刘杰, 卞春松, 等. 中国88个马铃薯审定品种SSR指纹图谱构建与遗传多样性分析[J]. 作物学报, 2009, 35(8): 1451-1457. |
| [21] |
薛艳, 李保成, 张新宇, 等. 新疆早熟棉品种SSR分子标记体系的建立[J]. 石河子大学学报:自然科学版, 2011(1): 1-5. |
| [22] |
赵亮, 蔡彩平, 梅鸿献, 等. 用于区别不同棉花品种基因组特征的微卫星位点筛选[J]. 作物学报, 2012, 38(10): 1810-1817. |
| [23] |
孙志栋, 王学德, 倪西源, 等. 棉花DNA提取方法的探讨[J]. 浙江农业学报, 2004, 16(4): 177-181. |
| [24] |
Park YH, Alabady MS, Ulloa M, et al. Genetic mapping of new cotton fiber loci using EST-derived microsatellites in an interspecific recombinant inbred(RIL)cotton population[J]. Molecular Genetics and Genomics, 2005, 274(4): 428-441. DOI:10.1007/s00438-005-0037-0 |
| [25] |
石建斌, 侯万伟, 刘玉皎, 等. 蚕豆种质资源清蛋白遗传多样性分析[J]. 植物遗传资源学报, 2012, 13(1): 22-28. |
| [26] |
聂新辉, 尤春源, 李晓方, 等. 新陆早棉花品种DNA指纹图谱的构建及遗传多样性分析[J]. 作物学报, 2014, 40(12): 2104-2117. |
| [27] |
刘峰, 赵佩, 徐子初, 等. 一种适用于SSR-PCR的棉花干种子DNA快速简便提取方法[J]. 分子植物育种, 2010, 8(5): 981-986. |
| [28] |
黄滋康. 中国棉花品种及其系谱[M]. 北京: 中国农业出版社, 2007.
|
| [29] |
贺道华, 邢宏宜, 李婷婷, 等. 92份棉花资源遗传多样性的SSR分析[J]. 西北植物学报, 2010, 30(8): 1557-1564. |