2. 中国科学院大学,北京100049
2. University of Chinese Academy of Sciences, Beijing 100049
胚胎发育晚期丰富蛋白在种子发育后期阶段不断积累而得名[1]。LEA蛋白分布极为广泛,植物的其他组织和器官经非生物胁迫和ABA能诱导LEA蛋白的合成[2, 3],此外在蛭形轮虫、细菌、昆虫、线虫中都有发现LEA蛋白的存在[4-6]。LEA蛋白不仅能提升生命体对逆境胁迫的耐受性[7-9],而且LEA蛋白的异源表达也能提升转化子对逆境胁迫的耐受性。Wang等[10]将小麦TaLEA3导入羊草(Leymus chinensis plants)后,发现转基因羊草对干旱的耐受性明显提升。所以,LEA蛋白不仅是非生物胁迫环境因素选择的靶位点,也是局部研究植物适应机制的目标基因之一,克隆LEA明确其作用机制,对于转基因技术改良植物的抗逆性具有重要意义。
LEA蛋白根据不同的分类方法有不同的命名[11-13],Battaglia等[14]根据氨基酸同源性和保守基序将LEA蛋白分为7组。LEA_1、LEA_2、LEA_3、LEA_4、LEA_6和LEA_7组都有特定的保守基序,被视为典型LEA蛋白;而LEA_5组因为没有明显的保守基序或相似序列,被认为是非典型LEA蛋白。不同的LEA蛋白家族在功能和作用机制上并不具有明显的相似性,小麦中Em蛋白(LEA_1)体外可以防止柠檬酸合成酶的凝聚和失活[15];拟南芥中AtEm6(LEA_1)蛋白不仅是种子发育所必需,也能响应高盐胁迫[16-17]。LEA_2蛋白内在的无序结构,主要作为分子伴侣来发挥冷冻保护[18]。LEA_3可以响应多种胁迫,例如,水稻中OSLEA3(LEA_3)可以响应ABA诱导和高盐胁迫[19],玉米中的ZmLEA3(LEA_3)可以响应于高盐、低温、ABA诱导等多种途径[20-21]。LEA_5B家族的SAG21可以提升拟南芥对细菌病原体的耐受性和抗氧化能力,LEA_5A家族的Rab8可以提升玉米对干旱的耐受性[22]。
目前,LEA_5蛋白作为一类非同源蛋白,其功能机制尚且十分模糊。本研究从蒺藜苜蓿A17幼苗中克隆到一个编码胚胎发育晚期丰富蛋白的基因MtLEA5B。并通过生物信息学方法对其进行分析,运用RT-PCR和原核表达对其表达模式和对宿主菌在温度胁迫条件下的保护进行验证,为转基因技术改良植物的抗逆性提供参考奠定基础。
1 材料与方法 1.1 材料将蒺藜苜蓿A17种子用浓硫酸处理5-10 min,用双蒸水洗涤多次除去残余硫酸。处理后的种子置于含有1/2 MS固体培养基中,4℃春化2 d移至21℃、16 h/8 h光照条件下萌发。3 d后将已发芽的幼苗移栽到蛭石:营养土(3: 1)的混合基质中,在21℃、16 h/8 h光照条件下生长,每7 d用复合肥溶液浇灌1次。所用引物的合成由上海生物生工合成(上海)。
将生长3周的幼苗进行非生物胁迫和ABA处理。NaCl胁迫处理:用150 mmol/L NaCl水溶液浇灌培养基质,分别于0和8 h及1和3 d取样;ABA胁迫处理:将整株幼苗取出洗净砂石后,转移至铺有多层吸水纸的培养皿中,用0.05% Tween 20(V/V)的100 μmol/L脱落酸溶液喷洒,封盖防止植物脱水,分别于0、1、3和6 h取样;脱水胁迫处理:将幼苗取出洗净砂石,温室自然脱水,分别于0、4、8和12 h取样;低温胁迫处理:将幼苗转移至4℃、16 h/8 h光照培养箱中进行培养,分别于0和8 h及1和3 d,取样。取样后迅速液氮冷冻,-80℃冰箱保存,每组处理设3次生物学重复。
1.2.2 MtLEA5B的克隆和序列分析胁迫处理样本总RNA提取使用TRIzol®试剂(Invitrogen,USA),参照说明书使用Recombinant DNase I(RNase-free,TaKaRa,大连)去除基因组DNA。cDNA第一条链按PrimeScript ®RT reagent Kit(Perfect Real Time,TaKaRa,大连)的操作说明合成。以蒺藜苜蓿转录组cDNA为模板,MtLEA5BF/R为引物,用PyrobestTM DNA聚合酶(TaKaRa,大连)进行30次PCR扩增。使用PCR纯化试剂盒(上海生工生物,上海)纯化回收。
采用DNAMAN软件分析核酸序列;用ExPASy网站(http://web.expasy.org/translate/)分析开放阅读框OPF;用NCBI网站(http://www.ncbi.nlm.nih.gov/)进行核苷酸和蛋白质相似性检索;用SMART(http://smart.embl-heidelberg.de/)和Pfam(http://pfam.xfam.org/)进行蛋白质序列分析[23]。
1.2.3 MtLEA5B原核表达载体构建将上述PCR纯化产物经BamHⅠ和SacⅠ双酶切连接到pET30a载体中。重组质粒转化至大肠杆菌菌株(DH5α),筛选阳性克隆提取质粒,进行酶切鉴定,获得原核表达载体pET30a-MtLEA5B。
1.2.4 MtLEA5B蛋白的异源表达和热稳定性分析将质粒pET30a和pET30a-MtLEA5B转化至大肠杆菌BL21(DE3)菌株内。在含有50 mg/L卡那霉素的LB培养皿中筛选阳性克隆,接种于10 mL含有50 mg/L卡那霉素的LB液体培养基中,37℃,180 r/min震荡培养。12 h后,按照1: 100(V: V)转接到5 mL LB培养基中培养OD600=0.6-0.8。菌液中加入异丙基硫代半乳糖苷(Isopropyl-D-thiogalactopyranoside,IPTG)至最终浓度1.0 mmol/L。诱导3 h,取1 mL pET30a和pET30a-MtLEA5B菌液作为第一组,另取1 mL pET30a和pET30a-MtLEA5B菌液作为第二组。
将2组诱导样品8 000 r/min离心2 min,弃上清,用80 mL的灭菌双蒸水重悬,第一组直接100℃金属浴10 min;第二组加入20 μL 5×SDS,100℃金属浴10 min。15 000 r/min离心5 min,取上清,第1组加入20 μL 5×SDS。各取6 μL进行SDS-PAGE进行电泳。
1.2.5 异源表达大肠杆菌转化子抗逆性试验 1.2.5.1 定性试验将含有pET30a-MtLEA5B和pET30a载体的大肠杆菌进行液体培养和诱导,培养和诱导条件如1.2.4所述。培养诱导后的菌液OD600=0.9,用含有50 mg/L卡那霉素和1.0 mmol/L IPTG的LB液体培养基将诱导菌液分别稀释10、100和1 000倍,取原诱导菌液和稀释菌液进行胁迫处理试验。温度胁迫试验中分别用60℃金属浴温育30 min和-20℃冰箱冷冻24 h。将上述胁迫菌液和对照组菌液取10 μL滴加到含有1.0 mmol/L IPTG和50 mg/L卡那霉素的固体LB培养基中。37℃倒置培养24 h后进行观察。
1.2.5.2 定量分析大肠杆菌转化子的培养、IPTG诱导、胁迫条件以及LB固体培养基与1.2.5.1相同,当IPTG诱导菌液培养OD600=0.9,用含有50 mg/L卡那霉素和1.0 mmol/L IPTG的LB液体培养基稀释10倍,取100 μL均匀涂于固体LB培养基中,37℃倒置培养24 h进行菌落计数,公式如下:

以蒺藜苜蓿幼苗样品cDNA为模板进行扩增,获得MtLEA5B的cDNA序列,长度为285 bp;编码94个氨基酸(图 1-A),预测分子量为10.05 kD,理论等电点(pI)为9.40(https://web.expasy.org/cgi-bin/protparam/protparam)。基因序列在JCVI数据库(http://www.jcvi.org/)中显示MtLEA5B是位于第1染色体编码胚胎发育晚期丰富蛋白的基因;MtLEA5B蛋白富含丙氨酸(12.72%)和缬氨酸(11.70%),而半胱氨酸和组氨酸含量为0;DNAMAN软件显示平均亲水系数为-0.472(图 1-B)。
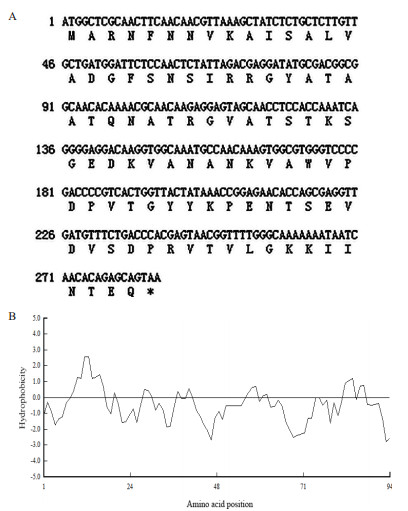
|
| 图 1 蒺藜苜蓿MtLEA5B的序列分析 A:蒺藜苜蓿LEA5B编码区序列和氨基酸序列;B:MtLEA5B蛋白质的疏水性预测 |
通过进行BlastP分析,MtLEA5B蛋白包含Pfam LEA_3结构域,并且与地三叶(Trifolium subterraneum)、大豆(Glycine max)、木豆(Cajanus cajan)和菜豆(Phaseolus vulgaris)豆科植物的LEA蛋白具有较高的相似性(图 2)。

|
| 图 2 蒺藜苜蓿MtLEA5B与其他植物LEA蛋白氨基酸序列比对分析 TsLEA:Trifolium subterraneum(GAU43746.1);GmLEA:Glycine max(NP_001237596.1);CcLEA:Cajanus cajan(XP_020204737.1);PvLEA:Phaseolus vulgaris(XP_007145200.1) |
通过对不同胁迫下MtLEA5B的半定量RT-PCR分析(图 3),发现MtLEA5B表达量上调,在蒺藜苜蓿幼苗中为诱导型表达。
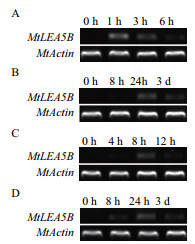
|
| 图 3 ABA诱导和非生物胁迫条件下MtLEA5B的RT-PCR分析 A:100 μmol/L ABA;B:150 mmol/L NaCl;C:温室脱水;D:4℃低温 |
重组质粒pET30a-MtLEA5B经酶切鉴定(图 4-A),转化至大肠杆菌表达菌株BL21(DE3),进行SDS-PAGE电泳检测。结果(图 4-B)显示,MtLEA5B蛋白原核表达理论值是10.05 kD,但是发现表达重组蛋白相比于蛋白理论分子量略大,并且诱导菌液直接在无菌水中煮沸,大部分蛋白都已变性,但目的蛋白含量未发生太大变化,表明MtLEA5B蛋白的高度偏向性导致其具有较高的抗性,在SDS变性溶剂和高温煮沸条件下仍然存在部分结构。
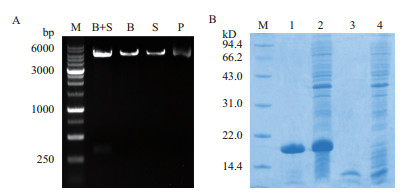
|
| 图 4 pET30a-LEA5B表达载体酶切鉴定(A)和SDS-PAGE电泳检测(B) M:1.0 kb-plus DNA Marker和低分子量蛋白质Marker;B+S:BamHⅠ+ SacⅠ双酶切;B:BamHⅠ单酶切;S:SacⅠ单酶切;P:pET30a-LEA5B质粒;1:含pET30a-LEA5B载体的BL21经IPTG诱导后沉淀中的总蛋白;2:含pET30a-LEA5B载体的BL21经IPTG诱导后上清液中的总蛋白;3:含pET30a载体的BL21经IPTG诱导后沉淀中的总蛋白;4:含pET30a-载体的BL21经IPTG诱导后上清液中的总蛋白 |
为鉴定大肠杆菌中过表达MtLEA5B是否对宿主菌具有保护作用。将含有pET30a-MtLEA5B和pET30a载体的大肠杆菌BL21(DE3)分别培养至OD600=0.9,经60℃高温和-20℃冷冻胁迫处理,发现含有pET30a-MtLEA5B载体的大肠杆菌生存状态明显增多(图 5-A)。
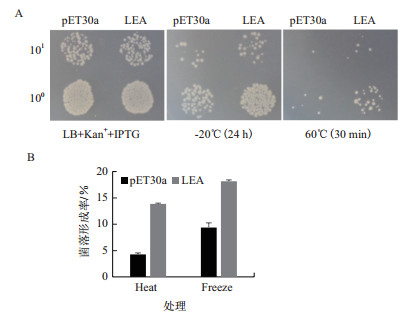
|
| 图 5 LEA5B蛋白过表达对大肠杆菌温度胁迫的保护 A:菌液滴板试验;B:菌落形成率统计 |
菌落计数结果(图 5-B)显示,在60℃高温胁迫处理后,含有BLpET30a载体的菌株菌落形成率仅有4.29%,含有pET30a-MtLEA5B载体的菌株菌落形成率为13.88%;在-20℃冷冻胁迫处理后,含有pET30a载体的菌株菌落形成率仅有9.39%,含有pET30a-MtLEA5B载体的菌株菌落形成率为18.16%。
3 讨论LEA蛋白与植物逆境胁迫耐受性密切相关,可以维持植物在极端环境条件下的正常生命代谢活动[24-27]。本研究在蒺藜苜蓿幼苗中克隆到定位于第1染色体上的MtLEA5B,编码LEA_5B类蛋白,经疏水氨基酸残基预测MtLEA5B蛋白具有较高的亲水性。MtLEA5B蛋白的氨基酸序列与地三叶、大豆、木豆等豆科植物具有高度一致性,表明LEA_5B蛋白具有种属特异性。其表达模式响应于高盐、脱水、温度胁迫和ABA处理,但MtLEA5B在不同胁迫条件下响应不尽相同,表明MtLEA5B参与蒺藜苜蓿对非生物胁迫抗性反应过程是复杂的,可能通过多种信号通路交叉进行。
虽然有实验结果对特定LEA蛋白功能和机制进行阐述,但LEA_5蛋白是一类疏水残基高于典型LEA蛋白的非同源蛋白,这些结果并不具有普适性。Baker和Galau等[28-29]发现LEA_5蛋白沸水孵化可能形成一种不溶的球形构象。本研究中,经沸水孵化后,大部分大肠杆菌细胞的内生蛋白质消失在上层清液与总蛋白提取的SDS样品缓冲中,但是MtLEA5B重组蛋白数量上并没有造成明显减少,推测原因是MtLEA5B重组蛋白质中富含的极性丙氨酸残基使具有较高热稳定性和亲水性,以及较短的氨基酸序列赋予它灵活的结构。此外,重组MtLEA5B蛋白的表观分子质量明显高于估计的分子质量,其原因可能是LEA蛋白分子的高亲水性或无序结构导致其在沸水和SDS中仍存在部分结构。
众多研究表明,异源表达LEA蛋白能够提升植物和细菌对非生物胁迫的耐受性[30-34,]。Rodriguez-Salazar等[35]研究发现LEA1蛋白响应于囊肿干燥且能提升维氏固氮菌高温耐受性。Wu等[36]发现过表达SmLEA可以增强大肠杆菌和丹参对干旱和高盐胁迫的耐受性。本研究在大肠杆菌中过表达MtLEA5B蛋白,明显增强了大肠杆菌对温度胁迫的耐受性。但是,MtLEA5B蛋白过表达在高盐和脱水条件下未发现有明显的保护作用,推测MtLEA5B对不同的胁迫有不同的响应机制[37-38]。此外有研究表明,LEA蛋白对植物的发育过程也具有调控作用[39-40]。因此MtLEA5B是否存在其他的调控机制尚且不明,需要进一步科研进行证实。
4 结论从蒺藜苜蓿中克隆获得MtLEA5B,其受外源激素ABA和高盐、脱水和温度胁迫的调控;其编码的蛋白质属于LEA_5B蛋白家族,在SDS变性剂和高温煮沸条件下仍能保持一定的结构。MtLEA5B在大肠杆菌转化子中超表达可以增强大肠杆菌对温度胁迫的耐受性。
| [1] |
Dure l, Greevway SC, Galau GA. Developmental biochemistry of cotton seed embryogenesis and germination:changing messenger ribonucleic acid populations as shown by in vitro and in vivo protein synthesis[J]. Biochemistry, 1981, 20(14): 4162-4168. DOI:10.1021/bi00517a033 |
| [2] |
Gram J, Bartels D. The molecular basis of dehydration tolerance in plant[J]. Annual Review of plant biology, 1999, 50(1): 571-599. DOI:10.1146/annurev.arplant.50.1.571 |
| [3] |
Battista JR, Park MJ, Mclemore AE. Incativation of two homologues of proteins presumed to be involved in the desiccation tolerance of plant sensitizes Deinococcus radiodurans R1 to desiccation[J]. Cryobiology, 2001, 43(2): 133-139. DOI:10.1006/cryo.2001.2357 |
| [4] |
Campos FC, Cuevas-Velazquez C, et al. Group 1 LEA proteins, an ancestral plant protein group, are also present in other eukaryotes, and in the archeae and bacteria domains[J]. Molecular Genetic Genomics, 2013, 288: 503-517. DOI:10.1007/s00438-013-0768-2 |
| [5] |
Wise MJ, Tunnacliffe A. POPP the question:what do LEA proteins do?[J]. Trends in Plant Science, 2004, 9(1): 13-17. DOI:10.1016/j.tplants.2003.10.012 |
| [6] |
Browne J, Tunacliffe A, Burnell A. Anhydrobiosis:plant desiccation gene found in a nematode[J]. Nature, 2002, 416(6876): 38. DOI:10.1038/416038a |
| [7] |
Bartels D, Salamini F. Desiccation tolerance in the resurrection plant Craterostigma Plantagineum. A contribution to the study of drought tolerance at the molecular level[J]. Plant Physiology, 2001, 127(4): 1346-1353. DOI:10.1104/pp.010765 |
| [8] |
李剑, 赵常玉, 张富生, 等. LEA蛋白与植物抗逆性[J]. 植物生理学通讯, 2010, 46(11): 1101-1108. |
| [9] |
zhao Y, wang Y, liu Q, et al. Cloning of a new LEA1 gene promoter from soybean and functional analysis in transgenic tobacco[J]. Plant Cell Tissue and Organ Culture, 2017, 130: 379-391. DOI:10.1007/s11240-017-1234-3 |
| [10] |
Wang L, Li X, Chen S, et al. Enhanced drought tolerance in transgenic Leymus chinensis TaLEA3[J]. Biotechnology Letter, 2009, 31(2): 313-319. DOI:10.1007/s10529-008-9864-5 |
| [11] |
Baker J, Van Dennsteele C, Dure L. Sequence and characterization of 6 lea proteins and their genes from cotton[J]. Plant Mol Biol, 1989, 12(5): 475-486. DOI:10.1007/BF00036962 |
| [12] |
Bray EA. Molecular responses to water deficit[J]. Plant Physiology, 1993, 103(4): 1035-1040. DOI:10.1104/pp.103.4.1035 |
| [13] |
Tunnacliffe A, Wise MJ. The continuing conundrum of the LEA proteins[J]. Naturwissenschaften, 2007, 94: 791-812. DOI:10.1007/s00114-007-0254-y |
| [14] |
Battaglia M, Olvera-Carrillo Y, Garciarrybio A, et al, The enigmatic LEA proteins and other hydrophilins[J]. Plant Physiology, 2008, 148: 6-24.
|
| [15] |
Goyal K, Walton LJ, Tunnacliffe A. LEA proteins prevent protein aggregation due to water stress[J]. Biochemistry Journal, 2005, 388(1): 151-157. DOI:10.1042/BJ20041931 |
| [16] |
Manfre AJ, Lanni LM, Marcotte WR. The Arabidopsis group 1 late embryogenesis abundant protein AtEM6 is required for normal seed development[J]. Plant Physiology, 2006, 140: 140-149. |
| [17] |
Tang W, Page M. Overexpression of the Arabidopsis AtEm6 gene enhances salt tolerance in transgenic rice cell lines[J]. Plant Cell Tissue and Organ Culture, 2013, 114: 339-350. DOI:10.1007/s11240-013-0329-8 |
| [18] |
Hand SC, Menzea MA, Tonerb M, et al. LEA proteins during water stress:not just for plants anymore[J]. Annual Review of Physiology, 73: 115-134. DOI:10.1146/annurev-physiol-012110-142203 |
| [19] |
Moons A, De Keyser A, Van Montagu M. A group 3 LEA cDNA of rice, responsive to abscisic acid, but not to jasmonic acid, shows variety-specific differences in salt stress response[J]. Gene, 1997, 1119(97): 197-204. |
| [20] |
Liu Y, Wang L, Cai GH, et al. Response of tobacco to the Pseudomonas syringae pv. tomato DC3000 is mainly dependent on salicylic acid signaling pathway[J]. FEMS Microbiology Letter, 2013, 344(1): 77-85. DOI:10.1111/femsle.2013.344.issue-1 |
| [21] |
Liu Y, Wang L, Xing X, et al. ZmLEA3, a multifunctional group 3 LEA protein from maize(Zea mays L.), is involved in biotic and abiotic stresses[J]. Plant Cell Physiology, 2013, 54(6): 944-959. DOI:10.1093/pcp/pct047 |
| [22] |
Amara L, Capellades M, Ludevid MD. Enhance water stress tolerance of transgenic maize plants over-expressing LEA Rab28 gene[J]. Plant Physiology, 2013, 170(9): 864-873. DOI:10.1016/j.jplph.2013.01.004 |
| [23] |
Larkin MA, Blackshields G, Brown NP, et al. Clustal W and Clustal X version 2. 0[J]. Bioinformatics Applications Note, 2001, 23(21): 2947-2948. |
| [24] |
Liu ZY, Li XL, Qi X, et al. Alfalfa science research by Chinese scholars science 1950:history and main topics[J]. Acta Prataculturas Sinica, 2015, 24(10): 58-69. |
| [25] |
Battaglia M, Covarrubias AA. Late Embryogenesis Abundant(LEA)proteins in Legumes[J]. Front Plant Sci, 2013, 4: 190. |
| [26] |
Kalemba EM, Bagniewska-Zadworna A, Ratajczak E. Multiple subcellular localizations of teins in the embryonic axes of common beech(Fagus sylvatica L.)seeds during maturation and dry storage[J]. Journal of Plant Growth Regulation, 2015, 34(1): 137-149. DOI:10.1007/s00344-014-9451-z |
| [27] |
Grahman LN, Chen L, Nazim S, et al. Interactions of intrinsically disordered Thellungiella salsuginea dehydrins TsDHN-1 and TsDHN-2 with membranes-synergistic effects of lipid composition and temperature on secondary structure[J]. Biochemistry and Cell biology, 2010, 88(5): 791-807. DOI:10.1139/O10-026 |
| [28] |
Baker I, Steele C, Dure L. Sequence and characterization of 6 LEA proteins and their genes from cotton[J]. Plant Mol Biol, 1988, 11: 277-291. DOI:10.1007/BF00027385 |
| [29] |
Galau GA, Wang HY, Hughes, et al. Cotton Lea5 and Lea74 encode atypical late embryogenesis-abundant proteins[J]. Plant Physiology, 1993, 101: 695-696. DOI:10.1104/pp.101.2.695 |
| [30] |
Liu R, Liu M, Liu J, et al. Heterologous expression of a Ammopiptanthus mongolocus late embryogensis abundant protein gene(Am-LEA)enhance Escherichia coli viability under cold and heat stress[J]. Plant Growth Regulation, 2010, 60(2): 163-168. DOI:10.1007/s10725-009-9432-6 |
| [31] |
Wang WG, Li R, Liu B, et al. Alternatively spliced transcripts of group 3 late embryogenesis abundant protein from Pogonatherum paniceum confer different abiotic stress tolerance in Escherichia coli[J]. Journal of Plant Physiology, 2012, 169(15): 1559-1564. DOI:10.1016/j.jplph.2012.06.017 |
| [32] |
Ochoa-Alfaro AE, Rodríguez-Kessler M, Pérez-Morales MB, et al. Functional characterization of an acidic SK3 dehydrin isolated from an Opuntia streptacantha cDNA library[J]. Planta, 2012, 235(3): 565-578. DOI:10.1007/s00425-011-1531-8 |
| [33] |
Lakshmi TV, Varalaxmi Y, et al. Metabolic engineering of SK2-type of dehydrinⅠ(DHN1)gene isolated from Sorghum bicolor enhances tolerance to water-deficit and NaCl stresses in transgenic tobacco[J]. Plant Omics, 2015, 8(6): 556. |
| [34] |
Shi J, Liu M, Chen Y, et al. Heterologous expression of the dehydrin-like protein gene AmCIP from Ammopiptanthus mongolicus enhance viability of Escherichia coli and tobacco under cold stress[J]. Plant Growth Regulation, 2015, 79(1): 1-10. |
| [35] |
Rodriguez-Salazar J, Moreno S, Espín g. LEA proteins are involved in cyst desiccation resistance and other abiotic stresses in Azotobacter vinelandii[J]. Cell Stress and Chaperones, 2017, 22: 397-408. DOI:10.1007/s12192-017-0781-1 |
| [36] |
Wu Y, Liu C, Kuang J, et al. Overexpression of SmLEA enhance salt and drought tolerance in Escherichia coli and Salvia miltiorrhiza[J]. Protoplasma, 2011, 1-9. |
| [37] |
Zhang L, Oht A, Takagi M, et al. Expression of plant group 2 and group 3 lea gene in Saccharomyces cerevisiae revealed functional divergence among LEA proteins[J]. Journal of Biochemistry, 2000, 127(4): 611-616. DOI:10.1093/oxfordjournals.jbchem.a022648 |
| [38] |
Lan T, Cai D, Zhang YZ. Expression of three different group soybean lea genes enhance stress tolerance in Escherichia coli[J]. Journal of Integrative Plant Biology, 2005, 42(5): 613-621. |
| [39] |
Park SC, Im YH, Jeong JC, et al. Sweetpotato late embryogensis abundant 14(IbLEA14)gene influences lignification and increases osmotic-and salt stress-tolerance of transgenic calli[J]. Planta, 2011, 233(3): 621-634. DOI:10.1007/s00425-010-1326-3 |
| [40] |
Salleh FM, Evans K, Goodall B, et al. A novel function for a redox-related LEA protein(SAG21/AtLEA5)in root development and biotic stress responses[J]. Plant, Cell & Environment, 2012, 35(2): 418-429. |





