2. 湖南农业大学南方粮油作物协同创新中心,长沙 410128;
3. 中国农业科学院蔬菜花卉研究所,北京 100081;
4. 贵州省江口县农牧科技局,贵州 554400
2. Southern Regional Collaborative Innovation Center for Grain and Oil Crops in China, Changsha 410128;
3. Institute of Vegetables and Flowers, Chinese Academy of Agricultural Sciences, Beijing 100081;
4. Jiangkou County Agriculture and Animal Husbandry Science and Technology Bureau of Guizhou Province, Guizhou 554400
植物在生长发育过程中受到外界生物因素及非生物因素的影响,通过长期进化过程,植物本身产生了一系列复杂的调控机制,miRNA对靶mRNA的沉默途径就是其中之一。microRNA(miRNA)是真核细胞中一类长度约为21-25 nt的非编码内源保守性单链小分子RNA,其作为一种转录后调控因子,通过对靶mRNA进行切割或造成翻译阻遏来抑制基因的表达[1]。研究发现,miRNA通过参与调控相关抗性基因的表达,对环境胁迫信号做出不同的响应[2-4]。MiR390是一种古老的高度保守的miRNAs,它的靶基因AGO7蛋白是RISC(RNA诱导沉默复合体)的重要组成部分,miR390进入RISC后对mRNA进行切割或翻译抑制使AGO7基因沉默[5]。目前,对植物miR390基因的研究大多集中在拟南芥和水稻等模式植物上,较多采用高通量测序技术结合生物信息学进行靶基因、作用位点和功能预测等方面的分析,运用实时定量PCR(Quantitative Real Time-PCR)、转基因技术等研究手段探究miR390与靶基因的互作关系。本文综述了miR390基因家族参与植物的生长发育过程和响应植物非生物胁迫中的作用,旨为阐明miRNA介导植物基因表达调控机制提供重要参考。
1 miR390基因家族简介 1.1 植物miR390的形成过程及作用机制植物miR390的形成过程是在细胞核中完成的,首先内源miRNA基因通过RNA聚合酶Ⅱ转录形成具有茎环结构的初级转录本(pri-miRNA),随后经SE(C2H2锌指蛋白SERRATE)-DCL1(Dicer-Like酶1)-HYL1(双链RNA结合蛋白HYPONASTIC LEAVESI)复合体切割形成前体小RNA(pre-miRNA)。pre-miRNA再次被该复合体切割产生miR390∷miRNA*二聚体。该二聚体在植物外运蛋白直系同源物5(Plant homolog of expor-tin-5,HASTY)的作用下从细胞核转运到细胞质中,然后该二聚体解旋释放出成熟的miR390,其在HYL1、HEN1(s-腺苷甲硫氨酸依赖的甲基转移酶)及DCL1的协助下与AGO7蛋白等组成RISC,进而对靶基因进行切割或抑制其翻译,另一条miRNA*逐步降解[6-7]。miR390作用其靶基因的调控途径与其他miRNAs的作用方式不同,它通过作用在TAS(Trans-acting siRNA gene)上诱导产生21个碱基长的tasiRNA(Trans-acting siRNA),再靶向作用于生长素因子ARF(Auxin response factor)基因[8-10]。在模式植物拟南芥中miR390被装载到包含有AGO7的沉默复合体(RISC)上,通过“Two-Hit”(两个miR390的靶向位点)的机制作用在AtTAS1上,然后诱导产生两个tasiARF(tasiRNA靶向ARF基因),这两个tasiARF序列相似,都可以作用在AtARF2/3/4基因上[5](图 1)。

|
| 图 1 植物中miR390的起源、合成及作用过程 |
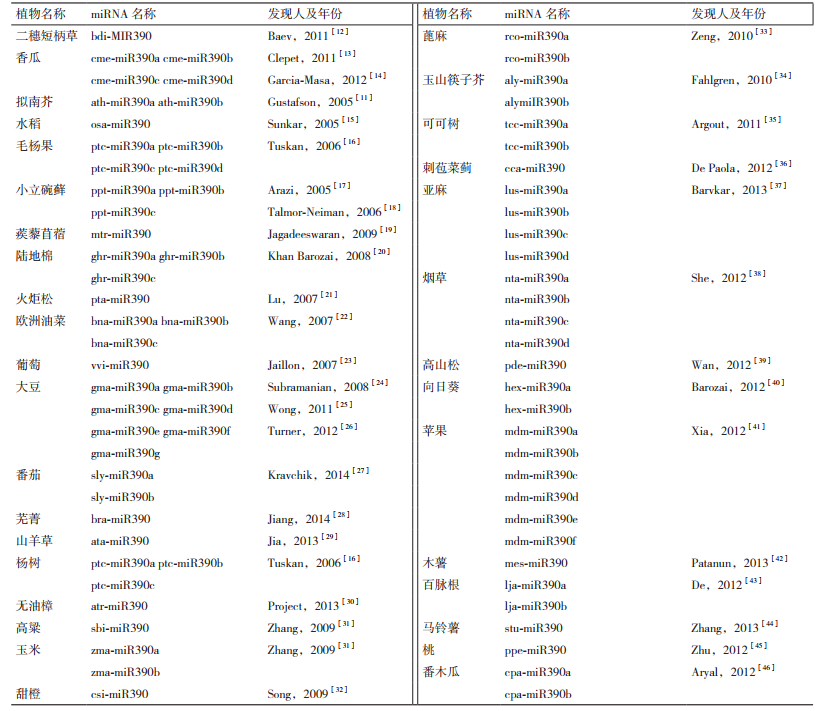
2005年,AM Gustafson等首次在拟南芥(Arabidopsis thaliana)中发现了两种miR390,即ath-miR390a和ath-miR390b,之后,miR390陆续在多种植物中被发现[11]。通过表达序列标签分析、高通量测序、miRNA微阵列分析、Northern杂交和克隆检测等方法,在二穗短柄草(Brachypodium distachyon)、香瓜(Cucumis melo)、拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、毛杨果(Populus trichocarp)、小立碗藓(Physcomitrella patens)、蒺藜苜蓿(Medicago truncatula)、马铃薯(Solanum tuberosum)、木薯(Manihot esculenta)、烟草(Nicotiana tabacum)、番茄(Solanum lycopersicum)、玉米(Zea mays)、高粱(Sorghum bicolor)和大豆(Glycine max)等多种植物中检测到了miR390,并且发现它在不同物种间具有高度的进化保守性。
英国曼彻斯特大学生命科学院2014年6月新发布的miRBase21.0版数据库(http://www.mirbase.org)记录了miR390基因家族的各个成熟序列。成熟的miR390含有21个碱基,具有5'端磷酸基和3'羟基结构,它的序列为5'aagcucaggagggauagcgcc3',是在Dicer酶的作用下由含有发夹结构的单链RNA前体经过加工后生成的。至今,该数据库中记录了miR390基因家族共有74个miRNA成员,分布于34个物种中(表 1)。其中,大豆中含有7种,苹果中含有6种,烟草等4种植物中含有4种;杨树等4种植物中含有3种;拟南芥等9种植物中含有2种;马铃薯等15种植物中只含有1种。在某些植物中,miR390的另一条臂也被加工产生miR390*,用-5p和-3p表示成熟序列来自于前体的5'和3'端,如stu-miR390a-5p来自于前体stu-miR390的5'端,stu-miR390-3p来自于前体stu-miR390的3'端。
生长素是主要的植物激素之一,参与植物生长发育的全方位调控。miR390通过作用于生长素信号参与调控植物的生长发育过程。它的靶基因之一ARF是一类能通过与生长素响应元件结合并促进或抑制基因表达的转录因子,在植物生长发育的不同时期和不同组织部位中普遍表达。miR390作用于TAS基因引发ta-siRNA的生物合成,再靶向作用于生长素应答因子ARF基因并调控其表达,进而引起植物一系列的表型变化[47]。在水稻中,根据Os-miR390的前体RNA结构特点设计引物,从基因组中克隆出其前体DNA片段,并将其构建在玉米U biI启动子驱动的表达载体中,转入水稻后得到过表达的Osta-siR2141植株,结果发现了异常的顶端分生组织及营养阶段发育迟缓等表型缺陷[48],并且miR390在根部及幼苗中的转录量较高[49],推测miR390参与水稻根部及幼苗的生长素信号转导过程,从而引发植株一系列表型缺陷。ARF基因与花芽分化、维管组织形成、胚的发育以及侧根发生等多个研究领域均有紧密关联[50]。研究发现,miR390能间接调控花期[51],它通过抑制ARF3和ARF4[52-53]转录因子来延长幼年期,从而推迟开花。进一步研究发现在拟南芥RNA聚合酶基因、DCL4基因和AGO7基因的功能缺失突变体中miR390不能正常合成,且表现提前进入成年期,并产生成年态叶片,这一结果论证了上述观点。钙和钙依赖性蛋白激酶(Calcium-dependent protein kinase,CDPK)在调节马铃薯结瘤过程中起着重要的作用,通过生物信息学的方法预测StCDPK1是miR390的转录后调节因子,Santin等[54]分析了其在马铃薯生命周期的不同组织和阶段中的表达发现,StCDPK1与马铃薯茎、根、匍匐茎到块茎转变期间以及块茎芽中的维管系统有紧密关联,qRT-PCR结果显示StCDPK1在马铃薯不同组织中普遍表达,并通过农杆菌转化法进一步证实。这些结果表明miR390调控StCDPK1的表达在马铃薯各组织中普遍表达且存在组织特异性,揭示了该激酶在马铃薯发育过程中的作用。Zhao等[55]发现油菜花中miR390参与了早期胚胎的发育过程。miR390-TAS3-ARF调控途径在植物叶等器官的极性发育及生长时期的转变上也发挥着调控作用[56-57],特别是对植物横向器官发育的调控尤为明显。如在侧根发育过程中,miR390-TAS3-ARF2-ARF3-ARF4可以定量控制侧根的生长[58]。番茄miR390的功能研究多围绕在果实的生长发育与成熟过程进行,通过高通量测序分析得到了番茄叶片和果实中miR390的表达情况发现,其在番茄幼小果实中的表达量远高于叶片、未开放的花以及成熟果实中,有研究对番茄果实发育的4个阶段miRNAs和降解组进行分析发现,miR390可以靶向降解TAS3a、TAS3b和肌动蛋白相关复合物亚基[59],说明miR390在番茄的果实发育过程中起到调控作用。以上研究表明,miR390能通过靶向作用多种调节因子广泛参与植物的生长发育过程,并在其中发挥作用。
3 miR390响应植物的非生物胁迫非生物胁迫是指由过度或不足的物理或化学条件引发的,对植物的生长、发育或繁殖都能产生不利影响的胁迫因子。miR390不仅可以通过调控靶标基因的表达,参与植物生长发育,反馈调节自身的代谢合成。还能通过参与调控相关抗性基因的表达,对重金属胁迫、盐胁迫、干旱胁迫和低温等胁迫信号做出不同的响应[4, 60]。
3.1 重金属胁迫常见的金属胁迫包括植物生理生化所必需的元素如铜(Cu)、铁(Fe)、锌(Zn)和非必需的元素如隔(Cd)、钴(Co)、汞(Hg)及铝(Al)等。在重金属胁迫下植物体内的活性氧大量积累,抗氧化酶系统与活性氧系统之间的平衡被打破,从而导致质膜、蛋白质和DNA的损伤,对植物体造成严重的伤害[61]。2000年来,miRNA与靶基因的互作关系是重金属胁迫应答的研究热点。镉是一种生物毒性很强且分布很广的重金属元素,水稻对镉的吸收性较强,镉进入水稻植株体内可向其籽粒等器官中转移并积累[62]。利用植物miRNA靶基因预测软件miRU和5iRU的方法证实水稻miR390的靶基因为1个富亮氨酸受体激酶LRR-RLK(Receptor-like protein kinase),然后通过RT-PCR发现,在镉处理下水稻植株根中RLK的表达显著上调[63],并且不同时间点miR390和RLK mRNA表现出此消彼长的一一对应关系,说明miR390调控RLK参与了水稻镉胁迫的应答,并且两者之可能存在负调控[64]。丁艳菲等[65]利用水稻miRNA芯片技术,在水稻幼苗根中筛选获得19种与镉胁迫应答相关的miRNA,其中,miR390的表达受到了显著的抑制,进一步研究把水稻miR390转化到水稻的愈伤组织中获得T2代种子,播种在含镉的培养液中发现,过表达miR390的水稻植株对镉敏感[66]。Ding等[67]研究发现镉胁迫下miR390过表达转基因株系OsHMA2和OsNramp5的表达增强,促进了水稻地上部分镉积累量的增加。目前镉胁迫下miR390与其靶基因的研究多集中在互作关系上,而具体的作用机制有待深入研究。
植株根系是直接与土壤接触的部分,土壤中的重金属物质通过根系进入到植株的各个组织器官中,研究miR390在重金属胁迫下的表达情况发现,其在根系中的变化较明显。Cd、Hg和Al能抑制植物根中miR390的表达,从而使得完整的TAS3转录本和tasiARFs的积累减少,最终抑制侧根的生长[68]。Cd胁迫下马蔺根系中miR390表达量下调[69]。在高浓度Cu处理的拟南芥植株中,miR390通过反式作用siRNAs的生物发生从而发挥它的作用,转而调控生长素响应因子,表现为主根的生长减少而短侧根的密度增加,并且主根和次生根根尖中有丝分裂活性、生长素和细胞因子在积累方面也发生了改变,推测miR390能响应Cu毒性有关[70-71]转而调控生长素响应因子。将亚麻抗性品种和敏感性品种的幼苗暴露于AlCl3溶液中4 h和24 h,结果显示miR390的表达发生变化且在敏感性和抗性品种中的改变是不同的,亚麻中miR390-TAS3能编码转录物,推测miR390能通过调控亚麻植物的生长过程在Al胁迫中起作用[72]。miR390在响应其他植物重金属胁迫时也有明显的改变,蒺藜苜蓿在铝胁迫下[73],水稻在砷胁迫下[74-75],甘蓝型油菜在镉胁迫下[76]miR390的表达量均有不同程度的下降。以上结果说明植株在受到重金属胁迫时,植株的表型及miR390的表达量发生变化,且在根系中表现明显。
3.2 干旱胁迫干旱胁迫下植株体内某些miRNAs会过量或低量表达,因此研究干旱胁迫下miRNAs做出的响应可以为农作物的逆境响应机制研究奠定基础。直接旱害(DNH)被认为是一种具有“应激效应”的干旱处理方式,驯化后旱害(DH)是一种经过干旱诱导后表达的处理方式,被认为是一种“驯化效应”[33],在研究木薯抗旱相关miRNA中发现,DNH和DH处理下miR390b在SC124(抗旱能力较高的木薯品种)和KU50(抗旱能力较低的木薯品种)的功能叶和根中都有表达,但整体上来看DH中miR390的表达量高于DNH[77]。对烟草进行干旱胁迫(DL)和重新浇水(WL)处理并以未处理(CL)做对照,结果显示miR390在不同干旱条件下表现出差异调节[78]。以上结果说明miR390响应干旱胁迫是一个复杂的过程,不同干旱条件下miR390的表达有一定差异。RNA依赖的RNA聚合酶(RNA-dependent-RNA polymerase,RdRp)是一类可被多种生物胁迫和非生物胁迫诱导的基因,目前为止,已在拟南芥、烟草、棉花、番茄、黄瓜和水稻等多种植物中被分离获得。研究发现,RDR6可通过反式作用miR390的靶基因TAS3促进ta-siRNA的生物合成,从而促进植物侧根的伸长[79]。当科丰一号大豆品种处于2叶期时,对其进行干旱处理(2% PEG 8000)6 h后,该基因在茎和叶中的表达量出现了短暂的降低,处理24 h后该基因在根中的表达量突然上升,而茎和叶中的表达量没有明显的变化[80]。根据这一结果预测miR390能响应植物的干旱胁迫且存在组织特异性。Sunkar等[81]发现miR390在拟南芥中也能响应干旱胁迫。以上研究可以推测miR390在干旱胁迫中发挥作用,且在植物不同组织和不同干旱条件下的表达量存在差异。
3.3 盐胁迫土壤盐渍化是一个全球性问题,严重制约了农业的发展。在盐胁迫下miRNAs的表达量会发生变化,Sunkar等[81]发现miR390在拟南芥遭受盐碱胁迫时均有表达。以草莓栽培品种全明星(Allstar)为实验材料,分别进行不同浓度的NaCl处理,发现不同盐胁迫处理下,miR390的表达量都受到了明显的抑制[82]。miR390能靶向作用相关基因,调控其表达,参与植物的盐胁迫过程。在玉米冠根组织的降解组文库中测序检测到miR390可靶向作用无机焦磷酸酶[83],而对高粱根和叶鞘的研究发现,盐胁迫初期无机焦磷酸酶水解活性明显增加,根和叶鞘的生长没有受到抑制;盐胁迫后期Na+开始向地上部分运输并在叶片中积累,此时,叶片液泡膜质子泵和Na+/H+逆向转运活性开始增加,根和叶鞘的Na/K比增加,无机焦磷酸酶水解活性下降。相应地,根和叶鞘的生长也下降[84]。同样,在烟草[85]、拟南芥[86]和大麦[87]等植物中都发现无机焦磷酸酶在盐胁迫下表达量发生改变。说明miR390靶向作用与无机焦磷酸酶,调控其表达,参与植物的盐胁迫,且随着胁迫时间的变化其表达量也会发生改变,最后导致植物生长上的变化。采用同源克隆的方法从大豆品种中分离出GmRDR6a和GmRDR6b,盐处理48 h之内GmRDR6a和GmRDR6b在根、茎、叶中的表达量均明显高于对照,且GmRDR6a在根、茎、叶中的表达量分别于盐处理12、24和48 h时达到最高;GmRDR6b在根、茎、叶中的表达量分别于盐处理6、12和24 h时达到最高[80]。这一结果说明miR390可靶向作用于RDR6,调控其表达,参与植物的盐胁迫,且在植物的多个组织中均有表达,但miR390表达量达到峰值所需要的时长存在差异。
3.4 低温胁迫低温是限制植物生长和分布的一种重要非生物胁迫,也是危害农作物生长的常见自然灾害之一。植物的低温胁迫包括冷害和冻害,都能对植物造成严重的伤害。因此,研究植物的抗寒性就显得尤为重要,研究发现miR390在响应低温胁迫中发挥作用。运用RT-PCR分析23个抗寒候选miRNA在木薯品种C4和KU50中的表达差异,结果显示,低温胁迫下miR390的表达量下降[88]。选用具有强耐冷和强抗冻能力的高山冰缘植物高山离子芥作为材料,通过高通量测序预测其低温胁迫下受调控的miRNAs,基因本体论(GO)比较他们靶基因的特征和表达模式,然后通过RT-PCR验证,结果发现miR390通过调控靶基因的表达对低温做出响应[89]。李贺等[90]研究发现miR390在低温胁迫处理的甜杨幼苗叶片中得到表达,并且miR390a在甜杨幼苗中的表达情况与其在毛果杨受4℃低温处理后经微阵列分析获得的表达谱结果极为相似,推测miR390与其靶基因的作用方式在两者中存在相似性。我们课题组对马铃薯材料进行了冷驯化及寒冻处理,通过高通量测序预测、RLM-RACE分析证实低温响应miR390的靶基因有2个富含亮氨酸重复序列类型受体蛋白激酶、1个内切1,3;1,4-β-D-葡聚糖酶、3个类受体丝氨酸/苏氨酸蛋白激酶和1个PGR5-类蛋白1B。实时定量PCR验证显示,冷驯化末期,miR390调控的关键靶向基因是富含亮氨酸重复序列型类受体蛋白激酶和内切1,3;1,4-β-D-葡聚糖酶,寒冻初期miR390调控的关键靶向基因是富含亮氨酸重复序列型类受体蛋白激酶和受体丝氨酸/苏氨酸蛋白激酶,寒冻后期miR390调控的关键靶向基因是受体丝氨酸/苏氨酸蛋白激酶。同时发现miR390在马铃薯的不同组织中表达量不同,存在组织特异性。
4 其他生物胁迫miR390在激素等其他生物胁迫中也发挥着重要作用。研究发现生物胁迫下拟南芥中miR390的表达下调[91],缺氧胁迫下表达上调[92]。miR390基因家族是一类与叶片衰老相关的暗诱导miRNAs,通过微阵列平台分析全暗处理的植株(DPs)和单独暗叶处理的植株(IDLs)发现miR390a的表达水平在全暗处理下相对较高(P < 0.01,信号强度 > 500)[93]。还有研究发现光敏感型miR390异构体能靶向调控四吡咯类物质合成的CPO I氧化酶和CHLP还原酶,而这两种酶在植物光合作用中起到重要的作用[94]。通过这两个研究可以推测miR390能靶向光合作用相关的酶,影响植物的光合作用,表现为叶片衰老。激素胁迫同样影响着miR390的表达,BA能显著促进“全明星”草莓试管苗中miR390的表达,表达量相差12.41倍;IAA也能显著提高其中miR390的表达,表达量相差62.71倍[95]。Yoon等[96]研究指出,在高IAA浓度下(10 μmol·L-1或50 μmol·L-1)处理12-24 h,拟南芥miR390的表达水平明显增加,而在低浓度IAA(10 nmol·L-1)下处理相同时间,表达量没有显著差异。推测miR390可响应不同浓度的激素胁迫,且在不同浓度下表达量存在差异。
5 展望miRNA通过调控靶基因的表达,广泛参与植物的胁迫应答,是当今植物逆境胁迫研究的热点。目前对miR390的研究仍然偏重于拟南芥和水稻等模式植物,小麦和马铃薯等其他主要农作物及园艺作物上的研究进展缓慢。虽然通过高通量测序技术结合生物信息学预测获得大量miRNA及其靶基因的组学信息,但目前仅能比较快速的应用实时定量PCR简单的探究miRNA及其靶基因的作用关系,而进一步研究常用的转植物基因技术步骤复杂,试验周期长,技术要求高,成为限制深入探究miRNA及其靶基因互作直接证据的主要瓶颈。基于相同的原因,目前对miR390的研究大多只停留在用实时定量PCR的方法间接分析层面,高效率、短时间且能直接证明植物miRNA作用其靶基因的离体或原位瞬时表达技术体系的开发,将为深入探讨miR390在响应植物非生物逆境胁迫的机理研究提供有力的手段。
| [1] |
魏强, 梁永宏, 李广林. 植物miRNA的进化[J]. 遗传, 2013, 35(3): 315-323. |
| [2] |
Sunkar R, Li YF, Jagadeeswaran G. Functions of microRNAs in plant stress responses[J]. Trends in Plant Science, 2012, 17(4): 196-203. DOI:10.1016/j.tplants.2012.01.010 |
| [3] |
Khraiwesh B, Zhu JK, Zhu J. Role of miRNAs and siRNAs in biotic and abiotic stress responses of plants[J]. Biochimica Et Biophysica Acta, 2012, 1819(2): 137-148. DOI:10.1016/j.bbagrm.2011.05.001 |
| [4] |
Sunkar R. MicroRNAs with macro-effects on plant stress responses[J]. Seminars in Cell & Developmental Biology, 2010, 21(8): 805-811. |
| [5] |
Xia R, Xu J, Meyers BC. The emergence, evolution, and diversifica-tion of the miR390-TAS3-ARF pathway in land plants[J]. Plant Cell, 2017, 29(6): 1232-1247. |
| [6] |
Wu G. Plant MicroRNAs and development[J]. 遗传学报, 2013, 40(5): 217-230. |
| [7] |
Karlsson P, Christie MD, Seymour DK, et al. KH domain protein RCF3 is a tissue-biased regulator of the plant miRNA biogenesis cofactor HYL1[J]. Proc Natl Acad Sci USA, 2015, 112(45): 14096-14101. DOI:10.1073/pnas.1512865112 |
| [8] |
马智明, 曹家树. 动植物microRNA的起源、合成及作用模式[J]. 中国细胞生物学学报, 2016(7): 857-863. |
| [9] |
Allen E, Xie Z, Gustafson AM, et al. microRNA-directed phasing during trans-acting siRNA biogenesis in plants[J]. Cell, 2005, 121(2): 207-221. DOI:10.1016/j.cell.2005.04.004 |
| [10] |
Leor W, Carles CC, Osmont KS, et al. A database analysis method identifies an endogenous trans-acting short-interfering RNA that targets the arabidopsis[J]. Proc Natl Acad Sci USA, 2005, 102(27): 9703-9708. DOI:10.1073/pnas.0504029102 |
| [11] |
Gustafson AM, Allen E, Givan S, et al. ASRP: the Arabidopsis small RNA project database[J]. Nucleic Acids Res, 2005, 33(Database issue): D637-640. |
| [12] |
Baev V, Milev I, Naydenov M, et al. Implementation of a De novo genome-wide computational approach for updating Brachypodium miRNAs[J]. Genomics, 2011, 97(5): 282-293. DOI:10.1016/j.ygeno.2011.02.008 |
| [13] |
Clepet C. RNA captor: A tool for RNA characterization[J]. PLoS One, 2011, 6(4): e18445. DOI:10.1371/journal.pone.0018445 |
| [14] |
Garciamas J, Benjak A, Sanseverino W, et al. The genome of melon(Cucumis melo L.)[J]. Proc Natl Acad Sci USA, 2012, 109(29): 11872-11877. DOI:10.1073/pnas.1205415109 |
| [15] |
Sunkar R, Girke T, Jain PK, et al. Cloning and characterization of microRNAs from rice[J]. Plant Cell, 2005, 17(5): 1397-1411. DOI:10.1105/tpc.105.031682 |
| [16] |
Tuskan GA, Difazio S, Jansson S, et al. The genome of black cottonwood, Populus trichocarpa(Torr. & Gray)[J]. Science, 2006, 313(5793): 1596-1604. DOI:10.1126/science.1128691 |
| [17] |
Arazi T, Talmorneiman M, Stav R, et al. Cloning and characterization of micro-RNAs from moss[J]. Plant Journal for Cell & Molecular Biology, 2005, 43(6): 837-848. |
| [18] |
Talmor-Neiman M, Stav R, Frank W, et al. Novel micro-RNAs and intermediates of micro-RNA biogenesis from moss[J]. Plant Journal for Cell & Molecular Biology, 2006, 47(1): 25-37. |
| [19] |
Jagadeeswaran G, Zheng Y, Li YF, et al. Cloning and characterization of small RNAs from Medicago truncatula reveals four novel legume-specific microRNA families[J]. New Phytologist, 2009, 184(1): 85-98. DOI:10.1111/nph.2009.184.issue-1 |
| [20] |
Khan Barozai MY, Irfan M, Yousaf R, et al. Identification of micro-RNAs in cotton[J]. Plant Physiology & Biochemistry Ppb, 2008, 46(8-9): 739-751. |
| [21] |
Lu S, Sun Y, Amerson HV. MicroRNAs in loblolly pine(Pinus taeda L.)and their association with fusiform rust gall development[J]. Plant Journal for Cell & Molecular Biology, 2007, 51(6): 1077-1098. |
| [22] |
Wang L, Wang MB, Tu JX, et al. Cloning and characterization of microRNAs from Brassica napus[J]. Febs Letters, 2007, 581(20): 3848-3856. DOI:10.1016/j.febslet.2007.07.010 |
| [23] |
Jaillon O, Aury JM, Noel B, et al. The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla[J]. Nature, 2007, 449(7161): 463-467. DOI:10.1038/nature06148 |
| [24] |
Subramanian S, Fu Y, Sunkar R, et al. Novel and nodulation-regulated microRNAs in soybean roots[J]. BMC Genomics, 2008, 9(1): 160-173. DOI:10.1186/1471-2164-9-160 |
| [25] |
Wong CE, Zhao YT, Wang XJ, et al. MicroRNAs in the shoot apical meristem of soybean[J]. J Exp Bot, 2011, 62(8): 2495-2506. DOI:10.1093/jxb/erq437 |
| [26] |
Turner M, Yu O, Subramanian S. Genome organization and characteristics of soybean microRNAs[J]. BMC Genomics, 2012, 13(1): 169-184. DOI:10.1186/1471-2164-13-169 |
| [27] |
Kravchik M, Sunkar R, Damodharan S, et al. Global and local perturbation of the tomato microRNA pathway by a trans-activated DICER-LIKE 1 mutant[J]. J Exp Bot, 2014, 65(2): 725-739. DOI:10.1093/jxb/ert428 |
| [28] |
Jiang J, Lv M, Liang Y, et al. Identification of novel and conserved miRNAs involved in pollen development in Brassica campestris ssp. chinensis by high-throughput sequencing and degradome analysis[J]. BMC Genomics, 2014, 15(1): 146-158. DOI:10.1186/1471-2164-15-146 |
| [29] |
Jia J, Zhao S, Kong X, et al. Aegilops tauschii draft genome sequence reveals a gene repertoire for wheat adaptation[J]. Nature, 2013, 496(7443): 91-95. DOI:10.1038/nature12028 |
| [30] |
Project AG. The Amborella genome and the evolution of flowering plants[J]. Science, 2013, 342(6165): 1241089. DOI:10.1126/science.1241089 |
| [31] |
Zhang LF, Chia JM, Kumari S, et al. A genome-wide characterization of microRNA genes in maize[J]. PLoS Genetics, 2009, 5(11): e1000716. DOI:10.1371/journal.pgen.1000716 |
| [32] |
Song CN, Fang JG, Li XY, et al. Identification and characterization of 27 conserved microRNAs in citrus[J]. Planta, 2009, 230(4): 671-685. DOI:10.1007/s00425-009-0971-x |
| [33] |
Zeng C, Wang W, Zheng Y, et al. Conservation and divergence of microRNAs and their functions in Euphorbiaceous plants[J]. Nucleic Acids Res, 2010, 38(3): 981-995. DOI:10.1093/nar/gkp1035 |
| [34] |
Fahlgren N, Jogdeo S, Kasschau KD, et al. MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana[J]. Plant Cell, 2010, 22(4): 1074-1089. DOI:10.1105/tpc.110.073999 |
| [35] |
Argout X, Salse J, Aury JM, et al. The genome of Theobroma cacao[J]. Nature Genetics, 2011, 43(2): 101-108. |
| [36] |
De Paola D, Cattonaro F, Pignone D, et al. The miRNAome of globe artichoke: conserved and novel micro RNAs and target analysis[J]. BMC Genomics, 2012, 13: 41. DOI:10.1186/1471-2164-13-41 |
| [37] |
Barvkar VT, Pardeshi VC, Kale SM, et al. Genome-wide identification and characterization of microRNA genes and their targets in flax(Linum usitatissimum): Characterization of flax miRNA genes[J]. Planta, 2013, 237(4): 1149-1161. DOI:10.1007/s00425-012-1833-5 |
| [38] |
She T, Yu W, Li Z, et al. Identification of wounding and topping responsive small RNAs in tobacco(Nicotiana tabacum)[J]. BMC Plant Biol, 2012, 12(1): 28-45. DOI:10.1186/1471-2229-12-28 |
| [39] |
Wan LC, Zhang H, Lu S, et al. Transcriptome-wide identification and characterization of miRNAs from Pinus densata[J]. BMC Genomics, 2012, 13(1): 132-142. DOI:10.1186/1471-2164-13-132 |
| [40] |
Barozai MY, Baloch IA, Din M. Identification of MicroRNAs and their targets in Helianthus[J]. Molecular Biology Reports, 2012, 39(3): 2523-2532. DOI:10.1007/s11033-011-1004-y |
| [41] |
Xia R, Zhu H, An Y, et al. Apple miRNAs and tasiRNAs with novel regulatory networks[J]. Genome Biology, 2012, 13(6): R47. DOI:10.1186/gb-2012-13-6-r47 |
| [42] |
Patanun O, Lertpanyasampatha M, Sojikul P, et al. Computational identification of microRNAs and their targets in cassava(Manihot esculenta Crantz.)[J]. Molecular Biotechnology, 2013, 53(3): 257-269. DOI:10.1007/s12033-012-9521-z |
| [43] |
De LA, Markmann K, Cognat V, et al. Two microRNAs linked to nodule infection and nitrogen-fixing ability in the legume Lotus japonicus[J]. Plant Physiology, 2012, 160(4): 2137-2154. DOI:10.1104/pp.112.204883 |
| [44] |
Zhang R, Marshall D, Bryan GJ, et al. Identification and characterization of miRNA transcriptome in potato by high-throughput sequencing[J]. PLoS One, 2013, 8(2): e57233. DOI:10.1371/journal.pone.0057233 |
| [45] |
Zhu H, Xia R, Zhao B, et al. Unique expression, processing regulation, and regulatory network of peach(Prunus persica)miRNAs[J]. BMC Plant Biol, 2012, 12(1): 149-166. DOI:10.1186/1471-2229-12-149 |
| [46] |
Rishi A, Yang X, Yu Q, et al. Asymmetric purine-pyrimidine distribution in cellular small RNA population of papaya[J]. BMC Genomics, 2012, 13(1): 682-695. DOI:10.1186/1471-2164-13-682 |
| [47] |
Garcia D, Collier S, Byrne MR. Specification of leaf polarity in Arabidopsis via the trans-acting siRNA pathway[J]. Current Biology, 2006, 16(9): 933-938. DOI:10.1016/j.cub.2006.03.064 |
| [48] |
Sunkar R, Girke T, Jian PK. Cloning and characterization of micro-RNAs from rice[J]. Plant Cell, 2005, 17(5): 1397-1411. DOI:10.1105/tpc.105.031682 |
| [49] |
Wang J, Gao X, Li L, et al. Overexpression of Osta-siR2141 caused abnormal polarity establishment and retarded growth in rice[J]. J Exp Bot, 2010, 61(6): 1885-1895. DOI:10.1093/jxb/erp378 |
| [50] |
梅梅, 王晓禹, 张晓林, 等. 植物生长素响应因子ARF研究进展[J]. 种子, 2017, 36(1): 47-54. |
| [51] |
Montgomery TA, Howell MD, Cuperus JT, et al. Specificity of ARGONAUTE7-miR390 interaction and dual functionality in TAS3 trans-acting siRNA formation[J]. Cell, 2008, 133(1): 128-141. DOI:10.1016/j.cell.2008.02.033 |
| [52] |
Garcia D. A miRacle in plant development: role of microRNAs in cell differentiation and patterning[J]. Seminars in Cell Developmental Biology, 2008, 19(6): 586-595. DOI:10.1016/j.semcdb.2008.07.013 |
| [53] |
Fahlgren N, Montgomery TA, Howell MD, et al. Regulation of AUXIN RESPONSE FACTOR3 by TAS3 ta-siRNA affects developmental timing and patterning in Arabidopsis[J]. Current Biology Cb, 2006, 16(9): 939-944. DOI:10.1016/j.cub.2006.03.065 |
| [54] |
Santin F, Bhogale S, Fantino E, et al. Solanum tuberosum StCDPK1 is regulated by miR390 at the posttranscriptional level and phosphorylates the auxin efflux carrier StPIN4 in vitro, a potential downstream target in potato development[J]. Physiologia Plantarum, 2016, 159(2): 244-261. |
| [55] |
Zhao YT, Wang M, Fu SX, et al. Small RNA profiling in two Brassica napus cultivars identifies microRNAs with oil production-and development-correlated expression and new small RNA classes[J]. Plant Physiology, 2012, 158(2): 813-823. DOI:10.1104/pp.111.187666 |
| [56] |
Cho SH, Coruh C, Axtell MJ. miR156 and miR390 regulate tasiRNA accumulation and developmental timing in Physcomitrella patens[J]. Plant Cell, 2012, 24(12): 4837-4849. DOI:10.1105/tpc.112.103176 |
| [57] |
Rhoades MW, Reinhart BJ, Lim LP, et al. Prediction of plant microRNA targets[J]. Cell, 2002, 110(4): 513-520. DOI:10.1016/S0092-8674(02)00863-2 |
| [58] |
Marin E, Jouannet V, Herz A, et al. miR390, Arabidopsis TAS3 tasiRNAs, and their AUXIN RESPONSE FACTOR targets define an autoregulatory network quantitatively regulating lateral root growth[J]. Plant Cell, 2010, 22(4): 1104-1117. DOI:10.1105/tpc.109.072553 |
| [59] |
于丽丽, 刘伟伟, 方媛, 等. 番茄LemiR390及其预测靶基因LeTAS3的鉴定与表达分析[J]. 园艺学报, 2015, 42(2): 271-279. |
| [60] |
Khraiwesh B, Zhu JK, Zhu J. Role of miRNAs and siRNAs in biotic and abiotic stress responses of plants[J]. Biochimica Et Biophysica Acta, 2012, 1819(2): 137-148. DOI:10.1016/j.bbagrm.2011.05.001 |
| [61] |
Romero-Puertas MC, Corpas FJ, Rodríguez-Serrano M, et al. Differential expression and regulation of antioxidative enzymes by cadmium in pea plants[J]. Journal of Plant Physiology, 2007, 164(10): 1346-1357. DOI:10.1016/j.jplph.2006.06.018 |
| [62] |
张金彪, 黄维南. 镉对植物的生理生态效应的研究进展[J]. 生态学报, 2000, 20(3): 514-523. |
| [63] |
Ding Y, Zhen C, Cheng Z. Microarray-based analysis of cadmium-responsive microRNAs in rice(Oryza sativa)[J]. J Exp Bot, 2011, 62(10): 3563-3573. DOI:10.1093/jxb/err046 |
| [64] |
曹玉婷, 丁艳菲, 朱诚. 类受体蛋白激酶与植物非生物胁迫应答[J]. 中国生物化学与分子生物学报, 2014, 30(3): 241-247. |
| [65] |
丁艳菲. 水稻镉胁迫应答相关microRNA的分离与功能研究[D]. 杭州: 浙江大学, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10335-1012338312.htm
|
| [66] |
丁艳菲, 刘海丽, 朱诚, 等. 水稻miR390在增加植物镉胁迫敏感性中的应用: 中国, 102250902 A[P]. 2011.
|
| [67] |
Ding Y, Ye Y, Jiang Z, et al. MicroRNA390 Is Involved in cadmium tolerance and accumulation in rice[J]. Front Plant Sci, 2016, 7(127): 235-244. |
| [68] |
Zhou ZS, Zeng HQ, Liu ZP, et al. Genome-wide identification of Medicago truncatula microRNAs and their targets reveals their differential regulation by heavy metal[J]. Plant Cell & Environment, 2012, 35(1): 86-99. |
| [69] |
刘凉琴, 宋爱萍, 张永侠, 等. 马蔺根系响应Cd胁迫的miRNA高通量测序分析[J]. 植物资源与环境学报, 2016, 25(3): 1-11. |
| [70] |
Lequeux H, Hermans C, Lutts S, et al. Response to copper excess in Arabidopsis thaliana: Impact on the root system architecture, hormone distribution, lignin accumulation and mineral profile[J]. Plant Physiol Biochem, 2010, 48(8): 673-682. DOI:10.1016/j.plaphy.2010.05.005 |
| [71] |
肖莉, 刘春, 向世鹏, 等. MicroRNAs在植物响应金属毒性中的作用[J]. 衡阳师范学院学报, 2013, 34(3): 113-117. |
| [72] |
Dmitriev AA, Kudryavtseva AV, Bolsheva NL, et al. miR319, miR390, and miR393 are involved in aluminum response in flax(Linum usitatissimum L.)[J]. Biomed Research International, 2017, 4975146. |
| [73] |
Chen X. Small RNAs in development-insights from plants[J]. Curr Opin Genet Dev, 2012, 22(4): 361-367. DOI:10.1016/j.gde.2012.04.004 |
| [74] |
Liu QP, Zhang HM. Molecular identification and analysis ofarsenite stress-responsive miRNAs in rice[J]. J Agric Food Chem, 2012, 60(26): 6524-6536. DOI:10.1021/jf300724t |
| [75] |
Zhang J, Zhang S, Han S, et al. Genome-wide identification ofmicroRNAs in larch and stage-specific modulation of 11 conserved microRNAs and their targets during somatic embryogenesis[J]. Planta, 2012, 236(2): 647-657. DOI:10.1007/s00425-012-1643-9 |
| [76] |
Zhou ZS, Song JB, Yang ZM, et al. Genome-wideidentification of Brassica napus microRNAs and their targets in response to cadmium[J]. J Exp Bot, 2012, 63(12): 4597-4613. DOI:10.1093/jxb/ers136 |
| [77] |
宋顺. 木薯抗旱相关microRNA差异表达分析[D]. 海南: 海南大学, 2010. http://cdmd.cnki.com.cn/Article/CDMD-10589-2010183787.htm
|
| [78] |
Chen Q, Li M, Zhang Z, et al. Integrated mRNA and microRNA analysis identifies genes and small miRNA molecules associated with transcriptional and post-transcriptional-level responses to both drought stress and re-watering treatment in tobacco[J]. BMC Genomics, 2017, 18(1): 62-77. DOI:10.1186/s12864-016-3372-0 |
| [79] |
Marin E, Jouannet V, Herz A, et al. miR390, Arabidopsis TAS3 tasiRNAs, and their AUXIN RESPONSE FACTOR targets define an autoregulatory network quantitatively regulating lateral root growth[J]. Plant Cell, 2010, 22(4): 1104-1117. DOI:10.1105/tpc.109.072553 |
| [80] |
吴冰月, 宋普文, 陈华涛, 等. 2个大豆RNA依赖的RNA聚合酶基因和的克隆与分析[J]. 南京农业大学学报, 2014, 37(3): 27-34. DOI:10.7685/j.issn.1000-2030.2014.03.004 |
| [81] |
Sunkar R, Jagadeeswaran G. In silico identification of conserved microRNAs in large number of diverse plant species[J]. BMC Plant Biol, 2008, 8(1): 37-49. DOI:10.1186/1471-2229-8-37 |
| [82] |
李贺, 毛健鑫, 戚华彩, 等. 草莓miR390基因及其启动子的鉴定与表达分析[J]. 果树学报, 2014, 31(3): 362-369. |
| [83] |
翟立红. 玉米冠根和雄穗中microRNA及Argonaute基因表达的研究[D]. 湖北: 华中农业大学, 2013. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2394844
|
| [84] |
王宝山, 邹琦. NaCl胁迫对高粱根、叶鞘和叶片液泡膜ATP酶和焦磷酸酶活性的影响[J]. 植物生理学报, 2000, 26(3): 181-188. |
| [85] |
郭兆奎. 烟草吸钾相关基因克隆与表达调控研究[D]. 哈尔滨: 哈尔滨工业大学, 2008. http://cdmd.cnki.com.cn/Article/CDMD-10213-2009224139.htm
|
| [86] |
王波. 拟南芥和盐生植物灰绿藜液泡膜焦磷酸酶基因与TIR1基因表达相关性分析[D]. 乌鲁木齐: 新疆大学, 2007. http://cdmd.cnki.com.cn/Article/CDMD-10755-2007165655.htm
|
| [87] |
佐拉. 野生大麦与栽培大麦耐盐性的生理及遗传差异研究[D]. 杭州: 浙江大学, 2014. http://cdmd.cnki.com.cn/article/cdmd-10335-1014377917.htm
|
| [88] |
薄维平. 木薯耐寒相关microRNA的差异表达分析[D]. 海南: 海南大学, 2010. http://kns.cnki.net/KCMS/detail/detail.aspx?filename=rdzx201008007&dbname=CJFD&dbcode=CJFQ
|
| [89] |
党春艳. 高山离子芥低温胁迫调控的miRNAs及其靶基因的表达分析[D]. 兰州: 兰州大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10730-1016190525.htm
|
| [90] |
孙润泽, 侯琦, 章文乐, 等. 甜杨低温响应microRNAs的克隆与分析[J]. 基因组学与应用生物学, 2011, 30(2): 204-211. |
| [91] |
Zhang W, Ga oS, Zhou X, Chellappan P, et al. Bacteria -responsive microRNAs regulate plant innate immunity by modulating plant hormone networks[J]. Plant Molecular Biology, 2011, 75: 93-105. DOI:10.1007/s11103-010-9710-8 |
| [92] |
Moldovan D, Spriggs A, Yang J, et al. Hypoxia -responsive microRNAs and transacting small interfering RNAs in Arabidopsis[J]. J Exp Bot, 2010, 61: 165-177. DOI:10.1093/jxb/erp296 |
| [93] |
Huo X, Wang C, Teng Y, et al. Identification of miRNAs associated with dark-induced senescence in Arabidopsis[J]. BMC Plant Biol, 2015, 15(1): 266-277. DOI:10.1186/s12870-015-0656-5 |
| [94] |
乔岩. 马铃薯光诱导糖苷生物碱代谢相关miRNAs的鉴定与功能分析[D]. 兰州: 甘肃农业大学, 2017. http://cdmd.cnki.com.cn/Article/CDMD-10733-1017824774.htm
|
| [95] |
李贺, 毛健鑫, 戚华彩, 等. 草莓miR390基因及其启动子的鉴定与表达分析[J]. 果树学报, 2014, 31(3): 362-336. |
| [96] |
Yoon EK, Yang JH, Lim J, et al. Auxin regulation of the microRNA390-dependent transacting small interfering RNA pathway in Arabidopsis lateral root development[J]. Nucleic Acids Res, 2010, 38: 1382-1391. DOI:10.1093/nar/gkp1128 |