酿酒酵母(Saccharomyces cerevisiae)广泛用于乙醇发酵生产,也是异源蛋白表达的重要宿主之一。利用酿酒酵母表达的外源蛋白包括抗体等药物蛋白[1]和纤维素酶等多种工业酶等[2]。因此,提高酿酒酵母外源蛋白表达水平相关研究受到普遍关注。纤维素酶属于糖苷水解酶家族,通过催化糖苷键水解从而解聚纤维素并释放葡萄糖,进而可用于生物燃料和生物基化学品的发酵生产[3]。将纤维素酶在酿酒酵母中表达,特别是分泌表达,可以将纤维素组分的酶水解及葡萄糖乙醇发酵整合,开发联合生物加工(Consolidated bioprocessing,CBP)策略[3],是纤维素乙醇生产创新技术开发的前沿。纤维素酶为复合酶系,主要包括内切葡聚糖酶(Endoglucanase,EG)、外切纤维素酶(Cellobiohydrolase,CBH)和β-葡萄糖苷酶(β-glucosidase,BGL),通过协同作用降解纤维素[4]。目前,酿酒酵母异源表达纤维素酶水平低,导致纤维素组分酶水解速率慢,是制约整个利用CBP生产纤维素乙醇过程的突出问题。虽然基于酿酒酵母细胞表面展示技术,异源表达纤维素酶可以锚定到细胞表面形成局部高浓度,在一定程度上解决纤维素组分水解速率慢的问题。但纤维素组分是非可水溶性的,与酿酒酵母细胞接触的比表面积很低,酿酒酵母细胞表面展示纤维素酶后,纤维素组分水解速率仍然无法与乙醇发酵相匹配[5]。因此研究纤维素酶的分泌生产是提高CBP效率的重要内容。此外,促进酶的分泌生产也有利于生产多种药用蛋白,因此引起了广泛关注。
酿酒酵母表达异源蛋白的效率受到多因素影响,包括酶基因的选择[6]、启动子强弱[7]等。此外,在酿酒酵母中异源蛋白表达和分泌不可避免会对细胞产生多种胁迫,如营养匮乏、能量供给不足及氧化胁迫等[8, 9]。所以,过表达胁迫耐受性基因可能对酿酒酵母外源蛋白分泌表达具有综合的促进作用。研究表明超氧化物歧化酶基因SOD1过表达可提高重组酵母纤维素酶的生产[10],与细胞胁迫相关的热激响应(Heat shock response,HSR)的激活能够提高酿酒酵母异源α-淀粉酶的生产[11]。因此,对胁迫耐受性相关基因进行深入研究,有可能进一步促进重组酵母纤维素酶的生产。
本课题组近期研究表明,编码线粒体核糖体蛋白家族的基因MRP8在乙酸耐受性提高的酿酒酵母突变株中表达量明显高于对照菌株,进一步研究证明,过表达MRP8可提高模式酿酒酵母菌株S288c乙酸胁迫耐受性[12]。目前对MRP8的功能了解很少,由于其过表达对酿酒酵母环境胁迫耐受性的促进作用,推测该基因有可能在重组酵母表达外源蛋白过程中也起到和SOD1类似的促进作用,但目前对其在纤维素酶等外源蛋白生产中的作用还没有研究报道。本文以异源表达CBH1酿酒酵母菌株为模式体系,研究MRP8过表达对CBH1合成与分泌的影响,为进一步研究利用胁迫相关基因改造促进酿酒酵母高效分泌表达其他纤维素酶,进而为开发纤维素乙醇生产CBP创新技术奠定基础。
1 材料与方法 1.1 材料 1.1.1 菌株、质粒和引物本实验中使用的酿酒酵母菌株、质粒和引物见表 1和表 2。重组酿酒酵母出发菌株S. cerevisiae Y294-CBH1为过表达Talaromyces emersonii来源的CBH1[6]的S. cerevisiae Y294衍生菌株,由南非西开普敦大学Riaan den Haan博士提供。
培养大肠杆菌Escherichia coli DH5α使用LB液体培养基(g/L):胰蛋白胨10,酵母粉5,氯化钠10;LB固体培养基在此基础上添加20 g/L琼脂粉。活化酿酒酵母及进行酶活评价使用YPD培养基(g/L):葡萄糖20,蛋白胨20,酵母粉10;YPD固体培养基在此基础上添加20 g/L琼脂粉。以上培养基均在115℃高压蒸汽灭菌20 min。
1.2 方法 1.2.1 菌株构建与活化以模式酿酒酵母S288c基因组为模板,用引物Xma Ⅰ MRP8-F/Sal Ⅰ MRP8-R扩增出MRP8基因序列。采用USER酶连接方法[15]快速构建得到含PGK1p-MRP8-CYC1t片段的质粒。质粒经测序验证正确后作为模板,利用引物19-PGK1p-F和19-CYC1t-R扩增获得用于酿酒酵母同源重组的线性化片段,其中包含酿酒酵母S288c染色体XV上整合位点(YORWdelta22)的上游同源臂、PGK1启动子、MRP8基因、CYC1终止子、以及整合位点的下游同源臂。利用醋酸锂转化法[16]将Cas9表达载体Cas9-NAT转至分泌产酶的重组酿酒酵母Y294-CBH1菌株中,在含有100 mg/L诺尔丝菌素的YPD固体平板上筛选得到转化子。再将YORWdelta22位点的gRNA表达载体pRS42H-gRNA-19和线性化片段转化至含有Cas9表达载体的转化子中,以含有100 mg/L诺尔丝菌素和300 mg/L潮霉素B的YPD固体平板筛选转化子。提取基因组DNA,用引物Ppgk1-F和Sal Ⅰ MRP8-R,以及Xma Ⅰ MRP8-F和Tcyc1-R扩增转化元件,验证转化子。将转化子在不含抗生素的YPD液体培养基中继代,每24 h转接一次,连续转接5次,丢失Cas9-NAT和pRS42H-gRNA-19质粒,获得重组菌株用于产酶实验。所获得的重组酵母菌株均保存于-80℃冰箱。
重组酵母菌株活化:取-80℃保存的重组酿酒酵母菌株在YPD液体培养基中30℃、150 r/min过夜培养至对数期,经二次活化后,用于后续发酵实验。
1.2.2 重组酵母酶活评价经二次活化的重组酿酒酵母菌株接种于含100 mL YPD培养基的250 mL三角瓶中,30℃、150 r/min过夜培养后离心收集菌体,再以初始OD600为1的起始接种量,于100 mL YPD培养基中,在150 r/min、30℃条件下培养。每隔24 h定时取样测定。所取发酵液6 000 r/min离心OD6005 min,留上清测定酶活。
酶活测定方法:参考文献方法测定[17],以0.02 g/L pNP(p-nitrophenyl)适当稀释绘制标准曲线,换算后得到相应酶活。一个酶活力单位定义为每分钟生成1 μg pNP所需的酶量。发酵液上清的酶活力以每mg细胞干重计量。
酵母细胞干重测定:收集不同时期酵母细胞,ddH2O洗两次后,分别稀释成不同浓度(每个浓度准备两份),一份用来测定OD600,一份用来测定细胞干重,从而制得OD600-干重标准曲线。发酵过程取样后测OD600,并根据标准曲线计算干重。
1.2.3 SDS-PAGE分析取500 μL培养96 h的发酵液,离心收集上清,冷冻干燥后,加25 μL ddH2O溶解,添加糖苷内切酶-H(Endoglycosidase H,Endo-H,New England Biolabs)37℃反应1 h用于去糖基化,以不添加Endo-H作为对照,进行SDS-PAGE。电泳结束后用考马斯亮蓝R-250染色,经脱色液(10% V/V冰醋酸,5% V/V乙醇)脱色后观察蛋白条带。
1.2.4 重组酵母胞内活性氧和ATP含量检测分别取12、24、48、72和96 h的酵母细胞,3 000 r/min离心3 min后,收集酵母细胞,然后用1 mol/L的PBS(磷酸盐缓冲液)洗涤两次,使用活性氧检测试剂盒(碧云天公司,S0033)和ATP检测试剂盒(碧云天公司,S0026)检测胞内活性氧及ATP的含量。
1.2.5 关键基因表达水平分析取4 mL培养24 h和48 h的发酵液,10 000 r/min离心2 min后收集酵母细胞,ddH2O洗两次,液氮速冻后,冻存于-80℃,用于RNA提取。使用总RNA抽提纯化试剂盒(Sangon Biotech)提取总RNA,然后用反转录试剂盒(Prime Script RT reagent Kit,TaKaRa)反转成cDNA作为模板,使用iQ™ SYBR® Green试剂盒(Bio-Rad)进行实时定量PCR(RT-qPCR)分析,以2-ΔΔCt方法比较转录量[18],以ALG9为内参基因。本实验中使用的实时定量引物见表 3。
培养二次活化后的菌株至对数生长期,将菌液OD600调至1.0,按照一定梯度稀释后,取2 µL菌液在对照条件,以及分别添加刚果红(Congo red)、衣霉素(Tunicamycin,TM)、二硫苏糖醇(Dithiothreitol,DTT)的YPD平板上点板培养,记录菌株生长情况。刚果红浓度分别为5、10、25、50、100、200、500 μg/mL;衣霉素浓度分别为0.5和0.8 μg/mL;二硫苏糖醇浓度为5 mmol/L。
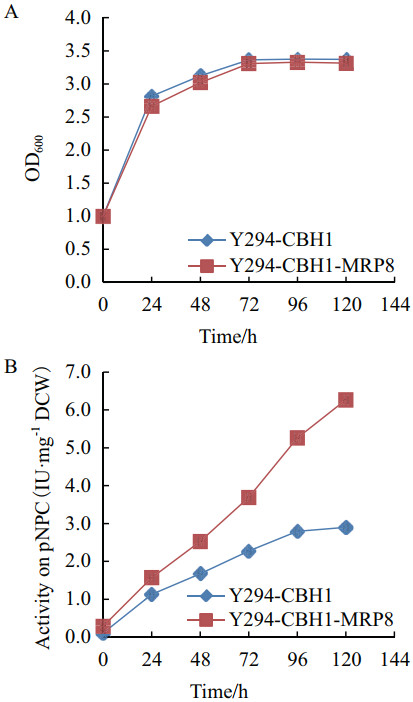
2 结果 2.1 菌株生长情况及酶活评价采用CRISPR-Cas9基因组编辑技术将MRP8定点整合到酿酒酵母XV染色体YORWdelta22位点,研究其过表达对纤维素酶生产的影响。如图 1-A所示,在30℃培养96 h时,Y294-CBH1-MRP8菌株的生物量OD600约为3.33,对照菌株约为3.37,表明生长无明显差异。酶活情况如图 1-B所示,在30℃培养96 h时,MRP8过表达菌株分泌产生的CBH1酶活达到了5.26 IU/mg DCW,与对照菌株相比提高了约80%,说明MRP8过表达显著提高了菌株CBH1生产。
|
| 图 1 MRP8过表达菌株和对照菌株的生长与CBH1酶活性比较 A和B分别为菌株Y294-CBH1和Y294-CBH1-MRP8在YPD培养基中的生长情况和CBH1酶活力 |
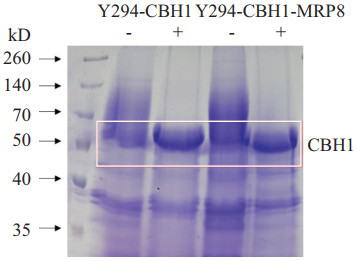
由于在酿酒酵母中生产异源蛋白,易产生糖基化现象,影响蛋白条带观察,故利用Endo-H进行去糖基化反应。SDS-PAGE分析结果如图 2所示,与对照菌株相比,重组菌株Y294-CBH1-MRP8菌株在CBH1预测分子量位置条带亮度更亮,推测CBH1蛋白浓度更高,与CBH1酶活高于对照菌株结果一致。
|
| 图 2 MRP8过表达菌株和对照菌株粗酶液SDS-PAGE蛋白电泳分析 菌株Y294-CBH1和Y294-CBH1-MRP8在YPD培养基中培养至96 h的蛋白电泳,其中+和-分别表示添加和不添加Endo-H的结果 |
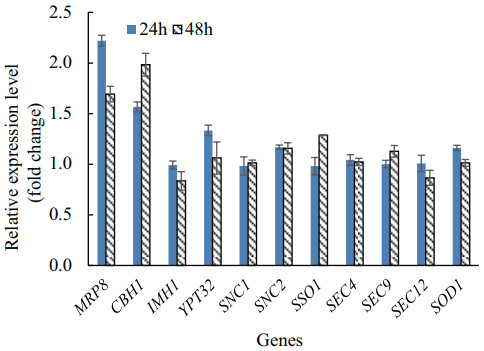
为了挖掘MRP8过表达提高酿酒酵母分泌表达CBH1酶活的内在机理,检测了重组酵母和对照菌株中与外源蛋白分泌相关基因的转录水平(图 3)。在发酵24 h时,基因几乎无显著变化,然而在发酵48 h时,CBH1表达上调2.0倍,但是与蛋白转运相关的基因IMH1和TYP32等表达都没有明显变化,提示MRP8过表达提高CBH1的生产是促进了CBH1酶基因转录,可能与外切纤维素酶在细胞内的转运和分泌无关,但还需要研究其他时间点和更多的基因,并结合后续的转录组数据进行深入分析。抗氧化酶SOD1基因转录也未发现明显变化。
|
| 图 3 MRP8过表达菌株和对照菌株相关基因转录水平比较 菌株Y294-CBH1和Y294-CBH1-MRP8在YPD培养基中培养24 h和48 h后提取RNA进行RT-qPCR分析的结果 |
对突变体及对照菌株胞内活性氧和ATP进行检测,结果如图 4所示。在发酵初期12 h时,与对照菌株相比,MRP8过表达菌株活性氧偏高,但ATP含量较低,这可能是由于异源蛋白的表达增强,使得细胞在发酵初期产生一定响应。从24 h开始,突变体的CBH1酶活明显高于对照菌株,但胞内活性氧和ATP无显著差异。
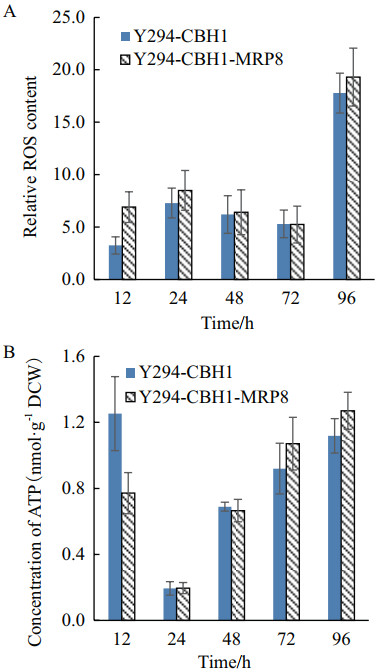
|
| 图 4 MRP8过表达菌株和对照菌株胞内活性氧和ATP含量比较 A和B分别为菌株Y294-CBH1和Y294-CBH1-MRP8在发酵不同时间点的胞内活性氧相对含量和胞内ATP的含量 |
评价突变体和对照菌株在添加刚果红、衣霉素或二硫苏糖醇条件下的生长情况,结果如图 5所示。刚果红敏感性与细胞壁完整性相关[19],在添加5-500 μg/mL刚果红条件下,MRP8过表达菌株的生长与对照菌株相比无明显差别,说明MRP8过表达提高外源蛋白分泌与细胞壁完整性无关。此外,额外添加衣霉素和二硫苏糖醇能够引起未折叠蛋白响应,从而造成内质网胁迫[20]。实验结果表明,MRP8过表达菌株和对照菌株在衣霉素平板中的生长基本一致,此外,突变体在DTT平板中生长略弱于对照,证实CBH1蛋白生产增强和未折叠蛋白响应无显著联系。
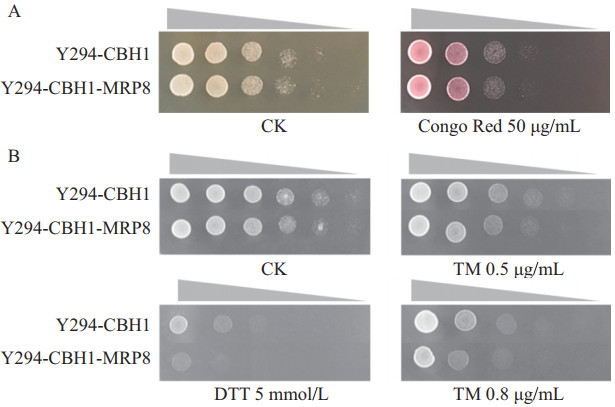
|
| 图 5 MRP8过表达菌株和对照菌株在胁迫条件下的生长情况比较 A和B分别为菌株Y294-CBH1和Y294-CBH1-MRP8在含刚果红(Congo red)、衣霉素(TM)或二硫苏糖醇(DTT)的YPD平板上30℃培养24 h的生长情况,CK表示不添加任何物质的对照条件 |
本研究证明,过表达MRP8促进外切纤维素酶CBH1的生产,且对菌株生长无影响。前期研究表明,蛋白质经过转录翻译后,在内质网上进行折叠加工,期间涉及的相关基因均会参与到此过程中[21]。此外,在蛋白质从内质网到高尔基体,从高尔基体到细胞膜的运输过程中同样涉及大量蛋白的参与[22-24]。在本研究中,蛋白分泌量增多,实时定量PCR分析MRP8过表达突变体CBH1转录量提高,但囊泡运输过程相关基因的转录水平无明显变化,说明MRP8过表达可能促进CBH1蛋白合成,而可能和增强蛋白分泌途径没有直接关系,但是相关研究还需要深入分析其他时间点和更多相关的关键蛋白基因后才能得到更准确的结论。未来将进行不同时间点重组酵母细胞全局转录组分析,得到更加全面的信息,进而深入揭示蛋白表达和分泌的分子机理。
MRP8在酿酒酵母细胞中的具体功能目前还不十分清楚。以往研究表明,MRP8转录受细胞壁损伤胁迫的诱导[25],其蛋白表达在厌氧条件下比好氧条件低[26]。此外,MRP8在过氧化氢和乙醇胁迫下受到泛素化修饰[27],说明该基因的表达与胁迫响应有关。蛋白的过量生产和分泌可能产生活性氧[9],但是检测发现MRP8过表达菌株ROS无明显变化,推测该基因过表达能降低活性氧的积累,从而导致ROS和对照水平相当。SOD1基因转录无明显差异,SOD酶活检测也未发现显著变化,推测活性氧的清除可能与其他抗氧化机制有关。蛋白合成过程中需要能量供应,检测胞内ATP含量发现,过表达菌株与对照水平基本一致,猜测是由于MRP8过表达促进能量代谢,使得CBH1蛋白合成提高所消耗的能量得到抵消。此外,MRP8过表达提高外源蛋白分泌与细胞壁完整性和内质网胁迫均无显著联系,其调控产酶的分子机理值得进一步深入挖掘。
本课题组前期对与生物燃料生产相关的环境胁迫耐受性基因机理进行了研究,发现了多个与酿酒酵母环境胁迫耐受性相关的基因,例如发现过表达组蛋白H4甲基化转移酶编码基因SET5可提高酿酒酵母对乙酸和氧化胁迫的耐受性[28]。本研究结果表明,其他与胁迫耐受性相关基因也可能用于提高纤维素酶的生产,未来可继续研究这些环境胁迫耐受性相关基因对重组蛋白生产的影响。后续也将着重研究MRP8过表达对其他酶生产的影响,从而促进重组酵母在多种药用蛋白和工业酶生产中的应用。
4 结论在重组酿酒酵母中过表达MRP8可提高外切纤维素酶的生产,与对照菌株相比,CBH1酶活提高了约80%。进一步对促进酶分泌的机理进行探讨,发现在MRP8过表达突变体中,CBH1转录水平高于对照菌株,但是与蛋白分泌相关的关键基因转录水平没有明显变化。过表达MRP8对细胞壁完整性和由未折叠蛋白响应引起的内质网胁迫无明显影响,其增强产酶机理与促进酶基因转录有关。
| [1] |
Ruijter JC, Koskela EV, Frey AD. Enhancing antibody folding and secretion by tailoring the Saccharomyces cerevisiae endoplasmic reticulum[J]. Microb Cell Fact, 2016, 15(1): 87. DOI:10.1186/s12934-016-0488-5 |
| [2] |
Davison SA, den Haan R, van Zyl WH. Heterologous expression of cellulase genes in natural Saccharomyces cerevisiae strains[J]. Appl Microb Biotech, 2016, 100(18): 8241-54. DOI:10.1007/s00253-016-7735-x |
| [3] |
Liu H, et al. Engineering microbes for direct fermentation of cellulose to bioethnol[J]. Crit Rev Biotech, 2018, 2018: 1-17. |
| [4] |
Zhao XQ, Xiong L, Zhang MM, et al. Towards efficient bioethanol production from agricultural and forestry residues:exploration of unique natural microorganisms in combination with advanced strain engineering[J]. Bioresour Technol, 2016, 215: 84-91. DOI:10.1016/j.biortech.2016.03.158 |
| [5] |
den Haan R, van Rensburg E, et al. Progress and challenges in the engineering of non-cellulolytic microorganisms for consolidated bioprocessing[J]. Curr Opin Biotech, 2015, 33: 32-38. DOI:10.1016/j.copbio.2014.10.003 |
| [6] |
Ilmén M, den Haan R, Brevnova E, et al. High level secretion of cellobiohydrolases by Saccharomyces cerevisiae[J]. Biotechnol Biofuels, 2011, 4(1): 30. DOI:10.1186/1754-6834-4-30 |
| [7] |
Bao JC, Huang MT, et al. Moderate expression of SEC16 increases protein secretion by Saccharomyces cerevisiae[J]. Appl Environl Microb, 2017, 83(14): e03400-16. |
| [8] |
Van RE, Den HR, Smith J, et al. The metabolic burden of cellulase expression by recombinant Saccharomyces cerevisiae Y294 in aerobic batch culture[J]. Appl Microb Biotech, 2012, 96(1): 197-209. DOI:10.1007/s00253-012-4037-9 |
| [9] |
Tyo K E, Liu Z, Petranovic D, et al. Imbalance of heterologous protein folding and disulfide bond formation rates yields runaway oxidative stress[J]. BMC Biology, 2012, 10(1): 16. DOI:10.1186/1741-7007-10-16 |
| [10] |
Kroukamp H, den Haan R, et al. Overexpression of native PSE1 and SOD1 in Saccharomyces cerevisiae improved heterologous cellulase secretion[J]. Appl Energ, 2013, 102(2): 150-156. |
| [11] |
Hou J, Österlund T, Liu Z, et al. Heat shock response improves heterologous protein secretion in Saccharomyces cerevisiae[J]. Appl Microb Biotech, 2013, 97(8): 3559-3568. DOI:10.1007/s00253-012-4596-9 |
| [12] |
万春, 万青青, 等. 过表达MRP8提高酿酒酵母乙酸耐性及乙醇发酵效率[J]. 生物加工过程, 2017, 15(5): 80-85. |
| [13] |
Zhang GC, Kong II, Kim H, et al. Construction of a quadruple auxotrophic mutant of an industrial polyploid Saccharomyces cerevisiae strain by using RNA-guided Cas9 nuclease[J]. Appl Environ Microbiol, 2014, 80(24): 7694-7701. DOI:10.1128/AEM.02310-14 |
| [14] |
Bai Flagfeldt D, Siewers V, Huang L, et al. Characterization of chromosomal integration sites for heterologous gene expression in Saccharomyces cerevisiae[J]. Yeast, 2009, 26(10): 545-551. DOI:10.1002/yea.v26:10 |
| [15] |
Jensen NB, Strucko T, Kildegaard KR, et al. EasyClone:method for iterative chromosomal integration of multiple genes in Saccharo-myces cerevisiae[J]. FEMS Yeast Res, 2014, 14: 238-248. DOI:10.1111/fyr.2014.14.issue-2 |
| [16] |
Gietz RD, Schiestl RH. High-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method[J]. Nat Protoc, 2007, 2(1): 31-34. DOI:10.1038/nprot.2007.13 |
| [17] |
La Grange DC, Pretorius IS, Claeyssens M, et al. Degradation of xylan to D-xylose by recombinant Saccharomyces cerevisiae co-expressing the Aspergillus niger β-xylosidase(xlnD)and the Trichoderma reesei xylanase Ⅱ(Xyn2)genes[J]. Appl Environ Microbiol, 2001, 67(12): 5512-5519. DOI:10.1128/AEM.67.12.5512-5519.2001 |
| [18] |
Teste MA, Duquenne M, Francois JM, et al. Validation of reference genes for quantitative expression analysis by real-time RT-PCR in Saccharomyces cerevisiae[J]. BMC Mol Biol, 2009, 10(1): 99. DOI:10.1186/1471-2199-10-99 |
| [19] |
Ram AFJ, Klis FM. Identification of fungal cell wall mutants using susceptibility assays based on Calcofluor white and Congo red[J]. Nature Protocols, 2006, 1(5): 2253. DOI:10.1038/nprot.2006.397 |
| [20] |
Tan SX, Teo M, Lam YT, et al. Cu, Zn superoxide dismutase and NADP(H)homeostasis are required for tolerance of endoplasmic reticulum stress in Saccharomyces cerevisiae[J]. Molecular Biology of the Cell, 2009, 20(5): 1493-1508. DOI:10.1091/mbc.e08-07-0697 |
| [21] |
Tang HT, Bao XM, et al. Engineering protein folding and transloca-tion improves heterologous protein secretion in Saccharomyces cerevisiae[J]. Biotechnol Bioeng, 2015, 112(9): 1872. DOI:10.1002/bit.25596 |
| [22] |
Tang HT, Song MH, He Y, et al. Engineering vesicle trafficking improves the extracellular activity and surface display efficiency of cellulases in Saccharomyces cerevisiae[J]. Biotechnol Biofuels, 2017, 10(1): 53. DOI:10.1186/s13068-017-0738-8 |
| [23] |
van Zyl JHD, den Haan R, van Zyl WH. Over-expression of native Saccharomyces cerevisiae exocytic SNARE genes increased heterologous cellulase secretion[J]. Appl Microb Biotech, 2014, 98(12): 5567-5578. |
| [24] |
van Zyl JHD, den Haan R, van Zyl WH. Overexpression of native Saccharomyces cerevisiae ER-to-Golgi SNARE genes increased heterologous cellulase secretion[J]. Appl Microb Biotech, 2016, 100(1): 505-518. DOI:10.1007/s00253-015-7022-2 |
| [25] |
Boorsma A, Nobel H, Riet B, et al. Characterization of the transcriptional response to cell wall stress in Saccharomyces cerevisiae[J]. Yeast, 2004, 21(5): 413-427. DOI:10.1002/(ISSN)1097-0061 |
| [26] |
Bruckmann A, Hensbergen PJ, Balog CIA, et al. Proteome analysis of aerobically and anaerobically grown Saccharomyces cerevisiae cells[J]. J Proteomics, 2009, 71(6): 662-669. DOI:10.1016/j.jprot.2008.11.012 |
| [27] |
Zhou W, Ryan JJ, Zhou H. Global analyses of sumoylated proteins in Saccharomyces cerevisiae:induction of protein sumoylation by cellular stresses[J]. J Biol Chem, 2004, 279(31): 32262-32268. DOI:10.1074/jbc.M404173200 |
| [28] |
Zhang MM, Zhao XQ, Cheng C, et al. Improved growth and ethanol fermentation of Saccharomyces cerevisiae in the presence of acetic acid by overexpression of SET5 and PPR1[J]. Biotechnol J, 2015, 10(12): 1903-1911. DOI:10.1002/biot.v10.12 |