2. 中国农业科学院农产品加工研究所,北京 100193
2. Institute of Food Science and Technology CAAS, Beijing 100193
保加利亚乳杆菌是一种重要的乳品工业发酵用菌株,是发酵过程中产酸、产风味物质的主要菌株。保加利亚乳杆菌在发酵过程中易受噬菌体污染导致发酵失败,这一直是困扰乳品工业的难题。所以研究乳酸菌抵御噬菌体攻击的机制,并筛选抗噬菌体侵染的菌株是发酵乳产业亟待解决的关键问题[1]。噬菌体是专以细菌作为宿主的一种生物体,通常生活在一些细菌高密度聚集的地方,乳制品发酵过程易受噬菌体侵染也正是因为给噬菌体提供了数量庞大的菌体宿主[2]。相应地,细菌与噬菌体在长期的斗争中也进化出了多种抵御噬菌体侵染的方式。如细菌可以通过在菌体表面生成胞外多糖类的生物膜[3],利用物理阻隔的方式阻止噬菌体进入菌体[4];也可以通过菌体表面的噬菌体受体基因沉默[5],进而改变细菌表面受体来抵抗噬菌体的感染等等。但这些防御系统都无法给细菌提供特异性免疫,而CRISPR(Clustered regularly interspaced shortpalindromic repeats)以及Cas(CRISPR-associated)蛋白构建的防御系统能够给宿主菌提供特异性免疫以抵抗噬菌体或质粒等外源基因的入侵[6],限制基因的水平转移[7],并且具有精准、高效、可遗传的优势,近年来受到广泛关注。
CRISPR-Cas系统存在于多数细菌和古细菌中,其作为后天获得性免疫系统能够抵抗噬菌体、质粒等外源DNA的侵染,并且能够通过不同的噬菌体感染而获得相应噬菌体的抗性[8]。CRISPR-Cas系统由重复序列(Direct Repeats,DR)、间隔序列(Spacer)、前导序列(Leading sequence)及Cas蛋白共同组成[9]。其中重复序列高度保守,具有回文序列,可以转录且形成茎环结构[10]。间隔序列与噬菌体、质粒有着同源性,记录着该菌株在长期进化过程中曾经遭受过哪些外源基因的侵入[11-13]。细菌在感染噬菌体后,噬菌体中的一段序列(Protospacer)被菌体CRISPR/cas系统所识别并被捕获整合到细菌基因组的CRISPR区,形成一个间隔序列,正是间隔序列给宿主提供了识别外源噬菌体的能力。研究证实,若删除间隔序列后,菌株抵抗噬菌体的能力随之消失[14]。前导序列与重复序列相连,能够识别新的间隔序列[15, 16],并启动crRNA(CRISPR RNAs)的转录[17]。CRISPR-Cas系统是针对噬菌体等外源基因的获得性免疫系统[18],并能将这种获得性免疫系统稳定遗传给后代[8, 16, 19, 20]
目前在乳酸菌上关于CRISPR-Cas系统防御噬菌体的系统研究只见于嗜热链球菌,在2015年,李婉、霍贵成等[21]已对嗜热链球菌基因组上的CRISPR的组成及抗噬菌体能力和作用机制进行了初步的研究和预测,而另外一株重要的发酵菌株—保加利亚乳杆菌CRISPR的相关研究目前较少。因此本文对保加利亚乳杆菌的CRISPR序列进行了系统分析,利用生物信息学方法对CRISPR区的DR序列进行了遗传进化分析并预测了其二级结构,同时对间隔序列也进行了同源性分析,为探明保加利亚乳杆菌抗噬菌体的分子机制奠定基础。
1 材料与方法 1.1 材料 1.1.1 实验菌株及培养基保加利亚乳杆菌LJJ-6来源于实验室(已在中国普通微生物菌种保藏管理中心进行专利保藏,保藏号CGMCC No.11346)、保加利亚乳杆菌ATCC11842来源于中国普通微生物菌种保藏管理中心(菌株编号CGMCC No.1.6970);其他6株分离自市售酸奶,培养基采用MRS肉汤培养基。
1.1.2 试剂与仪器细菌基因组提取试剂盒,北京天根生物技术有限公司;溶菌酶,Solabiro公司;TIANGEN 2×PCRMasterMix,天根生化科技有限公司;引物,华大基因科技有限公司;MRS肉汤,北京陆桥生物技术有限公司。LDZX-50KB立式压力蒸汽灭菌器,上海申安医疗器械厂;Sigma-3K15离心机,德国SIGMA科技有限公司;DYY-6C电泳仪,北京六一仪器厂;Fluor ChemFC2凝胶成像系统,美国Alpha公司;TP600-PCR扩增仪,TakARA Bio INC;DHP-90278型电热恒温培养箱,上海一恒科技有限公司;WD-9403C紫外仪,北京市六一仪器厂;HDL超净工作台,北京东联哈尔仪器制造有限公司。
1.2 方法 1.2.1 菌株的培养及基因组DNA的提取将冷冻保存的实验菌株与从酸奶中分离得到的保加利亚乳杆菌转接到MRS肉汤培养基中,37℃活化培养两代,每代培养18 h。菌株总DNA的提取按照细菌基因组提取试剂盒说明书进行。
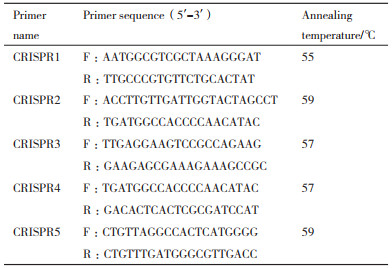
1.2.2 CRISPR序列扩增与测序在NCBI(http://www.ncbi.nlm.nih.gov/)上查找已公布全基因组序列的9株保加利亚乳杆菌基因组信息作为引物设计的模板,在9株保加利亚乳杆菌中每一株菌含一个或两个CRISPR系统,仅少部分含2个CRISPR系统。根据其CRISPR上下游序列的保守性利用Primer Premier 7.0设计了5种不同类型的CRISPR区扩增引物,确保了CRISPR位点扩增时不被遗漏。然后对8株实验菌株分别用这5种不同引物进行CRISPR一一扩增,将有扩增结果的条带进行测序验证。按试剂盒说明书建立PCR反应体系(50 μL):DNA模板2 μL;正向引物1 μL;反向引物1 μL;10×LongTaqBufferⅠ5 μL;dNTP Mixture(2.5mmol·L-1)4 μL;ddH2O 37 μL。
PCR反应循环设置:94℃预变性3 min,94℃变性30 sec,60℃退火30 sec,72℃延伸3 min,72℃终延伸5 min,37 cycles。结果检测:待反应结束后,取PCR反应产物5 μL,1%琼脂糖凝胶电泳检测,将目的条带单一的样本送公司测序。
1.2.3 CRISPR基因座活性分析对扩增的8株实验菌的CRISPR与NCBI中已公布全基因组序列的9株德氏乳杆菌标准菌的CRISPR序列利用CRISPR Finder(http://crispr.u-psud.fr/Server/)在线软件进行查找,统计标准菌株及实验菌株中每个CRISPR所含的独特间隔序列的数目,分析基因座活性。利用GraphPad Prism 7软件制作图。
1.2.4 CRISPR序列同源性分析对实验菌株保加利亚乳酸杆菌、标准菌株保加利亚乳酸杆的CRISPR区进行重复序列整理。将重复序列利用MEGA7软件下的邻接法(Neighbor-JoiningMethod),自展值(Bootstrap value)分析重复次数为1 000次构建系统发育树,对重复序列进行同源性分析。
1.2.5 重复序列二级结构预测将测序得到的重复序列转换成RNA序列后,在线提交RNAfoldWebServer(http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi)服务器,进行重复序列二级结构预测分析。
2 结果 2.1 CRISPR序列扩增与测序根据NCBI中已公布全基因组序列的德氏乳杆菌保加利亚乳酸杆菌为标准菌株设计引物,CRISPR区序列的扩增引物如表 1。用每一类型的CRISPR引物分别对8株实验菌进行一一扩增。扩增结果见图 1。在8株实验菌均含有一个可信CRISPR区,ATCC11842、GM在以CRISPR1为扩增引物的条件下,在大于2 000 bp附近均有清晰条带出现,而在其他引物扩增中均未获得扩增条带;LJJ-6在CRISPR2为扩增引物的条件下,在2 000 bp处有清晰单一条带,其他扩增引物均未扩增出相应的条带;SY、YL在CRISPR4为扩增引物的条件下,在1 000-2 000 bp之间均有清晰条带,且扩增效率高、特异性较强,其他引物扩增下均没有发现扩增结果;MN、JLB在CRISPR2引物扩增下,在800 bp附近均扩增出单一条带,且无非特异性条带出现,其他引物均未获得扩增条带;WD在CRISPR5为引物下出现了扩增结果,大约在500 bp附近,其余引物扩增下均无扩增结果。将扩增的单一条带送公司测序。
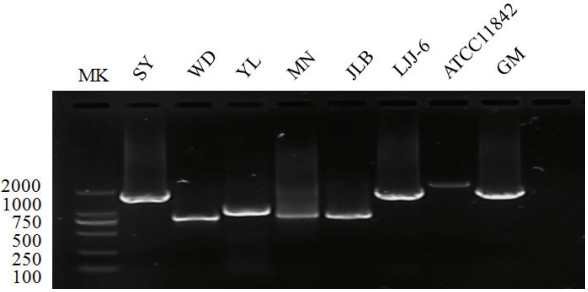
|
| 图 1 CRISPR序列扩增 |
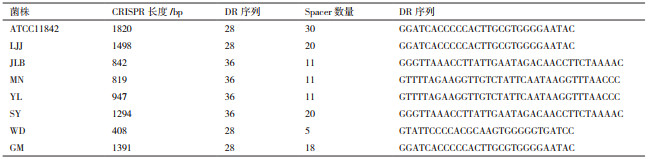
对测序结果分析整理,用CRISPR-Finder进行CRISPR序列查找鉴定,可知图 1中扩增条带均为CRISPR序列。CRISPR序列信息如表 2,其中CRISPR序列最长为1 820 bp,含有30个间隔序列,最短仅408 bp,含5个间隔序列。重复序列最长为36 bp,最短为28 bp。
9株保加利亚乳杆菌标准菌株及8株实验菌株,共17株菌各个CRISPR基因座所含的独特间隔序列数目散布图如图 2。其中CRISPR1基因座出现了5次,在基因组中出现频率较高,其间隔序列最多21个,平均约17个;CRISPR2基因座中间隔序列数目变化较大,最多为63个,最少只有5个,平均约33个;CRISPR3、CRISPR5在基因组中出现的频率较低,其基因座间隔序列数最多为11个,最少仅8个;CRISPR4基因座中的间隔序列数目最多为39个,最少15个,平均为25个。CRISPR6基因座中间隔序列最多为30个,最少18个,分布均匀每个基因座平均约23个间隔序列。
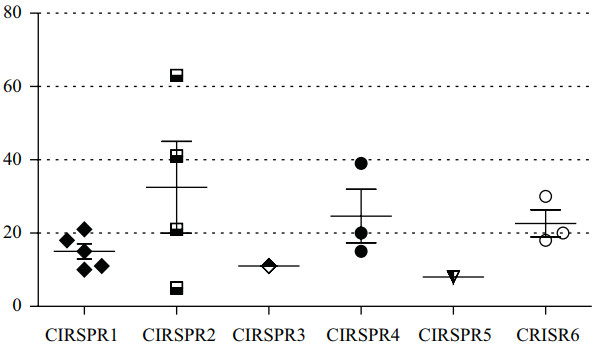
|
| 图 2 CRISPR重复-间隔序列的数目分布 |
由图 2可以看出,CRISPR2基因座中间隔序列数目的最大值和平均值均为最高,因此推测出保加利亚乳杆菌中CRISPR2基因座最为活跃,则对抗外来基因元件入侵的能力更强,CRISPR4基因座次之。而CRISPR3、CRISPR5基因座中间隔序列数目最低且在基因组中出现的频率最低,因此推测保加利亚乳杆菌中CRISPR3、CRISPR5基因座最不活跃。
2.4 CRISPR序列同源性分析将8株实验菌株的重复序列与NCBI中已公开全基因组序列的9株标准菌株保加利亚乳杆菌CRISPR重复序列归纳整理构建遗传进化树(图 3)。该进化树主要包含8个分支,WD菌株的DR与ND02、JCM1738标准菌株的DR位于同一分支;实验菌株MN、YL与标准菌株2038、DSM20080、KCTC1373的DR位于同一分支,且同源性较高;JLB、SY的DR与标准菌株ND04的DR位于同一分支上,LJJ-6与GM、ATCC11842的DR位于同一分支,同源性高度一致,在进化方向上相近。
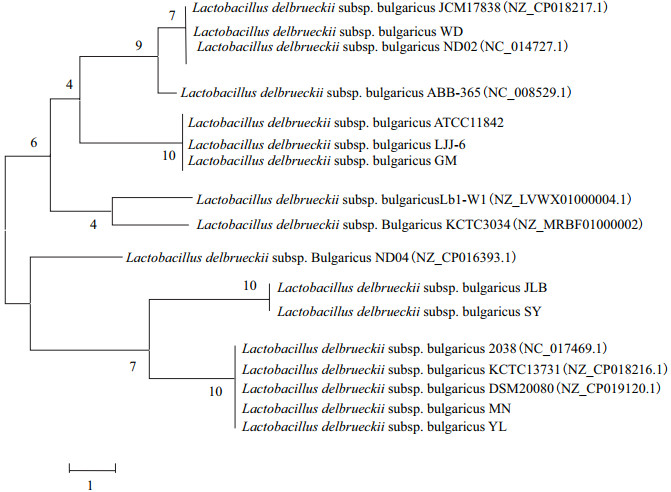
|
| 图 3 重复序列的遗传进化分析 |
通过对17株保加利亚乳杆菌CRISPR区的6种重复序列RNA二级结构预测时均发现,重复序列均可形成如图 4的茎环结构,根据茎环的数量特征可以分为两类,一类是如图 4(a)、(b)、(c)仅有1个茎区,2个大小不同的环部分配茎区两侧,以“环”为主。另一类是如图 4(d)、(e)、(f)形成多个茎、环,以“茎”为主,同时在序列比对分析还发现一个重复序列对应多种不同的间隔序列,而一个间隔序列只对应一个重复序列,二级结构的形成可能与新Spacer及cas编码蛋白的相互作用有关。据文献报道,新Spacer通常会插入CRISPR区前导序列与第1个DR之间[16, 17]。

|
| 图 4 重复序列二级结构预测 |
细菌CRISPR作为一种抵御噬菌体攻击的获得性防御机制,近年来已经成为研究领域的热点。在乳制品发酵行业中,CRISPR作为一种高效的抗噬菌体系统,已在嗜热链球菌中被证明[22]。对于每个独特的间隔序列都是从入侵的外源基因元件中捕获的,然后整合到自己的基因组中,形成一个获得性免疫记忆系统,产生特异性的记忆功能,在下次有携带相同序列或外源片段入侵时,它们会迅速识别,并募集体内的相关cas蛋白(比如cas9)对目标基因进行切割,产生相对应的抗性,来抵抗外源基因的入侵。所以通过基因改造CRISPR结构将已知侵染发酵的噬菌体保守序列插入发酵菌的CRISPR结构中,使之形成特异性的记忆免疫系统[23],在噬菌体侵染时会产生相应的抗性以抵御这些噬菌体的侵染,这对于开发抗噬菌体的发酵菌株具有较好的应用前景。本研究中8株保加利亚乳杆菌CRISPR的126个DR序列与126个spacer序列,分别来自8个不同厂家的商品化酸奶,由于发酵菌株的不同,且在长期进化过程中遇到的外源攻击元件也不同,因此造成菌株CRISPR序列的显著差异。将17个CRISPR结构进行归纳整理为6种不同类型的CRISPR系统,对每一类型的CRISPR基因座活性进行了预测分析,结果显示spacer序列丰富的其基因座活性高,可能的原因是CRISPR位点越丰富所产生的特异性识别就会越强,由于细菌CRISPR间隔序列来源于外源核酸片段,而核酸片段通常是指病毒噬菌体、质粒DNA。所以来源于噬菌体的外源片段越丰富,产生相应抗性的种类就越多,意味着细菌抗噬菌体能力越强。因此CRISPR基因座中间隔序列的数目和种类可在一定程度上反映该CRISPR基因座的活性程度[24]。CRISPR系统存在leader序列,repeat序列,spacer片段,cas基因,而leader序列与紧邻的末端repeat序列之间存在新spacer插入位点,推测可能为相关cas蛋白的结合位点[16]。本文研究了9株保加利亚乳杆菌标准菌株及8株实验菌株的17个CRISPR系统均发现较为保守的回文结构,该回文结构极可能为相关cas蛋白结合位点。在对8个实验菌株的CRISPR结构进行研究时发现,除了repeat数目较多的CRISPR结构有30个之外,有的仅有5个repeat序列的CRISPR结构。
据文献报道,独特的间隔序列是宿主防御入侵的噬菌体或质粒DNA形成的一种特异性免疫识别系统[23]。在对实验菌株间隔序列进行比对分析时发现,96条间隔(除已公布序列的ATCC11842菌株的30条)序列中仅15条完全比对上原间隔序列,比对率较低,与Mojica等[25]的研究报道,对4 500条spacer进行blast比对分析发现仅88条与原间隔序列且有较高的相似性。相关的原因据Edwards等[26]报道为:第一,spacer序列较短且异常多样化,能够比对到显著相似性外源片段的概率较低;第二,可能是由于对应的噬菌体数据库目前还不够完善,已知噬菌体的序列较少,还不能从现有数据库中检索到spacer序列相似的外源片段。此外还有49条间隔序列与自然界的质粒与噬菌体有着高相似性,且比对到来源于噬菌体的大于质粒的,说明很有可能CRISPR结构间有着基因水平转移的现象存在,乳酸菌抗噬菌体能力的强弱可能与CRISPR结构存在一定的联系,此外分析时还发现同一个重复序列对应着多个不同间隔序列,而一个间隔序列只对应一个重复序列,推测可能的原因是重复序列是一种识别机制,这还有待进一步研究的证明。这一点与焦雪等[27]的报道相似。同时对于同源性较高的菌株它们的间隔序列和重复序列也会存在很大的不一致性,这可能是由于它们的生长环境不一致造成的差异性。本研究还发现乳酸杆菌属的其他菌株Lactobacillus delbrueckii subsp. bulgaricus2038、KCTC13731、DSM20080与实验菌株Lactobacillus delbrueckii subsp. bulgaricusMN、YL的重复序列相同,但它们的亲缘关系是相对较远的,可能的原因是发生了水平基因转移CRISPR序列,这种情况在其他菌株中也有发现[28]。总之,本研究为探明保加利亚乳杆菌抗噬菌体的分子机制奠定基础,也为筛选、开发和改造抗噬菌体的发酵剂菌株提供了基础数据,具有较高的实际意义和应用价值。
4 结论本文以9株已公布全基因组序列的保加利亚乳杆菌和8株实验株为基础,对保加利亚乳杆菌的CRISPR进行了系统的生物信息分析,并对菌株的抗噬菌体侵染能力进行了预测和分析。结果发现CRISPR中间隔序列的高度可变性的特点。进一步发现CRISPR2基因座的活性最高,抗噬菌体能力较强。同时发现在ATCC11842同一株菌的2个不同CRISPR结构也是存在一定的差异,说明CRISPR结构的多态性变化。
| [1] |
Andrea Q, Sylvain M, Geneviève M, et al. Streptococcus thermophilus bacteriophages[J]. International Dairy Journal, 2010, 20(10): 657-664. DOI:10.1016/j.idairyj.2010.03.012 |
| [2] |
Klaenhammer TR, Fitzgerald GF. Bacteriophages and bacteriophage resistance[J]. Springer Netherlands, 1994, 106-168. |
| [3] |
ChaturongakulS, Ounjai P. Phage-host interplay:examples from tailed phages and Gram-negative bacterial pathogens[J]. Frontiers in Microbiology, 2014, 5. |
| [4] |
Simon J. Labrie, Julie E. Samson, Sylvain Moineau. Bacteriophage resistance mechanisms[J]. Nat Rev Microbiol, 2010, 8(5): 3317-327. |
| [5] |
Bikard D, Marraffini LA, et al. Innate and adaptive immunity in bacteria:mechanisms of programmed genetic variation to fight bacteriophages[J]. Current Opinion in Immunology, 2012, 24(1): 15-20. DOI:10.1016/j.coi.2011.10.005 |
| [6] |
Manica A, Zebec Z, Teichmann D, et al. In vivo activity of CRISPR-mediated virus defence in a hyperthermophilic archaeon[M]// Molecular Microbiology. : 481-491.
|
| [7] |
Wiedenheft B, Sternberg SH, Doudna JA. RNA-guided genetic silencing systems in bacteria and archaea[J]. Nature, 2012, 482(7385): 331. DOI:10.1038/nature10886 |
| [8] |
Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea[J]. Science, 2010, 327(5962): 167-170. DOI:10.1126/science.1179555 |
| [9] |
李婉, 边鑫, 王娜娜, 等. 乳酸菌CRISPR-Cas系统研究进展[J]. 中国乳品工业, 2016, 44(12): 22-23. DOI:10.3969/j.issn.1001-2230.2016.12.006 |
| [10] |
Kunin V, Sorek R, Hugenholtz P. Evolutionary conservation of sequence and secondary structures in CRISPR repeats[J]. Genome Biology, 2007, 8(4): R61. DOI:10.1186/gb-2007-8-4-r61 |
| [11] |
Wang J, Friedman G, Doyon Y, et al. Targeted gene addition to a predetermined site in the human genome using a ZFN-based nicking enzyme[J]. Genome Res, 2012, 22(7): 1316-1326. DOI:10.1101/gr.122879.111 |
| [12] |
Fyodor D. Urnov, Edward J. Rebar, Michael C. Holmes, et al. Genome editing with engineered zinc finger nucleases[J]. Nat Rev Genet, 2010, 11(9): 636-646. DOI:10.1038/nrg2842 |
| [13] |
Yanfang Fu, Jennifer A Foden, Cyd Khayter, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells[J]. Nat Biotechnol, 2013, 31(9): 822-826. DOI:10.1038/nbt.2623 |
| [14] |
Prashant Mali, John Aach, P Benjamin Stranges, et al. CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering[J]. Nat Biotechnol, 2013, 31(9): 833-838. DOI:10.1038/nbt.2675 |
| [15] |
Deveau, H. Barrangou, R. Garneau, et al. Phage Response to CRISPR-Encoded Resistance in Streptococcus thermophilus[J]. J Bacteriol, 2007, 190(4): 1390-1400. |
| [16] |
Barrangou, R, R. Fremaux, C. Deveau, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007, 315(5819): 1709-1712. DOI:10.1126/science.1138140 |
| [17] |
Rodolphe Barrangou. CRISPR-Cas systems and RNA-guided interference[J]. Wiley Interdisciplinary Reviews:RNA, 2013, 4(3): 267-278. DOI:10.1002/wrna.1159 |
| [18] |
Ruud. Jansen, Jan. D. A. van Embden, Wim. Gaastra, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Mol Microbiol, 2002, 43(6): 1565-1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [19] |
John van der Oost, Matthijs M. Jore, Edze R. Westra, et al. CRISPR-based adaptive and heritable immunity in prokaryotes[J]. Trends Biochem Sci, 2009, 34(8): 401-407. DOI:10.1016/j.tibs.2009.05.002 |
| [20] |
Deveau, H ., J. E. Garneau, S. Moineau. CRISPR/Cas System and Its Role in Phage-Bacteria Interactions[J]. Annual Review of Microbiology, 2010, 64(1): 475-493. DOI:10.1146/annurev.micro.112408.134123 |
| [21] |
李婉, 梁宏彰, 张丹青. 等, 嗜热链球菌CRISPR-Cas系统的检测[J]. 微生物学报, 2016(04): 680-688. |
| [22] |
李婉, 王娜娜, 张丹青, 霍贵成. 嗜热链球菌CRISPR序列的检测及原间隔序列预测[J]. 现代食品科技, 2016, 32(10): 252-258. |
| [23] |
Bruggemann, HolgerYoung, JacqueC. Dill, et al. Phage-Induced Expression of CRISPR-Associated Proteins Is Revealed by Shotgun Proteomics in Streptococcus thermophilus[J]. PLoS One, 2012, 7(5): e38077. DOI:10.1371/journal.pone.0038077 |
| [24] |
Datsenko KA, Pougach K, Tikhonov, et al. Molecular memory of prior infections activates the CRISPR/Cas adaptive bacterial immunity system[J]. Nat Commun, 2012, 3: 945. DOI:10.1038/ncomms1937 |
| [25] |
Mojica FJ, Díez-Villaseñor C, García-Martínez J, et al. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements[J]. Journal of Molecular Evolution, 2005, 60(2): 174-182. DOI:10.1007/s00239-004-0046-3 |
| [26] |
Edwards RA, Rohwer F. Viral metagenomics[J]. Nature Reviews Microbiology, 2005, 3(6): 504-510. DOI:10.1038/nrmicro1163 |
| [27] |
焦雪, 张培军, 冷东泽, 等. 温和气单胞菌CRISPR位点的检测与分析[J]. 中国兽医学报, 2017, 37(05): 839-843. |
| [28] |
Chakraborty S, Snijders AP, Chakravorty R, et al. Comparative network clustering of direct repeats(DRs)and cas genes confirms the possibility of the horizontal transfer of CRISPR locus among bacteria[J]. Molecular Phylogenetics and Evolution, 2010, 56: 878-887. DOI:10.1016/j.ympev.2010.05.020 |