2002年,Jansen等[1, 2]首次定义了存在于细菌或古细菌中的抵御噬菌体入侵感染的CRISPR/Cas系统。该系统由成簇规律间隔短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR)结构,以及CRISPR相关的蛋白质(CRSPR-associated protein,Cas9)组成[3, 4]。CRISPR/Cas9系统大体分为三个类型,其中的Ⅱ型CRISPR/Cas9系统仅需一种Cas9蛋白就能独立对靶DNA有效切割[5]。该系统依赖质粒转录的一段单链向导RNA(single guide RNA,sgRNA)和其序列互补的靶DNA形成RNA-DNA复合物来决定CRISPR/Cas9核酸酶特异性[6]。由于该系统设计简单,仅需完成一个克隆改变Cas9质粒的小段gRNA前序列即可特异性靶向切割,并且适用范围广阔,目前已有报道在动物[7-8]、植物[9-11]、微生物[12-13]和病毒[14-15]等都具有显著的切割活性。该系统靶向切割细胞基因组后,细胞利用自身同源性定向修复(Homology directed repair,HDR)机制实现带有同源臂的外源序列插入,是目前染色质定向改造主要方式之一。
基因FOXA2[16],曾用名是HNF3β,定位于人染色体中20p11,全长45 kb,包含3个外显子以及两个内含子。其蛋白长度为457个氨基酸。FOXA2蛋白属于叉头框(Fox)家族的成员。因该家族蛋白具有一组高度保守的翼状螺旋区域,主要特征是作为转录因子作用于DNA结合区域,所以又称为转录因子FOXA2。Fox家族蛋白最先被发现于胚胎发育的进程中,深入研究发现Fox家族蛋白在免疫调节,周期调控等多个生理过程中发挥着重要的作用。转录因子之一的FOXA2近年来被发现与能量代谢、胰腺发育、癌症与上皮间质转化(Epithelial-mesenchymal transition,EMT)等都有紧密的关系[17, 18]。
完成基因FOXA2的内源性示踪且不干扰基因本身的转录翻译与蛋白质修饰,是一种很好的追踪FOXA2基因表达开启与关闭的方法。对于寻找FOXA2基因的通路相关基因、互作蛋白、潜在靶向药物与抑制剂等都有很好的前景。在FOXA2的独立开放阅读框(Open Reading Frame,ORF)后,我们选择在该基因第3个外显子的蛋白质编码区(Coding sequence,CDS)后插入绿色荧光蛋白(Enhanced Green Fluorescent Protein,EGFP)EGFP Donor(IRES+EGFP Sequences)序列。内部核糖体进入位点序列(Internal ribosome entry site,IRES)功能是招募核糖体对后面EGFP mRNA进行翻译。本研究通过探索新型基因编辑CRISPR/Cas9的技术方法,实现对生物基因序列的高效编辑;通过构建基因FOXA2的绿色荧光蛋白示踪体系,为实现与FOXA2相关的代谢、肿瘤、免疫等深入研究提供便捷有效的材料工具。
1 材料与方法 1.1 材料CRISPR/Cas9真核表达载体pX330-U6-Chimeric_BB-CBh-hSpCas9(Addgene plasmid # 42230);HEK293T细胞购自中科院典藏细胞库、乳腺癌MCF-7细胞购自TACC;Bbs Ⅰ、Xba Ⅰ、EcoR Ⅰ限制性内切酶和T4连接酶购自Thermo公司;DMEM培养基购于GIBCO公司;胎牛血清(FBS)为Hyclone公司产品;Anti-FOXA2(ab60721)、Anti-β-actin(HRP)(ab49900)抗体购自abcam公司;Anti-Mouse(HRP)(Catalog170-6516)购自BIO-RAD公司;Anti-GFP(AF1483)、Anti-Rabbit(HRP)(A0208)购自Beyotime公司。
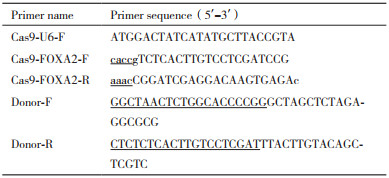
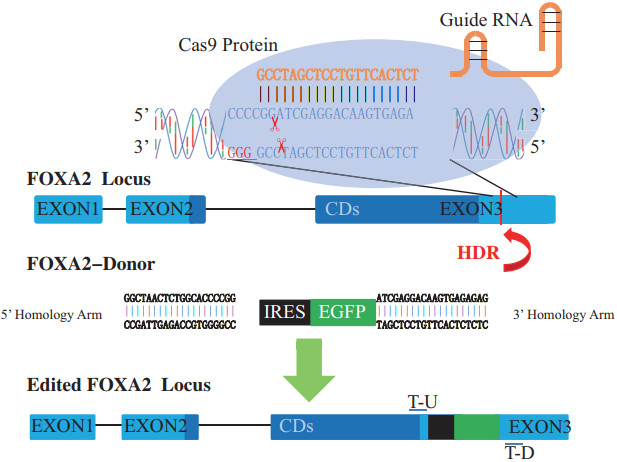
1.2 方法 1.2.1 Cas9/gRNA靶位点设计以及真核表达载体的构建根据真核表达载体pX330-U6-Chimeric_BB-CBh-hSpCas9识别的前间区序列邻近基序(Protospacer Adjacent Motif,PAM)位点NGG,质粒表达的gRNA特异性互补识别PAM位点及其上游20 bp核酸序列。Cas9核酸酶在PAM位点上游第3位和第4位碱基间切开以形成平末端切口。例如5' -NNNNNNNNNNNNNNNNN▲NNNNGG-3' 。gRNA靶位点的选择参考麻省理工学院在线设计网站http://crispr.mit.edu/,选取在线网站Score较高的序列。经HEK293T细胞中靶向位点活性检测,最终确定靶向位点,位于人染色体20p11,FOXA2基因第3个外显子中,CDS区后(图 1)。
|
| 图 1 CRISPR/Cas9靶向FOXA2插入IRES+EGFP |
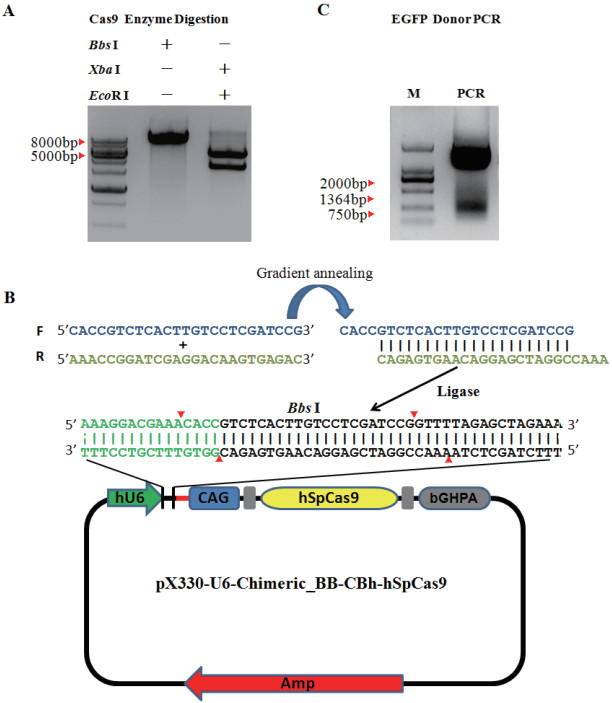
用Bbs Ⅰ酶切表达载体pX330-U6-Chimeric_BB-CBh-hSpCas9,以求表达载体质粒线性化。结合Bbs Ⅰ酶切后粘性末端碱基,设计靶向gRNA序列克隆引物:Cas9-FOXA2-F,Cas9-FOXA2-R(带下划线为粘性末端序列碱基)。将两条引物梯度退火至使双链互补结合。T4连接酶连接上述互补序列与已线性化的表达载体(图 2-B)。转化大肠杆菌感受态DH5α涂板,挑取单菌落,质粒小提。表达载体测序鉴定。测序引物为Cas9-U6-F。示踪核酸序列供体DNA片段EGFP Donor引物:Donor-F,Donor-R。Donor引物中带下划线为20 bp同源臂序列(表 1)。
|
| 图 2 靶向FOXA2质粒和EGFP Donor的克隆及鉴定 A:载体pX330-U6-Chimeric_BB-CBh-hSpCas9的酶切鉴定及线性化;B:靶向FOXA2的Cas9-FOXA2载体的克隆构建;C:EGFP Donor的克隆PCR |
取对数生长期的MCF-7细胞,传代至六孔板,细胞生长至铺板70%。采用Lipofectamine 2000转染靶向FOXA2基因的真核表达质粒pX330-U6-Chimeric_BB-CBh-hSpCas9(简写为Cas9-FOXA2)3 µg和DNA片段EGFP Donor 500 ng,阴性对照组转染空靶表达质粒Cas9-NG 3 µg及DNA片段EGFP Donor 500 ng。用含10%的FBS和1%双抗(工作浓度100 mg/L青霉素和100 mg/L链霉素)的DMEM完全培养基,于37℃、5%浓度CO2、细胞培养箱培养。48 h后汞灯下观察细胞荧光,拍照,并以绿色荧光为标志物,取阴性对照组做阴性参考进行流式细胞荧光分选技术(Fluorescence activated Cell Sorting,FACS)分选。以单孔单细胞为标准,梯度有限稀释分选后的细胞至96孔板。同上培养条件继续培养被编辑的单克隆细胞至细胞适当密度,传代富集。
1.2.3 基因组PCR鉴定于基因组序列靶向切口上游76 bp(引物末端碱基开始至切口处)设计引物T-U,切口下游62 bp(切口处至引物前端)设计引物T-D。理论上编辑成功的细胞检测PCR片段长度为1 504 bp。引物序列信息,见表 2。取FACS分选、富集的单克隆细胞提取基因组DNA。以不同单克隆细胞基因组DNA为模板,上述引物T-U和T-D,进行聚合酶链式反应(Polymerase Chain Reaction,PCR)。鉴定引物位点见图 1。
相较于正常细胞,被成功编辑的细胞MCF-7 KI-EGFP,理论上在细胞内具有示踪FOXA2基因开启或关闭的能力。肿瘤细胞的EMT是肿瘤细胞丧失上皮表型至获得间质表型并同时增强细胞迁移和侵袭能力的过程,此过程重要的调控基因FOXA2会伴随着细胞上皮型至间质型的转化表达量逐渐下降甚至部分关闭。表皮细胞生长因子(Epidermal Growth Factor,EGF)具有诱导肿瘤上皮型细胞向肿瘤间质型细胞转化的作用。因此我们设计使用EGF诱导编辑鉴定的上皮表型单克隆细胞株MCF-7 KI-EGFP来验证示踪体系。
细胞内转录水平(mRNA expression levels)验证基因FOXA2示踪体系。胰酶消化FACS分选、富集的单克隆细胞,并提取细胞总RNA。采用逆转录方法,将总RNA逆转录为cDNA再进行RT-PCR检测。测定对照组及实验组中细胞FOXA2表达量差异。
蛋白质印迹法(Western blot)验证基因FOXA2示踪体系。胰酶消化FACS分选、富集的单克隆细胞,并提取细胞总蛋白。正常MCF-7细胞总蛋白作为阴性对照。对照组和实验组上样相同蛋白量约30 µg,通过检测细胞骨架蛋白β-actin表达量差异确定上样平衡。测定对照组,实验组细胞总蛋白中FOXA2蛋白的表达量差异。
2 结果 2.1 表达载体以及同源重组片段的克隆、鉴定真核表达载体pX330-U6-Chimeric_BB-CBh-hSpCas9原始载体的酶切表明,被限制性核酸内切酶Bbs Ⅰ线性化的表达载体呈现一条8 506 bp的单链,被限制性核酸内切酶Xba Ⅰ、EcoR Ⅰ双酶切后呈现5 095 bp和3 411 bp双片段(图 2-A)。靶向FOXA2的真核表达载体质粒设计(图 2-B)。测序结果表明,靶向FOXA2基因的Cas9-FOXA2克隆成功。同源重组片段EGFP Donor的克隆及纯化(图 2-C)。
2.2 靶向FOXA2基因座及同源性定向修复插入报告基因根据实验目的,结合靶位点设计网站筛选出的靶向位点,在FOXA2第三号外显子中CDs区后插入绿色荧光蛋白报告基因(图 1)。在细胞面积铺板70%且处于对数生长期的MCF-7细胞中脂质体转染法转染Cas9-FOXA2表达质粒和EGFP Donor报告基因片段。阴性对照组转染pX330-U6-Chimeric_BB-CBh-hSpCas9(Cas9-NG)原始质粒载体和EGFP Donor报告基因片段。48 h后观察绿色荧光,经FACS分选,传代后的混合细胞集群荧光显著增多(图 3)。根据荧光细胞数量占全细胞比例统计,MCF-7细胞转染效率 > 20%。

|
| 图 3 在MCF-7细胞FOXA2基因中插入绿色荧光蛋白并流式分选富集 A和B为阴性对照组;C和D为未分选的实验组;E和F为分选后的实验组 |
取FACS分选后的细胞,于96孔板梯度稀释(图 4-A)。A1孔为分选后的混合细胞,从A1孔至H1孔依次梯度稀释10倍;再从第1列至第12列,依次稀释10倍。旋转摇动96孔板致使单细胞停落于孔径中央区域,并小心放置于细胞培养箱静止培养。于细胞完全贴壁后开始分列前,统计具有单细胞的孔径,做好标记。培养基添加双抗并提高血清浓度至15%。等待细胞生长分裂,以求编辑的单克隆细胞的富集,观察有绿色荧光的孔径细胞,做好编号1 #至10 #。传代扩培,至细胞数量增至1×106,取5×105细胞传代扩培,剩余细胞提取基因组DNA。以上述基因组DNA为模板,KI位点前后引物T-U和T-D(见表 2)扩增跨界片段(EGFP Donor片段及其前后部分基因组序列片段)(图 4-B),并纯化该PCR序列,测序结果显示1 #单克隆细胞于靶位点发生有效同源重组。

|
| 图 4 FACS分选的阳性细胞梯度有限稀释及单细胞基因组PCR鉴定 从A1到H1,从第1列到第12列的有限梯度稀释;B:单克隆细胞的PCR鉴定 |
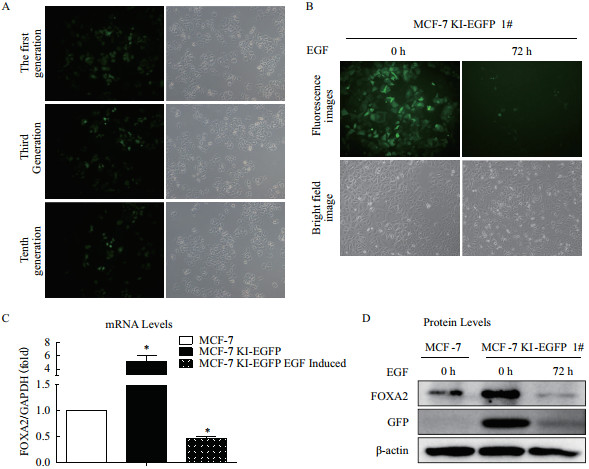
取第1代、第3代、第10代冻存的编辑成功的细胞株,复苏。拍照荧光图及明场图,观察细胞荧光稳定性。结果(图 5)显示,绿色荧光传代具有相对的稳定性,绿色荧光蛋白表达示踪FOXA2具有准确相关性。
|
| 图 5 基因编辑细胞传代稳定性及诱导验证 A:细胞系传代稳定性;B:单克隆细胞株MCF-7 KI-EGFP 1 #的EGF诱导验证;C:FOXA2的mRNA水平检测;D:FOXA2蛋白质水平的Western blot检测 |
取正常MCF-7细胞传代铺板60 mm盘,2盘,作为对照组。鉴定成功的第1代单克隆MCF-7 KI-EGFP细胞1 #传代铺板60 mm盘,4盘,为实验组。传代密度为50%。待细胞融合度80%,对单克隆MCF-7 KI-EGFP 1 #细胞2盘,进行EGF诱导处理。其余不做处理。EGF工作浓度为100 ng/ml。诱导时间72 h。
MCF-7 KI-EGFP单克隆细胞FOXA2转录水平验证。收取对照细胞MCF-7、MCF-7 KI-EGFP 1 #及EGF诱导MCF-7 KI-EGFP 1 #三盘细胞,提取总RNA并逆转录为cDNA,进行RT-PCR检测。检测结果(图 5-B)显示,相较于正常MCF-7细胞,编辑的单克隆细胞1 #,FOXA2基因mRNA表达量有明显上调;相较于对照组MCF-7细胞,1 #细胞在EGF诱导处理后,FOXA2基因mRNA表达量明显下调。
MCF-7-KI-EGFP单克隆细胞FOXA2基因翻译水平验证。收取对照组MCF-7、MCF-7 KI-EGFP 1 #以及EGF诱导的MCF-7 KI-EGFP 1 #三盘细胞,并提取总蛋白,进行蛋白质免疫印迹实验。数据(图 5-C)显示,相较于正常MCF-7细胞,单克隆细胞MCF-7 KI-EGFP 1 #,具有明显的FOXA2蛋白表达上调趋势;EGF诱导处理的MCF-7 KI-EGFP 1 #细胞,相较于正常MCF-7细胞,FOXA2蛋白表达下调;相较于未处理的1 #细胞,EGF诱导处理的1 #细胞绿色荧光蛋白表达明显减少。
3 讨论FOXA2作为分化因子,在胚胎细胞向肝细胞特定分化中具有重要作用[19, 20];在调节肝脂质代谢过程中,Foxa2受胰岛素和胰高血糖素的诱导调节,而被AKT信号磷酸化出核,P300乙酰化或HDACs/Sirt1去乙酰化,从而行使转录调节下游基因功能[21];在乳腺癌、肺腺癌等疾病中,FOXA2激活细胞间粘附因子E-cadherin的转录表达,并且抑制ZEB2等实现抑制肿瘤EMT进程[17, 18]。实现基因FOXA2的示踪,对于上下游通路基因研究、疾病模型模拟、肿瘤发生发展研究具有重要的意义。本研究在乳腺癌细胞系MCF-7中实现对基因FOXA2的示踪,旨在实现肿瘤相关信号通路发生发展过程的探索。
基因编辑CRISPR/Cas9技术,是近十年快速发展起来的靶向核酸新型技术,作为核酸酶,并经人工gRNA改造后理论上可以靶向切割任意匹配核酸序列。基因编辑前期发展方向,多是围绕特定基因修饰细胞株及动物转基因模型建立展开。随着研究深入,技术进步目前已实现在临床应用于急性淋巴白血病、肺癌等人体T淋巴细胞改造[22]。
在乳腺癌上皮表型MCF-7细胞中,靶向该细胞特定基因非编码基因组区域研究基因区域性功能显示其有效和便捷性[23]。且MCF-7细胞对药筛、诱导等具有很好的响应性,是合适的乳腺腔管型肿瘤研究模型,因此我们选取MCF-7作为靶向研究细胞株。在挑选具有高SgRNA活性靶向位点阶段,我们根据网站http://crispr.mit.edu/筛选出4个高分位点,克隆出4个位点的靶向载体并进行HEK293T细胞的转染、靶向区域的PCR以及T7E1核酸错配内切酶的鉴定,来确定Cas9靶向载体在293T细胞的切割效率,并选取靶向切割活性最高的SgRNA位点。在基因FOXA2蛋白质编码区后插入IRES+EGFP序列且不破坏FOXA2终止密码子,因此FOXA2的CDS区绿色荧光蛋白核酸序列共处一个基因座,共用一套转录增强区域和抑制区域,保证EGFP和FOXA2转录水平同步进行。和IRES序列作为招募核糖体的内部核糖体进入位点序列(Internal ribosome entry site,IRES),帮助EGFP绿色荧光蛋白序列结合核糖体从而开始翻译进程,其自身携带的转录起始和终止密码子保证蛋白质完整结构的正常翻译。利用该技术实现对特定基因内源性表达示踪,具有其它外源示踪体系无法相比的高效和准确性,且原理简单,技术通用性强。该类细胞株在研究中方便直观,可实现动态性的检测活体细胞或组织中基因表达,这是传统技术研究方法的空白区。
4 结论本研究利用基因编辑CRISPR/Cas9技术在乳腺癌细胞系MCF-7中定点插入绿色荧光蛋白序列实现对FOXA2的示踪。实验从靶向位点序列的筛选验证、材料构建、细胞株制备及分选鉴定等多方面详细描述了研究过程。实验结果表明,经细胞模型EGF诱导验证,绿色荧光蛋白表达水平能够有效示踪FOXA2表达开启和关闭。
| [1] |
Jansen R, Embden JD, Gaastra W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Mol Microbiol, 2002, 43(6): 1565-1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [2] |
Andersson AF, Banfield JF. Virus population dynamics and acquired virus resistance in natural microbial communities[J]. Science, 2008, 320(5879): 1047-1050. DOI:10.1126/science.1157358 |
| [3] |
Bhaya D, Davison M, Barrangou R. CRISPR-Cas systems in bacteria and archaea:versatile small RNAs for adaptive defense and regulation[J]. Annu Rev Genet, 2011, 45: 273-297. DOI:10.1146/annurev-genet-110410-132430 |
| [4] |
Fineran PC, Dy RL. Gene regulation by engineered CRISPRCas systems[J]. Curr Opin Microbiol, 2014, 18: 83-89. DOI:10.1016/j.mib.2014.02.007 |
| [5] |
Makarova KS, Haft DH, Barrangou R, et al. Evolution and classification of the CRISPR-Cas systems[J]. Nature Reviews Microbiology, 2011, 9(6): 467-477. DOI:10.1038/nrmicro2577 |
| [6] |
Chylinski K, Makarova KS, Charpentier E, et al. Classification and evolution of type Ⅱ CRISPR-Cas systems[J]. Nucleic Acids Res, 2014, 42(10): 6091-6105. DOI:10.1093/nar/gku241 |
| [7] |
Wang H, Yang H, Shivalila CS, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas mediated genome engineering[J]. Cell, 2013, 153(4): 910-918. DOI:10.1016/j.cell.2013.04.025 |
| [8] |
Wan H, Feng C, Teng F, et al. One-step generation of p53 gene biallelic mutant Cynomolgus monkey via the CRISPR/Cas system[J]. Cell Res, 2015, 25(2): 258-261. DOI:10.1038/cr.2014.158 |
| [9] |
Johnson RA, Gurevich V, Filler S, et al. Comparative assessments of CRISPR-Cas nucleases'cleavage efficiency in planta[J]. Plant Mol Biol, 2015, 87(1-2): 143-156. DOI:10.1007/s11103-014-0266-x |
| [10] |
Endo M, Mikami M, Toki S. Multigene knockout utilizing offtarget mutations of the CRISPR/Cas9 system in rice[J]. Plant Cell Physiol, 2015, 56(1): 41-47. DOI:10.1093/pcp/pcu154 |
| [11] |
Jiang W, Zhou H, Bi H, et al. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis, tobacco, sorghum and rice[J]. Nucleic Acids Res, 2013, 41(20): e188. DOI:10.1093/nar/gkt780 |
| [12] |
Jiang Y, Chen B, Duan C, Sun B, et al. Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system[J]. Appl Environ Microbiol, 2015, 81(7): 2506-14. DOI:10.1128/AEM.04023-14 |
| [13] |
van Pijkeren JP, Britton RA. Precision genome engineering in lactic acid bacteria[J]. Microb Cell Fact, 2014, 13(Suppl1): S10. |
| [14] |
Yuen KS, Chan CP, Wong NH, et al. CRISPR/Cas9-mediated genome editing of Epstein-Barr virus in human cells[J]. J Gen Virol, 2015, 96(Pt 3): 626-636. |
| [15] |
Seeger C, Sohn J A. Targeting Hepatitis B Virus With CRISPR/Cas9[J]. Mol Ther Nucleic Acids, 2014, 3: e216. DOI:10.1038/mtna.2014.68 |
| [16] |
Tan Y, Adami G, Costa RH. Maintaining HNF6 Expression Prevents AdHNF3beta-Mediated Decrease in Hepatic Levels of Glut-2and Glycogen[J]. Hepatology, 2002, 35(4): 790-8. DOI:10.1053/jhep.2002.32482 |
| [17] |
Tang Y, Shu G, Yuan X, et al. FOXA2 functions as a suppressor of tumor metastasis by inhibition of epithelial-to-mesenchymal transition in human lung cancers[J]. Cell Res, 2011, 21(2): 316-26. DOI:10.1038/cr.2010.126 |
| [18] |
Zhang Z, Yang C, Gao W, et al. FOXA2 attenuates the epithelial to mesenchymal transition by regulating the transcription of E-cadherin and ZEB2 in human breast cancer[J]. Cancer Lett, 2015, 361(2): 240-50. DOI:10.1016/j.canlet.2015.03.008 |
| [19] |
Goldman O, Han S, Hamou W, et al. Endoderm generates endothelial cells during liver development[J]. Stem Cell Reports, 2014, 3(4): 556-65. DOI:10.1016/j.stemcr.2014.08.009 |
| [20] |
Alder O, Cullum R, Lee S, et al. Hippo signaling influences HNF4A and FOXA2 enhancer switching during hepatocyte differentiation[J]. Cell Rep, 2014, 9(1): 261-71. DOI:10.1016/j.celrep.2014.08.046 |
| [21] |
von Meyenn F, Porstmann T, Gasser E, et al. Glucagon-Induced Acetylation of Foxa2 Regulates Hepatic Lipid Metabolism[J]. Cell Metab, 2013, 17(3): 436-47. DOI:10.1016/j.cmet.2013.01.014 |
| [22] |
Cyranoski D. CRISPR gene-editing tested in a person for the first time[J]. Nature, 2016, 539(7630): 479. DOI:10.1038/nature.2016.20988 |
| [23] |
Korkmaz G, Lopes R, Ugalde AP, et al. Functional genetic screens for enhancer elements in the human genome using CRISPR-Cas9[J]. Nat Biotechnol, 2016, 34(2): 192-8. DOI:10.1038/nbt.3450 |