2. 深圳国家基因库合成与编辑平台,深圳 518083
2. Genome Synthesis and Editing Platform, China National GeneBank, Shenzhen 518083
小鼠免疫系统涉及种类众多、功能特性各异的多种类型细胞,揭示这些免疫细胞分化发育的机制是免疫学研究的核心工作之一。在小鼠中,造血干细胞(Hematopoietic stem cells,HSC)是其他血细胞的始祖,HSC通过分化发育可以建立整个血细胞系统(图 1-A)[1-2]。近年来单细胞转录组测序和CRISPR/Cas基因编辑技术快速发展,为免疫学研究提供了新方法。如能对分离的小鼠造血干细胞实施基因编辑,再通过体内移植或体外诱导促使其分化发育;通过细胞表面抗原分析或单细胞转录组分析,能快捷地研究各类免疫细胞分化发育的分子机制。为此,需要建立针对造血干细胞和其他分化度较低的前体免疫细胞的基因编辑技术方法。
|
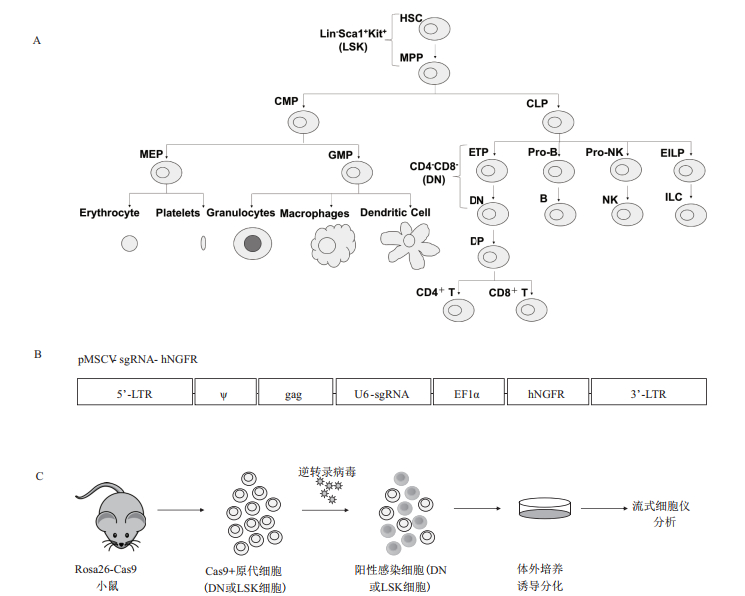
| 图 1 实验设计说明 A:造血干细胞分化发育简要示意图[1-2],相关细胞大小比例及形状不代表真实情况。造血干细胞分化后,主要形成两大分支:髓系细胞和淋巴系细胞(HSC:Hematopoietic stem cell,造血干细胞;MPP:Multipotent progenitor,多潜能祖细胞;CMP:Common myeloid progenitor,共同髓系前体细胞;CLP:Common lymphoid progenitor,共同淋巴系前体细胞;MEP:Megakaryocyte-erythroid progenitor,“巨核细胞/红细胞前体细胞;GMP:Granulocyte-macrophage progenitor,粒细胞/巨噬细胞前体细胞;DC:Dendritic cell,树突状细胞;ETP:Early T-cell Precursor,早期T细胞前体细胞;DN:CD4-CD8- Double negative cell,DN细胞;DP:CD4+CD8+ double positive T cell,DP细胞;EILP:Early innate lymphoid progenitor,固有淋巴样细胞前体细胞;ILC:Innate lymphoid cell,固有淋巴样细胞;NK:Natural killer cell,自然杀伤细胞);B:sgRNA载体结构示意图。sgRNA由U6启动子驱动表达,hNGFR报告基因由EF1α启动子驱动表达;C:实验操作方法示意图。首先从Cas9敲入小鼠体内分离得到DN细胞或LSK细胞,通过感染逆转录病毒获得阳性感染细胞。在体外培养并诱导分化后,使用流式细胞仪分析基因编辑结果 |
目前,针对人类CD34+造血干细胞(CD34+ Hematopoietic stem/progenitor cell,CD34+ HSPC)实施基因编辑的报道已有多项,但针对小鼠造血干细胞实施基因编辑的研究鲜有报道。人类对CD34+ HSPC的研究通常采用磁珠法,即从脐带血或外周血中分离造血干细胞,使用电转或病毒载体(如慢病毒载体或腺相关病毒载体)实施遗传转化,通过基因编辑实现基因治疗等多种目的[3-10]。在小鼠造血干细胞中实施基因编辑仅有少量报道。2014年,Heckl等[11]从小鼠骨髓中分离LSK细胞,并使用慢病毒载体实施基因编辑敲除了Smc3、Ezh2等基因;随后将LSK细胞移植到受体小鼠体内,在长期培养后构建了小鼠急性髓系白血病模型。2016年,Gundry等[4]从小鼠骨髓中分离到Kit+骨髓细胞,并通过电转实施了基因编辑。此外,有多项报道介绍了针对树突状细胞、B细胞、T细胞及巨噬细胞等分化度高的免疫细胞实施基因编辑的研究成果[12-13]。相关工作极大的拓展了CRISPR/Cas技术在免疫学研究中的应用。本研究从Cas9敲入小鼠骨髓中分离得到LSK细胞,并从其胸腺中分离得到DN细胞。LSK细胞和DN细胞都是包含多种细胞类型的混合细胞。LSK细胞包含造血干细胞HSC和轻度分化的MPP细胞等,其细胞表面分子特征为Lin-、Sca1+、Kit+。DN细胞包含ETP(DN1)细胞、DN2细胞、DN3细胞和DN4细胞。针对LSK细胞和DN细胞,用小鼠干细胞病毒(Mouse stem cell virus,MSCV)作为载体对Tcf7(其编码的蛋白为T cell factor 1,TCF1)、GFP和CD4基因实施了基因编辑。通过体外培养和诱导分化,LSK细胞和DN细胞被诱导分化为下游细胞,在分化的下游细胞中检测到基因编辑效果。
1 材料与方法 1.1 材料HEK293T细胞、OP9细胞、OP9-DL细胞和NIH3T3细胞均为本实验留存细胞。实验室所使用的OP9细胞稳定感染了含有GFP标记基因的逆转录病毒(MSCV)。而OP9-DL细胞也感染了逆转录病毒,但其中含有Notch信号配体和GFP标记基因。纯合子SpCas9敲入小鼠购自The Jackson Laboratory公司。pLenti-CRISPR v2载体购自Addgene公司(#52961)。pMSCV-IRES-hNGFR载体和pMSCV-IRES-Thy1.1载体为本实验室留存载体。流式细胞术抗体购自eBioscience公司或BioLegend公司。引物购自IDT公司。
1.2 方法 1.2.1 sgRNA载体构建选取4个基因Luciferase(Luc)、GFP、CD4和Tcf7,使用美国麻省理工学院在线CRISPR/Cas设计软件(http://crispr.mit.edu8079/?)设计相应的sgRNA(表 1)。按照pLenti-CRISPR v2载体操作说明[14-15],完成sgRNA的克隆构建工作。通过PCR方法从pLenti-CRISPR-sgRNA载体上克隆U6-sgRNA-EF1α基因片段,使用Bgl Ⅱ和Nco Ⅰ双酶切方法将该基因片段亚克隆至逆转录病毒载体pMSCV-hNGFR中,从而得到pMSCV-U6-sgRNA-EF1α-hNGFR载体。sgRNA载体上包含人hNGFR(human CD271,human nerve growth factor receptor)表面抗原报告基因,可以使用Anti-hNGFR抗体染色并检测(图 1-B)。使用无内毒素质粒大提试剂盒(QIAGEN)抽提质粒备用。
HEK293T细胞和NIH3T3细胞使用DMEM培养基(Thermo Fisher Scientific),培养基中额外加入10%胎牛血清(Atlanta Biologicals),Pen-Strep抗生素和L-Glutamine(Thermo Fisher Scientific)。DMEM培养基相关成分混合均匀后过滤除菌,置于4℃冰箱备用。OP9细胞和OP9-DL细胞使用MEM-α培养基(Thermo Fisher Scientific),培养基中额外加入10%胎牛血清,Pen-Strep抗生素和L-Glutamine。MEM-α培养基相关成分混合均匀后过滤除菌,置于4℃冰箱备用。
1.2.3 病毒包装及滴度测定第1天晚上,在10 cm培养皿中种上400万HEK293T细胞。第2天早上,使用Lipofectamine 3000(Thermo Fisher Scientific)进行转染。对于sgRNA病毒,每个10 cm平皿转染5 μg pMSCV-U6-sgRNA-EF1α-hNGFR质粒和5 μg逆转录病毒包装质粒(本实验室留存)。转染24 h后换液,加入5 mL新鲜配制的DMEM培养基。换液后作为零点开始计时,24 h后第1次收取病毒并加入5 mL新鲜培养基,48 h后第2次收取病毒。收集病毒后,立即使用Retro-X Concentrator(Clontech)对病毒进行浓缩处理,然后放入-80冰箱冻存。
病毒滴度测定。第1天晚上,在6孔板中种上NIH-3T3细胞,每孔20万个细胞。第2天使用“培养基-病毒混合液”替换旧培养基:对于每个孔,使用1 948 μL新鲜培养基,加入50 μL浓缩后的病毒和2 μL的Polybrene(终浓度为10 μg/mL)。混合均匀并连续培养48 h后,使用Anti-hNGFR抗体染色并经流式细胞仪分析确定病毒滴度。
1.2.4 OP9细胞中基因编辑在6孔板中种上10万个OP9细胞,加入Cas9病毒(本实验室留存)和GFP-sgRNA-1病毒。培养3 d后,取样品使用流式细胞仪分析OP9细胞中GFP表达量。培养7 d后,取样品使用流式细胞仪分析OP9细胞中GFP表达量。持续培养,并每隔3 d检测GFP表达状况变化。培养3周后,使用流式细胞仪将Thy1.1+hNGFR-(Cas9+sgRNA-),Thy1.1+hNGFR+(Cas9+sgRNA+),Thy1.1-hNGFR-(Cas9-sgRNA-)和Thy1.1-hNGFR+(Cas9-sgRNA+)4个细胞群体分选出来并单独培养。使用荧光显微镜观察4个细胞群体的GFP表达情况。抽提Thy1.1+hNGFR+(Cas9+sgRNA+)细胞的基因组DNA,克隆GFP片段并测序。
1.2.5 DN细胞基因编辑及检测取纯合子Cas9敲入小鼠(敲入位点为Rosa26,pCAG-hSpCas9-P2A-EGFP,后简称Rosa26-Cas9小鼠)[16],解剖提取其胸腺分离出胸腺中的细胞(GFP+)。使用Biotin标记的Anti-CD4抗体和Anti-CD8抗体(eBioscience)与胸腺细胞在冰上孵育20 min,之后使用BioMag® Streptavidin磁珠(QIAGEN)分离得到DN细胞。使用sgRNA(Luc-sgRNA-1,GFP-sgRNA-2,CD4-sgRNA-1)病毒感染DN细胞,而后37℃培养过夜。第2天早上除去病毒,并将DN细胞转移到预先辐射处理过(30-Gray)的OP9-DL细胞上进行共培养,在培养基中加入IL-7和Flt-3细胞因子(PEPROTECH),诱导DN细胞分化为下游T细胞。每隔3 d更换一半培养基并补充细胞因子。连续培养7 d后,随后进行流式细胞仪分析(图 1-C)。
1.2.6 LSK细胞基因编辑及检测取纯合子Cas9敲入小鼠,解剖并分离其骨髓。使用ACK细胞裂解液(ThermoFisher)裂解红细胞。加入Mouse-Rat血清,冰上孵育20 min。加入使用Biotin标记的Anti-Mac1、Anti-Ter119和Anti-Gr1抗体与骨髓细胞在冰上孵育30 min。之后使用PBS缓冲液将未结合的抗体洗掉。将骨髓细胞与BioMag® Streptavidin磁珠混合,在4℃冷房中上下旋转孵育30 min。使用磁铁将磁珠清除。使用Anti-Sca1,Anti-Kit及Anti-Linage Cocktail(Lin)抗体对分离的骨髓细胞进行染色,冰上孵育20 min后使用PBS缓冲液将未结合的抗体洗掉。使用流式细胞仪将Lin-Sca1+Kit+细胞分选出来,并立即感染sgRNA病毒(TCF1-sgRNA-1,TCF1-sgRNA-2,GFP-sgRNA-2)或者空载体病毒(MSCV-Thy1.1)。补充必要的血清、Pen-Strep双抗、L-Glutamine及细胞因子(IL3、IL6、Flt3和SCF,PEPROTECH),而后37℃培养过夜。第2天早上离心后除去病毒液,在MEM-α培养基中加入血清、Pen-Strep双抗、L-Glutamin及细胞因子(IL3、IL6、Flt3和SCF),而后37℃培养24 h。由于sgRNA病毒包含有hNGFR报告基因,而空载病毒包含Thy1.1报告基因,抗体孵育后使用流式细胞仪进行第2次分选,分别将hNGFR(sgRNA)阳性的细胞,以及Thy1.1(对照组,无sgRNA)阳性的细胞分选出来。将hNGFR+细胞和Thy1.1+细胞按照3:1的比例混合。之后,将混合的细胞转移到预先辐射(30 Gray)处理过的OP9-DL细胞上进行共培养,在培养基中加入IL-7和Flt-3细胞因子。将hNGFR+细胞和预先辐射(30 Gray)处理过的OP9细胞上进行共培养。每隔3 d更换一半培养基并补充相应的细胞因子,连续培养两周。之后,进行流式细胞仪分析(图 1-C)。
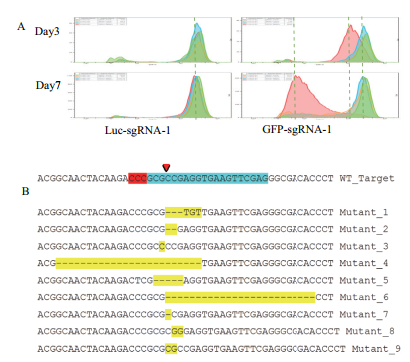
2 结果 2.1 MSCV-CRISPR系统测试为了检验新构建的基于MSCV载体的CRISPR/Cas系统是否能够进行基因编辑,选择GFP为靶标基因,在持续表达GFP的OP9细胞系中进行敲除GFP的测试。将Cas9病毒和GFP-sgRNA-1病毒共感染到OP9细胞,7 d后通过流式细胞仪分析,只有Thy1.1+hNGFR+(Cas9+sgRNA+)细胞群体的GFP强度显著下降(图 2-A)。使用流式细胞仪将Cas9+sgRNA+细胞群体分离出,克隆GFP靶点片段并测序,发现在靶点处有短的缺失或插入等变异(图 2-B)。这些结果表明,在OP9细胞中,由MSCV载体介导的基因编辑真实可信。
|
| 图 2 OP9细胞中基因编辑结果 A:流式细胞仪分析GFP表达强度变化的结果(Thy1.1+hNGFR-(Cas9+sgRNA-),Thy1.1+hNGFR+(Cas9+sgRNA+),Thy1.1-hNGFR-(Cas9-sgRNA-)和Thy1.1-hNGFR+(Cas9-sgRNA+)4个细胞群体GFP表达的检测结果被叠加在一起。其中Cas9+sgRNA+双阳性细胞群体为红色。左侧为对照组,感染了Luc-sgRNA-1病毒,在连续培养3 d和7 d后双阳性细胞GFP强度无显著变化。右侧为GFP敲除实验组,感染的GFP-sgRNA-1病毒,在连续培养7 d后双阳性细胞GFP强度显著下降);B:培养3周后,将Cas9+sgRNA+细胞群体sgRNA靶点区域DNA克隆测序,发现靶点处有短的缺失或插入突变(上方为野生型基因序列,其中蓝色高亮显示序列为靶点序列。红色三角为理论切割位点。下方为突变体克隆序列,黄色高亮显示部分为发生变异的序列) |
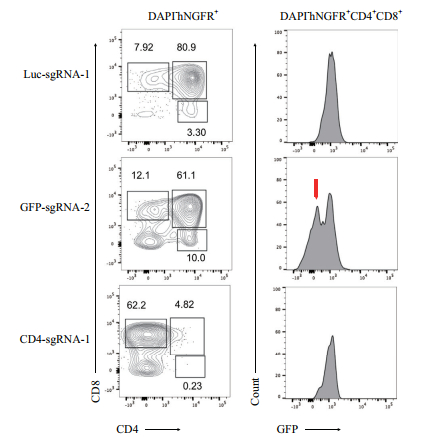
相对于从小鼠骨髓分离LSK细胞而言,从小鼠胸腺中分离DN细胞操作十分容易,且易于获取大量的细胞。因此,我们选用Cas9敲入小鼠,首先从中分离了DN细胞(GFP+)进行实验。分别感染了3种sgRNA(Luc-sgRNA-1,GFP-sgRNA-2,CD4-sgRNA-1)以靶向荧光素酶基因(Luc)、GFP和CD4。与Luc-sgRNA-1和CD4-sgRNA-1基因编辑结果比较发现,只有GFP-sgRNA-2基因编辑后出现GFP-细胞群体。与Luc-sgRNA-1和GFP-sgRNA-2基因编辑结果比较发现,只有CD4-sgRNA-1基因编辑后下游细胞中CD4+细胞比例大幅下降,同时CD8+细胞比例却大幅增加(图 3)。这一结果表明,使用Cas9敲入小鼠,以逆转录病毒作为载体,可以在小鼠原代DN细胞中实现基因编辑。
|
| 图 3 DN细胞体外基因编辑结果 在Rosa26-Cas9小鼠的DN细胞中实施基因编辑,通过诱导DN细胞分化并连续培养7 d后检测基因编辑结果。在CD4敲除实验中,与Luc-sgRNA-1和GFP-sgRNA-2组相比,CD4-sgRNA-1组中CD4+细胞几乎消失且CD4+CD8+细胞比例大幅度下降到4.82%,同时CD8+细胞比例却大幅上升到62.2%。而在GFP敲除实验中,与Luc-sgRNA-1和CD4-sgRNA-1组相比,GFP-sgRNA-2组中约有一半细胞变为GFP-细胞 |
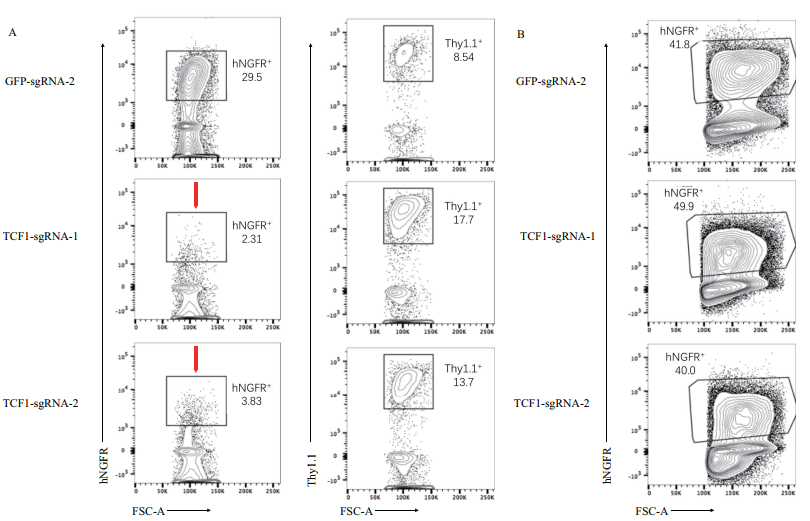
在上述实验基础上,针对LSK细胞进行了基因编辑测试。我们使用Cas9敲入小鼠进行相关实验,并通过流式细胞仪分选出LSK细胞。随后,分别感染了3种sgRNA(TCF1-sgRNA-1,TCF1-sgRNA-2,GFP-sgRNA-2)以靶向Tcf7和GFP。由于敲除TCF1后,LSK细胞不能正常分化为下游T细胞[17]。因此,我们加入了感染空病毒(MSCV-Thy1.1)的LSK细胞作为内参。体外培养时,使用3:1的比例进行共培养。在GFP-sgRNA-2基因编辑组中,经过2周体外培养后,hNGFR阳性细胞与Thy1.1细胞比例变为3.5:1(29.5%:8.54%)。这一比例与初始培养时3:1的比例接近。而在TCF1-sgRNA-1基因编辑组中,hNGFR阳性细胞与Thy1.1细胞比例变为1:7.7(2.31%:17.7%)。在TCF1-sgRNA-2基因编辑组中,hNGFR阳性细胞与Thy1.1细胞比例变为1:3.6(3.83%:13.7%)(图 4-A)。将感染了Tcf7基因sgRNA病毒的LSK细胞与OP9细胞共培养时,LSK细胞会发育为髓系免疫细胞。由于TCF1对于髓系免疫细胞发育非必需,因此当TCF1被敲除后,骨髓系免疫细胞发育不受影响[17]。在实验中,在GFP-sgRNA-2,TCF1-sgRNA-1,TCF1-sgRNA-2三个基因编辑组中,都有高比例的hNGFR阳性细胞(41.8%:49.9%:40.0%)(图 4-B)。这些hNGFR阳性细胞都有高比例的Mac1阳性细胞(结果未展示)。这表明,使用MSCV病毒为载体可以在Cas9敲入小鼠的LSK细胞中有效实施基因编辑。
|
| 图 4 LSK细胞体外基因编辑结果 A:Rosa26-Cas9小鼠的LSK细胞分离后感染MSCV病毒,将感染了sgRNA病毒的LSK细胞(hNGFR+)与转入空病毒的LSK细胞(Thy1.1+)按照3:1比例混合后与OP9-DL细胞共培养2周,诱导分化为下游T细胞(Thy1.2+CD25+)。左侧为每个混合样品中hNGFR+细胞的比例,右侧为每个混合样品中Thy1.1+细胞的比例。可以看到,在TCF1-sgRNA-1和TCF1-sgRNA-2基因编辑组中,hNGFR+细胞比例分别为2.31%和3.83%。由于流式细胞仪分选得到的细胞不纯,在长时间培养过程中会产生高比例细胞即非hNGFR+又非Thy1.1+;(B)Rosa26-Cas9小鼠的LSK细胞分离后感染sgRNA病毒,之后与OP9细胞共培养2周,诱导分化为下游骨髓系细胞(Mac1+)。在3组细胞中,有类似比例的hNGFR+细胞 |
本研究从Cas9敲入小鼠中分离相关造血干细胞和免疫前体细胞,通过逆转录病毒(MSCV)作为载体实施了基因编辑。在能够感染MSCV病毒的情况下,这一方法可能适用于大鼠等亲缘关系较近的哺乳动物。虽然使用MSCV病毒载体可以高效感染LSK和DN细胞,但病毒稳定整合到细胞基因组中,并持续不断地表达可能会对LSK细胞和DN细胞分化发育造成不利影响,并引发更多的脱靶效应。未来可以考虑构建可以被诱导表达的CRISPR/Cas系统,或者采用非整合病毒载体来介导实施基因编辑。此外,从Cas9敲入小鼠中分离相应细胞实施基因编辑,在一定程度上限制了这一方法的应用。未来可以构建同时含有Cas9和sgRNA表达元件的病毒载体,将有助于解决这一问题。
在LSK细胞中实施基因编辑并在体外诱导其分化,这一技术平台的建立有助于研究造血干细胞早期分化发育分子机制。由于造血干细胞在免疫系统中具有最高的分化潜能,在其中实施基因编辑,其下游细胞都会继承相同的遗传学特征。这使得我们在对特定基因实施敲除后,可以在更广的范围内研究该基因对免疫系统其他细胞分化发育的影响。本研究中只介绍了在体外环境中针对LSK和DN细胞实施基因编辑的研究结果,缺乏小鼠个体水平实验研究结果。如果能够将这一技术方法延伸,将相关基因编辑的LSK细胞移植到受体小鼠体内,并利用体内环境诱导编辑后的LSK细胞分化发育重建免疫系统,这将极大提升这一方法的价值和意义。此外,近年来单细胞组学技术快速发展,高通量单细胞转录组分析技术方法也成功建立[18-19]。综合运用这些技术方法将推动免疫细胞发育研究快速向前发展。
4 结论从Cas9敲入小鼠中分离原代LSK细胞或DN细胞,使用逆转录病毒作为载体将sgRNA表达元件导入到相应细胞中引发基因编辑。在DN细胞中成功敲除GFP和CD4表面抗原的表达;在LSK细胞中成功敲除T细胞分化关键因子TCF1的表达。
| [1] |
Blank U, Karlsson S. TGF-β signaling in the control of hematopoietic stem cells[J]. Blood, 2015, 125(23): 3542-3550. DOI:10.1182/blood-2014-12-618090 |
| [2] |
王振, 丁晓丹, 张岩, 等. 胚胎干细胞向造血细胞分化的研究进展[J]. 中国细胞生物学学报, 2013, 35(7): 907-915. DOI:10.11844/cjcb.2013.07.9002 |
| [3] |
Mandal PK, Ferreira LM, Collins R, et al. Efficient ablation of genes in human hematopoietic stem and effector cells using CRISPR/Cas9[J]. Cell Stem Cell, 2014, 15(5): 643-652. DOI:10.1016/j.stem.2014.10.004 |
| [4] |
Gundry MC, Brunetti L, Lin A, et al. Highly efficient genome editing of murine and human hematopoietic progenitor cells by CRISPR/Cas9[J]. Cell Rep, 2016, 17(5): 1453-1461. DOI:10.1016/j.celrep.2016.09.092 |
| [5] |
Dever DP, et al. CRISPR/Cas9 β-globin gene targeting in human haematopoietic stem cells[J]. Nature, 2016, 7629: 384-389. |
| [6] |
Hoban MD, Lumaquin D, Kuo CY, et al. CRISPR/Cas9-mediated correction of the sickle mutation in human CD34+ cells[J]. Mol Ther, 2016, 24(9): 1561-1569. DOI:10.1038/mt.2016.148 |
| [7] |
De Ravin SS, et al. CRISPR-Cas9 gene repair of hematopoietic stem cells from patients with X-linked chronic granulomatous disease[J]. Sci Transl Med, 2017, 9(372): pii: ecah3480. DOI:10.1126/scitranslmed.aah3480 |
| [8] |
Xu L, Yang H, Gao Y, et al. CRISPR/Cas9-mediated CCR5 ablation in human hematopoietic stem/progenitor cells confers HIV-1 resistance in vivo[J]. Mol Ther, 2017, 25(8): 1782-1789. DOI:10.1016/j.ymthe.2017.04.027 |
| [9] |
刘改改, 李爽, 韦余达, 等. 利用CRISPR/Cas9技术对人多能干细胞进行高效基因组编辑[J]. 遗传, 2015(11): 1167-1173. |
| [10] |
璩良, 李华善, 等. CRISPR/Cas9系统的分子机制及其在人类疾病基因治疗中的应用[J]. 遗传, 2015, 37(10): 974-982. |
| [11] |
Heckl D, et al. Generation of mouse models of myeloid malignancy with combinatorial genetic lesions using CRISPR-Cas9 genome editing[J]. Nat Biotechnol, 2014, 32(9): 941-946. DOI:10.1038/nbt.2951 |
| [12] |
Parnas O, Jovanovic M, Eisenhaure TM, et al. A genome-wide CRISPR screen in primary immune cells to dissect regulatory networks[J]. Cell, 2015, 162(3): 675-686. DOI:10.1016/j.cell.2015.06.059 |
| [13] |
Chu VT, Graf R, Wirtz T, et al. Efficient CRISPR-mediated mutagenesis in primary immune cells using CrispRGold and a C57BL/6 Cas9 transgenic mouse line[J]. Proc Natl Acad Sci USA, 2016, 113(44): 12514-12519. DOI:10.1073/pnas.1613884113 |
| [14] |
Sanjana NE, Shalem O, Zhang F. Improved vectors and genome-wide libraries for CRISPR screening[J]. Nat Methods, 2014, 11(8): 783-784. DOI:10.1038/nmeth.3047 |
| [15] |
Shalem O, et al. Genome-scale CRISPR-Cas9 knockout screening in human cells[J]. Science, 2014, 343(6166): 84-87. DOI:10.1126/science.1247005 |
| [16] |
Platt RJ, et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling[J]. Cell, 2014, 159(2): 440-455. DOI:10.1016/j.cell.2014.09.014 |
| [17] |
Weber BN, Chi AW, Chavez A, et al. A critical role for TCF-1 in T-lineage specification and differentiation[J]. Nature, 2011, 476(7358): 63-68. DOI:10.1038/nature10279 |
| [18] |
Papalexi E, Satija R. Single-cell RNA sequencing to explore immune cell heterogeneity[J]. Nat Rev Immunol, 2017. |
| [19] |
Stubbington MJT, Rozenblatt-Rosen O, Regev A, et al. Single cell transcriptomics to explore the immune system in health and disease[J]. Science, 2017, 358(6359): 58-63. DOI:10.1126/science.aan6828 |




