1987年,日本微生物学家石野良纯偶然间发现了细菌中存在某种异常重复序列[1],到2012年,Jinek等[2]首次在体外用sgRNA(single guide RNA)引导Cas9(CRISPR-associated,Cas)蛋白对DNA进行切割,证实采用规律成簇的间隔短回文重复(Clustered regularly interspaced short palindromic repeat,CRISPR)系统进行基因编辑的可行性,从此拉开了CRISPR/Cas9系统蓬勃发展的序幕。到今天为止,有关CRISPR/Cas9技术的研究越来越多,该系统的结构、原理、功能已经逐渐的清晰明了,其应用也不仅局限在微生物,适用范围已经扩展到植物细胞、动物细胞、动物基因组编辑等方面,而且在临床上也已经有了成功的案例。作为新一代的基因编辑工具,CRISPR/Cas9系统拥有其他编辑工具不可比拟的优势,其应用范围广,经济可行,容易操作,有望成为未来主要的基因编辑工具。
1 CRISPR系统研究进展 1.1 CRISPR系统的作用原理CRISPR系统源自于细菌、古细菌,是原核生物中用来防御质粒、病毒等核酸入侵而建立的自身免疫系统[3]。目前根据核酸剪切酶的功能和序列的不同将CRISPR系统分为不同亚型[4],其中CRISPR/Cas9系统是目前应用最为广泛的系统。在原核生物中,CRISPR/Cas9系统由CRISPR基因座、Cas9蛋白和tracrRNA(Transactivating crRNA)构成,其中CRISPR基因座由前导区序列(Leader)、串联重复序列(Repeats)和间隔序列(Spacers)组成[5]。前导区序列长300-500 bp,位于基因座上游,该序列没有开放阅读框,且在同一物种间高度保守[6];重复序列同样高度保守,具有稳定的茎-环结构,长度一般在23-50 bp[7];间隔序列是该系统中起关键作用的序列,它转录出一段crRNA(CRISPR-derived RNA),成熟的crRNA识别外源靶序列,引导具有切割活性的Cas9蛋白与外源靶序列结合发挥切割功能。tracrRNA是连接crRNA与Cas9蛋白的桥梁,使三者形成稳定的复合体。在CRISPR/Cas9系统中Cas9蛋白是CRISPR系统中具有切割酶活性的蛋白[8],它具有两个核酸酶结合域,分别切割两条DNA链,即HNH结构域切割外源序列中crRNA的互补链,RuvC结构域切割非互补链[9],形成DNA双链断裂(Double strand break,DSB)[2]。当外源核酸首次入侵时,细菌体识别外源核酸序列中一段特定的序列并整合为CRISPR基因座的间隔序列,当质粒或病毒再次入侵细菌时,该间隔序列便会转录出一段crRNA,同时tracrRNA也被转录出来,与crRNA形成双链RNA并连接到Cas9蛋白形成稳定的复合体,通过crRNA识别外源核酸引导Cas9蛋白将外源基因敲除。
随着CRISPR系统不断的研究发展,CRISPR/Cas9系统也被广泛应用于真核生物中。在真核细胞应用中,CRISPR/Cas9系统由两个关键元件组成,即识别靶基因的sgRNA与发挥切割酶活性的Cas9蛋白。sgRNA是由tracrRNA与gRNA结合成一条sgRNA,如图 1所示。在作用于基因时,首先sgRNA与靶基因互补配对引导Cas9蛋白定位到靶序列上,然后Cas9蛋白识别由NGG序列构成的前间区序列邻近基序(Protospacer adjacent motif,PAM),此时Cas9蛋白激活,将靶基因切断。在真核细胞内基因被Cas9蛋白破坏后会启动两种修复途径,即非同源末端连接(Non-homologous end joining,NHEJ)与同源重组(Homologous-directed repair,HDR)修复,NHEJ修复途径是直接将断裂的双链连接,这样的修复方式会造成碱基的缺失,破坏基因开放阅读框,使该基因发生移码或无义突变,导致基因失活,实现基因敲除;HDR修复途径则需要引入一条修复模板,该模板与DNA一条链连接,通过碱基互补配对便可以将模板整合到靶序列上,通过这种修复方式可以实现基因敲入[10]。目前CRISPR系统已经在斑马鱼、小鼠、猪、拟南芥等多个物种间实现了基因编辑[11-13]。
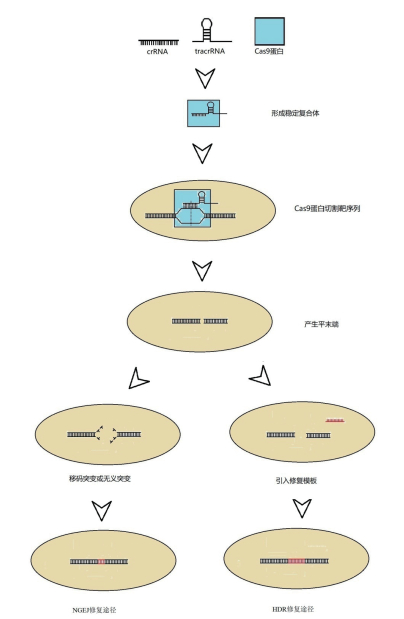
|
| 图 1 真核细胞中CRISPR/Cas9系统应用原理 |
利用CRISPR/Cas9系统可以实现对基因特异位点的切割,通过NHEJ修复途径实现对位点基因的敲除,效率很高,是CRISPR/Cas9系统作为基因修饰工具最广泛的应用。在实现基因敲除时只需要设计20 bp的sgRNA就可以实现对基因特异位点的切割,实现单基因敲除。此前利用单基因敲除已经实现对多个物种的基因编辑,包括哺乳动物如小鼠、大鼠、猪等,甚至在灵长类动物也有成功的报道[14]。
随着CRISPR/Cas9系统的迅速发展,学者发现该技术同样可以用于多基因的敲除,只需要同时设计多条sgRNA,结合Cas9蛋白作用于细胞就可以实现多基因敲除。Manjunath等[15]指出在治疗人类免疫缺陷病毒(HIV-1)感染时,利用CRISPR技术在体外将HIV-1的宿主细胞辅助受体CCR5或CXCR4基因敲除,再将CCR5或CXCR4基因敲除的造血干细胞回输给该患者,这些输送回体内的骨髓干细胞便可以分化产生抵抗HIV-1感染的CD4+T细胞。另外,Khalili等[16]也实现了另一个治疗方法:设计建靶向HIV-1的LTR基因sgRNA,建立稳定的sgRNA/Cas9细胞系,发现该细胞系可以对抗新的HIV-1感染,并且能够快速地阻止HIV-1基因整合到宿主基因组。这些CRISPR/Cas9系统的应用充分体现出它在基因敲除时发挥的重要作用以及为某些传染病、遗传病等疾病的治疗提供更加便捷的技术。
CRISPR/Cas9系统作为“全能”的基因编辑技术,在定点基因敲入方面也展现出巨大的应用前景。这种基因敲入的方式所利用的是同源重组(HDR)修复方式,同源重组在基因修复中的发生率很低,但通过CRISPR/Cas9技术诱发DNA双链断裂可以明显提高同源重组反应发生的概率,在Cas9蛋白切割的同时导入一段两侧与内源基因相似或相同的外源基因,便能够把该外源基因导入基因组。2013年,Mali等[17]利用改造的CRISPR/Cas9系统成功激活细胞HDR修复途径,实现外源基因的定点插入。之后Ma等[18]在Mecp2基因中敲入了两个loxp位点,成功构建了条件敲除大鼠。目前利用CRISPR/Cas9系统已经实现了细胞水平基因敲入,但动物水平的成功报导并不多,特别是猪等大型动物,对于基因敲入的转入效率有待进一步研究。
1.3 基于CRISPR系统的基因调控CRISPR/Cas9系统近几年备受关注,发展至今已形成诸多成熟的应用技术。CRISPR/Cas9系统介导的基因调控通过干预Cas9蛋白,使其两个核酸酶结构域失去切割酶的活性,这样的Cas9蛋白称为dCas9(dead Cas9)蛋白。虽然该蛋白不能切割DNA链,但仍然能够结合到靶位点,通过空间位阻效应阻碍RNA聚合酶的结合与延伸。Qi等[19]研究表明,采用dCas9系统在处理细胞时,dCas9蛋白与靶基因结合,可以阻碍靶序列的转录达到基因沉默的效果。在CRISPR/Cas9系统中,若设计的sgRNA特异性较弱容易发生错配,会使Cas9蛋白切割靶序列以外的基因,对基因组造成不可逆损伤。而dCas9蛋白并不发挥切割酶活性,不会影响基因组结构,并且在研究中发现dCas9蛋白与作用靶点结合的过程是可逆的,当移除dCas9蛋白后,靶序列的基因功能可以恢复[20]。
在真核细胞中转录调控需要多个调控因子共同作用,调节途径比较复杂,学者以tracrRNA的茎环或者dCas9蛋白为媒介连接转录激活因子或者转录抑制的功能蛋白,建立了CRISPR激活(CRISPRa)与CRISPR抑制(CRISPRi)。KRAB(Krüppel-assoc-iated box)是一种转录抑制因子[21],Parsi等[22]将dCas9蛋白与转录抑制因子KRAB连接,形成dCas9-KRAB复合体,成功应用在人体胚胎干细胞中抑制蛋白的表达。Maeder等[23]将dCas9蛋白与转录激活因子VP16连接,应用于人类细胞成功促进人类细胞VEGFA and NTF3基因的转录。如果将sgRNA作为一个基因定位的工具,dCas9蛋白作为连接蛋白的桥梁,便可以连接与基因相关的功能蛋白作用于目标序列,从而研究蛋白对基因调控的影响,有效地促进基因与蛋白相互关系的研究进展。
1.4 脱靶效应改善措施CRISPR/Cas9系统最大的不足便是脱靶效应(Off-target effects),指Cas9蛋白除了切割靶序列以外,还作用于与靶序列相似的其他序列。sgRNA定位靶序列时允许最多3个碱基的错配,甚至当序列长度不等于20 bp时也可能发生错配,其中PAM序列对碱基错配的影响最大,远PAM端的碱基突变比近PAM端的碱基突变更易发生碱基错配导致脱靶[24]。为了减少脱靶效应研究人员采取了很多改善方法,如双切酶(Double-nickase)措施策略。Trevino等[25]从Cas9蛋白入手,保持sgRNA功能结构不变,使Cas9蛋白的某一个核酸酶结构域失活。在切割DNA链时,Cas9蛋白只能在sgRNA互补的DNA链上产生缺口。在该系统中,需要在靶基因的两条链上分别设计sgRNA,且两个切割位点靠得越近,DNA双链断裂的机会越大。双切口酶策略需要两条sgRNA同时与靶序列碱基配对才能产生DNA双链断裂,这样便极大的增加了CRISPR/Cas9系统的特异性,减少脱靶效应的发生。另一种减少脱靶效应的策略是将dCas9蛋白与FokⅠ核酸酶结合,形成融合蛋白,利用FokⅠ核酸酶切割DNA链的特性,即只有当两个FokⅠ核酸酶靠近形成二聚体时,才会发挥切割酶的活性。在应用时,同样需要在靶序列附近设计一对sgRNA,sgRNA引导dCas9-FokⅠ融合蛋白在靶序列附近形成二聚体,切割双链DNA,实现基因敲除。只有当两条sgRNA分别与靶序列结合时才能发挥剪切酶的活性,极大的降低脱靶效应的发生,其效率可达90%以上[26-27]。
1.5 Cas9蛋白加工修饰CRISPR/Cas9系统主要由sgRNA与Cas9蛋白发挥作用,sgRNA作为精确的定位元件,保证该复合体准确定位到靶序列,Cas9蛋白作用于靶序列发挥功能蛋白的作用。学者将目光聚焦在Cas9蛋白上,通过替换Cas9蛋白或者加工修饰,从而优化CRISPR/Cas9系统。Zetsche等[28]将Cas9蛋白替换成Cpf1蛋白,CRISPR/Cpf1系统属于Ⅱ型CRISPR家族,Cpf1行使核酸酶活性只需要1条gRNA,并不需要tracrRNA的辅助,而且Cpf1切割DNA后形成5'黏性末端,更适用于基因修饰,目前Cpf1已广泛应用于在哺乳动物的基因修饰。CRISPR/saCas9系统则适用于腺相关(AAV)病毒,它与CRISPR/Cas9系统拥有不同的PAM序列,以腺病毒为载体转染细胞时采用saCas9蛋白切割靶序列效果较好[29]。CRISPR/Cas9系统采用sgRNA引导Cas9蛋白准确定位到靶序列上,实现对基因的修饰,这种特性使得该技术的开发与应用充满了无限的可能性,也为蛋白与基因的研究提供了更多的思路。
1.6 基于CRISPR/Cas9系统的临床应用CRISPR/Cas9系统作为第三代基因编辑工具,简洁的在线设计,简便的实验操作,作用于靶基因切割作用强,目前多个实验团队已经将其作为基因编辑工具应用于临床研究并取得成功。张锋团队开发出SHERLOCK(Specific high sensitivity enzymatic reporter UnLOCKing),它由gRNA与Cas13a蛋白组成,且有一个特点即当Cas13a蛋白切开特异RNA后,就开始疯狂切开它能遇到的任何RNA,利用这一特性。研究人员将该系统用于核酸检测,在不同的病毒上测试了它的特异性,灵敏度可达阿摩尔级。之后研究人员又把该系统应用于检测癌症突变,该系统同样以阿摩尔级的灵敏度识别出肿瘤的特有突变[30]。利用该系统的高灵敏度,鉴定人类基因组中多个单核苷酸多态性以及对于各种疾病病原体的检测等都会有很高的应用价值。该系统无论对于科学还是临床医学都是很强大的工具,其研究与应用领域非常广泛,不仅对临床多种疾病的诊断会有很大的帮助,而且会在药物的开发中发挥重要作用。
2 CRISPR系统在小鼠中的应用2013年,张锋等(Wang)[13]利用CRISPR/Cas9系统首次实现对小鼠基因的编辑,并且是多基因同时敲除。从此开启了采用CRISPR/Cas9定点编辑基因技术作用于小鼠基因组的研究。之后沈彬通过在Cas9和核定位序列(Nuclear localization sequence,NLS)之间加一个32个氨基酸的Linker,解决了Cas9在哺乳动物细胞中入核难的问题,并在EGFP基因上设计靶点,共注射Cas9和sgRNA后成功敲除了小鼠的EGFP基因[31]。利用CRISPR/Cas9系统进行单/多基因位点同时敲除获得突变小鼠,为利用CRISPR/Cas9技术应用于小鼠的研究奠定了基础。
2.1 建立动物模型目前小鼠已成为各个实验室哺乳动物研究的首选物种,基因突变小鼠作为遗传学研究的重要工具,在基因功能和遗传病的研究中扮演重要角色。之前通过靶向基因插入的方法获取转基因动物模型费时费力,很大程度上限制了遗传学研究的发展。利用CRISPR/Cas9技术构建基因突变小鼠模型操作简便,耗时短,极大地加快了遗传学研究的进展[13]。癌症是目前医学面临的重要难题,癌症基因组项目已证实癌症的发生伴随着癌症相关基因的突变。在CRISPR/Cas9技术出现之前要获得突变模型需要耗费大量的时间,致使癌症基因的研究缓慢。Xue等和Lawrence利用CRISPR/Cas9技术针对肝脏的肿瘤抑制基因p10和p53,构建该目的基因的CRISPR/Cas9的表达质粒,利用立体动力注入法将质粒传递到肝脏组织,成功抑制了抑癌基因的表达,为肝癌研究奠定了基础[32-33]。另外染色体重排也是引起癌症的又一个主要原因[34],Maddalo等[35]采用CRISPR/Cas9技术,设计克隆的亚基因组靶向EML4和ALK位点sgRNA/Cas9,用腺病毒作为载体,高效感染成年小鼠肺上皮细胞,生成EML4-ALK肺癌小鼠模型,为人类癌症基因工程小鼠癌症模型的构建提供了新的技术,为剖析相关肿瘤发生的分子机制,研究药物的敏感性和耐药性提供了强大的基础平台。此外,汪启翰等[36]也利用CRISPR/Cas9技术构建血友病小鼠模型,在小鼠FⅨ基因设计靶位点sgRNA,并与体外转录Cas9mRNA同时显微注射到C57BL/6品系小鼠的受精卵,获得突变小鼠,突变率高达85%,且突变具有可遗传性,并证实CRISPR/Cas9系统能够高效地进行基因编辑。
2.2 疾病治疗为了研究这项技术在疾病治疗中是否可以发挥作用,Wu等[37]选择小鼠白内障遗传疾病模型的突变基因,设计了该基因的sgRNA,与Cas9共注射到突变小鼠的受精卵中,在F1代小鼠的结果中发现l/3的小鼠的白内障症状得到治愈,并且可以稳定传递给下一代,说明CRISPR/Cas9技术对小鼠白内障的治疗有效。该技术还被应用于病毒感染性疾病的治疗:全球超过两亿多的乙肝病毒感染者,由于缺乏治疗严重影响了这些人的生活。目前临床上采用清除感染者肝细胞内的HBV共价闭合环状DNA(cccDNA)作为治愈慢性乙型肝炎的关键。Kennedy等[38]采用CRISPR/Cas9技术,设计靶向Cas9/sgRNA复合体作用HBV感染的细胞,结果发现该复合体有效地抑制HBVcccDNA的形成、降低乙肝表面抗原分泌、使乙型肝炎病毒突变失活以及减少抗原的分泌和病毒的复制和感染,导致cccDNA HepaRG细胞退化。
3 展望CRISPR系统作为第三代基因编辑工具,从DNA识别到实现靶序列敲除与敲入,从作用原理到应用,从实验设计到敲除效率均要优于锌指核酸酶(Zinc finger nucleases,ZFNs)和转录激活因子样效应物核酸酶(Transcription activator-like effector nucleases,TALEN)技术,有望成为未来的主要基因编辑工具,正如该技术开发人员所说:“CRISPR技术的应用仅仅受我们想象力的约束。”
该技术最大的缺点就是脱靶效应,对于不同的物种,脱靶率也不一样,需要找出导致脱靶的原因,如何让Cas9蛋白准确的切割靶序列,减少脱靶效率是该技术应用面临的主要难题。CRISPR系统作为一种基因编辑的工具,对于其机制的研究正在不断的深入,应用范围也在不断的扩展,包括CRISPR/C2c2、SGN等工具都是适应不同的实验条件发展而来的基因编辑技术。利用CRISPR系统基因编辑的强大技术,可以为基因的功能、调控方式,以及对于蛋白质作用机制的研究提供更加便捷的手段。目前,对于CRISPR系统的机制研究目前仍处于基础阶段,应用范围也有待不断深入。例如,Type Ⅰ CRISPR系统与Type Ⅲ CRISPR系统的作用机制理论报导并不多,另外利用CRISPR系统基因编辑的功能应用于基因改造的研究也处于起步阶段,如何更好的发挥CRISPR系统的作用还有待进一步的研究,以及对于目前临床难以治愈的遗传性疾病、病毒性疾病等方面探索新的治疗方法,实现CRISPR系统从实验室到临床应用,目前的报导并不多,这也有待于学者进一步的改进CRISPR系统的安全性与实用性。总之,CRISPR系统目前已经在分子生物学领域发挥了巨大的作用,但是CRISPR系统的应用还需不断地深入探索,其潜力仍需进一步的开发。
| [1] |
Ishino Y, Shinagawa H, Makino K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product[J]. J Bacteriol, 1987, 169(12): 5429-5433. DOI:10.1128/jb.169.12.5429-5433.1987 |
| [2] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [3] |
Mojica FJ, Diez-Villasenor C, Garcia-Martinez J, et al. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements[J]. J Mol Evol, 2005, 60(2): 174-182. DOI:10.1007/s00239-004-0046-3 |
| [4] |
Makarova KS, Haft DH, Barrangou R, et al. Evolution and classification of the CRISPR-Cas systems[J]. Nat Rev Microbiol, 2011, 9(6): 467-477. DOI:10.1038/nrmicro2577 |
| [5] |
Jansen R, Embden JD, Gaastra W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Mol Microbiol, 2002, 43(6): 1565-1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [6] |
Alkhnbashi OS, Shah SA, Garrett RA, et al. Characterizing leader sequences of CRISPR loci[J]. Bioinformatics, 2016, 32(17): i576-i585. DOI:10.1093/bioinformatics/btw454 |
| [7] |
Lander ES. The Heroes of CRISPR[J]. Cell, 2016, 164(1-2): 18-28. DOI:10.1016/j.cell.2015.12.041 |
| [8] |
Haurwitz RE, Jinek M, Wiedenheft B, et al. Sequence- and structure-specific RNA processing by a CRISPR endonuclease[J]. Science, 2010, 329(5997): 1355-1358. DOI:10.1126/science.1192272 |
| [9] |
Nishimasu H, Ran FA, Hsu PD, et al. Crystal structure of Cas9 in complex with guide RNA and target DNA[J]. Cell, 2014, 156(5): 935-949. DOI:10.1016/j.cell.2014.02.001 |
| [10] |
Sander JD, Joung JK. CRISPR-Cas systems for editing, regulating and targeting genomes[J]. Nat Biotechnol, 2014, 32(4): 347-355. DOI:10.1038/nbt.2842 |
| [11] |
Hwang WY, Fu Y, Reyon D, et al. Efficient genome editing in zebrafish using a CRISPR-Cas system[J]. Nat Biotechnol, 2013, 31(3): 227-229. DOI:10.1038/nbt.2501 |
| [12] |
Li JF, Norville JE, Aach J, et al. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9[J]. Nat Biotechnol, 2013, 31(8): 688-691. DOI:10.1038/nbt.2654 |
| [13] |
Wang H, Yang H, Shivalila CS, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering[J]. Cell, 2013, 153(4): 910-918. DOI:10.1016/j.cell.2013.04.025 |
| [14] |
Niu Y, Shen B, Cui Y, et al. Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos[J]. Cell, 2014, 156(4): 836-843. DOI:10.1016/j.cell.2014.01.027 |
| [15] |
Manjunath N, Yi G, Dang Y, et al. Newer gene editing technologies toward HIV gene therapy[J]. Viruses, 2013, 5(11): 2748-2766. DOI:10.3390/v5112748 |
| [16] |
Khalili K, Kaminski R, Gordon J, et al. Genome editing strategies: potential tools for eradicating HIV-1/AIDS[J]. J Neurovirol, 2015, 21(3): 310-321. DOI:10.1007/s13365-014-0308-9 |
| [17] |
Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [18] |
Ma Y, Zhang X, Shen B, et al. Generating rats with conditional alleles using CRISPR/Cas9[J]. Cell Res, 2014, 24(1): 122-125. DOI:10.1038/cr.2013.157 |
| [19] |
Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022 |
| [20] |
Gilbert LA, Larson MH, Morsut L, et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes[J]. Cell, 2013, 154(2): 442-451. DOI:10.1016/j.cell.2013.06.044 |
| [21] |
Margolin JF, Friedman JR, Meyer WK, et al. Kruppel-associated boxes are potent transcriptional repression domains[J]. Proc Natl Acad Sci USA, 1994, 91(10): 4509-4513. DOI:10.1073/pnas.91.10.4509 |
| [22] |
Parsi KM, Hennessy E, Kearns N, et al. Using an inducible CRISPR-dCas9-KRAB effector system to dissect transcriptional regulation in human embryonic stem cells[J]. Methods Mol Biol, 2017, 1507: 221-233. DOI:10.1007/978-1-4939-6518-2 |
| [23] |
Maeder ML, Linder SJ, Cascio VM, et al. CRISPR RNA-guided activation of endogenous human genes[J]. Nat Methods, 2013, 10(10): 977-979. DOI:10.1038/nmeth.2598 |
| [24] |
Hsu PD, Scott DA, Weinstein JA, et al. DNA targeting specificity of RNA-guided Cas9 nucleases[J]. Nat Biotechnol, 2013, 31(9): 827-832. DOI:10.1038/nbt.2647 |
| [25] |
Trevino AE, Zhang F. Genome editing using Cas9 nickases[J]. Methods Enzymol, 2014, 546: 161-174. DOI:10.1016/B978-0-12-801185-0.00008-8 |
| [26] |
Tsai SQ, Wyvekens N, Khayter C, et al. Dimeric CRISPR RNA-guided FokⅠ nucleases for highly specific genome editing[J]. Nat Biotechnol, 2014, 32(6): 569-576. DOI:10.1038/nbt.2908 |
| [27] |
Guilinger JP, Thompson DB, Liu DR. Fusion of catalytically inactive Cas9 to FokⅠ nuclease improves the specificity of genome modification[J]. Nat Biotechnol, 2014, 32(6): 577-582. DOI:10.1038/nbt.2909 |
| [28] |
Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system[J]. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038 |
| [29] |
Ran FA, Cong L, Yan WX, et al. In vivo genome editing using Staphylococcus aureus Cas9[J]. Nature, 2015, 520(7546): 186-191. DOI:10.1038/nature14299 |
| [30] |
Gootenberg JS, Abudayyeh OO. Nucleic acid detection with CRISPR-Cas13a/C2c2[J], 2017, 356(6336): 438-442. http://europepmc.org/abstract/MED/28408723
|
| [31] |
沈彬. 利用CRISPR/Cas9进行基因编辑[D]. 南京: 南京大学, 2014. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2949519
|
| [32] |
Xue W, Chen S, Yin H, et al. CRISPR-mediated direct mutation of cancer genes in the mouse liver[J]. Nature, 2014, 514(7522): 380-384. DOI:10.1038/nature13589 |
| [33] |
Lawrence MS, Stojanov P, Mermel CH, et al. Discovery and saturation analysis of cancer genes across 21 tumour types[J]. Nature, 2014, 505(7484): 495-501. DOI:10.1038/nature12912 |
| [34] |
Blasco RB, Karaca E, Ambrogio C, et al. Simple and rapid in vivo generation of chromosomal rearrangements using CRISPR/Cas9 technology[J]. Cell Rep, 2014, 9(4): 1219-1227. DOI:10.1016/j.celrep.2014.10.051 |
| [35] |
Maddalo D, Manchado E, Concepcion C P, et al. In vivo engineering of oncogenic chromosomal rearrangements with the CRISPR/Cas9 system[J]. Nature, 2014, 516(7531): 423-427. DOI:10.1038/nature13902 |
| [36] |
汪启翰, 怀聪, 孙瑞林, 等. 利用CRISPR/Cas系统快速高效构建血友病乙小鼠模型[J]. 遗传, 2015(11): 1143-1148. |
| [37] |
Wu Y, Liang D, Wang Y, et al. Correction of a genetic disease in mouse via use of CRISPR-Cas9[J]. Cell Stem Cell, 2013, 13(6): 659-662. DOI:10.1016/j.stem.2013.10.016 |
| [38] |
Kennedy EM, Bassit LC, Mueller H, et al. Suppression of hepatitis B virus DNA accumulation in chronically infected cells using a bacterial CRISPR/Cas RNA-guided DNA endonuclease[J]. Virology, 2015, 476: 196-205. DOI:10.1016/j.virol.2014.12.001 |




