2. 湖北天勤生物科技有限公司,武汉 430064
2. Hubei Topgene Biotechnology Co., Ltd., Wuhan 430064
随着人类医学研究的发展,更广泛深入地了解疾病病理并确定治疗策略已非常迫切。建立动物模型是现代医学领域研究人类疾病的重要方法,选定动物与人类之间的物种差异直接决定了该动物模型的有效性。迄今为止,人类在小鼠等啮齿类动物模型的构建上做了大量研究[1-2],但不管在生理状况还是遗传调控方面,啮齿类动物与人类都存在较大差异,这也极大程度限制了小鼠等动物在人类疾病动物模型中的应用。与其它动物相比,非人灵长类动物(Nonhuman primate,NHP)在进化发育、生理生化及病理方面和人类最为接近。2011年,中国科学家完成了食蟹猴和恒河猴基因组的比较分析[3],猕猴与人类基因组同源性高达93%[4]。由此可见,非人灵长类动物是研究人类疾病的理想动物模型。
模仿人类疾病的非人灵长类动物模型很难以自发突变的方式获得。因此,通过基因编辑的方法获得携带人类致病基因的动物模型对研究人类疾病非常关键。传统的转基因方法不仅效率低,且需要大量胚胎干细胞,费时费力。由于非人灵长类动物较长的性成熟周期和较低的生育率,利用传统的转基因方法对其进行基因修饰难度极大,相关模型的构建进展非常缓慢。CRISPR/Cas9是继ZFNs和TALENs技术之后迅速发展起来的第三代基因组编辑技术,其成本低、制作简单、效率更高,迅速成为了各研究领域有效的基因编辑工具。随着应用的深入,CRISPR/Cas9的脱靶效应、嵌合突变等突出问题也受到研究者的重视,并做了一系列改进。本文就基因编辑技术在非人灵长类动物中的应用研究、存在的突出问题及应对策略作简要概述,为进一步研究人类疾病非人灵长类动物模型建立提供借鉴与参考。
1 传统的转基因方法和TALENs基因编辑技术在非人灵长类动物中的应用 1.1 传统的转基因方法建立非人灵长类动物模型构建转基因灵长类动物的方法主要利用逆转录病毒载体法,其原理是将目的基因重组到逆转录病毒载体上,通过感染宿主细胞,将外源目的基因整合到宿主基因组中。自2001年美国科学家利用逆转录病毒载体法获得世界上第一只携带绿色荧光蛋白的转基因非人灵长类动物ANDi以来[5],研究者们通过慢病毒法建立了多个模仿人类神经疾病的非人灵长类动物模型。所获得的转基因猴模型,都表现出所模拟的人类疾病相关主要临床症状[6-8]。在第一代转基因非人灵长类动物的基础上,还孕育出第二代携带相同基因修饰并具有同样临床症状的动物模型,进一步证明这种遗传改造可以实现生殖系传递[8-9]。
虽然研究者通过逆转录病毒载体法在非人灵长类动物模型构建中取得了一定进展,但是这些基因修饰位点都是随机的,具有不确定性,无法获得精确基因修饰的非人灵长类动物模型。转基因方法较低的效率也制约了其应用,虽然Niu等[10]利用以猴免疫缺陷病毒(SⅣ)为基础的慢病毒载体成功构建了EGFP转基因恒河猴,转化效率比之前的报道有较大提高,但在生长周期较长的非人灵长类动物中应用进展仍然非常缓慢,研究者迫切需要更精确高效的基因编辑技术来进行相关研究。
1.2 ZFNs和TALENs基因编辑技术建立非人灵长类动物模型锌指核酸内切酶(ZFNs)在结构上包括锌指蛋白(ZFP)结构域和Fok Ⅰ切割结构域[11],类转录激活效应因子核酸酶(TALENs)是由转录激活因子样效应物(TALE)代替锌指作为DNA结合域与FokⅠ切割域组成的基因编辑工具[12]。ZFNs和TALENs分别通过锌指蛋白和类转录激活因子效应物识别DNA序列,并在特定位点对DNA进行切割,形成双链断裂,随后诱导细胞内的修复机制,在非同源末端连接(Non-homologous end joining,NHEJ)或同源重组(Homologous Recombination,HR)修复机制下实现基因敲除或敲入。
构建和设计ZFNs花费时间较长,成本很高,而且脱靶效应非常严重。至今尚没有ZFNs在非人灵长类动物中研究应用的报道。相对于ZFNs,TALENs技术设计相对简单,成本较低,而且DNA序列设计特异性高,脱靶效应较低。2014年,中国科学家Liu等[13]首次利用TALENs技术,获得了可导致雷特综合征(Rett syndrome,RTT)的MECP2突变基因的雌性食蟹猴。RTT食蟹猴与RTT病人在基因型和表型上都有很多相似性,对人类研究RTT的发病机制和治疗方法有非常重要的意义[14]。常染色体隐性遗传性小头畸形(Microcephaly,MCPH)是一种神经系统发育障碍疾病,Ke等[15]利用TALENs技术,构建了携带双等位基因MCPH1突变的食蟹猴,并表现出小头畸形病的临床特征。为更好地研究MCPH1致病机制提供了有效的动物模型。
TALENs技术已经在实际研究中有了较多报道。但是,TALENs基因编辑技术针对不同靶位点需要设计不同的识别蛋白,涉及巨大的蛋白改造工作,耗时耗力且成本高,对多基因位点打靶更是极不便利。这些局限性都严重制约了它的广泛应用。
2 CRISPR/Cas9基因编辑技术在非人灵长类动物中的应用CRISPR/Cas系统作为大多数细菌和古菌的获得性免疫系统,由规律成簇间隔短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR)和相关的Cas蛋白组成,通过RNA介导,利用短的非编码RNA(crRNA),指导Cas内切酶蛋白到达目的位点,对靶基因进行特异性切割[16]。随着对系统功能和机制的深入了解,CRISPR/Cas系统被分为两个大类(class1和class2),进而又可分为6个类型,共16个亚型[17-18]。class1包括Ⅰ、Ⅲ和Ⅳ三个类型,这类CRISPR/Cas系统利用多个蛋白组成的复合效应体对靶基因进行干扰,干扰机制比较复杂。class2系统则是利用具有多结构域的效应蛋白单体干扰靶基因,包括Ⅱ、Ⅴ和Ⅵ三个类型。其中,Ⅱ型CRISPR/Cas系统典型的效应蛋白为Cas9,其切割位点位于靶序列3'端PAM序列上游3 bp,形成平末端的DNA双链断裂。Ⅴ型CRISPR/Cas系统的效应蛋白为Cpf1,它不需要反式激活crRNA便可实现靶位点切割,切割位点可形成5'突出的粘性末端[19-20],Cpf1蛋白在基因编辑研究中也极具应用潜力。Ⅵ型CRISPR/Cas系统的效应蛋白为C2c2,它具有RNA指导的单链RNA酶切活性,为RNA编辑提供了新工具[18, 21]。
Ⅱ型CRISPR/Cas9系统发现较早,机制简单且易于操作,是目前机制研究最清楚和应用最广泛的一套体系[22-24]。CRISPR/Cas9基因编辑方法为人类进一步研究非人灵长类动物模型建立,找到治疗人类遗传性疾病更好更有效的治疗方法提供了技术支持。
2.1 非人灵长类动物模型多基因敲除的实现已报道的基因编辑技术在非人灵长类动物中的研究,都只实现了单基因敲除。2014年2月,来自云南省灵长类生物医学重点实验室、南京大学模式动物中心和南京医科大学的研究人员利用CRISPR/Cas9技术,首次获得了携带定向突变基因的孪生食蟹猴,并同时实现了两个基因(Ppar-γ和Rag1)的敲除。在随后对新生猴DNA样本分析检测中未发现脱靶效应[25]。同年12月,该联合实验室在对所获基因工程猴的后续研究中发现,靶向基因突变性状在食蟹猴的各个组织细胞中均有表达,包括生殖系细胞,基因修饰极有可能通过生殖细胞遗传到下一代[26]。这项研究的突破,为以后构建模仿多基因突变疾病的非人灵长类动物模型奠定了技术基础。
2.2 非人灵长类动物疾病模型的建立CRISPR/Cas9技术自2013年问世以来,研究者已经建立了多个模仿人类疾病的非人灵长类动物中模型,研究速度大幅提升。杜氏肌萎缩症疾病(Duchenne muscular dystrophy,DMD)是一种X染色体连锁隐性遗传疾病,因肌蛋白dystrophin的基因突变造成其功能丧失而引起。发病率为活产男婴的1 /30 000。2015年,Chen等[27]使用CRISPR/Cas9技术靶向突变猕猴的肌营养不良蛋白基因,分析因难产而死亡猴的肌肉组织发现,dystrophin蛋白表达明显下降或消失,并表现出类似于DMD病人早期出现的肌肉细胞退化的病理特征。对所获基因编辑恒河猴模型DNA样本分析检测中未发现明显的脱靶效应。同年,Kang等[28]利用CRISPR/Cas9技术获得了DAX1基因缺失的食蟹猴模型,DAX1突变可引发X连锁先天性肾上腺发育不全(Adrenal hypoplasia congenita,AHC)和促性腺激素分泌不足的性腺功能低下症(Hypogonadolropic hypogonadism,HH),这只DAX1基因突变猴表现出肾上腺发育缺陷和异常的睾丸结构的临床表征与AHC-HH患者非常相似。Wan等[29]优化了CRISPR/Cas9技术,不经过种系交配而一步直接获得p53(肿瘤抑制基因)突变的食蟹猴模型。Midic等[30]获得了导致β地中海贫血症的珠蛋白HBB突变猕猴模型,经分析并未发现脱靶效应(表 1)。
CRISPR/Cas9技术凭借其高效准确且设计操作简便等诸多优点,已成为近几年最受关注的基因编辑技术。虽然CRISPR/Cas9系统至今才兴起短短几年时间,却已经取得了很多划时代的突破性进展。随着对CRISPR/Cas9系统了解不断深入,一些突出问题引起了研究者的重视。
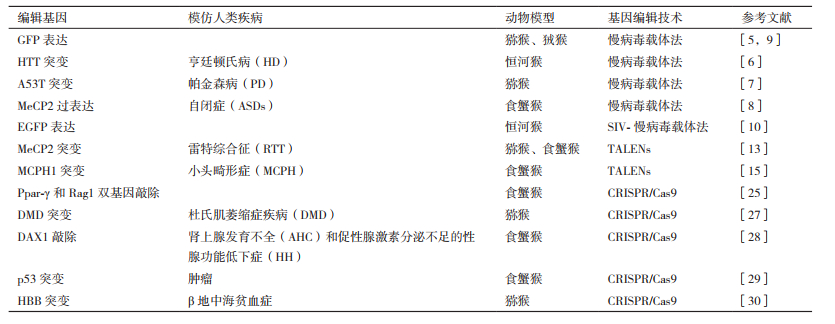
3.1 CRISPR/Cas9脱靶效应可能引入非预期突变2014年初,Nature Methods杂志将TALEN和CRIS-PR/Cas9技术评为2014年值得关注技术,同时也明确指出了CRISPR/Cas9的脱靶现象[31]。多个研究组的研究也证实了CRISPR/Cas9系统有着较高的脱靶活性[32-34]。虽然CRISPR/Cas9系统在非人灵长类动物的应用研究中尚未发现脱靶效应,但是利用CRISPR/Cas9系统对人类三核胚胎进行人类珠蛋白HBB基因编辑的研究中却发现有脱靶效应的存在[35]。这不得不让我们反思CRISPR/Cas9系统未在非人灵长类动物中发生脱靶效应的原因,可能我们对猴类基因组的研究还不够深入导致。但是建立非人灵长类动物模型研究人类疾病,脱靶效应是亟待解决的问题。为了提高CRISPR/Cas9系统的打靶效应,研究者做了大量研究工作,可以指导我们建立更加精准打靶的非人灵长类动物模型(图 1)。
|
| 图 1 提高CRISPR/Cas9系统在非人灵长类动物模型中打靶效应的优化策略 |
Ran等[36]通过分别突变Cas9蛋白的两个核酸酶结构域关键催化残基D10A或H840A发现,Cas9切口酶突变体D10A Cas9,配合一对gRNA,在目标基因位置配对切口,双切口使脱靶效应降低了50-1 500倍。尽管引入一对Cas9双切口酶比野生型Cas9蛋白的靶点切割特效性更高,但是仍然存在着脱靶现象。在此基础上,将D10A和H840A两个催化残基同时突变,获得失去切割活性但仍保留DNA结合活性的dCas9,将dCas9与非特异性的核算内切酶FokⅠ形成融合蛋白(dCas9-FokⅠ)。只有当两个融合蛋白单体彼此靠近形成二聚体时才能对靶基因进行切割,大大降低了脱靶效应[37-38]。张锋团队[39]对Cas9蛋白结构分析,Cas9酶约有1 400个氨基酸,用丙氨酸替代其中3个氨基酸位点,获得的增强型Cas9(eSpCas9)有精确的靶向功能,脱靶效应减少到无法检测的水平。Joung团队[40]替换Cas9蛋白4条链,获得高保真型Cas9(SpCas9-HF1)。该突变核酸酶与非特异性序列结合力更弱,增强靶向序列与Cas9蛋白结合的竞争力,可以将超过85%的靶序列切割,但是却检测不到非特异性序列切割。
3.1.2 Cas9蛋白表达调控优化Cas9蛋白在体内长时间的表达和持续活性会增大脱靶效应的风险。研究者利用Cas9蛋白的“调控开关”,有目的性地开启和关闭Cas9表达,降低脱靶效应。Tan等[41]将Cas9和雌激素受体结合域ERT2融合(Cas9-ERT2),通过4-羟基他莫昔芬(4-HT)化合物诱导,可逆调控Cas9蛋白表达。同样,将Cas9蛋白分成两个失活片段paCas9,每个失活片段融合Magnet蛋白。蓝光的照射或切断,能将Magnet蛋白结合或分开,从而可逆地调控Cas9蛋白活性[42]。Rauch等[43]发现了Cas9的抑制蛋白AcrⅡA2和AcrⅡA4。AcrⅡA4能够阻断Cas9酶活性,减少了Cas9在细胞中长时间的持续活性带来的脱靶效应,使脱靶发生率降低了4倍,而靶位点的基因编辑未受到影响。
3.1.3 gRNA核苷酸序列设计优化现在有大量的数据库可以对目的序列BLAST进行脱靶预测,并通过数据库分析提高gRNA的靶向特异性[44-45]。Hsu等[46]通过建立计算机模型,计算出最优化的gRNA核苷酸链长度,可以降低脱靶效应。Seung等[47]通过设计一系列长度不同的gRNA,发现gRNA不仅能影响Cas9蛋白活性,还能影响脱靶效应的发生。Fu等[48]也进一步证实了缩短gRNA核苷酸序列,不但没有影响gRNA的靶向功能,还使脱靶率降低了5 000倍。缩短一对gRNA的核苷酸序列,配合上述dCas9-FokⅠ融合蛋白使用,使CRISPR/Cas9系统的特异性大幅提高[49]。
3.2 CRISPR/Cas9嵌合突变造成遗传异质性(Heterozygous)CRISPR/Cas9技术在非人灵长类动物中引起的嵌合突变是另一个值得关注的问题。在已报道获取的CRISPR/Cas9基因修饰新生猴,基因组测序结果发现具有多种基因型,表明所获基因修饰猴为嵌合体[25],DMD猴模型的嵌合突变率达到87%[27]。由于非人灵长类动物生长周期长,通过多代交配筛选到纯靶向基因修饰的动物模型需要花费大量时间和经费。嵌合突变也会严重影响靶基因功能研究和相关疾病病理分析,因此减少嵌合体的产生尤为必要。
引起嵌合突变的原因主要有两种:其一,Cas9修饰靶位点后,由于分裂细胞之间DNA修复活性存在较大差异,导致不同细胞、组织之间发生不同程度的靶基因突变;其二,双链DNA断裂在非同源末端连接的修复过程中,会发生碱基随机插入和缺失(Insertion/deletion,indel),导致靶位点indel各不相同,形成多基因型嵌合突变体。目前研究表明Cas9蛋白的持续表达可能会增加嵌合突变,使靶基因编辑发生在细胞分裂前的单细胞阶段,是比较有效降低嵌合突变的方法之一。Tu等[50]通过在Cas9蛋白N末端连接泛素蛋白酶体,能促进Cas9蛋白在食蟹猴胚胎中降解,结合胚胎分裂方法,缩短Cas9半衰期能够减少嵌合突变并提高打靶效率。在小鼠中,用电转化或显微注射法将Cas9 mRNA和sgRNA注入受精卵中,产生了大量的嵌合突变体。用Cas9蛋白和sgRNA电转化法替代上述方法,在受精卵单细胞分裂前完成基因编辑,嵌合突变率大幅降低[51]。同时,在嵌合突变的检测方法上,传统的Sanger基因组测序、T7EN1等技术存在仪器和人为误差,导致突变位点检测存在遗漏。建立更严格有效的全基因组深度测序法,实现对靶基因嵌合突变的全面评估分析,对建立更有效精准的非人灵长类动物模型也有指导性作用[52-54]。
3.3 CRISPR/Cas9基因敲入效率较低目前利用CRISPR/Cas9系统对非人灵长类动物进行基因编辑,DNA发生同源重组修复(HR)的几率非常低,仅在利用Cas9技术对食蟹猴p53基因编辑过程中,成功实现了小片段DNA序列敲入和基因定点突变[29]。然而,大片段基因序列靶向敲入在非人灵长类动物中尚无成功报道。研究者试图通过阻断非同源末端连接的修复途径(NHEJ),以期提高HR几率。抗癌药物Scr7可与NHEJ信号通路中的关键酶Ligase Ⅳ结合,阻止DNA的NHEJ途径。Maruyama等[55]将Scr7添加到基因编辑的小鼠受精卵中,显著提高了小鼠模型的同源重组修复。Aida等[56]通过优化CRISPR/Cas系统,使其包含Cas9蛋白、CRISPR RNA(crRNA)以及反义激活crRNA三个部分,实现了小鼠基因组中较长基因盒的靶向敲入。这些研究思路也可以指导我们应用于非人灵长类动物模型中。
4 结语由于人体的复杂性与伦理方面的限制,以人体本身作为实验对象来研究疾病机制及医学治疗策略是极其缓慢且困难的。例如,2016年,我国科学家利用CRISPR/Cas9系统对人类三核胚胎进行HBB基因修饰[35],虽然三核胚胎不能发育成正常人类胚胎,但是在基因编辑技术还不是非常完善的背景下,这个实验在国际伦理上引起了巨大争议。同年,中国科学家利用CRISPR/Cas9技术敲除人类淋巴细胞的PD-1基因,将修饰后的细胞重新注入患者体内进行人类肺癌临床治疗实验[57],这次基因治疗只针对放化疗和其他治疗手段均无效的志愿者,因此治疗对象比较局限。最近,Nature杂志发布了美国科学家利用CRISPR/Cas9技术对正常的人类胚胎进行编辑,成功修复了遗传性肥厚型心肌病的致病MYBPC3突变基因,引起巨大轰动[58],但是对人体实验的伦理约束极大限制了实验结果的进一步验证。目前,国际上有望出台基因编辑技术在人体中的应用细则。但是,禁止对人类生殖系进行基因编辑是人类普遍的共识。
建立模仿人类疾病的有效动物模型,可以避免直接进行人体实验的风险。作为与人类生理生化上相似度最高的动物,非人灵长类动物建立人类疾病模型能够更真实有效地模拟人类疾病并研究疾病的治疗策略,是替代人体模型的极佳选择。但是,由于非人灵长类动物较长的生长周期和高昂的饲养费用,特别是缺乏高效的基因修饰技术,造成非人灵长类动物疾病模型的研究困难重重。近年来,得益于基因编辑技术的重大突破,才使得该领域的研究逐渐有了起色(表 1)。不过目前已报道的非人灵长类动物疾病模型,大部分都局限于模仿人类神经性遗传疾病的范畴。借鉴其它动物疾病模型的相关研究思路和技术路线,可建立疾病覆盖面更广的非人灵长类动物模型,如治愈白内障模型[59],或是通过多个抑癌基因突变构建癌症模型[60-61]等。在此基础上,进一步分析非人灵长类动物模型的疾病演进机制,对相关模型开展治疗方法的探索与评估[62-63],将为人类征服重大疾病、提高生存质量和预期寿命作出巨大贡献(图 2)。
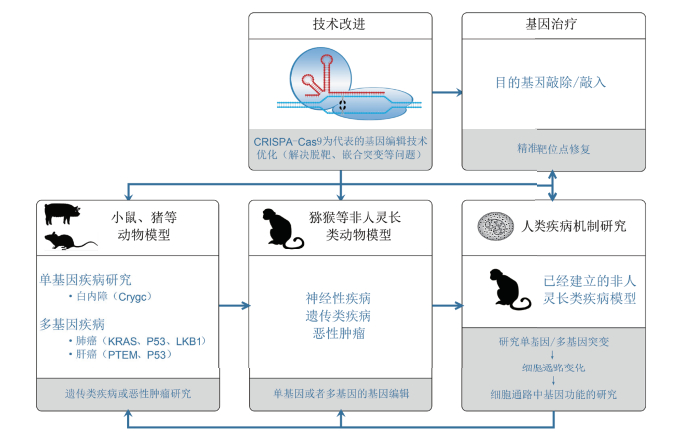
|
| 图 2 利用CRISPR/Cas9基因编辑技术构建非人灵长类动物疾病模型的研究策略 |
我国是世界上拥有最丰富非人灵长类动物资源的国家之一,目前在运用基因编辑技术构建非人灵长类动物模型的研究领域中处于世界领先水平。不过,随着对基因编辑技术应用的不断深化与扩展,脱靶效应、嵌合突变等突出问题应该引起足够重视。相信随着基因编辑技术的不断完善,研究人员将有更大的创新空间,非人灵长类动物疾病模型必然会在人类医学健康研究领域发挥重要作用。
| [1] |
Van DBL. Genetic rodent models of amyotrophic lateral sclerosis[J]. J Biomed Biotechnol, 2011, 2011(6): 348765. |
| [2] |
Hoke A, Ray M. Rodent models of chemotherapy-induced peripheral neuropathy[J]. ILAR J, 2014, 54(3): 273-281. DOI:10.1093/ilar/ilt053 |
| [3] |
Yan G, Zhang G, Fang X, et al. Genome sequencing and comparison of two nonhuman primate animal models, the cynomolgus and Chinese rhesus macaques[J]. Nat Biotechnol, 2011, 29(11): 1019-1023. DOI:10.1038/nbt.1992 |
| [4] |
Gibbs RA, Rogers J, Katze MG, et al. Evolutionary and biomedical insights from the rhesus macaque genome[J]. Science, 2007, 316(5822): 222-234. DOI:10.1126/science.1139247 |
| [5] |
Chan AW, Chong KY, Martinovich C, et al. Transgenic monkeys produced by retroviral gene transfer into mature oocytes[J]. Science, 2001, 291(5502): 309-312. DOI:10.1126/science.291.5502.309 |
| [6] |
Yang SH, Cheng PH, Banta H, et al. Towards a transgenic model of Huntington's disease in a non-human primate[J]. Nature, 2008, 453(7197): 921-924. DOI:10.1038/nature06975 |
| [7] |
Niu Y, Guo X, Chen Y, et al. Early Parkinson's disease symptoms in α-synuclein transgenic monkeys[J]. Hum Mol Genet, 2015, 24(8): 2308-2317. DOI:10.1093/hmg/ddu748 |
| [8] |
Liu Z, Li X, Zhang JT, et al. Autism-like behaviours and germline transmission in transgenic monkeys overexpressing MeCP2[J]. Nature, 2016, 530(7588): 98-102. DOI:10.1038/nature16533 |
| [9] |
Sasaki E, Suemizu H, Shimada A, et al. Generation of transgenic non-human primates with germline transmission[J]. Nature, 2009, 459(7246): 523-527. DOI:10.1038/nature08090 |
| [10] |
Niu Y, Yu Y, Bernat A, et al. Transgenic rhesus monkeys produced by gene transfer into early-cleavage-stage embryos using a simian immunodeficiency virus-based vector[J]. Proc Natl Acad Sci U S A, 2010, 107(41): 17663-17667. DOI:10.1073/pnas.1006563107 |
| [11] |
Carroll D, Wahl LM. Genome engineering with zinc-finger nucleases[J]. Genetics, 2011, 188(4): 773-782. DOI:10.1534/genetics.111.131433 |
| [12] |
Cermak T, Doyle EL, Christian M, et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting[J]. Nucleic Acids Res, 2011, 39(12): e82. DOI:10.1093/nar/gkr218 |
| [13] |
Liu H, Chen Y, Niu Y, et al. TALEN-mediated gene mutagenesis in rhesus and cynomolgus monkeys[J]. Cell Stem Cell, 2014, 14(3): 323-328. DOI:10.1016/j.stem.2014.01.018 |
| [14] |
Chen Y, Yu J, Niu Y, et al. Modeling rett syndrome using TALEN-edited MECP2 mutant cynomolgus monkeys[J]. Cell, 2017, 169(5): 945-955. DOI:10.1016/j.cell.2017.04.035 |
| [15] |
Ke Q, Li W, Lai X, et al. TALEN-based generation of a cynomolgus monkey disease model for human microcephaly[J]. Cell Res, 2016, 26(9): 1048-1061. DOI:10.1038/cr.2016.93 |
| [16] |
Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea[J]. Science, 2010, 327(5962): 167-170. DOI:10.1126/science.1179555 |
| [17] |
Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems[J]. Nat Rev Microbiol, 2015, 13(11): 722-736. DOI:10.1038/nrmicro3569 |
| [18] |
Abudayyeh OO, Gootenberg JS, Konermann S, et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector[J]. Science, 2016, 353(6299): aaf5573. DOI:10.1126/science.aaf5573 |
| [19] |
Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system[J]. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038 |
| [20] |
Fonfara I, Richter H, Bratovic M, et al. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA[J]. Nature, 2016, 532(7600): 517-521. DOI:10.1038/nature17945 |
| [21] |
Liu L, Li X, Wang J, et al. Two distant catalytic sites are responsible for C2c2 RNase activities[J]. Cell, 2017, 168(1-2): 121-134. DOI:10.1016/j.cell.2016.12.031 |
| [22] |
Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [23] |
Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [24] |
Nishimasu H, Ran FA, Hsu PD, et al. Crystal structure of Cas9 in complex with guide RNA and target DNA[J]. Cell, 2014, 156(5): 935-949. DOI:10.1016/j.cell.2014.02.001 |
| [25] |
Niu Y, Shen B, Cui Y, et al. Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos[J]. Cell, 2014, 156(4): 836-843. DOI:10.1016/j.cell.2014.01.027 |
| [26] |
Chen Y, Cui Y, Shen B, et al. Germline acquisition of Cas9/RNA-mediated gene modifications in monkeys[J]. Cell Res, 2015, 25(2): 262-265. DOI:10.1038/cr.2014.167 |
| [27] |
Chen Y, Zheng Y, Kang Y, et al. Functional disruption of the dystrophin gene in rhesus monkey using CRISPR/Cas9[J]. Hum Mol Genet, 2015, 24(13): 3764-3774. DOI:10.1093/hmg/ddv120 |
| [28] |
Kang Y, Zheng B, Shen B, et al. CRISPR/Cas9-mediated Dax1 knockout in the monkey recapitulates human AHC-HH[J]. Hum Mol Genet, 2015, 24(25): 7255-7264. DOI:10.1093/hmg/ddv425 |
| [29] |
Wan H, Feng C, Teng F, et al. One-step generation of p53 gene biallelic mutant Cynomolgus monkey via the CRISPR/Cas system[J]. Cell Res, 2015, 25(2): 258-261. DOI:10.1038/cr.2014.158 |
| [30] |
Midic U, Hung PH, Vincent KA, et al. Quantitative assessment of timing, efficiency, specificity and genetic mosaicism of CRISPR/Cas9-mediated gene editing of hemoglobin beta gene in rhesus monkey embryos[J]. Hum Mol Genet, 2017, 26(14): 2678-2689. DOI:10.1093/hmg/ddx154 |
| [31] |
Rusk N. CRISPRs and epigenome editing[J]. Nat Methods, 2014, 11(1): 28. |
| [32] |
Fu Y, Foden JA, Khayter C, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells[J]. Nat Biotechnol, 2013, 31(9): 822-826. DOI:10.1038/nbt.2623 |
| [33] |
Mali P, Aach J, Stranges PB, et al. CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome take engineering[J]. Nat Biotechnol, 2013, 31(9): 833-838. DOI:10.1038/nbt.2675 |
| [34] |
Pattanayak V, Lin S, Guilinger JP, et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity[J]. Nat Biotechnol, 2013, 31(9): 839-843. DOI:10.1038/nbt.2673 |
| [35] |
Liang P, Xu Y, Zhang X, et al. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes[J]. Protein Cell, 2015, 6(5): 363-372. DOI:10.1007/s13238-015-0153-5 |
| [36] |
Ran FA, Hsu PD, Lin CY, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J]. Cell, 2013, 154(6): 1380-1389. DOI:10.1016/j.cell.2013.08.021 |
| [37] |
Guilinger JP, Thompson DB, Liu DR. Fusion of catalytically inactive Cas9 to FokⅠ nuclease improves the specificity of genome modification[J]. Nat Biotechnol, 2014, 32(6): 577-582. DOI:10.1038/nbt.2909 |
| [38] |
Shengdar QT, Nicolas W, Cyd K, et al. Dimeric CRISPR RNA-guided FokⅠ nucleases for highly specific genome editing[J]. Nat Biotechnol, 2014, 32(6): 569-576. DOI:10.1038/nbt.2908 |
| [39] |
Slaymaker IM, Gao L, Zetsche B, et al. Rationally engineered Cas9 nucleases with improved specificity[J]. Science, 2016, 351(6268): 84-88. DOI:10.1126/science.aad5227 |
| [40] |
Kleinstiver BP, Pattanayak V, Prew MS, et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects[J]. Nature, 2016, 529(7587): 490-495. DOI:10.1038/nature16526 |
| [41] |
Liu KI, Ramli MN, Woo CW, et al. A chemical-inducible CRISPR-Cas9 system for rapid control of genome editing[J]. Nat Chem Biol, 2016, 12(11): 980-987. DOI:10.1038/nchembio.2179 |
| [42] |
Nihongaki Y, Kawano F, Nakajima TP, et al. Photoactivatable CRISPR-Cas9 for optogenetic genome editing[J]. Nat Biotechnol, 2015, 33(7): 755-760. DOI:10.1038/nbt.3245 |
| [43] |
Rauch BJ, Silvis MR, Hultquist JF, et al. Inhibition of CRISPR-Cas9 with Bacteriophage Proteins[J]. Cell, 2017, 168(1-2): 150-158. DOI:10.1016/j.cell.2016.12.009 |
| [44] |
Lee CM, Cradick TJ, Fine EJ, et al. Nuclease target site selection for maximizing on-target activity and minimizing off-target effects in genome editing[J]. Mol Ther, 2016, 24(3): 475-487. DOI:10.1038/mt.2016.1 |
| [45] |
Rastogi A, Murik O, Bowler C, et al. PhytoCRISP-Ex: a web-based and stand-alone application to find specific target sequences for CRISPR/CAS editing[J]. BMC Bioinformatics, 2016, 17(1): 261. DOI:10.1186/s12859-016-1143-1 |
| [46] |
Hsu PD, Scott DA, Weinstein JA, et al. DNA targeting specificity of RNA-guided Cas9 nucleases[J]. Nat Biotechnol, 2013, 31(9): 827-832. DOI:10.1038/nbt.2647 |
| [47] |
Seung WC, Sojung K, Yongsub K, et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases[J]. Genome Res, 2014, 24(1): 132-141. DOI:10.1101/gr.162339.113 |
| [48] |
Fu Y, Sander JD, Reyon D, et al. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs[J]. Nat Biotechnol, 2014, 32(3): 279-284. DOI:10.1038/nbt.2808 |
| [49] |
Wyvekens N, Topkar VV, Khayter C, et al. Dimeric CRISPR RNA-guided FokⅠ-dCas9 nucleases directed by truncated gRNAs for highly specific genome editing[J]. Hum Gene Ther, 2015, 26(7): 425-431. DOI:10.1089/hum.2015.084 |
| [50] |
Tu Z, Yang W, Yan S, et al. Promoting Cas9 degradation reduces mosaic mutations in non-human primate embryos[J]. Sci Rep, 2017, 7(1): 42081. |
| [51] |
Hashimoto M, Yamashita Y, Takemoto T. Electroporation of Cas9 protein/sgRNA into early pronuclear zygotes generates non-mosaic mutants in the mouse[J]. Dev Biol, 2016, 418(1): 1-9. DOI:10.1016/j.ydbio.2016.07.017 |
| [52] |
Platt RJ, Chen S, Zhou Y, et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling[J]. Cell, 2014, 159(2): 440-455. DOI:10.1016/j.cell.2014.09.014 |
| [53] |
Sakurai T, Watanabe S, Kamiyoshi A, et al. A single blastocyst assay optimized for detecting CRISPR/Cas9 system-induced indel mutations in mice[J]. BMC Biotechnol, 2014, 14(1): 69. DOI:10.1186/1472-6750-14-69 |
| [54] |
Tsai SQ, Zheng Z, Nguyen NT, et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases[J]. Nat Biotechnol, 2015, 33(2): 187-197. DOI:10.1038/nbt.3117 |
| [55] |
Kim D, Bae S, Park J, et al. Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells[J]. Nat Methods, 2015, 12(3): 237-243. DOI:10.1038/nmeth.3284 |
| [56] |
Maruyama T, Dougan SK, Truttmann MC, et al. Increasing the efficiency of precise genome editing with CRISPR-Cas9 by inhibition of nonhomologous end joining[J]. Nat Biotechnol, 2015, 33(5): 538-542. DOI:10.1038/nbt.3190 |
| [57] |
Aida T, Chiyo K, Usami T, et al. Cloning-free CRISPR/Cas system facilitates functional cassette knock-in in mice[J]. Genome Biol, 2015, 16(1): 87. DOI:10.1186/s13059-015-0653-x |
| [58] |
David Cyranoski. CRISPR gene-editing tested in a person for the first time[J]. Nature, 2016, 539(7630): 479. DOI:10.1038/nature.2016.20988 |
| [59] |
Ma H, Marti-Gutierrez N, Park SW, et al. Correction of a pathogenic gene mutation in human embryos[J]. Nature, 2017, 548(7668): 413-419. DOI:10.1038/nature23305 |
| [60] |
Wu Y, Liang D, Wang Y, et al. Correction of a genetic disease in mouse via use of CRISPR-Cas9[J]. Cell Stem Cell, 2013, 13(6): 659-662. DOI:10.1016/j.stem.2013.10.016 |
| [61] |
Liu Y, Qi X, Zeng Z, et al. CRISPR/Cas9-mediated p53 and pten dual mutation accelerates he patocarcinogenesis in adult hepatitis B virus transgenic mice[J]. Sci Rep, 2017, 7(1): 2796. DOI:10.1038/s41598-017-03070-8 |
| [62] |
Reardon S. Leukaemia success heralds wave of gene-editing therapies[J]. Nature, 2015, 12; 527(7577): 146-147. |
| [63] |
Wang Z, Pan Q, Gendron P, et al. CRISPR/Cas9-derived mutations both inhibit HIV-1 replication and accelerate viral escape[J]. Cell Rep, 2016, 15(3): 481-489. DOI:10.1016/j.celrep.2016.03.042 |