2. 福建农林大学福建省农业生态过程与安全监控重点实验室,福州 350002
2. Fujian Provincial Key Laboratory of Agroecological Processing and Safety Monitoring, Fujian Agriculture and Forestry University, Fuzhou 35002
细菌的外膜蛋白(Outer membrane proteins,OMPs)是一类位于革兰氏阴性细菌外膜上的蛋白质,可协助细菌摄取所需的营养物,促进细菌的生长,并在细菌的生理与病理机制中扮演着重要作用[1-4]。外膜蛋白的合成、转运与正确折叠需要经历一个复杂的过程,先在细胞质内合成具有N-末端信号的前体,经由SEC系统介导穿过细胞内膜,并在分子伴侣(Skp,SurA)的作用下经过周质空间,最终在BAM(β-barrel assembly machinery)复合物的协助下正确地折叠并整合到外膜中形成β-桶状结构[5-7]。BAM复合物一般由一个外膜蛋白BamA和4个脂蛋白BamB、BamC、BamD与BamE组成[8-10]。前期研究发现大肠杆菌的BamA属于Omp85家族蛋白,它与BamD是BAM系统中必不可少的组成部分,在外膜蛋白转运中起着至关重要的作用[7, 11]。BamB、BamC、BamD的同源物仅存在于革兰氏阴性菌,单独突变bamB、bamC和bamE基因,可引起少数外膜蛋白的错误装配。而三个重要性相对较弱的脂蛋白BamB、C、E对外膜蛋白转运的影响也各不相同,bamB的缺失对外膜蛋白的合成产生较大的影响[11-14]。此外,大肠杆菌BamA:B:C:D:E形成核心复合物,并在表面活性剂的作用下,可以分解为BamA:B、BamC:D和BamC:D:E三种复合物形式,而bamC的缺失会导致这些复合物的稳定性下降,并导致BamB与BamD对蛋白酶的敏感性增加[16]。同时在野生株中BamA易受外源的蛋白酶K的影响,bamE的缺失在一定程度可以降低其影响[17]。但在不同细菌中,BAM系统的复合物组成及其功能也存在一定差异。
目前对于BAM系统的研究大都集中于大肠杆菌及脑膜炎双球菌等少数几种细菌中,在其他细菌中的相关研究还鲜有报道。嗜水气单胞菌ATCC 7966是一株属于O:1血清型且具有一定毒性的模式菌株[18],BAM系统在该菌株中如何发挥调控作用尚不明确。本研究利用同源重组交换的方法成功构建嗜水气单胞菌bamA、bamB和bamD突变株,并结合质谱及Western blotting技术,研究这3个重要的蛋白对多个外膜蛋白表达及转运的影响,从而深化对BAM复合物在嗜水气单胞菌外膜转运系统中作用的认识。
1 材料与方法 1.1 材料 1.1.1 菌株、质粒、引物嗜水气单胞菌Aeromonas hydrohila ATCC 7966由实验室保存,感受态细胞E.coli S17-1λpir与自杀载体pUTKm1均由福建农林大学张燎原老师惠赠。根据Uniprot数据库中bamA、bamB、bamD的基因序列设计相应引物(表 1)。
限制性内切酶Kpn I、Sac I、Sca I和T4 DNA连接酶购自赛默飞世科技有限公司;DNA聚合酶和DNA marker为TaKaRa公司产品;胶回收试剂盒和质粒提取试剂盒购自Omega公司;基因组小量制备试剂盒购自天根生物科技公司;琼脂糖购自生工生物工程有限公司;卡那霉素(Kan)氨苄青霉素(Amp)均购自生工生物工程有限公司。
1.1.3 培养基胰蛋白胨10 g/L,酵母提取物5 g/L,NaCl 10 g/L。
1.2 方法 1.2.1 PCR扩增以提取的嗜水气单胞菌ATCC 7966基因组DNA为模板,利用相关引物进行PCR扩增反应,PCR产物经过1%琼脂糖凝胶电泳检测,检测后将获得的特异性DNA条带用胶回收试剂盒回收。
1.2.2 基因突变株的构建根据BamA、B、D基因序列分别设计引物(引物序列详见表 1),以嗜水气单胞菌为模板,PCR扩增片段,扩增后的片段进行双酶切(Sac I、Sca I),经再次回收后与酶切后的自杀载体pUTKm1连接。连接产物转化至E.coli S17-1λpir,从而完成同源重组载体的构建。双亲本杂交参考张燎原方法进行操作[18]。
双亲本杂交利用的原理为同源重组单交换,目的是将同源重组载体整合进入嗜水气单胞菌的基因组中,具体操作如下:分别过夜培养嗜水气单胞菌ATCC 7966和E.coli S17-1 λpir/pUT-bamA、bamB、bamD,以1:20(V/V)的接种量转接至新鲜LB液体培养基进行培养,OD600nm至0.8(约3 h),取100 μL嗜水气单胞菌培养液和400 μL E.coliS17-1λpir/pUT-bamA、bamB、bamD培养液至离心管中进行混合,在4℃条件下进行离心(4 000 r/min),加入100 μL新鲜LB液体培养基重悬浮菌体,取50 μL重悬浮菌液点至预先准备好的放有0.22 μm膜的平板上,正置培养12 h后,用1 mL LB液体培养基将膜上培养的菌体洗涤下来,取100 μL洗涤下的菌液涂布在含终浓度100 μg/mL卡那霉素和100 μg/mL氨苄青霉素的LB平板上进行筛选,长出的菌落经特异性引物进行PCR验证。
1.2.3 膜蛋白的提取分别挑取嗜水气单胞菌ATCC 7966野生株与突变株的单克隆菌落于5 mL的LB培养基,30℃震荡过夜培养,以1:100(V/V)转接到100 mL LB液体培养基中扩大培养,30℃培养至OD600nm为1.0,8 000 ×g离心10 min收集菌体,超声破碎菌体,离心收集上清。将所得上清经0.45 μm滤膜过滤,滤液于超速离心机中以29 300 r/min离心60 min,沉淀用适量的PBS缓冲液溶解,-20℃保存。
1.2.4 差异蛋白的SDS-PAGE分析与质谱鉴定取经超速离心法得到野生株与突变株的膜蛋白进行SDS-PAGE分析,分析条件为恒压90 V,分离胶浓度为10%,上样量为20 μL电泳至溴酚蓝行至电泳槽下端为止,电泳后用考马斯亮蓝R250染色。将蛋白胶上所选择的差异条带切下,先用100 μL Milli-Q水震荡洗涤两次,再加入脱色液(50 mmol/L碳酸氢铵/乙腈=1:1)至胶粒蓝色褪去;加入150 μL纯乙腈脱水至胶粒完全变白,真空干燥;加入100 μL的10 mmol/L二硫苏糖醇(DTT),56℃水浴60 min;冷却到室温后,吸干,快速加入100 μL的55 mmol/L碘乙酰胺(IAA),置于暗室45 min;后依次用25 mmol/L碳酸氢铵、50%乙腈和100%乙腈,脱水到胶粒完全变白为止,真空抽干;后用Trypsin酶液消化过夜,加入2%三氟乙酸(TFA)终止反应,使TFA终浓度为0.1%,振荡混匀,离心,酶解的肽段用LC-LTQ XL质谱仪鉴定蛋白。
1.2.5 突变株bamA、B、D的Western blot分析将敲除菌中提取的膜蛋白,经SDS-PAGE电泳分离,进行印记杂交,将凝胶、滤纸、PVDF膜置于盛有转膜缓冲液的容器中,浸泡5 min,恒流1.3 mA,转膜30 min,将PVDF膜置于5%脱脂牛奶中封闭60 min,加入相应外膜蛋白抗体(1:2 000稀释)4℃孵育过夜,洗涤。后加入辣根过氧化物酶标记羊抗兔抗体,室温孵育60 min,洗涤,显色。
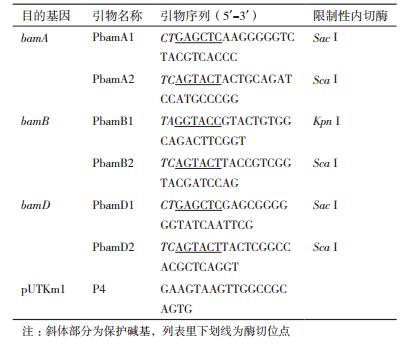
2 结果 2.1 bamA(AHA_1181)、bamB(AHA_1762)、bamD(AHA_4074)基因片段的扩增如图 1所示,利用DNA聚合酶从嗜水气单胞菌ATCC 7966基因组DNA中PCR扩增约1 060 bp、750 bp、580 bp的目的基因片段。对照Marker可以看出PCR扩增出bamA(1 060 bp)、bamB(750 bp)、bamD(580 bp)的特异性条带,确定PCR扩增出的条带为所需的基因片段。
|
| 图 1 bamA、bamB、bamD基因PCR产物琼脂糖凝胶电泳检测结果 1-3:分别为bamA、bamB、bamD的PCR扩增产物,M:核酸分子量标准 |
PCR产物经限制性内切酶完全消化后,与载体酶切后回收的片段连接,转化感受态细胞E. coli S17-1λpir,在50 μg/mL卡那霉素LB平板上挑选克隆。通过酶切(图 2)和测序鉴定,获得正确重组子自杀载体pUTKm-bamA-D,再通过双亲本接合转移,在100 μg/mL氨苄青霉素和100 μg/mL卡那霉素抗性LB平板上筛选,得到接合转移子。

|
| 图 2 pUT-bamA-D双酶切电泳图 1-3:分别为bamA、bamB、bamD的双酶切电泳结果,M:核酸分子量标准 |
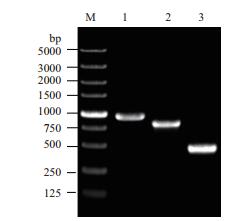
经过抗性平板的初步筛选,筛选到bamA、bamB、bamD突变菌株。为了进一步确定bamA、B、D基因是否已经被插入突变,分别在BamA、B、D的上游设计一条引物(PbamA1、PbamB1、PbamD1),在自杀载体上设计一条引物(P4)。以野生株和突变株的嗜水气单胞菌基因组DNA为模板,进行PCR扩增。结果(图 3)发现可以分别扩增出特定长度的片断,而对照为阴性图 3。同时将扩增产物送铂尚生物技术(上海)有限公司测序,测序结果显示正确。综上结果证实pUT-bamA、pUT-bamB、pUT-bamD载体已经插入嗜水气单胞菌ATCC 7966基因组中。
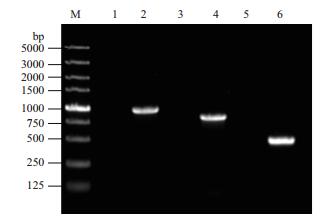
|
| 图 3 突变株bamA、B、D的PCR验证 1、3、5:以野生株DNA为模板的PCR扩增;2、4、6:分别以突变株bamA、bamB、bamD阳性菌落DNA为模板的PCR扩增 |
同野生株嗜水气单胞菌外膜蛋白进行比较,突变菌株bamA、bamB、bamD存在差异表达蛋白(图 4),对这些差异蛋白进行质谱分析,共鉴定到7个差异蛋白,其中5个为外膜蛋白,2个为位于内膜上的蛋白酶(表 2)。
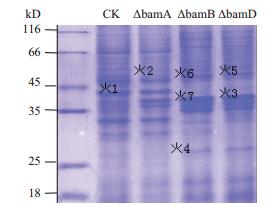
|
| 图 4 嗜水气单胞菌野生株与突变株膜蛋白的SDS-PAGE图谱 |
为了研究bamA、B、D突变后的变化,选取实验室已有的Ferrichome receptor(A0KQZ1)、Maltoporin(A0KHF6)、Colicin I receptor(A0KQ46)、TonB-dependent copper receptor(A0KFG8)外膜蛋白抗体,研究突变菌株bamA、B、D在胞浆和膜蛋白水平上,对蛋白的表达与转运的影响。结果发现,与野生株嗜水气单胞菌相比较,bamA、bamD突变后所有的相关蛋白都处于下调的状态,特别对Colicin I receptor与Maltoporin、TonB-dependent copper receptor三种外膜蛋白的影响最大。通过对细菌胞浆组分的western blotting结果分析,发现所有的检测蛋白也都下调,而且基因的缺失对Ferrichome receptor与Colicin I receptor、TonB-dependent copper receptor的表达影响较大,对Maltoporin的表达影响较小。但不同突变株对不同外膜蛋白的影响有差别,例如Colicin I receptor在bamD突变菌中胞浆中的表达明显降低,而Maltoporin则在bamB突变菌中显著下调(图 5)。

|
| 图 5 野生株(CK)与bamA、bamB、bamD突变株在膜蛋白水平与胞浆蛋白水平上多个相关蛋白的Western blotting A、B:分别为野生株(CK)与突变株在膜蛋白、胞浆蛋白水平上多个外膜蛋白的Western blotting |
细菌的外膜蛋白位于细胞与外界环境的交界处,在信号传导、毒力侵染、细菌耐药、抗逆性等多个生物学功能中起重要作用。因此,BAM系统复合物的正常运转可确保外膜蛋白的正确折叠和转运,对维持细菌的正常生长至关重要。Charlson等[13-14, 19]在大肠杆菌中发现BamA和BamD是细菌外膜蛋白折叠和转运的必须蛋白,bamB基因的突变,可以使BAM复合物受到损伤,部分外膜蛋白的分泌减少;而Fardini等[12]则认为沙门氏菌中的BamB并不是一个重要蛋白,当敲除bamB基因后,仅对BAM复合物系统造成轻微损伤。此外,研究发现脑膜炎双球菌中bamC和bamE的突变,对BAM复合物系统影响并不大,并不是细菌生存所需的关键蛋白[20]。
鉴于以往未见嗜水气单胞菌中BAM系统对外膜蛋白转运影响的报道,本研究利用分子生物学方法敲除该菌BAM系统中的重要组成亚基bamA,bamB和bamD。首先比较这些敲除菌株对细菌膜蛋白的表达影响,SDS-PAGE发现7条差异条带,质谱鉴定结果显示至少有5个外膜蛋白,并利用Western blotting验证部分蛋白的变化。结果发现,嗜水气单胞菌bamA、bamB和bamD的缺失突变均引起本研究中选取的4个外膜蛋白在细胞膜水平上的表达下降,除了BamD在Ferrichrome receptor的转运中起较为重要的作用以外,BamA、BamB和BamD对其他外膜蛋白转运均起重要的作用;研究还发现,在细菌胞浆组分中,不同BAM复合物亚基似乎对特定外膜蛋白表达的影响也有所不同,例如BamB蛋白在外膜蛋白Maltoporin的表达中起决定性作用,BamA次之;Colicin I receptor和Ferrichrome receptor的表达,则主要取决于BamD蛋白;而BamA,BamB和BamD在TonB-dependent copper receptor与BamD的表达中起相近的贡献,提示BAM系统的变化不仅影响外膜蛋白在膜蛋白组分上的转运,还可能通过某种途径影响外膜蛋白的调控表达。值得提出的是,在大肠杆菌中敲除bamA或者bamD基因以后,均导致细菌外膜蛋白不能正确折叠进入外膜而导致细菌死亡,而本研究中敲除嗜水气单胞菌的相应基因后只引起细菌生长略缓慢(数据未显示),并未造成细菌的致死,这可能与试验中pUTKm载体介导的突变并未完全无痕敲除目的基因有关,也有可能由这两个蛋白在BAM系统中的作用与大肠杆菌等细菌的差异导致。以上研究与在大肠杆菌中相应复合物功能的结论有略微不同,提示嗜水气单胞菌BAM转运系统可能还具有特定的外膜蛋白转运与折叠特性。
4 结论本研究成功构建了bamA、bamB、bamD缺失突变株,SDS-PAGE分析发现7条差异条带,质谱技术鉴定获得至少5个差异外膜蛋白,进一步的Western blot分析发现bamA、bamB和bamD的缺失均能不同程度地影响嗜水气单胞菌外膜蛋白的转运。
| [1] |
Rollauer SE, Sooreshjani MA, Noinaj N, et al. Outer membrane pro- tein biogenesis in Gram-negative bacteria[J]. Philosophical Tran-sactions of the Royal Society of London, 2015, 370(1679): 1-10. |
| [2] |
Kim SW, Oh MH, Jun SH, et al. Outer membrane Protein A plays a role in pathogenesis of Acinetobacter nosocomialis[J]. Virulence, 2016, 7(4): 413-426. DOI:10.1080/21505594.2016.1140298 |
| [3] |
Dalbey R E, Kuhn A. Protein traffic in Gram-negative bacteria how exported and secreted proteins find their way[J]. Microbiology Review, 2012, 36(6): 1023-1045. |
| [4] |
赵巧云, 谢勇. 幽门螺旋杆菌外膜蛋白与黏附机制的研究进展[J]. 上海交通大学学报, 2017, 37(2): 249-252. |
| [5] |
Romanhernandez G, Bernstein HD. An in vitro assay for outer membrane protein assembly by the BAM complex[J]. Methods Mol Biol, 2015, 1329: 203-213. DOI:10.1007/978-1-4939-2871-2 |
| [6] |
Noinaj N, Rollauer SE, Buchanan SK. The β-barrel membrane protein insertase machinery from Gram-negative bacteria[J]. Current Opinion in Structure Biology, 2015, 31: 35-42. DOI:10.1016/j.sbi.2015.02.012 |
| [7] |
Misra R, Stikeleather R, Gabriele R. In vivo roles of BamA, BamB and BamD in the biogenesis of BamA, a core protein of the β-barrel assembly machine of Escherichia coli[J]. Journal of Molecular Biology, 2015, 427(5): 1061-1074. DOI:10.1016/j.jmb.2014.04.021 |
| [8] |
Han L, Zheng J, Wang Y, et al. Structure of the BAM complex and its implications for biogenesis of outer-membrane proteins[J]. Nature Structural & Molecular Biology, 2016, 23(3): 192-196. |
| [9] |
Gu Y, Li H, Dong H, et al. Structural basis of outer membrane protein insertion by the BAM complex[J]. Nature, 2016, 531(7592): 64-69. DOI:10.1038/nature17199 |
| [10] |
Plummer AM, Fleming KG. BamA alone accelerates outer membrane protein folding in vitro through a catalytic mechanism[J]. Biochemistry, 2015, 54(39): 6009-6011. DOI:10.1021/acs.biochem.5b00950 |
| [11] |
Doerrler WT, Raetz CR. Loss of outer membrane proteins without inhibition of lipid export in an Escherichia coli YaeT mutant[J]. Journal of Biology Chemistry, 2005, 280(30): 27679-27687. DOI:10.1074/jbc.M504796200 |
| [12] |
Fardini Y, Trotereau J, Bottreau E, et al. Investigation of the role of the BAM complex and SurA chaperone in outer-membrane protein biogenesis and type Ⅲ secretion system expression in Salmonella[J]. Microbiology, 2009, 155: 1613-1622. DOI:10.1099/mic.0.025155-0 |
| [13] |
Hsieh PF, Hsu CR, Chen CT, et al. The Klebsiella pneumoniae YfgL (BamB) lipoprotein contributes to outer membrane protein biogenesis type-1 fimbriae expression, anti-phagocytosis, and in vivo virulence[J]. Virulence, 2016, 7(5): 587-601. DOI:10.1080/21505594.2016.1171435 |
| [14] |
Rigel NW, Schwalm J, Ricci DP, et al. BamE modulates the Escherichia coli beta-barrel assembly machine component BamA[J]. Journal of Bacteriology, 2012, 194(5): 1002-1008. DOI:10.1128/JB.06426-11 |
| [15] |
Webb CT, Selkrig J, et al. Dynamic association of BAM complex modules includes surface exposure of the lipoprotein BamC[J]. Journal of Molecular Biology, 2012, 422(4): 545-555. DOI:10.1016/j.jmb.2012.05.035 |
| [16] |
Rigel NW, Schwalm J, Ricci DP, et al. BamE modulates the Escherichia coli beta-barrel assembly machine component BamA[J]. Journal of Bacteriology, 2012, 194(5): 1002-1008. DOI:10.1128/JB.06426-11 |
| [17] |
Yu HB, Zhang YL, Lau YL, et al. Identification and characterization of putative virulence genes and gene clusters in Aeromonas hydrophila PPD134/91[J]. Applied Environmental Microbiology, 2005, 71(8): 4469-4477. DOI:10.1128/AEM.71.8.4469-4477.2005 |
| [18] |
张燎原, 徐泉明, 孙建. 粘质沙雷氏菌中乙偶姻合成途径调控基因的鉴定[J]. 江西农业学报, 2012, 3(44): 706-711. |
| [19] |
Charlson ES, Werner JN, et al. Differential effects of yfgL mutation on Escherichia coli outer membrane proteins and lipopolysaccha-ride[J]. Journal of Bacteriology, 2006, 188: 7186-7194. DOI:10.1128/JB.00571-06 |
| [20] |
Volokhina EB, Beckers F, Tommassen J, et al. The beta-barrel outer membrane protein assembly complex of Neisseria meningitidis[J]. Journal of Bacteriology, 2009, 191(22): 7074-7085. DOI:10.1128/JB.00737-09 |