萜类化合物(Terpenoids)是指由异戊二烯(Isoprene,C5)为基本结构单元组成的一类天然化合物。萜类作为植物体内一类重要的次生代谢产物,由初生代谢途径衍生而来,具有聚异戊二烯的碳骨架,根据异戊二烯的数目可以分为单萜(Monoter-pene,C10)、倍半萜(Sesquiterpene,C15)、二萜(Diterpene,C20)、三萜(Triterpene,C30)和多萜。萜类化合物普遍存在于各种植物中,对植物的生长发育、防御等有重要的作用,同时也是梨和苹果等花果实芳香物质的来源之一。植物中的萜类化合物根据其生理功能的不同可以分为初生代谢物和次生代谢物。初生代谢物对植物的生长发育必不可少,包括胡萝卜素、类胡萝卜素、甾体、多聚萜醇和植物激素等,次生代谢物的种类繁多,在植物与环境的相互作用、植物与昆虫和动物之间的相互作用以及植物体之间的信号传递发挥重要的作用。一些萜类成分具有很高的药用价值和医疗价值,如倍半萜成分青蒿素是目前治疗疟疾的最佳药物[1],紫杉醇是抗肿瘤的有效药物等[2]。萜类化合物通过类异戊二烯途径产生,研究表明,植物中类异戊二烯至少存在2条合成途径,包括经典的甲羟戊酸途径和丙酮酸/磷酸甘油醛途径[3-5]。
萜类合成酶作为萜类合成途径的关键酶是目前比较关注的研究方向之一,在拟南芥(Arabidopsis thaliana)[6]、水稻(Oryza sativa)[7]、番茄(Solanum lycopersicum)[8]、杨树(populus trichocarpa)[9]、大冷杉(Abies grandis)[10]、葡萄(Vitis vinifera L.)[11]和苹果(Malus domestica)[12]等植物中均有报导。梨基因组测序的完成和序列释放为梨萜类合成途径中各种关键酶的研究提供了可能[13]。Bohlmann等[14]在1998年根据萜类合成酶氨基酸序列的相关性把来自烟草、荞麦等多个物种共33条萜类合成酶基因分为6个亚家族;Trapp等[15]在2001年根据基因结构把被子植物的萜类合成酶基因分为三类。
梨果实的内在品质主要决定于果实的香气和风味,其中,果实香气成分主要由其挥发性的产物组成,而萜类化合物是其挥发性产物的重要组成部分。在高等植物中,TPS基因均以基因家族的形式存在。梨作为一种重要的经济作物,在基因组水平对其TPS基因家族的研究成果还非常匮乏。本研究通过整合梨CDS、全基因组数据和蛋白组数据等,结合生物信息学和分子生物学方法,对梨萜类合成酶基因家族进行各种分析,包括梨萜类合成酶基因家族成员的鉴定,理化性质和二、三级结构的预测,保守基序分析和组织表达模式分析等,旨在为梨TPS基因家族成员各基因的具体生物学功能和改良梨果实品质的后续研究提供理论基础。
1 材料与方法 1.1 材料梨(Pyrus bretschneideri)全基因组数据下载于http://gigadb.org/dataset/100083。TPS蛋白质序列来源于NCBI蛋白质数据库(GenBank),苹果(Malus domestica)AAO22848.2;拟南芥(Arabidopsis thaliana)AAO85539.1,AAG09310.1,AAO85533.1;大冷杉(Abies grandis)AAB70907.1;玉米(Zea mays)AAA73960.1;南瓜(Cucurbita maxima)AAD04292.1,AAB39482.1;金鱼草(Antirrhinum majus)AAO42614.1;番茄(Solanum lycopersicum)AAC39431.1;黄瓜(Cucumis sativus)BAB19275.1。供试材料品种为西洋梨,取自于山东农业大学果树实验基地。取3年生野生西洋梨新生幼叶、幼芽、根和茎,用锡箔纸包好,液氮速冻,-80℃保存备用。
1.2 方法 1.2.1 梨TPS基因家族成员的鉴定从梨(Pyrus bretschneideri)全基因组数据库(http://gigadb.org/dataset/100083)下载基因组和蛋白组数据,构建本地BLAST数据库[16],以GenBank中已注册的50条TPS蛋白序列作为查询序列执行本地BLASTP搜索,搜索的e值设置为(e-10)。搜索出的萜类合成酶同源序列使用保守域预测软件Pfam和SMART(http://smart.embl-heidelberg.de/)在线工具检测候选蛋白质序列[17],使用Pfam数据库的序列号PF01397(萜类合成酶N末端结构域)和PF03936(金属离子结合结构域)鉴定梨TPS基因。
1.2.2 梨TPS基因家族的蛋白理化性质和亚细胞定位分析使用ExPASy在线工具(http://www.expasy.org/proteomics)对鉴定出的所有蛋白序列进行氨基酸数目、分子量、等电点以及开放阅读框等的预测。使用SOPMA(http://www.expasy.org/proteomics)进行TPS蛋白二级结构预测。通过WOLF PSORT(http://www.genscript.com/wolf-psort.html)和Targetp1.1共同进行TPS基因的亚细胞定位分析。
1.2.3 梨TPS基因家族系统进化树的构建使用Clustal X方法对梨TPS氨基酸序列进行多重序列比对,使用MEGA6.0软件进行邻接法系统进化树的构建,校验参数Bootstrap重复1 000次,对获得的进化树进行亚族分类和进化分析[18]。
1.2.4 TPS蛋白保守域和三级结构预测使用在线软件MEME(http://meme-suite.org/tools/meme)对TPS蛋白保守域进行分析[19]。根据已知的梨TPS蛋白完整序列,通过在线工具SWISS-MODEL进行同源建模获得梨TPS蛋白的三级结构模型[20, 21]。
1.2.5 TPS基因家族成员的基因结构和染色体定位分析根据从梨全基因组数据库下载得到的外显子、内含子和梨基因组定位信息从基因库中提取待分析的蛋白相关信息,按GSDS所需格式整理,提交至GSDS在线网站进行基因结构分析。在梨基因组数据库中检索每一条TPS基因在染色体上的起始位置、终止位置和每一条蛋白在染色体上的位置信息。
1.2.6 梨TPS基因的组织表达分析使用TIANGEN公司产品RNAprep Pure Plant Kit提取西洋梨根、茎、新生幼叶、幼芽总RNA。使用反转录试剂盒FastQuant RT Kit(With gDNase)合成cDNA。设计实时荧光定量PCR引物(表 1)。采用梨ACTIN(AF386514)作为内参基因。方法按照CWBIO公司UltraSYBR Mixture(Low ROX)说明书进行。反应体系为2×UltraSYBR Mixture(Low ROX)9 μL、引物1(10 μmol/L)0.5 μL、引物2(10 μmol/L)0.5 μL、cDNA 1 μL和ddH2O 9 μL。反应条件为95℃ 10 min;95℃ 15 s,60℃ 30 s,40个循环,溶解曲线的温度是65℃-95℃。
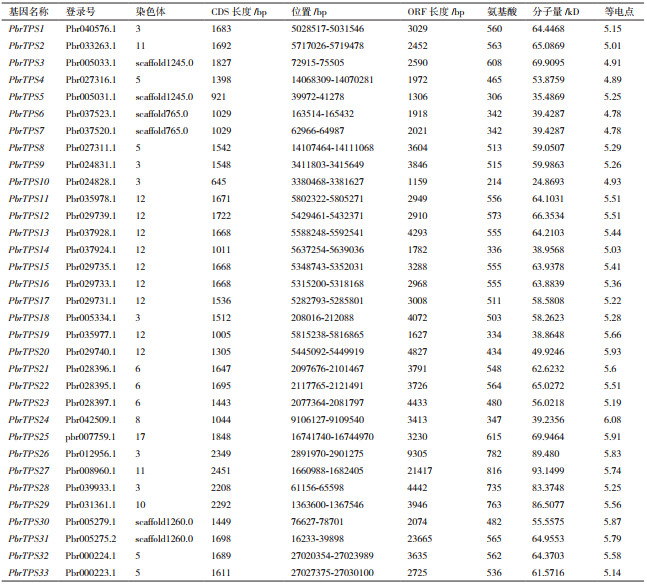
以GenBank中已经注册的50条TPS基因作为查询序列初步筛选得到49个候选蛋白序列。使用Pfam和SMART确定保守结构域,删除不含有典型结构域PF01397(萜类合成酶N末端结构域)、PF03936(金属离子结合结构域)的蛋白序列,最终得到33个TPS基因家族成员,少于拟南芥(40个)和番茄(44个)。参考拟南芥中TPS的命名方式,将梨TPS分别命名为PbrTPS1-PbrTPS33。利用ExPASy提供的在线工具对各条基因的基本信息进行预测,包括分子量、等电点、基因位置和CDS长度等(表 2),结果表明,梨TPS的CDS长度范围为645-2 451,ORF长度范围为1 159-23 665。通过分析发现,鉴定得到33条梨TPS基因家族成员的等电点均小于7,推测大多数的梨TPS基因可能编码弱酸性蛋白,在酸性的亚细胞环境中发挥作用。
染色体定位结果表明,33个TPS基因家族成员分别定位在梨的8条染色体上。分析发现,定位到第12染色体上的TPS基因序列数量最多。各有一条分别定位在第8、第10和第17染色体。其中,PbrTPS3、PbrTPS5、PbrTPS6、PbrTPS7、PbrTPS30和PbrTPS31定位在scaffold上(表 2)。
2.2 TPS基因家族成员二级结构和亚细胞定位分析Protscale预测结果表明33条TPS基因家族成员均属于亲水性蛋白。使用SOPAM程序(http://www.expasy.org/proteomics)对33条蛋白序列进行二级结构预测,结果(表 3)表明,梨TPS蛋白序列均由α-螺旋、无规卷曲和延伸链等二级结构元件组成。除PbrTPS24、PbrTPS25、PbrTPS27和PbrTPS30以无规卷曲为主要二级结构存在外,另外29条蛋白序列的二级结构主要形式均以α-螺旋形式出现。
使用WOLF PSORT和Targetp1.1工具共同进行亚细胞定位预测,结果(表 3)表明,33条TPS蛋白大部分定位于细胞质,但是也有少数分别定位于内质网、叶绿体、细胞核等部位。
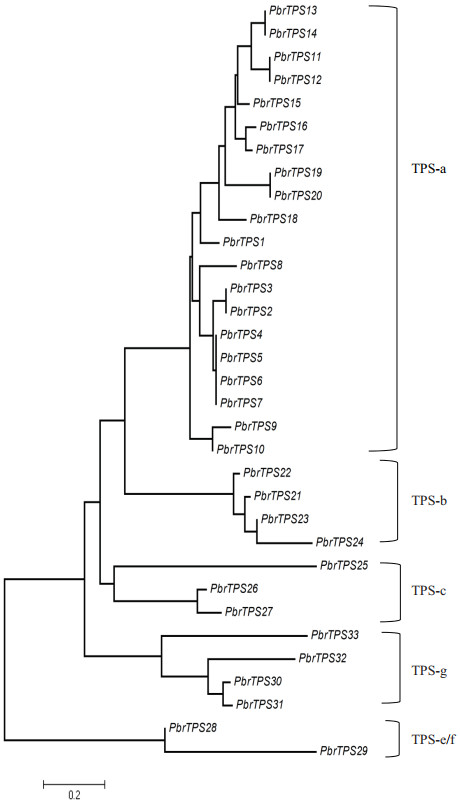
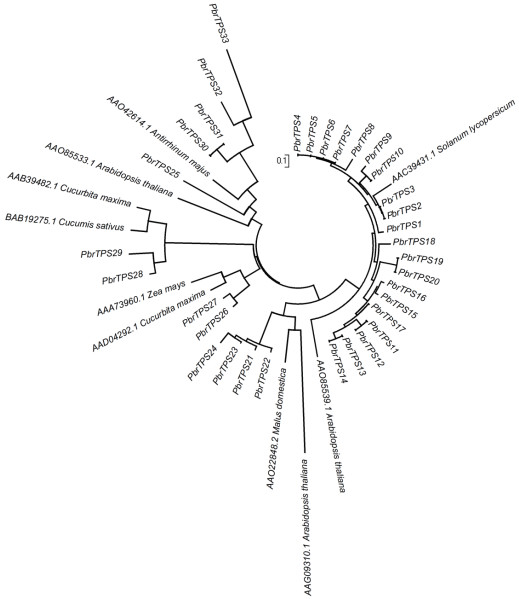
2.3 梨TPS基因家族成员系统进化和基因结构分析 2.3.1 梨TPS蛋白系统进化分析为了进一步了解梨TPS基因家族成员的系统进化关系,使用软件MAGA6.0采用邻接法对已经发现的33条梨TPS蛋白序列构建系统进化树进行进化聚类和分析,参照Bohlmann等[14]对TPS基因家族的分类方法对梨TPS基因家族进行亚家族分类,发现33个梨TPS蛋白可以分为5个亚族(图 1)。同时使用这33条序列与NCBI已经注册的拟南芥(Arabidopsis thaliana)、苹果(Malus domestica)、番茄(Solanum lycopersicum)等植物中的TPS-a、TPS-b、TPS-c、TPS-g和TPS-e/f亚家族各成员共同构建系统进化树,发现仅有33条序列进行分类的结果相同(图 2)。因此确认各亚家族含有的TPS基因家族成员的数量各不相同。其中TPS-a亚家族包含的成员数量最多,与葡萄[22]和中粒咖啡[23]等植物研究过程中发现的规律相似。
|
| 图 1 梨TPS基因家族系统进化树 |
|
| 图 2 梨与其他植物TPS蛋白的系统进化树 |
使用GSDS网站对基因结构进行预测。结果(图 3)表明,基因结构特征和亚家族之间具有对应关系,同一亚家族成员基因结构之间非常相似,内含子数量3-15个,但多数含有4-7个。

|
| 图 3 梨TPS基因家族基因结构分析 |
进一步分析发现,TPS-a亚族的外显子数量均比较多而且密集,除Pbr024828.1之外,TPS-a亚族和TPS-b亚族包含4-7个外显子,而TPS-c亚族和TPS-g亚族的内含子外显子组成则比较分散,但是各亚族内的组成结构都比较相似,因此,推测各组内的基因家族成员间在进化关系上也较近。
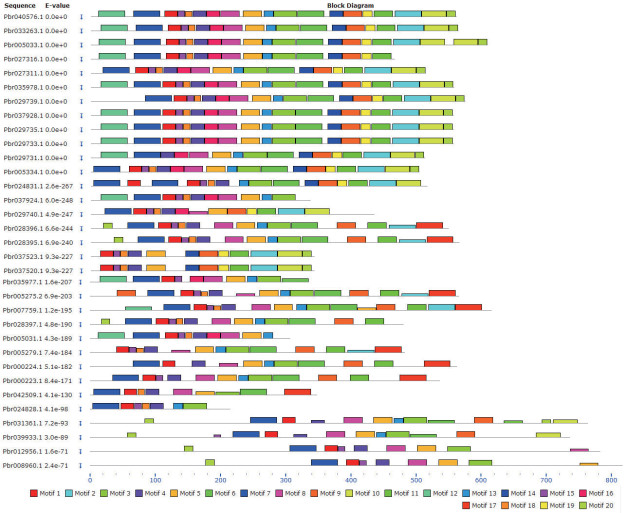
2.4 梨TPS基因家族成员的保守基序分析为了进一步分析梨TPS蛋白序列在各个亚族上的差异,使用MEME在线软件对TPS基因家族的保守结构域进行预测,最终获得20个保守基序(图 4)。分析发现,TPS-a亚族蛋白大多含有较多的基序数目,在TPS-a亚族中多数第一个保守序列是Motif12,其对应的序列为DTFDVHRPKPSATFSPSIWGDHFJSYASLZVDAELEQHVQE,而以Motif20为结束的保守基序对应的序列是KDLIASTLVEPVPL。另外,TPS-a中保守基序个数相对较多,而TPS-e/f的Motif个数相对较少。除PbrTPS5外,其余TPS基因家族成员中均含有Motif1和Motif3这两个保守基序,因此,这2个位点所包含的序列DLYDLYTVALRFRLLRQHGYNVS和YYFWSLGVYFEPQYSFARKILTKVTAJVTIIDDIYD在梨TPS蛋白中是高度保守的。Motif1、Motif7、Motif12构成了靠近N端的TPS保守结构域序列,Motif2、Motif10、Motif20构成了靠近C端的保守基序。
|
| 图 4 梨TPS基因家族保守基序分析 |
对发现的20个保守基序进行Pfam和SMART注释分析后共对4个基序进行了命名,分别是基序2、基序3、基序6和基序7(图 5),SMART验证结果显示基序7是萜类合成酶N末端结构域PF01397所包含的基序,基序2、3、6是金属离子结合结构域PF03936所包含的基序,剩余的Motif保守基序是未知的功能元件。
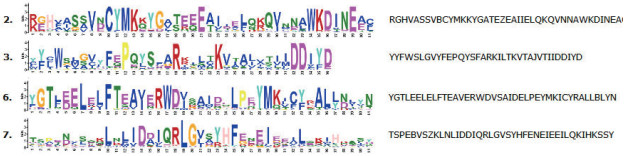
|
| 图 5 梨TPS氨基酸序列Logo图谱 |
蛋白质的生物学功能很大程度上取决于蛋白的空间结构,因此蛋白结构分析对于其功能的研究具有重要意义。使用在线工具SWISS-MODEL对梨TPS蛋白的三级结构进行同源建模。在对33个梨TPS蛋白进行同源建模后,发现TPS-a、TPS-c、TPS-g和TPS-e/f亚家族内的蛋白质在空间结构上都高度相似,各亚族内蛋白质具有高度保守的结构特征,因此分别在上述4个TPS亚家族中各选择2个作为代表性的序列进行同源建模(图 6),对TPS-b亚家族所有成员进行三级结构分析后发现得到的三维结构具有差异(图 6),推测TPS-a、TPS-c、TPS-g和TPS-e/f亚家族内蛋白质具有相似的功能而TPS-b亚家族成员在发挥的功能上可能有所差异。
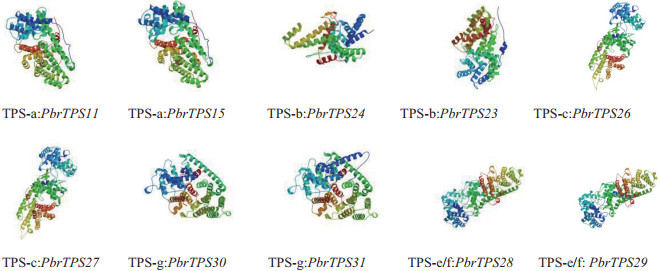
|
| 图 6 梨TPS蛋白三级结构 |
基于梨TPS基因的序列相似性和三级结构分析结果,对上述已经进行三级结构分析的10条序列进行组织表达模式分析。结果(图 7-A)显示,除PbrTPS24外,其余9个PbrTPS在根、茎、幼叶和幼芽中均有表达,且呈现出多种相对表达模式。另外,PbrTPS11、PbrTPS15、PbrTPS27、PbrTPS30和PbrTPS31在茎中的表达量最高,PbrTPS26、PbrTPS28和PbrTPS29在根中的表达量最高,PbrTPS23和PbrTPS24在叶中高表达(图 7-B)。
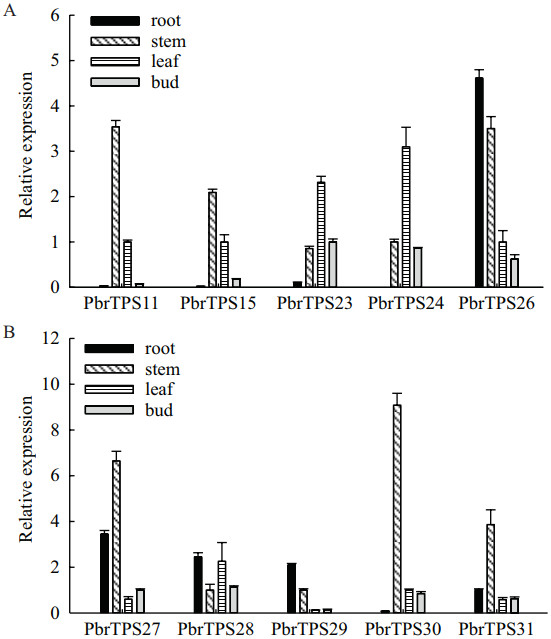
|
| 图 7 PbrTPS在梨不同组织中的表达分析 A:PbrTPS11、PbrTPS15、PbrTPS23、PbrTPS24和PbrTPS26在不同器官中的表达;B:PbrTPS27、PbrTPS28、PbrTPS29、PbrTPS30和PbrTPS31在不同器官中的表达 |
萜类合成酶是萜类化合物合成途径中的关键酶。近年来,不同物种基因组测序相继完成,使得从基因组的角度分析和研究基因家族成为可能[22-24]。目前,多个物种中的TPS基因家族成员已经被鉴定出来,在模式植物拟南芥[6]中发现了40个TPS基因家族成员,在番茄[8]中发现了44个TPS基因家族成员。在对拟南芥全基因组分析得到的40个TPS分析后发现,其中30个基因在序列相关性、基因结构和系统发育上非常相似,推测可能是由于在其进化过程中序列上发生了改变,出现了基因的片段重复或者串联重复等,进而导致不同酶结构和功能的不同[6]。本研究对梨与其它植物TPS蛋白的系统进化树研究,同样发现大多数处于同一个亚族的TPS基因不仅在进化上关系较近,同源建模结果显示蛋白质三维结构也非常相似,推测其编码的蛋白质功能也非常相似。二级结构预测结果显示,梨TPS蛋白二级结构序列均由α-螺旋、无规卷曲和延伸链等形式组成,α-螺旋和无规卷曲是其主要组成元件,这与在苹果[12]等植物中不同二级结构元件的比例和分布规律相似,推测不同来源的植物中TPS基因可能发挥相似的功能。Yoshikuni等[25]研究表明,功能专一性的酶是由功能复杂的酶进化而来,少量氨基酸之间的替换可以促进酶功能上的进化,因此,在同一个物种中通过基因进化出现多种不同萜类合成酶很有可能促进萜类代谢下游产物种类的增多。研究表明,TPS基因家族不仅广泛的存在于各种植物中,在不同植物基因组中其基因家族成员的数目差异也较大。对多种已经完成测序的植物中的TPS基因家族成员进行分类分析后发现,除大豆外,其他双子叶植物和单子叶植物的TPS基因家族中TPS-a亚家族成员数目最多[26, 27],在本实验中发现共有20个TPS基因属于TPS-a亚家族,占梨中TPS基因的大多数,因此推测在梨中,倍半萜类化合物种类和数量最为丰富,而在本研究中并没有发现TPS-d基因家族的成员,研究表明TPS-d基因家族是裸子植物特有的相对独立的基因家族分支[23],而梨树属于被子植物,这可能是在梨基因组中没有鉴定到TPS-d亚家族成员的主要原因。
目前,TPS基因家族的研究主要集中于苹果、拟南芥和番茄等植物中,而关于梨TPS基因家族的研究尚未见到报导。本研究根据梨与拟南芥中TPS基因的同源性进行了命名,所获得的TPS基因数目低于苹果中已经发现的TPS基因数目,推测可能是由于梨的全基因组学研究的不完全性导致,但仍有可能在梨中其自身的TPS基因家族成员数目相对较少。使用MEME在线工具进行保守基序预测,最终对4个基序进行命名。另外,梨TPS基因在染色体上呈随机不均匀分布,这也与TPS基因在其他物种染色体上的分布规律相似,如蒺藜苜蓿的23条TPS基因在第1、4、8染色体上均没有分布[28]。目前研究结果表明能够正常发挥功能的萜类合成酶基因长度一般最少在300 bp以上,如红豆杉中的紫杉二烯合成酶基因编码303个氨基酸[29],而在本研究发现PbrTPS10仅由214个氨基酸编码,推测可能是由于基因组进化过程中发生了片段缺失现象,但是其是否能够发挥萜类合成酶的正常功能,还需要后续的实验进一步验证。在对不同基因家族成员的组织表达模式进行分析发现,萜类合成酶基因的表达存在组织特异性,例如PbrTPS30在茎中的表达量比幼叶和幼芽中高9倍左右,PbrTPS24在根中基本不表达,这可能是由于萜类化合物存在物种和组织特异性,因此萜类合成酶基因的表达也存在组织特异性。
萜类合成酶是合成萜类代谢终产物的关键酶,是萜类化合物合成过程中的直接催化者,因此萜类合成酶催化的反应步骤是植物萜类代谢途径中的靶位点。由于植物体内大部分与萜类合成相关的酶表达量较低,相应的萜类化合物产量较低很难提取和纯化,因此需要在深入的了解萜类代谢途径的同时,更多的鉴定和分离出萜类合成过程中的各种关键酶,进而通过基因工程技术提高目的萜类化合物的生物合成水平。梨树是重要的经济作物,同时也具有重要的生态价值和观赏价值。梨果实中的芳香物质是评价果实内在品质的重要指标之一。近年来研究表明,果实中的挥发性物质主要包括醇类、醛类和脂类等,目前对西洋梨的研究大多集中于果实采摘后生理特性和贮藏技术的研究,而对其香气成分的研究相对较少。萜类合成酶与果实中各种挥发性萜类物质的种类和含量密切相关,因此,研究梨TPS基因家族各成员对提高果实品质也具有重要意义。
4 结论基于梨基因组数据库共鉴定获得33个梨萜类合成酶基因,定位于梨的8条染色体上,聚类为5个亚家族,其中,TPS-a亚家族成员数量最多,包括20个TPS基因,并且未发现TPS-d基因家族成员,各亚家族TPS蛋白三级结构高度相似。TPS基因家族成员存在组织特异性表达。
| [1] |
Brown GD. The biosynthesis of artemisinin(Qinghaosu)and the phytochemistry of Artemisia annua L. (Qinghao)[J]. Molecules, 2010, 15(11): 7603-7698. DOI:10.3390/molecules15117603 |
| [2] |
Cragg GM. Paclitaxel(Taxol):a success story with valuable lessons for natural product drug discovery and development[J]. Medicinal Research Reviews, 1998, 29(49): 315-331. |
| [3] |
Rohmer M. The discovery of a mevalonate-independent pathway for isoprenoid biosynthesis in bacteria, algae and higher plants[J]. Natural Product Reports, 1999, 16(5): 565-574. DOI:10.1039/a709175c |
| [4] |
Schwender J, Zeidler J, Gröner R, et al. Incorporation of 1-deoxy-D-xylulose into isoprene and phytol by higher plants and algae[J]. FEBS letters, 1997, 414(1): 129-134. DOI:10.1016/S0014-5793(97)01002-8 |
| [5] |
Rohmer M, Knani M, Simonin P, et al. Isoprenoid biosynthesis in bacteria:a novel pathway for the early steps leading to isopentenyl diphosphate[J]. Biochemical Journal, 1993, 295(2): 517-524. DOI:10.1042/bj2950517 |
| [6] |
Aubourg S, Lecharny A, Bohlmann LJ. Genomic analysis of the terpenoid synthase(AtTPS)gene family of Arabidopsis thaliana[J]. Molecular Genetics and Genomics, 2002, 267(6): 730-745. DOI:10.1007/s00438-002-0709-y |
| [7] |
Chen H, Li G, Köllner TG, et al. Positive Darwinian selection is a driving force for the diversification of terpenoid biosynthesis in the genus Oryza[J]. BMC Plant Biology, 2014, 14(1): 239. DOI:10.1186/s12870-014-0239-x |
| [8] |
Falara V, Akhtar TA, Nguyen TT, et al. The tomato terpene synthase gene family[J]. Plant Physiology, 2011, 157(2): 770-789. DOI:10.1104/pp.111.179648 |
| [9] |
Irmisch S, Jiang Y, Chen F, et al. Terpene synthases and their contribution to herbivore-induced volatile emission in western balsam poplar(Populus trichocarpa)[J]. BMC Plant Biology, 2014, 14(1): 270. DOI:10.1186/s12870-014-0270-y |
| [10] |
Zavarin E, Snajberk K, Critchfield WB. Terpenoid chemosystematic studies of Abies grandis[J]. Biochemical Systematics & Ecology, 1977, 5(2): 81-93. |
| [11] |
Matarese F, Scalabrelli G, D'Onofrio C. Analysis of the expression of terpene synthase genes in relation to aroma content in two aromatic Vitis vinifera varieties[J]. Functional Plant Biology, 2013, 40(6): 552-565. DOI:10.1071/FP12326 |
| [12] |
Nieuwenhuizen NJ, Green SA, Chen X, et al. Functional genomics reveals that a compact terpene synthase gene family can account for terpene volatile production in apple[J]. Plant Physiology, 2013, 161(2): 787-804. DOI:10.1104/pp.112.208249 |
| [13] |
Wu J, Wang Z, Shi Z, et al. The genome of the pear(Pyrus bretschneideri Rehd.)[J]. Genome Res, 2013, 23(2): 396-408. DOI:10.1101/gr.144311.112 |
| [14] |
Bohlmann J, Meyer-Gauen G, Croteau R. Plant terpenoid synthases:Molecular biology and phylogenetic analysis[J]. Proc Natl Acad Sci USA, 1998, 95(8): 4126-4133. DOI:10.1073/pnas.95.8.4126 |
| [15] |
Trapp SC, Croteau RB. Genomic organization of plant terpene synthases and molecular evolutionary implications[J]. Genetics, 2001, 158(2): 811-832. |
| [16] |
Altschul SF, Gish W, Miller W, et al. Basic local alignment search tool[J]. Journal of Molecular Biology, 1990, 215(3): 403-410. DOI:10.1016/S0022-2836(05)80360-2 |
| [17] |
Schultz J, Milpetz F, Bork P, et al. SMART, a simple modular architecture research tool:identification of signaling domains[J]. Proc Natl Acad Sci USA, 1998, 95(11): 5857-5864. DOI:10.1073/pnas.95.11.5857 |
| [18] |
Tamura K, Peterson D, Peterson N, et al. MEGA5:Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology & Evolution, 2014, 28(10): 2731-2739. |
| [19] |
Bailey TL, Boden M, Buske F A, et al. MEME suite:tools for motif discovery and searching[J]. Nucleic acids research, 2009, 37(Web Server issue): 202-208. |
| [20] |
Kiefer F, Arnold K, Künzli M, et al. The SWISS-MODEL repository and associated resources[J]. Nucleic Acids Research, 2009, 37(Database issue): 387-392. |
| [21] |
Martin DM, Fäldt J, Bohlmann J. Functional characterization of nine Norway Spruce TPS genes and evolution of gymnosperm terpene synthases of the TPS-d subfamily[J]. Plant Physiology, 2004, 135(4): 1908-1927. DOI:10.1104/pp.104.042028 |
| [22] |
Martin DM, Aubourg S, Schouwey MB, et al. Functional annotation, genome organization and phylogeny of the grapevine(Vitis vinifera)terpene synthase gene family based on genome assembly, FLcDNA cloning, and enzyme assays[J]. BMC Plant Biology, 2010, 10(1): 1-22. DOI:10.1186/1471-2229-10-1 |
| [23] |
程甜, 魏强, 李广林. 中粒咖啡萜类合成酶基因家族的生物信息学分析[J]. 植物学报, 2016, 51(2): 235-250. DOI:10.11983/CBB15022 |
| [24] |
王淋, 乌云塔娜, 叶生晶. 杜仲EuHMGRS基因家族的鉴定及生物信息学分析[J]. 经济林研究, 2013, 31(4): 16-24. |
| [25] |
Yoshikuni Y, Keasling JD. Pathway engineering by designed divergent evolution[J]. Current Opinion in Chemical Biology, 2007, 11(2): 233-239. DOI:10.1016/j.cbpa.2007.02.033 |
| [26] |
Chen F, Tholl D, Bohlmann J, et al. The family of terpene synthases in plants:a mid-size family of genes for specialized metabolism that is highly diversified throughout the kingdom[J]. Plant Journal for Cell & Molecular Biology, 2011, 66(1): 212-229. |
| [27] |
Liu J, Huang F, Wang X, et al. Genome-wide analysis of terpene synthases in soybean:functional characterization of GmTPS3[J]. Gene, 2014, 544(1): 83-92. DOI:10.1016/j.gene.2014.04.046 |
| [28] |
Parker MT, Zhong Y, Dai X, et al. Comparative genomic and transcriptomic analysis of terpene synthases in Arabidopsis and Medicago[J]. Systems Biology Iet, 2014, 8(4): 146-153. DOI:10.1049/iet-syb.2013.0032 |