DNA甲基化是表观遗传学的重要内容之一,在植物生长发育、防御、逆境胁迫等方面起着极其重要的作用[1]。DNA甲基化现象在高等植物中普遍存在,但不同植物种类甲基化水平存在差异,一般基因组DNA的30%-50%胞嘧啶处于甲基化状态[2]。其中大部分的甲基化都发生在CpG二核苷酸和CpNpG三核苷酸中,其他位点较少[1]。植物中DNA甲基化修饰主要发生在转座子和重复序列上,主要作为基因组防御系统来维持基因组的稳定,一方面沉默内源的转座子等“自私元件(Selfish DNA elements)”,另一方面可以抵抗外源病毒的入侵[3]。作为表观遗传的重要修饰途径之一,DNA甲基化与基因表达调控[4]、基因组印记[5]和转基因沉默[6]等生物学过程密切相关。Zilberman等[3]指出基因转录受甲基化影响,轻度甲基化的基因适量表达,本身发生去甲基化的基因表达将会增强。DNA甲基化在植物中的研究已有大量报道,其中拟南芥的研究最深入,为其他作物的DNA甲基化研究提供了参考。
木薯(Manihot esculenta Crantz)是大戟科(Euphorbiaceae)木薯属(Maihot P. Mill.)多年生植物,贮藏根淀粉含量在27%-34%之间,被誉为“淀粉之王”,是世界三大薯类作物之一,也是世界第六大粮食作物,具有重要的实用价值和经济价值,为热带、亚热带近10亿人口提供基本食粮,是我国重要的工业淀粉和生物质能源原料[7]。提高块根淀粉含量,揭示淀粉合成机理是木薯研究的重点之一。DNA甲基化在植物生长发育过程中起着重要作用,本研究通过分析木薯块根不同发育时期的基因组DNA甲基化变化情况,旨为木薯块根淀粉合成机理提供表观方面的依据。
1 材料与方法 1.1 材料本研究采用生长于中国热带农业科学院热带作物品种资源研究所国家木薯种质资源圃的华南5号(SC5)和Cas36-12两个木薯(Manihot esculenta Crantz)种质。其中,SC5块根淀粉含量较高,而Cas36-12的淀粉含量较低。SC5和Cas36-12种植于2016年3月,分别选取其形成期(植后4个月)、膨大期(植后7个月)及成熟期(植后10个月)块根作为研究材料。每个材料每个生育期随机选取3个块根,洗干净切成小块混合保存于-80℃冰箱中,取3个重复。
1.2 方法 1.2.1 DNA提取与甲基化敏感扩增多态性(MSAP)分析木薯块根总DNA采用TianGen植物基因组DNA提取试剂盒进行提取,提取方法参考说明书。MSAP分析中酶切、连接、预扩和选扩参照薛晶晶等[8]的方法分析,采用EcoRⅠ、MspⅠ和HpaⅡ作为限制性内切酶,双酶切组合分别选用EcoRⅠ/MspⅠ和EcoRⅠ/HpaⅡ(NEB),接头和引物序列如表 1所示。
采用PerkinElmer公司的LabChip GX Touch 24微流控毛细管电泳系统,使用HT DNA 1K微流控芯片进行DNA甲基化条带的分离,操作步骤参照DNA 1K Reagent Kit说明书进行。具体操作流程如下:
(1)芯片准备。使用前将芯片及试剂盒置于室温20 min;制备胶-染料溶液(Gel-Dye),向1管DNA Gel Matrix中加入13 μL DNA Dye Concentrate,旋涡混匀并将混合液转入两个带有滤膜的离心管(Spin filters),室温9 300 r/min离心7.5 min,丢弃滤膜,已过滤好的胶-染料混合液在4℃避光保存,可放置不超过三周。用去离子水分别清洗芯片上的孔1,3,4,7,8,10两次。往孔3,7,8,10中加入50 μL Gel-Dye,往孔4中加入50 μL的DNA Marker。用70%的异丙醇清洁芯片窗口的两面,将芯片放入LabChip GX Touch仪器前确保孔1是空的。
(2)DNA ladder和Buffer Tube准备。在0.2 mL Ladder Tube中先后加入108 μL的1X PCR buffer和12 μL HT DNA ladder,混匀;往0.75 mL Buffer Tube中加入750 μL 1X PCR Buffer;将Ladder Tube,Buffer Tube分别放入LabChip GX Touch样品板托架上的ladder和buffer管槽中。
(3)DNA样品的准备。将本研究获得的选扩增PCR产物20 μL与20 μL 1 X PCR buffer等量混合加入到96孔板中,并将96孔板置于LabChip GX Touch样品板上。
(4)按照仪器的操作说明进行仪器的运行。仪器运行结束后采用LabChip GX Reviewer 5.2分析软件进行数据分析,将获到的峰图转化为条带,有峰位点“Yes”记为“1”,无峰位点“No”记为“0 ”。根据“1,0”统计结果,利用Excel分析其发育过程中的甲基化变化情况。
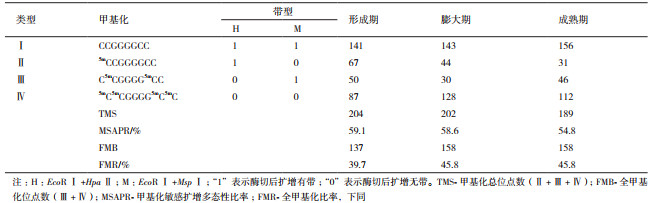
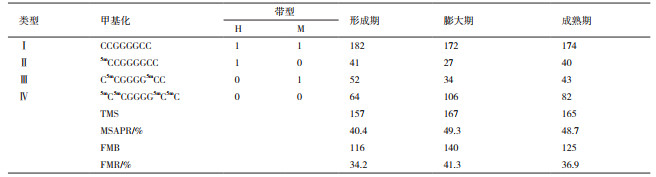
2 结果 2.1 SC5和Cas36-12不同发育期基因组DNA甲基化水平分析本研究利用MSAP技术分析木薯SC5和Cas36-12的形成期、膨大期及成熟期的块根基因组DNA 5' -CCGG位点胞嘧啶的甲基化水平和甲基化遗传模式。MSAP方法的关键在于胞嘧啶的甲基化状态会影响同裂酶HpaⅡ、MspⅠ的酶切反应,经PCR扩增出的多态性片段能够反映该位点的甲基化状态及程度(表 2和表 3)。因此,此方法被广泛用于动植物基因组甲基化分析。
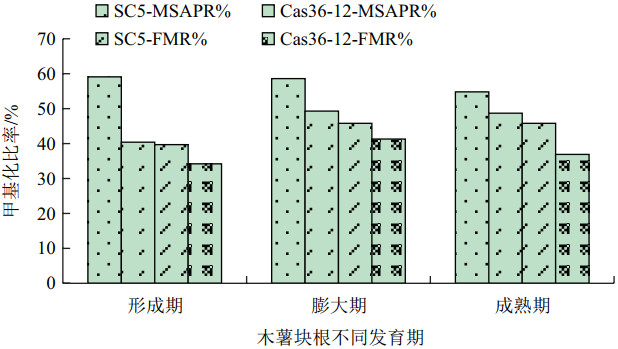
利用E1、E2分别与HM1-HM7配对组成的14对选择性引物对SC5、Cas36-12的形成期、膨大期及成熟期的块根进行MSAP分析。结果显示:SC5中共检测到345个DNA甲基化位点,Cas36-12中共检测到339个DNA甲基化位点。SC5中DNA甲基化水平(MSAPR%)随着块根的生长缓慢下降,由形成期的59.1%下降到成熟期的54.8%,全甲基化率(FMR%)呈现上升趋势,由39.7%上升至45.8%,且膨大期与成熟期相同(表 2,图 1)。而Cas36-12的MSAPR%和FMR%在膨大期时最大,达到49.3%和41.3%,至成熟期下降为48.7%和36.9%,但整体水平高于形成期的40.4%和34.2%(表 3,图 1)。SC5整个发育期的MSAPR%和FMR%均比Cas36-12高,且两个木薯品种在整个发育期的甲基化水平变化存在很大差异,推测可能与这两个木薯品种的块根淀粉含量差异巨大有关,且膨大期是整个块根形成的关键期。
|
| 图 1 SC5、Cas36-12不同发育期块根DNA甲基化水平变化 |
甲基化状态变化主要是指甲基化带型的多态性变化,即SC5和Cas36-12块根发育过程中的甲基化模式发生改变。将统计的多态性带型分为3种类型,第一类(A-C)为不发生变化带型,表明整个发育过程中,甲基化模式保持不变。第二类(D-I)为去甲基化类型,表明随着块根不断生长,基因组DNA甲基化水平降低,其中D、E、F为完全去甲基化,即所有被甲基化的胞嘧啶均发生去甲基化,G、H、I为部分去甲基化,即部分被甲基化的胞嘧啶发生去甲基化。第三类(J-O)为甲基化带型,表明随着块根生长,基因组DNA甲基化水平升高,其中J、K、L为重新甲基化,即前一个生长期没有甲基化,下一个生长期出现甲基化位点;M为全甲基化,即前一个生长期为单链外甲基化,下一个生长期变为双链内甲基化;N、O为超甲基化,即在前一个生长期甲基化位点的基础上,下一个生长期又有新的位点被甲基化(表 4和表 5)。
SC5块根生长过程中,膨大期和成熟期不发生变化的甲基化状态占49%和49.3%,分别有14.8%和15.7%的多态性带型发生了去甲基化,25.5%和9.6%的多态性带型发生了甲基化。其中,在整个发育过程中,全甲基化(0 1)带型和半甲基化(1 0)带型间没有变化。第二类(去甲基化类型)中,从形成期到膨大期过程中,发生变化的甲基化模式主要以完全去甲基化为主;从膨大期到成熟期过程中,发生变化的甲基化模式主要以部分去甲基化为主。在第三类(甲基化类型)中,发生变化的甲基化模式主要以超甲基化为主。比较SC5块根发育三个关键时期的甲基化变化情况,从形成期到膨大期,主要以发生甲基化为主;而从膨大期到成熟期,主要以去甲基化为主。
Cas36-12块根生长过程中,膨大期和成熟期不发生变化的甲基化状态占42.5%和47.5%,发生去甲基化的多态性带型有13.3%和15.3%,发生甲基化的有24.5%和9.4%。其中,在整个发育过程中全甲基化(0 1)带型和半甲基化(1 0)带型间的转变基本为0。第二类(去甲基化类型)中,膨大期与形成期相比,甲基化模式主要以完全去甲基化为主;而膨大期到成熟期的甲基化模式主要以部分去甲基化为主。第三类(甲基化类型)中,形成期到膨大期发生变化的甲基化模式主要以超甲基化为主,而膨大期到成熟期既有重新甲基化也有超甲基化的发生。比较Cas36-12块根发育3个关键时期的甲基化变化情况,从形成期到膨大期,主要以发生甲基化为主;从膨大期到成熟期,主要以去甲基化为主。
比较SC5和Cas36-12块根发育3个关键时期的甲基化变化情况,发现两份材料甲基化变化的整体趋势是一致的。形成期到膨大期主要以发生甲基化为主;而膨大期到成熟期,主要以去甲基化为主。推测在木薯生长过程中,基因组DNA的表观修饰具有普遍规律性。
3 讨论真核生物中,表观遗传通过调控染色质构象进而影响基因表达来调控植物的发育,其调控机制主要包括DNA甲基化、组蛋白修饰和小RNA干扰等,在植物的生长发育过程中起着至关重要的作用。种子萌发、开花、植株生长、程序性死亡等不同生长阶段、不同生长特性的植物DNA甲基化变化是不同的。因此,研究不同植物不同生长状态下的DNA甲基化变化对表观遗传学在植物中的功能都具有重要意义。研究表明:基因的甲基化会受转录水平影响,基因甲基化抑制基因表达,而基因的去甲基化会使转录水平增加[6]。而高等植物DNA甲基化在不同植物和组织中差异较大,一般基因组DNA有30%-50%的胞嘧啶处于甲基化状态,大多数低于50%[9, 10]。本研究以两份淀粉含量差异较大的木薯品种(SC5和Cas36-12)的不同生长期的块根作为实验材料,分析其不同生长期的DNA甲基化水平,结果发现,SC5的DNA甲基化水平高于Cas36-12,且超过了50%,与茶树[11]、牡丹[12]等的DNA甲基化水平相近,高于水稻[9]、拟南芥(35%-43%)[13]、棉花(41%)[10]等所报道的DNA甲基化水平。与Wang等[14]通过BS-seq测序对木薯与拟南芥的整体甲基化水平比较结果是一致的(木薯的甲基化水平高于拟南芥)。SC5和Cas36-12两个品种的DNA甲基化水平在3个生长期的差异较大,与吕亚[15]对这两个品种块根代谢相关蛋白的表达趋势是一致的,这可能和木薯基因组高度杂合的特性有关,与不同木薯品种块根淀粉代谢机理有关。
本研究采用的甲基化敏感扩增多态性(MSAP)技术是一种改良的AFLP技术,主要根据对甲基化敏感性不同的2种限制性内切酶HpaⅡ和MspⅠ对5' -CCGG-3' 位点进行酶切,2种酶均能识别并切割CCGG序列,但是2种酶对该位点的敏感性不同,可切割产生不同片段,通过扩增这些片段来揭示基因组的甲基化情况,是一种DNA甲基化检测的经典方法。该方法优点是简单易操作,使用方便,缺点是分辨率较低、假阳性较高且只能调查CCGG位点甲基化,获得的甲基化位点数较少,未能覆盖整个基因组,且选择性引物的多少对获得的甲基化位点也有一定影响。毛细管电泳(CE)具有高的分析效率、速度、微量、自动化和可定量等优势。利用MSAP技术结合毛细管电泳分析木薯块根的甲基化情况,可靠性较好。为了准确了解木薯块根发育过程中的甲基化变化,本实验下一步需要合成更多选择性引物,来检测其扩增结果,同时利用液相色谱法、亚硫酸盐测序、免疫共沉淀等方法验证。
4 结论本研究采用MSAP技术结合CE分析SC5和Cas36-12的DNA甲基化水平。SC5的DNA甲基化水平高于Cas36-12,且超过了50%。SC5和Cas36-12块根发育3个关键期的甲基化变化趋势一致。形成期到膨大期主要以发生甲基化为主;而膨大期到成熟期,主要以去甲基化为主。
| [1] |
Vanyushin BF, Ashapkin VV. DNA methylation in higher plants:past, present and future[J]. Biochim Biophys Acta, 2011, 1809(8): 360-368. DOI:10.1016/j.bbagrm.2011.04.006 |
| [2] |
Chan SW, Henderson IR, Jacobsen SE. Gardening the genome:DNA methylation in Arabidopsis thaliana[J]. Nat Rev Genet, 2005, 6(5): 351-360. DOI:10.1038/nrg1601 |
| [3] |
Zilberman D, Gehring M, Tran RK, et al. Genome-wide analysis of Arabidopsis thaliana DNA methylation uncovers an inter-dependence between methylation and transcription[J]. Nat Genet, 2007, 39(1): 61-69. DOI:10.1038/ng1929 |
| [4] |
Wang W, Zhao X, Pan Y, et al. DNA methylation changes detected by methylation-sensitive amplified polymorphism in two contrasting rice genotypes under salt stress[J]. J Genet Genomics, 2011, 38(9): 419-424. DOI:10.1016/j.jgg.2011.07.006 |
| [5] |
Jullien PE, Kinoshita T, Ohad N, et al. Maintenance of DNA methylation during the Arabidopsis life cycle is essential for parental imprinting[J]. Plant Cell, 2006, 18(6): 1360-1372. DOI:10.1105/tpc.106.041178 |
| [6] |
Adams KL, Percifield R, Wendel JF. Organ-specific silencing of duplicated genes in a newly synthesized cotton allotetraploid[J]. Genetics, 2004, 168(4): 2217-2226. DOI:10.1534/genetics.104.033522 |
| [7] |
薛晶晶, 陈松笔. 木薯环指蛋白基因MeRFP8克隆及表达[J]. 福建农林大学学报:自然科学版, 2017, 46(1): 73-80. |
| [8] |
Xue JJ, Li KM, Chen SB. DNA Methylation Analysis of Cassava(Manihot esculenta Crantz)SC8 and Its Autotetraploid in Response to Cold Stress[J]. Journal of Food Science and Engineering, 2017, 7(6): 297-304. |
| [9] |
潘雅姣, 傅彬英, 王迪, 等. 水稻干旱胁迫诱导DNA甲基化时空变化特征分析[J]. 中国农业科学, 2009, 42(9): 3009-3018. DOI:10.3864/j.issn.0578-1752.2009.09.001 |
| [10] |
李雪林, 林忠旭, 聂以春, 等. 盐胁迫下棉花基因组DNA表观遗传变化的MSAP分析[J]. 作物学报, 2009, 35(4): 588-596. |
| [11] |
周艳华, 曹红利, 岳川, 等. 冷驯化不同阶段茶树DNA甲基化模式的变化[J]. 作物学报, 2015, 41(7): 1047-1055. |
| [12] |
盖树鹏, 张风, 张玉喜, 等. 低温解除牡丹休眠进程中基因组DNA甲基化敏感扩增多态性(MSAP)分析[J]. 农业生物技术学报, 2012, 20(3): 261-267. |
| [13] |
Cervera M T, Ruiz-Garcia L, Martinez-Zapater J M. Analysis of DNA methylation in Arabidopsis thaliana based on methylation-sensitive AFLP markers[J]. Mol Genet Genomics, 2002, 268(4): 543-552. DOI:10.1007/s00438-002-0772-4 |
| [14] |
Haifeng Wang, Getu Beyene, Jixian Zhai, et al. CG gene body DNA methylation changes and evolution of duplicated genes in cassava[J]. PNAS, 2015, 112(44): 13729-13734. DOI:10.1073/pnas.1519067112 |
| [15] |
吕亚. 基于糖木薯块根不同发育期的蛋白质组学研究[D]. 海南: 海南大学, 2016.
|
| [16] |
Jaligot E, Beule T, Rival A. Methylation-sensitive RFLPs:characterisation of two oil palm markers showing somaclonal variation-associated polymorphism[J]. Theor Appl Genet, 2002, 104(8): 1263-1269. DOI:10.1007/s00122-002-0906-4 |
| [17] |
McClelland M, Nelson M, Raschke E. Effect of site-specific modification on restriction endonuclease and DNA modification methyltransferases[J]. Nucleic Acids Research, 1994, 22(17): 3640-3659. DOI:10.1093/nar/22.17.3640 |