植物在响应生物和非生物胁迫过程中逐渐形成一系列复杂的应对机制,其中相关基因的转录激活或抑制起着极其重要的作用[1]。许多基因均可以被逆境所诱导,特别是一些重要的转录因子家族成员,如WRKY、NAC、MYB等[2]。WRKY作为一种重要的转录因子家族,在拟南芥中共有72个成员[3]。WRKY转录因子的命名主要是由于其家族成员都最少含有一个WRKY结构域,即由约60个氨基酸组成的一段保守序列,其中最保守的是WRKYGQK这7个氨基酸。根据WRKY结构域和靠近C端的一个锌指结构(C2H2或C2HC)的不同,WRKY转录因子家族可以被分为3个亚族,并且与靶基因的结合通常被认为是通过W盒(W-box,TTGACC/T)顺式作用元件来进行[4]。
植物WRKY转录因子家族成员已被报道参与各种生物和非生物胁迫应答[5, 6]。水稻OsWRKY45存在2个等位基因OsWRKY45-1和OsWRKY45-2,在抵抗白叶枯病时表现出相反的表型[7]。拟南芥WRKY33的过量表达可以显著增加植物对灰霉病和黑斑病的抵抗力[8],MPK3/6通过磷酸化来激活WRKY33[9]。WRKY25和WRKY33参与拟南芥对盐害和热害的应答[10, 11]。另外,部分WRKY转录因子还参与拟南芥的生长发育调控,如叶片衰老[12]、根的发育[13]以及种皮的发育[14]等。
关于植物中WRKY互作蛋白的研究,目前鲜有报道。除了上述MPK外,植物体内的14-3-3蛋白也可以与一些WRKY蛋白形成复合物,从而介导上游激酶对WRKY的磷酸化并影响WRKY蛋白的稳定性和/或转录活性,拟南芥至少存在8个WRKY蛋白可以与其形成复合物[15]。一些VQ蛋白也可以与WRKY蛋白形成二聚体,如VQ16、VQ21和VQ22都可以与AtWRKY33蛋白的WRKY结构域C端结合,进而激活AtWRKY33的DNA结合活性[16]。另外,WRKY蛋白之间也可以发生相互作用。拟南芥中WRKY54和WRKY70都可以作为叶片衰老的负调控因子来延缓叶片的衰老,WRKY30可以分别与AtWRKY54和AtWRKY70发生互作,推测WRKY30可能参与植物的衰老调控网络[17]。Yang等[18]在胡桃中发现JrWRKY2和JrWRKY7可以形成同源或异源二聚体来响应一些非生物逆境。因此,探究WRKY蛋白与其他成员之间的互作情况,有利于深化对WRKY蛋白相关调控模式的认识。
在前期研究中,发现拟南芥中WRKY31的转录本水平能被热害以及低钾的短暂处理所抑制,暗示其可能参与对以上胁迫的快速响应,但具体的功能机制还有待研究。为了探究WRKY31如何调控植物对一些非生物胁迫的响应,本研究通过进行亚细胞定位和转录活性分析实验,了解其定位及转录作用。通过酵母双杂交和双分子荧光互补试验,了解WRKY31能够与自身以及WRKY6、WRKY42的互作关系,对深入阐明该转录因子的功能与机制奠定基础。
1 材料与方法 1.1 材料 1.1.1 植物材料实验所用材料为拟南芥(Arabidopsis thaliana)生态型Col-0;烟草为本氏烟草(Nicotiana benthamiana)。
1.1.2 菌种和载体大肠杆菌(Escherichia coli)菌株为DH5α;酵母菌(Saccharomyces cerevisiae)菌株为AH109;农杆菌(Agrobacterium tumefaciens)菌株为GV3101。酵母双杂交(Y2H)载体为pGBKT7和pGADT7(购自Clontech公司)。
1.1.3 实验引物使用的普通引物主要通过手动设计,并使用http://sg.idtdna.com/calc/analyzer进行Tm值检测;实时定量引物使用DNASTAR Primerselect软件设计,具体信息见表 1。
拟南芥或烟草种子先用70%乙醇浸泡处理1 min,再用次氯酸钠处理2 min,最后用灭菌后的蒸馏水清洗5-6次,播种于1/2MS培养基(添加1%蔗糖)平板上,移入4℃冰箱培养2-3 d,后移至温室中萌发。待生长7 d后将幼苗移至Pindstrup基质中(购自上海品氏托普公司)。温室植物生长的光周期为14 h/10 h(光照/黑暗),光照强度为120 μE/m2s,白天温度为22℃,夜间为20℃,湿度控制在60%-70%。
1.2 方法 1.2.1 原生质体的分离与双荧光素酶报告分析拟南芥原生质体的分离主要参照Niu等[19]描述的方法,即取生长4周龄的拟南芥叶片,使用纤维素酶和离析酶处理分离得到原生质体,并使用PEG4000介导转化法将质粒载体转入分离的原生质体中。将AtWRKY31的编码区克隆与效应载体pRT107-GAL4BD连接。转入原生质体培养24 h,用GloMax™ 20/20 Luminometer荧光计(Promega)检测LUC和REN荧光素酶的活性。
1.2.2 实时荧光定量PCR(qRT-PCR)将拟南芥Col-0种子植于培养皿中竖直生长7 d,然后将其移入多种处理培养基中或放置于特定温度的培养箱中,处理包括50 μmol/L ABA、125 mmol/L NaCl、25% PEG8000、低磷(LP,10 μmol/L PO43-)、低氮(LN,0.25 mmol/L NH4++NO3-)、低钾(LK,20 μmol/L K+)、10 μmol/L百草枯(MV)、5%葡萄糖(glucose)、冷害(4℃)和热害(38℃),继续竖直生长1、3和12 h后,每个处理收取10棵生长一致的拟南芥幼苗(使用3个生物学重复),液氮冻存。使用Trizol(生工,上海)进行RNA提取,具体步骤参照说明书。提取的RNA首先使用NanoDrop 1000(Thermo Scientific,USA)进行浓度检测,并通过电泳检测RNA完整性。然后使用Reverse Transcriptase M-MLV(RNase H-)和Oligo(dT)18(Fermentas)进行反转录,使用总RNA量为5 μg。qRT-PCR反应体系为10倍稀释的cDNA样品、2×SYBR Mixture(康为世纪,北京)和正反向引物,混合均匀,放入qPCR仪CFX96(Bio-Rad)上进行反应。每个反应使用2个技术重复和3个生物学重复。拟南芥内参基因使用UBQ10和UBC21,使用SPSS进行数据显著性分析(*P≤0.05,**P≤0.01)。
1.2.3 酵母双杂交将AtWRKY31的编码区序列克隆到pGBKT7与pGADT7载体上并进行测序验证。酵母感受态细胞AH109参考Clontech公司的Yeast Protocol Handbook方法制备,将测序正确的质粒通过PEG4000介导的方法依次转入酵母感受态细胞中,随后,将100 μL重悬菌液涂于缺陷型酵母合成培养基(SD-LW)上,培养箱中倒置培养2-3 d。将菌落分别接种于不同的酵母合成缺陷培养基(SD-LW、SD-LWH+5 mmol/L 3-AT和ESD-LWHA)上,放入30℃培养箱中培养2 d,此后需要每天观察菌落生长情况,正常情况下4-5 d后可进行X-gal染色测试。
滴定测试试验,选取二缺合成培养基(SD-LW)上的菌落接种于约4 mL YEPD液体培养基中振荡培养(30℃)12-14 h,随后测定菌液的OD600值,再用无菌水将OD600稀释至0.5,再按梯度进行稀释(1/10、1/100、1/1 000)。最后将稀释后的菌液吸取2 μL点于上述3种不同的缺陷培养基上,于30℃培养箱倒置培养4-5 d,根据菌落生长情况判断互作强度并进行X-gal染色,及时拍照记录。
1.2.4 亚细胞定位和双分子荧光互补实验将AtWRKY31的编码区克隆构建入pCsGFPBT载体,验证无误后转入农杆菌GV3101菌种中,摇菌过夜培养后,重悬于含有乙酰丁香酮的培养基(10 mmol/L MES-KOH(pH 5.6)、10 mmol/L MgCl2和150 μmol/L acetosyringon)中,注射生长32 d的烟草叶片背面。2 d后,取注射后的烟草叶片制片,并用激光共聚焦显微镜(LSM510,Germany)观察绿色荧光。
双分子荧光互补实验,首先将AtWRKY31和与其互作蛋白基因的编码区分别连入35S-SPYNE(R)173和35S-SPYCE(M)载体[20],然后转入农杆菌GV3101中,同上经过培养重悬后进行注射生长32 d的烟草叶片。2 d后取注射后的烟草叶片在激光共聚焦显微镜下观察黄色荧光(Olympus,Japan)。
2 结果 2.1 AtWRKY31的克隆与亚细胞定位分析通过RT-PCR扩增得到AtWRKY31的编码区(CDS)序列,并构建到含有GFP报告基因的pCsGFPBT载体中。经过测序验证,确认获得AtWRKY31的CDS序列,长度为1 614 bp。蛋白序列比对表明,AtWRKY31编码的蛋白含有一个WRKY结构域,具有C2H2型锌指结构,且与WRKY42和WRKY6同属WRKY基因家族的第二亚族中的b组(Group IIb)(图 1)。通过农杆菌介导的方法使WRKY31在烟草叶片中表达,然后使用激光扫描共聚焦显微镜观察WRKY31-GFP融合蛋白的定位情况,发现其仅定位于细胞核内(图 2)。

|
| 图 1 WRKY转录因子家族3个成员氨基酸序列多重比对 保守和非保守氨基酸分别使用黑色和灰色代替,其中最保守的WRKY结构域用下划线标出,锌指结构中保守的氨基酸残基使用三角形标出,序列比对使用ClustalX.83软件 |

|
| 图 2 AtWRKY31-GFP融合蛋白的亚细胞定位 GFP:绿色荧光视野;Bright:明视野;Merge:前两个视野的合并,标尺为50 μm |
为了研究AtWRKY31的调控机制,首先对AtWRKY31的转录活性进行了检测。将AtWRKY31编码区与pRT107-GAL4-BD载体上的GAL4-BD结构域进行融合,报告质粒分别含有5次串联重复的W盒以及连接有CaMV35S启动子的W盒驱动的萤火虫荧光素酶基因,内参质粒为CaMV35S启动子驱动的海肾荧光素酶基因(图 3-A)。将各个报告质粒以及内参质粒一起共转入拟南芥原生质体中,通过测定LUC和REN荧光素酶活性,来分析其转录活性(图 3-B)。从LUC与REN的比值可以看出,AtWRKY31的值明显的低于对照,因此是转录抑制子。

|
| 图 3 AtWRKY31在原生质体内的转录活性分析 A:实验用载体的结构示意图;B:测定LUC和REN荧光素酶活性。不同小写字母代表不同配对间的差异显著(P≤0.05) |
为了了解WRKY31可能的生物学功能,分析了它在多种非生物逆境处理条件下的表达量变化情况(图 4)。在热害处理时,WRKY31的表达量在3个时间点都显著的下调,在低氮条件下,其表达量在2个时间点出现显著降低,另外,在盐害、脱落酸(ABA)、百草枯(MV)、低钾和葡萄糖处理条件下,分别有一个时间点表达量出现显著的变化。
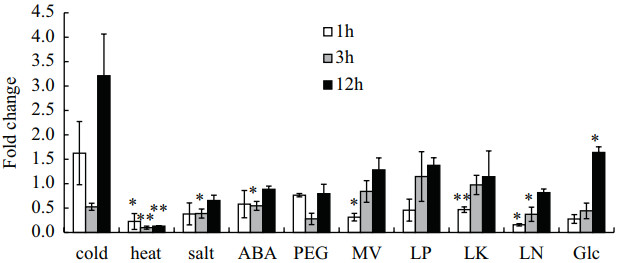
|
| 图 4 qRT-PCR分析WRKY31在各种逆境下的表达量变化 Cold:冷害处理;heat:热害处理;salt:盐处理;ABA:脱落酸;PEG:干旱胁迫;MV:活性氧胁迫;LP:磷缺乏;LK:钾缺乏;LN:氮缺乏;Gln:葡萄糖处理。每个数据都使用3个生物学重复,星号代表与1相比,不同处理下的显著性差异(*P≤0.05;**P≤0.01) |
前期有文献报道,WRKY家族中WRKY30可以与WRKY54和WRKY70通过互作来调控植物叶片的衰老。鉴于AtWRKY31是转录抑制子,通过酵母双杂交检测了AtWRKY31是否与其自身互作,结果验证了我们的假设(图 5,第一行)。此外,筛选了WRKY31与其他在进化上相邻的Group IIb成员之间的互作情况,发现WRKY31可以分别与自身以及WRKY6、WRKY42和自身发生互作,并且在酵母体内验证了AtWRKY31是一个转录抑制子(图 5,最后一行)。
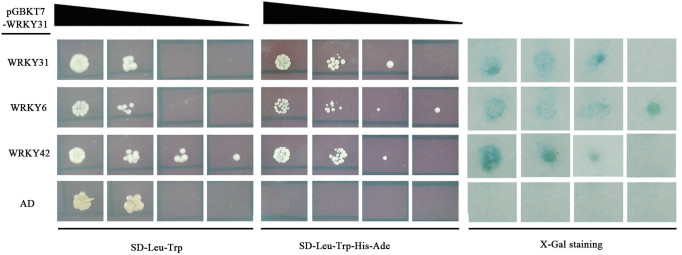
|
| 图 5 酵母双杂交分析拟南芥中WRKY31的互作蛋白 将转入不同质粒的酵母菌种AH109分别涂布于不同的培养基平板上,并使用X-Gal染料进行染色,AD代表pGADT7载体 |
为了进一步证实WRKY31与自身以及其他2个WRKY蛋白互作的真实性,在本氏烟草叶片通过瞬时表达,进行了基于YFP的双分子荧光互补实验(图 6)。可以看出,在WRKY31与自身以及其他互作蛋白配对中均能看到明显的YFP的黄色荧光,而在阴性对照中没有观察到荧光,进一步说明了互作的真实性。

|
| 图 6 双分子荧光互补实验验证拟南芥中WRKY31蛋白与其他成员之间的互作 YFP:黄色荧光视野;Bright:明视野;Merge:2个视野的合并;YFPn:黄色荧光蛋白N端序列;YFPc:黄色荧光蛋白C端序列,标尺为50 μm |
干旱、盐害、热害和营养缺乏等非生物胁迫,严重影响植物的生长发育和作物的产量。已有的报道表明,WRKY转录因子家族的成员在调控植物对生物以及非生物逆境响应方面,起着十分重要的作用[21]。克隆得到的WRKY转录因子家族成员AtWRKY31的功能还没有被报道,但其同一亚族的部分成员已经被研究。比如,AtWRKY6通过抑制AtPHO1的表达来负调控植物对低磷的应答[22]。另外也有报道AtWRKY6参与调控拟南芥中硼和砷酸盐的转运[23]。AtWRKY45和AtWRKY42这2个WRKY转录因子分别调控AtPHT1;1、AtPHT1;1与AtPHO1,进而影响植物体内磷的相对平衡[24, 25]。拟南芥中许多转录因子家族成员之间都存在功能的冗余性,而作为同属于一类亚类家族的WRKY31可能也具有与AtWRKY6和AtWRKY42同样的功能[26]。通过对拟南芥的WRKY31进一步研究,发现其在多种非生物逆境处理的条件下,表达量显著的下调。转录活性分析表明AtWRKY31是一个转录抑制子,但是不具有典型的类似EAR(Ethylene-responsive element binding factor-associated amphiphilic repression)的LxLxL或DLNxxP(x代表任意氨基酸残基)型基序(Motif)[27],暗示可能是具有新型转录抑制结构域的转录抑制子。酵母双杂交和荧光互补实验发现WRKY31可以与WRKY42、WRKY6发生互作,进一步暗示了WRKY31可能参与植物体内的离子转运网络。在植物体内,蛋白互作调控发挥着重要的作用,如在植物抗病应答中,MAPK3/6就是通过磷酸化作用来激活下游蛋白的活性[28]。另外,拟南芥中bHLH转录因子家族中一些成员也通过形成同源或异源二聚体的形式来激活或抑制自身的活性[29, 30]。关于WRKY31是否通过与WRKY42、WRKY6互作来影响植物体内的磷转运或参与其他调控网络,还需要进一步去探究。
4 结论以模式生物拟南芥为研究材料,克隆得到WRKY31,其表达受多种逆境胁迫的抑制。WRKY31定位于核内且作为一个转录抑制子发挥作用;WRKY31蛋白可以与其自身和同一家族的成员WRKY42和WRKY6发生互作。推测WRKY31可能通过与WRKY6和WRKY42转录因子互作的方式来发挥调控作用。
致谢 BiFC载体由Jorg Kudla教授提供。LUC相关载体质粒由Yoko Nishizawa教授(National Institute of Agrobiological Sciences,Japan)和张劲松研究员(中科院遗传发育所)提供,在此表示感谢。| [1] |
Qu LJ, Zhu YX. Transcription factor families in Arabidopsis:major progress and outstanding issues for future research[J]. Current Opinion Plant Biology, 2006, 9(5): 544-549. DOI:10.1016/j.pbi.2006.07.005 |
| [2] |
Wang H, Wang H, Shao H, et al. Recent advances in utilizing transc-ription factors to improve plant abiotic stress tolerance by transgenic technology[J]. Frontiers in Plant Science, 2016, 7(248): 1-13. |
| [3] |
Eulgem T, Rushton PJ, Robatzek S, et al. The WRKY superfamily of plant transcription factors[J]. Trends in Plant Science, 2000, 5(5): 199-206. DOI:10.1016/S1360-1385(00)01600-9 |
| [4] |
Rushton PJ, Somssich IE, Ringler P, et al. WRKY transcription factors[J]. Trends in Plant Science, 2010, 15(5): 247-258. DOI:10.1016/j.tplants.2010.02.006 |
| [5] |
Pandey SP, Somssich IE. The role of WRKY transcription factors in plant immunity[J]. Plant physiology, 2009, 150(4): 1648-1655. DOI:10.1104/pp.109.138990 |
| [6] |
Chen L, Song Y, Li S, et al. The role of WRKY transcription factors in plant abiotic stresses[J]. Biochimica et Biophysica Acta, 2012, 1819(2): 120-128. DOI:10.1016/j.bbagrm.2011.09.002 |
| [7] |
Tao Z, Liu H, Qiu D, et al. A pair of allelic WRKY genes play opposite roles in rice-bacteria interactions[J]. Plant Physiology, 2009, 151(2): 936-948. DOI:10.1104/pp.109.145623 |
| [8] |
Zheng Z, Qamar SA, Chen Z, et al. Arabidopsis WRKY33 transcription factor is required for resistance to necrotrophic fungal pathogens[J]. The Plant Journal, 2006, 48(4): 592-605. DOI:10.1111/tpj.2006.48.issue-4 |
| [9] |
Mao G, Meng X, Liu Y, et al. Phosphorylation of a WRKY transcription factor by two pathogen-responsive MAPKs drives phytoalexin biosynthesis in Arabidopsis[J]. The Plant Cell, 2011, 23(4): 1639-1653. DOI:10.1105/tpc.111.084996 |
| [10] |
Jiang Y, Deyholos MK. Functional characterization of Arabidopsis NaCl-inducible WRKY25 and WRKY33 transcription factors in abiotic stresses[J]. Plant Molecular Biology, 2009, 69(1/2): 91-105. |
| [11] |
Li S, Fu Q, Chen L, et al. Arabidopsis thaliana WRKY25, WRKY26, and WRKY33 coordinate induction of plant thermotolerance[J]. Planta, 2011, 233(6): 1237-1252. DOI:10.1007/s00425-011-1375-2 |
| [12] |
Miao Y, Smykowski A, Zentgraf U. A novel upstream regulator of WRKY53 transcription during leaf senescence in Arabidopsis thaliana[J]. Plant Biology(Stuttg), 2008, 10(1): 110-120. |
| [13] |
Grunewald W, Smet ID, Daniel R, et al. Transcription factor WRKY23 assists auxin distribution patterns during Arabidopsis root development through local control on flavonol biosynthesis[J]. Proceedings of the National Academy Sciences of the USA, 2011, 109(1): 1554-1559. |
| [14] |
Johnson CS, Kolevski B, Smyth DR. TRANSPARENT TESTA GLABRA2, a trichome and seed coat development gene of Arabidopsis, encodes a WRKY transcription factor[J]. The Plant Cell, 2002, 14(6): 1359-1375. DOI:10.1105/tpc.001404 |
| [15] |
Chang IF, Curran A, Woolsey R, et al. Proteomic profiling of tandem affinity purified 14-3-3 protein complexes in Arabidopsis thaliana[J]. Proteomics, 2009, 9(11): 2967-2985. DOI:10.1002/pmic.v9:11 |
| [16] |
Lai Z, Li Y, Wang F, et al. Arabidopsis sigma factor binding proteins are activators of the WRKY33 transcription factor in plant defense[J]. The Plant Cell, 2011, 23(10): 3824-3841. DOI:10.1105/tpc.111.090571 |
| [17] |
Besseau S, Li J, Palva ET. WRKY54 and WRKY70 co-operate as negative regulators of leaf senescence in Arabidopsis thaliana[J]. Journal of Experimental Botany, 2012, 63(7): 2667-2679. DOI:10.1093/jxb/err450 |
| [18] |
Yang G, Zhang W, Liu Z, et al. Both JrWRKY2 and JrWRKY7 of Juglans regia mediate responses to abiotic stresses and abscisic acid through formation of homodimers and interaction[J]. Plant Biology(Stuttg), 2017, 19(2): 268-278. |
| [19] |
Niu F, Wang C, Yan J, et al. Functional characterization of NAC55 transcription factor from oilseed rape(Brassica napus L.)as a novel transcriptional activator modulating reactive oxygen species accumulation and cell death[J]. Plant Molecular Biology, 2016, 92(1/2): 89-104. |
| [20] |
Waadt R, Kudla J. In planta visualization of protein interactions using bimolecular fluorescence complementation(BiFC)[J]. Nature Protocols, 2008, 3(4): 588. DOI:10.1038/nprot.2008.16 |
| [21] |
Bakshi M, Oelmuller R. WRKY transcription factors:Jack of many trades in plants[J]. Plant Signaling and Behavior, 2014, 9(2): e27700. DOI:10.4161/psb.27700 |
| [22] |
Chen YF, Li LQ, Xu Q, et al. The WRKY6 transcription factor modulates PHOSPHATE1 expression in response to low Pi stress in Arabidopsis[J]. The Plant Cell, 2009, 21(11): 3554-3566. DOI:10.1105/tpc.108.064980 |
| [23] |
Castrillo G, Sanchez-bermejo E, Lorenzo L, et al. WRKY6 transcription factor restricts arsenate uptake and transposon activation in Arabidopsis[J]. The Plant Cell, 2013, 25(8): 2944-2957. DOI:10.1105/tpc.113.114009 |
| [24] |
Su T, Xu Q, Zhang FC, et al. WRKY42 modulates phosphate homeostasis through regulating phosphate translocation and acquisition in Arabidopsis[J]. Plant Physiology, 2015, 167(4): 1579-1591. DOI:10.1104/pp.114.253799 |
| [25] |
Wang H, Xu Q, Kong YH, et al. Arabidopsis WRKY45 transcription factor activates PHOSPHATE TRANSPORTER1;1 expression in response to phosphate starvation[J]. Plant Physiology, 2014, 164(4): 2020-2029. DOI:10.1104/pp.113.235077 |
| [26] |
Wu WS, Lai FJ. Functional redundancy of transcription factors explains why most binding targets of a transcription factor are not affected when the transcription factor is knocked out[J]. BMC Systems Biology, 2015, 9(S6): 1-9. |
| [27] |
Kagale S, Links MG, Rozwadowski K. Genome-wide analysis of ethylene-responsive element binding factor-associated amphiphilic repression motif-containing transcriptional regulators in Arabidopsis[J]. Plant Physiology, 2010, 152(3): 1134. |
| [28] |
Li G, Meng X, Wang R, et al. Dual-level regulation of ACC synthase activity by MPK3/MPK6 cascade and its downstream WRKY transcription factor during ethylene induction in Arabidopsis[J]. PLoS Genetic, 2012, 8(6): e1002767. DOI:10.1371/journal.pgen.1002767 |
| [29] |
Zhang J, Liu B, Li M, et al. The bHLH transcription factor bHLH104 interacts with IAA-LEUCINE RESISTANT3 and modulates iron homeostasis in Arabidopsis[J]. The Plant Cell, 2015, 27(3): 787-805. DOI:10.1105/tpc.114.132704 |
| [30] |
Rybel B, Moller B, Yoshida S, et al. A bHLH complex controls embryonic vascular tissue establishment and indeterminate growth in Arabidopsis[J]. Developmental Cell, 2013, 24(4): 426-437. DOI:10.1016/j.devcel.2012.12.013 |





