2. 沈阳师范大学生命科学学院,沈阳 110034;
3. 沈阳师范大学教学实验中心,沈阳 110034
2. College of Life Science, Shenyang Normal University, Shenyang 110034;
3. Experimental Teaching Center, Shenyang Normal University, Shenyang 110034
近年来,有关位点特异性核酸酶(Site-specific nuclease,SSN)的基础理论研究取得了突破性进展,使精准基因编辑技术在生物学、医学和农业等领域得到了广泛应用,也取得了令人瞩目的成就。位点特异性核酸酶在生物体的基因编辑中提供了十分精准有效的DNA切割方法[1],其中,锌指核酸酶(Zine finger endonuclease,ZFN)以及类转录激活因子效应物核酸酶(Transcription activator-like effector nuclease,TALENs)已被广泛应用于基因工程技术中。这两种核酸酶的切割原理相同,均是将DNA结合蛋白与核酸内切酶Fok Ⅰ融合完成双链特异性切割,但两种酶DNA结合结构域较为复杂,操作上技术难度相对大,耗时长且容易脱靶[2-4],成为制约其发展的障碍。2013年,Cathomen[5]基于细菌的一种获得性免疫系统改造而成人工核酸内切酶CRISPR Cas 9,在真核和原核生物中进行精准基因编辑尝试,获得巨大成功。这种全新的基因组定点编辑技术以制作简单、成本低、作用高效[5]等优势迅速发展,并成功应用于各种原核生物和真核生物中,包括大肠杆菌[6]、酿酒酵母[7]、链霉菌属[8]、高等植物[9]、家蚕[10]、果蝇[11]和人类细胞[12-14]等。本文从CRIS-PR/Cas的来源、结构、作用机理及影响CRISPR/Cas打靶效率的因素等方面进行总结,旨为深刻理解其工作原理和在精准基因编辑中应用提供参考。
1 CRISPR/Cas系统的研究历史1987年,日本科学家在大肠杆菌K12的碱性磷酸酶基因附近发现串联间隔重复序列[15],但由于其功能无法确定,并未引起科学家的注意。1995年,西班牙科学家Mojica等[16]在地中海极嗜盐菌(Haloferax mediaterranei)和沃尔卡尼极嗜盐菌(Haloferax volcanii)中第二次发现了类似的结构。Mojica敏感地意识到在亲缘关系如此远的微生物中存在如此相似的结构,可能有着尚未发现的细胞功能,引起了许多研究人员的极大关注。2002年,Coffey等和Jansen等[17-18]把串联重复序列正式命名为成簇间隔规律短回文重复序列(Clustered regularly short palindromic repeats,CRISPR),但其生物学功能仍未阐明。2005年,法国的Bolotin和Vergnaud、西班牙的Mojica三位科学家领导的3个实验室[19-21]几乎同时报道CRISPR中的间隔序列(Spacer)与侵染细菌的病毒或噬菌体具有很高的同源性,并推测CRISPR系统可能与细菌的免疫防御机制有关。2007年,Barrangou等[22]首次发现嗜热链球菌(Streptococcus thermophilus)能够通过CRISPR/Cas系统抵抗噬菌体的入侵,证实了CRISPR/Cas可以获得性的提供细菌对抗噬菌体的能力。2012年,Sternberg等[23]发现了Ⅱ型CRISPR系统,该系统中只需要一个160 kD的Cas9蛋白,同时利用RNA就可以完成识别和切割靶向DNA序列。这一防御系统的发现激发了科研工作者对于其在基因编辑作用中的研究热情,利用和优化CRISPR系统对基因进行精准编辑的探索研究越来越多,科研成果也层出不穷,精准编辑功能在细菌、真菌和哺乳动物中都得到了认证,是新一代基因编辑技术。
2 CRISPR/Cas系统的基本结构随着生物信息学的发展及对越来越多微生物基因组测序结果进行分析,人们发现在已知物种中有90%的古细菌和40%的细菌的基因组中含有CRISPR位点,而且绝大多数的细菌和古细菌中至少含有一个CRISPR基因座,极少数的古细菌中含有上百个CRISPR基因座,说明了CRISPR/Cas系统对于细菌免疫防御机制是十分重要的[24]。CRISPR/Cas基因座由3个部分组成,5'端为tracrRNA基因,中间是Cas家族蛋白,3'端为CRISPR基因座,主要由一个前导序列、23-50 bp的重复序列和间隔序列串联组成,间隔序列由17-84 bp组成,平均长度约为36 bp。
Cas蛋白来自于细菌具有切割DNA的活性蛋白,除了探究已知的细菌Cas蛋白差异之外,环境中存在着很多不为人知或不可培养的微生物种类,研究人员从土壤,地下水,酸性矿泉水等富含微生物的环境中取材,将这些微生物的宏基因组信息与CRISPR和Cas1序列进行比对,发现了两类新型的CRISPR-Cas系统,将其命名为CRISPR-CasX和CRISPR-CasY,其中CasX蛋白约有980个氨基酸,CasX蛋白约有1 200个氨基酸[25]。可见CRISPR-Cas系统在微生物领域存在的普遍性,具有巨大的基因编辑潜能。
CRISPR-Cas系统中Cas基因进化速度最快,根据单亚基和多亚基的crRNA效应复合物定义可以将CRISPR-Cas系统分为两大类:ClassⅠ和ClassⅡ。ClassⅠ包括3个亚型:TypeⅠ、TypeⅢ和TypeⅣ,ClassⅡ包括两个亚型:TypeⅡ和TypeⅤ[26]。研究表明,ClassⅡ效应蛋白Cpf1是一类新型的CRISPR-Cas DNA核酸内切酶,Cpf1只包含RuvC结合结构域但不包含HNH结合结构域[27]。Cpf1可以介导基因组编辑的翻译后调节,Cas9和Cpf1的gRNA序列有明显差异,研究人员设计同时带有Cpf1的5'-支架序列和Cas9的3'-端序列融合版的引导RNA(fgRNA),fgRNA能够通用于Cpf1和Cas9两种类型的核酸酶[28]。Zhang等[29]发现具有基因编辑潜力的C2c2位于Leptotrichia shahii中ClassⅡ系统的TypeⅥ上。研究表明C2c2能够调控RNA指导的单链RNA的断裂,深度测序发现了C2c2是RNA指导的ssRNA发生断裂效应物。Zhang等[30]证明了来自化脓链球菌的Cas9酶(SpCas9)和来自金黄色葡萄球菌的Cas9核酸酶(SaCas9)具有相当的DNA靶向精确度。这些高效Cas9蛋白的发现,对靶向基因表达调控提供了更加高效的基因编辑方法。
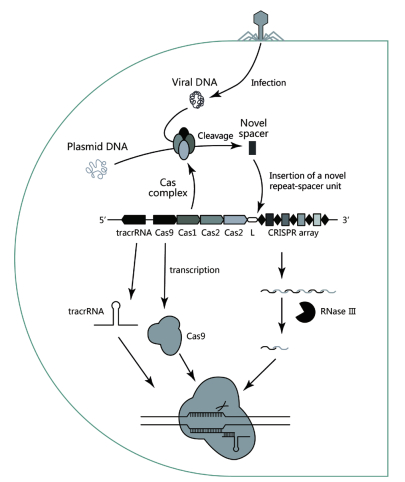
3 Ⅱ型CRISPR/Cas系统的作用机理Ⅱ型CRISPR/Cas系统行使功能主要分为3个阶段(图 1):即新间隔序列的获取、CRISPR位点的转录与加工、CRISPR/Cas系统对外源DNA的降解。
|
| 图 1 Ⅱ型CRISPR-Cas9系统抵御外源噬菌体的获得性免疫机制 |
第一阶段新间隔序列(Spacer)的获取可分为3个过程,当噬菌体首次侵染携有CRISPR系统的细菌中时,宿主首先扫描并识别入侵核酸的PAM序列,将临近PAM序列的外源DNA作为候选的原间隔序列(Protospacer)。一般来说,临近间隔序列的几个碱基比较保守,被称为(Protospacer adjacent motifs,PAM)序列,通常为NGG[31];接着在Cas1和Cas2等蛋白质的作用下,将入侵DNA切割成长度约为48 bp的核酸片段,最后通过相关蛋白质的作用,将其中一个核酸片段整合到前导序列和第一个重复序列之间,形成新的间隔序列,与这个序列同源的病毒DNA称为原间隔序列。
第二阶段,当相同的外源DNA再次侵入时,CRISPR基因座首先转录成长的前体CRISPR RNA(pre-crRNA),然后在RNaseⅢ的参与下被切割为长度不等的核酸小片段,即包含一段间隔序列和部分重复序列的成熟crRNA[32]。crRNA与反转录激活RNA(tracrRNA)通过碱基互补配对与Cas9蛋白形成靶向复合物crRNP,其在降解外源DNA的阶段起到重要作用[13, 33-34]。
第三阶段是对外源DNA的干扰。复合体crRNP通过间隔序列寻找到与外源DNA互补的靶序列,此时Cas9起到切割双链DNA的作用。在Ⅱ型CRISPR系统中,Cas9蛋白的HNH与RuvC核酸酶结构域能够分别切割间隔序列的互补链与非互补链,导致双链DNA断裂(Double strand breaks,DSBs)被降解,从而抵御了外源DNA的入侵[35]。
4 CRISPR/Cas9系统的应用优势近年来,有关利用CRISPR/Cas9进行精准基因编辑的应用研究发展十分迅速,已在微生物、植物、动物及癌症基因治疗等领域得到了广泛的应用,与以前的基因编辑方法相比,展现出巨大应用潜力和应用优势。
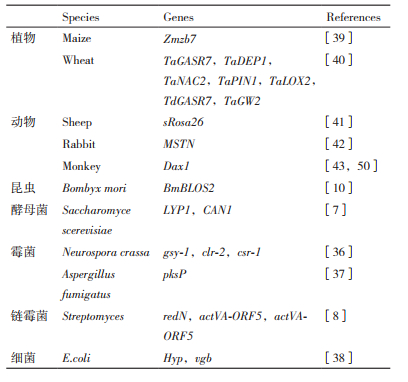
4.1 CRISPR/Cas9系统在生物育种中的应用在微生物学研究领域,应用该技术在粗糙脉孢菌(Neurospora crassa)[36]中将clr-2启动子替换为β微管蛋白启动子增强了纤维素酶的表达;Fuller等[37]在烟曲霉菌(Aspergillus fumigatus)中高效靶标酮化合物合酶基因,并进一步验证了组成型表达的Cas9核酸酶本身对烟曲霉生长和毒力没有危害作用;Wu等[38]成功利用CRISPR/Cas9技术在产气肠杆菌中敲除NADH脱氢酶基因簇nuoC/D和nuoC/D/E基因,得到了双敲除和三敲除菌株,在摇瓶水平和发酵罐水平表征菌株,结果表明在厌氧发酵罐中氢气的产量是原始菌株的1.95倍,产氢量得到了很大的提高,为生物制氢提供了新的思路。
在植物学研究领域,Feng等[39]将玉米的标记基因Zmzb7进行改造优化产量,结果显示CRISPR/Cas9系统在玉米的常染色质和异染色质区域都可以进行高效的基因编辑;Zhang等[40]通过CRISPR/Cas9瞬时表达获得了初代非转基因纯合子小麦突变株;Tian等[41]成功敲除了西瓜中的八氢番茄红素脱氢酶,结果显示CRISPR/Cas9系统在西瓜中的基因编辑效率达到100%。这些研究表明CRISPR/Cas9基因编辑系统为植物种子改良提供了新路径。
在动物学研究领域,利用CRISPR/Cas9技术已经成功对绵羊[42]、兔子[43]等中小型哺乳类动物进行了基因编辑,这些研究成果为以中小型动物模型,探究人类疾病的发病机制提供参考。为了获得患有蕾特氏症的动物模型,科学家将恒河猴(Macaca mulatta)和食蟹猴(Macaca fascicularis)的MESP2基因成功敲除,为人类研究神经系统疾病提供了实验模型[44]。
4.2 CRISPR/Cas9系统在基因治疗中的应用基因治疗是治疗病毒性疾病的热点,许多癌症都是由病毒引起,如肝癌由乙肝病毒引起,宫颈癌由人乳头瘤病毒引起,艾滋病因感染HⅣ-1病毒引起等。CRISPR/Cas9来源于细菌自身的防御系统[45],用该系统抵御并清除外来病毒感染抑制病毒生长甚至可治愈病症。Khalili等[46]发现Cas9/LTR-gRNA在细胞中表达时能够在HⅣ-1病毒整合前明显阻止其感染细胞。该项技术对于治愈病毒感染的疾病有着重要的指导意义。Maddalo等[47]运用CRISPR/Cas9基因编辑技术在小鼠体内敲除了EML4-ALK融合基因,对于晚期非小细胞肺癌临床治疗提供了新的方向。Xie等[48]敲除了血红蛋白β(HBB)基因对人类β-地中海贫血症的发生机制进行了探究。值得一提的是,Rong等[49]证明Cas9切口酶可以精确靶标人体胚胎干细胞中的长链DNA模板,其编辑效率大约在5%。这预示着该技术在不久的将来会对优化人类受精卵起到深远的影响。
表 1例举了部分已成功利用CRISPR/Cas9进行基因编辑的实例。从这些例子及研究者的文献研究报告中可以看出,CRISPR/Cas9基因编辑系统不仅设计简单、使用范围广,而且操作步骤简洁、实验周期短、验证过程容易,具有十分广阔的应用前景。作为新一代的基因编辑工具,在解析基因功能,基因新性功能的探索,疾病的基因治疗,生物基因改造等方面是强有力的方法,是探索生命的奥秘强有力的分子工具。
CRISPR/Cas9系统是一种方便快捷的基因改造方法,其通过sgRNA中的N20序列寻找并识别靶位点,如果需要进行多位点修饰,可以设计多条sgRNA[51-53]。但是最新的研究表明sgRNA的错配为5个碱基时,切割仍能够发生,这就导致了CRISPR/Cas9系统存在较高的脱靶效应[54-55]。为了降低CRISPR/Cas9的脱靶效应,研究人员对影响CRISPR/Cas9打靶效率的因素进行了深入研究并提出了一系列解决方法。
5.1 影响CRISPR/Cas9系统脱靶效应的因素及解决方法sgRNA结构和组成能够引起脱靶效应[56]。研究表明增加[57]或减少[58]2-3个核苷酸的sgRNA能够减少错配率从而增加靶序列的专一性。John等[59]创造了新的sgRNA设计规则并建立了人体和小鼠的全基因库,用基因库设计出的sgRNA能够有效减少CRISPR/Cas9系统的脱靶效率。
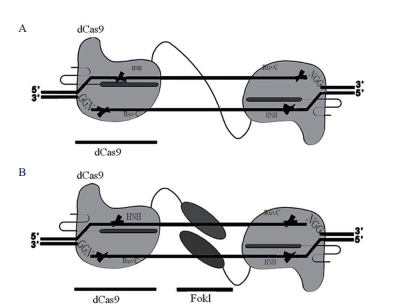
Cas9蛋白的结构对打靶效率也有着重要的影响。野生型Cas9蛋白具有切割双链DNA,的活性,形成双链断裂(Double-strand Breaks,DSBs)从而增加脱靶效应的几率。Frock等[60]将Cas9蛋白改造为Cas9 nickase(nCas9),nCas9能够使Cas9蛋白的HNH或RuvC核酸酶结构域失活而形成单链断裂(Single-strand Breaks,SSBs),单链缺口会通过高保真的碱基切除修复方式进行修复以减少脱靶效应;Qi等[61]把Cas9蛋白改造为Dead Cas9(dCas9)(图 2-A),dCas9能够使HNH和RuvC两个活性位点均失活,将dCas9与sgRNA在细胞中共表达,sgRNA介导dCas9与DNA结合可以抑制基因的起始转录从而减少脱靶效应。科学家还发现将dCas9与FokⅠ核酸酶结合形成融合蛋白(FokⅠ-dCas9)(图 2-B),可以增加CRISPR/Cas9的靶专一性,通过一对sgRNA和dCas9s识别靶序列,此时FokⅠ二聚体切割DNA,这种方法虽然能降低脱靶效应,但同时也会降低其打靶活力,并且对于FokⅠ二聚体来说N20的长度对其活力有着十分重要的影响,因此限制了基因组中潜在的靶序列数量[62-63]。Slaymaker等[64]采用有针对性的深测序和无偏全基因组脱靶效应分析评估Cas9的切割效率,证明了增强型化脓链球杆菌Cas9突变体(eSpCas9)能够减弱脱靶效应并且更加精确地进行切割。
|
| 图 2 CRISPR-Cas9介导的基因编辑机制 A:dCas9使RuvC和HNH两个结合结构域失活;B:dCas9-FokⅠ融合形成FokⅠ二聚体切割双链 |
此外,PAM序列的长度也是解决脱靶效应的策略之一。金黄色酿脓葡萄球菌(Staphylococcus aureus)Cas9蛋白(SaCas9)识别的PAM区为的NNGRRT(NNGAAT、NNGAGT、NNGGAT和NNGGGT),嗜热链球菌(Streptococcus thermophilus)Cas9蛋白(StCas9)的PAM序列为NGGNG和NNAGAAW,脑膜炎双球菌(Neisseria meningitidis)Cas9蛋白(NmCas9)的PAM序列为NNNNGATT,实验表明这些较长的PAM序列能够有效地减少脱靶效应[65-67]。这表明更长的PAM序列在全基因组中,特异性更高,潜在的脱靶位点少,为进一步增强靶序列的专一性创造了条件。
5.2 同源重组率对CRISPR/Cas9打靶效应的影响当Cas9蛋白切割双链DNA形成双链断裂后,细胞会采取非同源末端连接机制(Nonhomologous end joining,NHEJ)或同源重组(Homologous recombination,HR)修复受损的细胞,但是非同源末端连接修复机制会引发由碱基的插入或删除从而导致基因突变,同源重组修复机制能够精确的对细胞进行修复,增强CRISPR/Cas9系统的特异性。在真核生物中研究人员发现KU70、KU80和DNA连接酶Ⅳ是NHEJ通路中的关键因子。德国MDC研究所的研究人员使用DNA连接酶Ⅳ抑制剂SCR7进行了基因沉默,并表达了腺病毒蛋白E1B55K和E4orf6,抑制KU70和DNA连接酶Ⅳ能够将同源定向修复(Homology directed repair,HDR)效率提高4-5倍。在人类和小鼠细胞系中腺病毒蛋白与Cas9系统共表达时,HDR效率提高了8倍,大幅度抑制了NHEJ活性[68]。Whitehead研究所的研究人员在对受精卵进行基因编辑时添加了SCR7抑制剂,这项研究将CRISPR/Cas的HDR率提高了19倍[69]。另一方面,研究人员还发现供体DNA的长度也会影响同源重组率。Lin等[70]发现至少需要15 nt的同源臂长度才能实现同源重组。而Rong等[71]比较了引入长度为1 kb的上下游同源臂与引入单侧同源臂在人类胚胎干细胞中的同源重组效率,实验表明引入标靶基因的双侧通同源臂后的HDR效率更高。
6 CRISPR/Cas9系统应用前景特别是近年来研究人员利用CRISPR/Cas9系统进行精准基因编辑,也取得了令人瞩目的研究成果,无数成功的例子和高水平文章的发表显示了其强大应用潜力。尽管CRISPR/Cas9系统仍存着如打靶效率低、靶专一性不强等技术局限,但仍说明CRISPR/Cas9系统仍需完善,有巨大的研究空间需要探索。许多研究人员从CRISPR/Cas9系统组成结构分析和攻关尝试,改良sgRNA的结构,增强靶特异性是最直接的手段;也有研究人员致力于探索Cas9的结构及功能,筛选和改造特异性更强的Cas蛋白,来解决脱靶效应问题,同时寻找提高同源重组率的方法对提高打靶效率也有一定的作用。相信通过研究人员的努力和尝试,及随着生物技术的发展,CRISPR/Cas9系统应用中存在的技术局限,终将被攻克,CRISPR/Cas新一代基因编辑系统在农业,医学及生物等各个领域将具有更广阔的应用前景,发挥更重要的作用。
| [1] |
Gaj T, Gersbach CA, Barbas CF. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering[J]. Trends Biotechnol, 2013, 31: 397-405. DOI:10.1016/j.tibtech.2013.04.004 |
| [2] |
Capecchi MR. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century[J]. Nat Rev Genet, 2005, 6(6): 507-512. DOI:10.1038/nrg1619 |
| [3] |
Carroll D. Genome engineering with targetable nucleases[J]. Annu Rev Biochem, 2014, 83: 409-439. DOI:10.1146/annurev-biochem-060713-035418 |
| [4] |
Yu YJ, Bradley A. Engineering chromosomal rearrangements in mice[J]. Nat Rev Genet, 2001, 2(10): 780-790. DOI:10.1038/35093564 |
| [5] |
Mussolino C, Cathomen T. RNA guides genome engineering[J]. Nat Biotechnol, 2013, 31(3): 208-209. DOI:10.1038/nbt.2527 |
| [6] |
Jiang W, Bikard D, Cox D, et al. Marraffini LA. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nat Biotechnol, 2013, 31: 233-239. DOI:10.1038/nbt.2508 |
| [7] |
DiCarlo JE, Norville JE, Mali P, et al. Church GM. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems[J]. Nucleic Acids Res, 2013, 41: 4336-4343. DOI:10.1093/nar/gkt135 |
| [8] |
Cobb RE, Wang Y, Zhao H. High-efficiency multiplex genome editing of Streptomyces species using an engineered CRISPR/Cas system[J]. ACS Synthetic Biology, 2014, 4(6): 723-728. |
| [9] |
Shan Q, Wang Y, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system[J]. Nat Biotechnol, 2013, 31: 686-688. DOI:10.1038/nbt.2650 |
| [10] |
Wang Y, Li Z, Xu J, et al. The CRISPR/Cas System mediates efficient genome engineering in Bombyx mori[J]. Cell Res, 2013, 23: 1414-1416. DOI:10.1038/cr.2013.146 |
| [11] |
Yu Z, Ren M, Wang Z, et al. Highly efficient genome modifications mediated by CRISPR/Cas9 in Drosophila[J]. Genetics, 2013, 195: 289-291. DOI:10.1534/genetics.113.153825 |
| [12] |
Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339: 819-823. DOI:10.1126/science.1231143 |
| [13] |
Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339: 823-826. DOI:10.1126/science.1232033 |
| [14] |
Zhang Q, Rho M, Tang H, et al. CRISPR-Cas systems target a diverse collection of invasive mobile genetic elements in human microbiomes[J]. Genome Biology, 2013, 14(4): 1-15. |
| [15] |
Ishino Y, Shinagawa H, Makino K, et al. Nucleotide sequence of the lap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product[J]. J Bacteriol, 1987, 169(12): 5249-5433. |
| [16] |
Mojica FJ, Ferrer C, Juez G, et al. Long stretches of short tandem repeats are present in the largest replicons of the Archaea Haloferax mediterranei and Haloferax volcanii and could be involved in replicon partitioning[J]. Mol Microbiol, 1995, 17(1): 85-93. DOI:10.1111/mmi.1995.17.issue-1 |
| [17] |
Coffey A, Ross RP. Bacteriophage-resistance systems in dairy starter strains, molecular analysis to application[J]. Anton Leeuw, 2002, 82(1): 303-321. |
| [18] |
Jansen R, Embden JDAV, Gaastra W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Mol Microbiol, 2002, 43(6): 1565-1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [19] |
Bolotin A, Quinquis B, et al. Clustered regularly interspaced short palindrome repeats(CRISPRs)have spacers of extrachromosomal origin[J]. Microbiology, 2005, 151: 2551-2561. DOI:10.1099/mic.0.28048-0 |
| [20] |
Pourcel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies[J]. Microbiology, 2005, 151: 654-663. |
| [21] |
Mojica FJ, Díez-Villaseñor C, García-Martinez J, et al. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements[J]. Journal of Molecular Evolution, 2005, 60(2): 174-182. DOI:10.1007/s00239-004-0046-3 |
| [22] |
Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007, 315: 1709-1712. DOI:10.1126/science.1138140 |
| [23] |
Sternberg SH, Haurwitz RE, Doudna JA. Mechanism of substrate selection by a highly specific CRISPR endoribonuclease[J]. RNA, 2012, 18(4): 661-672. DOI:10.1261/rna.030882.111 |
| [24] |
Burstein D, Harrington LB, et al. New CRISPR-Cas syst-ems from uncultivated microbes[J]. Nature, 2017, 542(7640): 237-241. |
| [25] |
Grissa I, Vergnauud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J]. BMC Bioinformatics, 2007, 8: 172. DOI:10.1186/1471-2105-8-172 |
| [26] |
Sergey S, Abudayyeh OO, Makarova KS, et al. Discovery and Functional Characterization of Diverse Class 2 CRISPR-Cas Systems[J]. Molecular Cell, 2015, 60(3): 385. DOI:10.1016/j.molcel.2015.10.008 |
| [27] |
Schunder E, Rydzewski K, Grunow R, et al. First indication for a functional CRISPR/Cas system in Francisella tularensis[J]. Int J Med Microbiol, 2013, 303(2): 51-60. DOI:10.1016/j.ijmm.2012.11.004 |
| [28] |
Kweon J, Jang AH, Kim DE, et al. Fusion guide RNAs for orthogonal gene manipulation with Cas9 and Cpf1[J]. Nat Commun, 2017, 8(1): 1723. DOI:10.1038/s41467-017-01650-w |
| [29] |
Abudayyeh OO, Gootenberg JS, Konermann S, et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector[J]. Science, 2016, 353(6299): aaf5573. DOI:10.1126/science.aaf5573 |
| [30] |
Ran FA, Cong L, et al. In vivo genome editing using Staphylococcus aureus Cas9[J]. Nature, 2015, 520(7546): 186. DOI:10.1038/nature14299 |
| [31] |
Gasiunas G, Barrangou R, Horvath P, et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria[J]. Proc Natl Acad Sci USA, 2012, 109(39): E2579-2586. DOI:10.1073/pnas.1208507109 |
| [32] |
Pougach K, Semenova E, Bogdanova E, et al. Transcription, processing and function of CRISPR cassettes in Escherichia coli[J]. Mol Microbiol, 2010, 77(6): 1367-1379. DOI:10.1111/j.1365-2958.2010.07265.x |
| [33] |
Sander JD, Joung JK. CRISPR-Cas systems for editing regulating and targeting genomes[J]. Nat Biotechnol, 2014, 32(4): 347-355. DOI:10.1038/nbt.2842 |
| [34] |
Nishimasu H, Ran FA, Hsu PD, et al. Crystal structure of Cas9 in complex with guide RNA and target DNA[J]. Cell, 2014, 156(5): 935-949. DOI:10.1016/j.cell.2014.02.001 |
| [35] |
Jinek M, Chylisnki K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [36] |
Matsu-ura T, Baek M, Kwon J, et al. Efficient gene editing in Neurospora crassa with CRISPR technology[J]. Fungal Biology and Biotechnology, 2015, 2(1): 1-7. DOI:10.1186/s40694-014-0011-x |
| [37] |
Fuller KK, Chen S, Loros JJ, et al. Development of the CRISPR/Cas9 system for targeted gene disruption in Aspergillus fumigatus[J]. Eukaryotic Cell, 2015, 14(11): 1073-1080. DOI:10.1128/EC.00107-15 |
| [38] |
Wu Y, Hao Y, Wei X, et al. Impairment of NADH dehydrogenase and regulation of anaerobic metabolism by the small RNA RyhB and NadE for improved biohydrogen production in Enterobacter aerogenes[J]. Biotechnology for Biofuels, 2017, 10(1): 248. DOI:10.1186/s13068-017-0938-2 |
| [39] |
Feng C, Yuan J, Wang R, et al. Efficient targeted genome modification in maize using CRISPR/Cas9 system[J]. Journal of Genetics and Genomics, 2016, 43(1): 37-43. DOI:10.1016/j.jgg.2015.10.002 |
| [40] |
Zhang Y, Liang Z, Zong Y, et al. Efficient and transgene-free genome editing in wheat through transient expression of CRISPR/Cas9 DNA or RNA[J]. Nat Commun, 2016, 7: 12617. DOI:10.1038/ncomms12617 |
| [41] |
Tian S, Jiang L, Gao Q, et al. Efficient CRISPR/Cas9-based gene knockout in watermelon[J]. Plant Cell Reports, 2016, 36: 1-8. |
| [42] |
Wu M, Wei C, Lian Z, et al. Rosa26-targeted sheep gene knock-in via CRISPR-Cas9 system[J]. Sci Rep, 2016, 6: 24360. DOI:10.1038/srep24360 |
| [43] |
Lv Q, Yuan L, Deng J, et al. Efficient generation of myostatin gene mutated rabbit by CRISPR/Cas9[J]. Sci Rep, 2016, 6: 25029. DOI:10.1038/srep25029 |
| [44] |
Liu H, et al. TALEN-mediated gene mutagenesis in rhesus and cyn-omolgus monkeys[J]. Cell Stem Cell, 2014, 14(3): 323-328. DOI:10.1016/j.stem.2014.01.018 |
| [45] |
Lu XJ, Xue HY, Ke ZP, et al. CRISPR-Cas9: a new and promising player in gene therapy[J]. Journal of Medical Genetics, 2015, 52(5): 289-296. DOI:10.1136/jmedgenet-2014-102968 |
| [46] |
Khalili K, Kaminski R, Gordon J, et al. Genome editing strategies: potential tools for eradicating HIV-1/AIDS[J]. Journal of Neurovirology, 2015, 21(3): 310-321. DOI:10.1007/s13365-014-0308-9 |
| [47] |
Maddalo D, Manchado E, Concepcion CP, et al. In vivo engineering of oncogenic chromosomal rearrangements with the CRISPR/Cas9 system[J]. Nature, 2015, 524(7566): 502. |
| [48] |
Xie F, Ye L, Chang JC, et al. Seamless gene correction of β-thalassemia mutations in patient-specific iPSCs using CRISPR/Cas9 and piggyBac[J]. Genome Res, 2014, 24(9): 1526. DOI:10.1101/gr.173427.114 |
| [49] |
Rong Z, Zhu S, Yang X, et al. Homologous recombination in human embryonic stem cells using CRISPR/Cas9 nickase and a long DNA donor template[J]. Protein & Cell, 2014, 5(4): 258. |
| [50] |
Kang Y, Zheng B, Shen B, et al. CRISPR/Cas9-mediated Dax1 knockout in the monkey recapitulates human AHC-HH[J]. Human Molecular Genetics, 2015, 24(25): 7255-7264. DOI:10.1093/hmg/ddv425 |
| [51] |
Qi L, Larson M, Gilbert L, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152: 1173-1183. DOI:10.1016/j.cell.2013.02.022 |
| [52] |
Cho SW, Kim S, Kim JM, et al. Targeted genome enginerring in human cells with the Cas9 RNA-guide guided endounuclease[J]. Nat Biotechnol, 2013, 31: 230-232. DOI:10.1038/nbt.2507 |
| [53] |
Wang H, Yang H, Shivalila C, et al. One-step generation of mice carting mutations in multiple genes by CRISPR/Cas-mediated genome engineering[J]. Cell, 2013, 153: 910-918. DOI:10.1016/j.cell.2013.04.025 |
| [54] |
Hsu PD, Scott DA, et al. DNA targeting specificity of RNA-guided Cas9 nucleases[J]. Nat Biotechnol, 2013, 31: 827-832. DOI:10.1038/nbt.2647 |
| [55] |
Pattanayak V, Lin S, Guilinger JP, et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity[J]. Nat Biotechnol, 2013, 31: 839-843. DOI:10.1038/nbt.2673 |
| [56] |
Cho SW, Kim S, Kim Y, et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases[J]. Genome Res, 2014, 24: 132-141. DOI:10.1101/gr.162339.113 |
| [57] |
Kim D, Bae S, Park J, et al. Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells[J]. Nature Methods, 2015, 12(3): 237-243. DOI:10.1038/nmeth.3284 |
| [58] |
Fu Y, Sander JD, Reyon D, et al. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs[J]. Nat Biotechnol, 2014, 32: 279-284. DOI:10.1038/nbt.2808 |
| [59] |
John GD, Nicolo F, Meagan S, et al. Optimized sgRNA design to maximize activity minimize off-target effects of CRISPR-Cas9[J]. Nat Biotechnol, 2016, 34: 184-191. DOI:10.1038/nbt.3437 |
| [60] |
Frock RL, Hu J, Meyers R M, et al. Genome-wide detection of DNA double-stranded breaks induced by engineered nucleases[J]. Nat Biotechnol, 2015, 33: 179-186. DOI:10.1038/nbt.3101 |
| [61] |
Qi L, Larson M, Gilbert L, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022 |
| [62] |
Guilinger JP, Thompson DB, Liu DR. Fusion of catalytically inactive Cas9 to FokⅠ nuclease improves the specificity of genome modification[J]. Nat Biotechnol, 2014, 32(6): 287-305. |
| [63] |
Aouida M, Eid A, Ali Z, et al. Efficient fdCas9 synthetic endonuclease with improved specificity for precise genome engineering[J]. PLoS One, 2015, 10(7): e0133373. DOI:10.1371/journal.pone.0133373 |
| [64] |
Slaymaker IM, Gao L, Zetsche B, et al. Rationally engineered Cas9 nucleases with improved specificity[J]. Science, 2016, 351: 84-88. DOI:10.1126/science.aad5227 |
| [65] |
Ran FA, Cong L, Yan WX, et al. In vivo genome editing using Staphylococcus aureus Cas9[J]. Nature, 2015, 520(7546): 202-204. DOI:10.1038/nature14278 |
| [66] |
Müller M, Lee CM, Gasiunas G, et al. Streptococcus thermophilus CRISPR-Cas9 systems enable specific editing of the human genome[J]. Molecular Therapy, 2016, 24(3): 636-644. DOI:10.1038/mt.2015.218 |
| [67] |
Lee CM, Cradick TJ, Gang B. The Neisseria meningitides CRISPR/Cas9 system enables specific genome editing in mammalian cells[J]. Molecular Therapy, 2016, 24(3): 645-654. DOI:10.1038/mt.2016.8 |
| [68] |
Chu VT, Weber T, et al. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells[J]. Nat Biotechnol, 2015, 33(5): 543-548. DOI:10.1038/nbt.3198 |
| [69] |
Maruyama T, Dougan SK, Truttmann MC, et al. Increasing the efficiency of precise genome editing with CRISPR-Cas9 by inhibition of nonhomologous end joining[J]. Nat Biotechnol, 2015, 33(5): 538-542. DOI:10.1038/nbt.3190 |
| [70] |
Lin S, Staahl B, Alla RK, et al. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery[J]. Elife Science, 2014, 3(6): e04766-e04766. |
| [71] |
Rong Z, Zhu S, Yang X, et al. Homologous recombination in human embryonic stem cells using CRISPR/Cas9 nickase and a long DNA donor template[J]. Protein & Cell, 2014, 5(4): 258. |