当前,环境污染、能源危机和粮食短缺等这些世界性的难题亟待解决[1-3]。纤维素作为自然界中分布最为广泛且最为丰富的一类可再生资源,是由葡萄糖通过β-1,4-糖苷键链接而成的碳水化合物,将其加以开发利用对于解决上述问题具有十分重要的现实意义[4-6]。近年来,已经开发了一些纤维素资源利用技术,但仍存在转化利用率低、操作时间长、成本高等局限性[7-9]。研究发现,生物降解是利用纤维素最经济环保的手段,即通过微生物等产生的纤维素酶催化水解纤维素[10-14]。因此,纤维素酶的相关研究已成为当前研究的一大热点。
纤维素酶是一类可用于多种工业领域如医药、纺织制造、环保、食品加工、生物能源等的酶制剂[15-17],能够催化水解纤维素生成葡萄糖,根据其性质和功能可分为3大类:(1)内切葡聚糖酶(endo-1,4-D-glucanase,EC 3.2.1.4),该酶可随机水解纤维素分子链中的β-1,4-糖苷键,形成新的还原末端;(2)外切葡聚糖酶(exo-1,4-β-D-glucanase,EC 3.2.1.91),该酶沿纤维素链末端水解,依次释放纤维二糖;(3)β-葡萄糖苷酶(β-D-glucosidase,EC 3.2.1.21),该酶主要将由内切葡聚糖酶和外切葡聚糖酶水解产生的纤维二糖、寡糖及低聚糖继续水解生成葡萄糖[18-21]。纤维素酶的产生菌主要包括真菌、细菌等[22-27],其中真菌产生的纤维素酶酶系结构较完整,且多为胞外酶,便于后期的分离提取[28-31],但其生产成本高,仍是阻碍纤维素酶广泛应用的主要因素,体现在因菌株自身的特性和培养条件等使得纤维素酶的活力相对较低、所需发酵时间相对较长等方面[32-35]。因此,寻找高产纤维素酶真菌菌株、优化其产酶条件是利用纤维素酶水解纤维素的关键步骤。
我国真菌资源十分丰富,截止到2010年为止,已发现的真菌总数达14 060种[36]。薄孔菌属(Antrodia),隶属于真菌界(Fungi),担子菌门(Basidiomycota),伞菌纲(Agaricomycetes),多孔菌目(Polyporales),拟层孔菌科(Fomitopsidaceae),是重要的褐腐真菌(Brown rot fungi),同时其中一些种类也是纤维素酶的主要产生菌[37-39]。竹生薄孔菌(Antrodia bambusicola Y.C. Dai & B.K. Cui)是由Cui等[40]于2011年发表的一个新种,主要分布于我国安徽、福建等地,只生长在竹子上,目前还未见有报道对其产纤维素酶能力进行深入探索。
因此,本研究主要是将竹生薄孔菌进行液体发酵培养,测定其产纤维素酶能力,并利用响应面Plackett-Burman设计、最陡爬坡设计、Box-Behnken设计对其培养基进行了优化,旨在为天然纤维素酶的进一步开发利用提供理论依据。
1 材料与方法 1.1 材料 1.1.1 供试菌株竹生薄孔菌(Antrodia bambusicola)菌株Dai 11901,采自安徽省黄山市黄山风景区的竹子,经分离纯化获得,现保藏于北京林业大学微生物研究所。
1.1.2 试剂琼脂粉、Tween 80、CoCl2·6H2O和3,5-二硝基水杨酸(3,5-dinitrosalicylic acid,DNS)购自Sigma;其他试剂为分析纯。
1.1.3 培养基固体培养基:葡萄糖20.0 g/L,酵母浸粉5.0 g/L,琼脂粉20.0 g/L,KH2PO4 1.0 g/L,MgSO4·7H2O 0.5 g/L,ZnSO4·7H2O 0.05 g/L,pH 5.0,1×105 Pa高压灭菌30 min,待指温可触时加入终浓度为0.01 g/L已过滤除菌的维生素B1,充分混匀备用。
基础液体培养基:葡萄糖20.0 g/L,酵母浸粉5.0 g/L,KH2PO4 1.0 g/L,MgSO4·7H2O 0.5 g/L,ZnSO4·7H2O 0.05 g/L,pH 5.0,1×105 Pa高压灭菌30 min,待指温可触时加入终浓度为0.01 g/L已过滤除菌的维生素B1,充分混匀备用。
产纤维素酶培养基:葡萄糖10.0 g/L,蛋白胨5.0 g/L,麸皮10.0 g/L,Tween 80 3.0 mL/L,KH2PO4 1.0 g/L,MgSO4·7H2O 0.5 g/L,CoCl2·6H2O 0.03 g/L,FeSO4·7H2O 0.03 g/L,CaCl2 0.5 g/L,ZnSO4·7H2O 0.05 g/L,pH 5.0,1×105 Pa高压灭菌30 min,待指温可触时加入终浓度为0.01 g/L已过滤除菌的维生素B1,充分混匀备用。
1.2 方法 1.2.1 活化及培养于固体平板培养基上活化,28℃恒温培养箱培养10 d备用。
1.2.2 种子发酵及液体培养取250 mL三角瓶分装100 mL基础液体培养基,接种5个直径1 cm菌饼,于摇床28℃、150 r/min振荡培养9-10 d。内切式匀浆机将种子发酵菌液制成悬液,充分振荡备用。
将种子发酵悬液以5.0%(体积比)的接种量加入含100 mL产纤维素酶培养基的250 mL三角瓶中,于摇床28℃、150 r/min振荡培养,设3个重复。
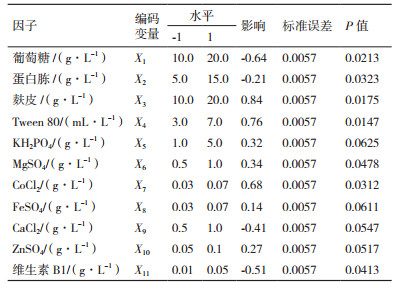
1.2.3 响应面设计优化产纤维素酶培养基Plackett-Burman设计:利用Plackett-Burman设计对可能影响竹生薄孔菌产纤维素酶的11个因子,即葡萄糖(X1)、蛋白胨(X2)、麸皮(X3)、Tween 80(X4)、KH2PO4(X5)、MgSO4(X6)、CoCl2(X7)、FeSO4(X8)、CaCl2(X9)、ZnSO4(X10)和维生素B1(X11),进行筛选试验。每个因子取高(+1)、低(-1)两个水平,见表 1。试验设计12,重复3次,响应值为纤维素酶活性,见表 2。试验设计、数据分析及模型建立采用Design Expert 8.0.6软件进行。对于单因子试验,该模型可描述为:
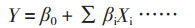 (1)
(1)
其中,Y为响应值,即竹生薄孔菌纤维素酶活性,β0为截距,βi为线性系数,Xi为编码变量。
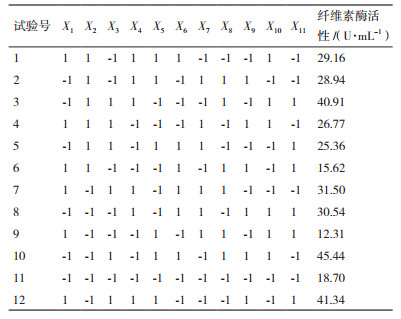
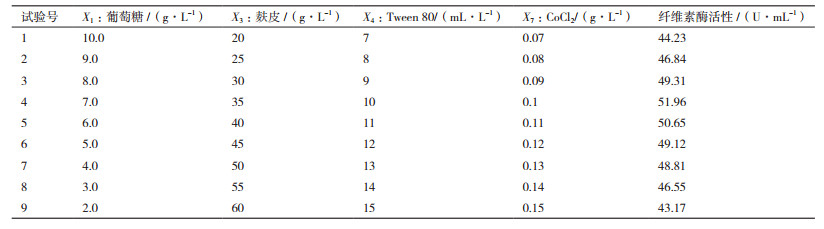
最陡爬坡设计:在Plackett-Burman设计试验结果的基础上进行最陡爬坡设计。根据显著因子对竹生薄孔菌纤维素酶活性的影响效应大小设定步长及变化方向,快速逼近最佳响应区域。因子的取值根据各因子效应的正负和大小确定,将正效应因子的值逐步增加,负效应因子的值逐步减小,见表 3。
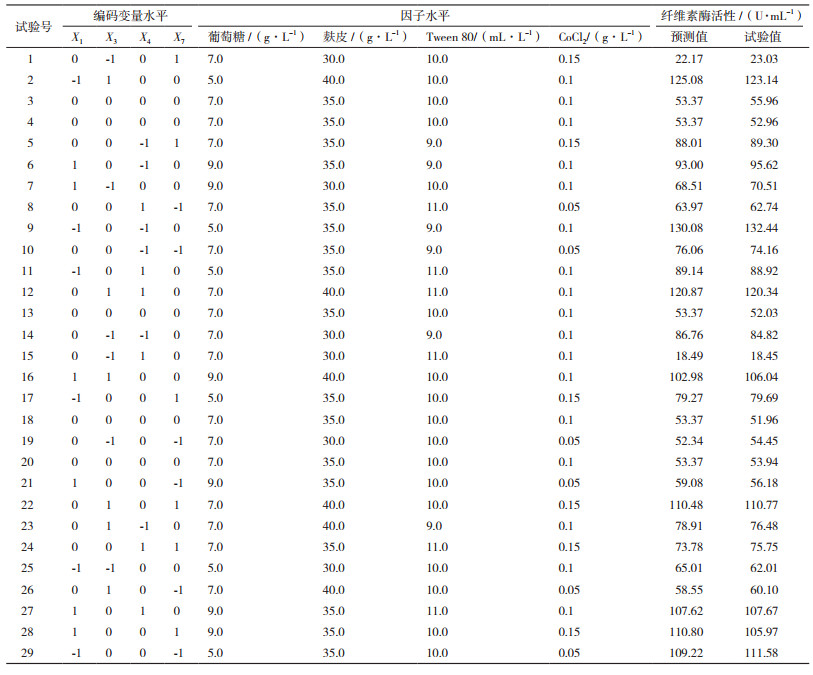
Box-Behnken设计:基于Plackett-Burman设计与最陡爬坡设计试验结果,利用Box-Behnken设计确定影响竹生薄孔菌纤维素酶活性主要因子的水平。根据已确定的4个主要因子及中心实验值,以纤维素酶为响应值,设计四因子三水平试验29,中心点5,见表 4。试验设计、数据分析及模型建立采用Design-Expert 8.0.6软件进行。试验结果经二次回归拟合,得到包括一次项、二次项、交互项的二次方程,分析各因子的主效应和交互效应,最后在一定水平范围内求取最佳值。试验因子可编码为:
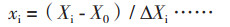 (2)
(2)
其中,xi为编码变量,Xi为因子水平,X0为因子Xi在中心点的水平,∆Xi为因子步长。
则二次多元回归方程被描述为:
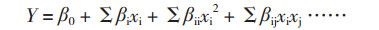 (3)
(3)
其中,Y为响应值,即竹生薄孔菌纤维素酶活性,xi和xj均为编码变量,β0为截距,βi、βii和βij分别为一次项、二次项和交互项系数。
1.2.4 样品制备培养一定时间后取样,将整瓶培养物抽滤,所得培养液经12 000 r/min离心20 min,上清液即为纤维素酶粗酶液。
1.2.5 纤维素酶活性测定采用DNS法测定滤纸纤维素酶活性[41]。
DNS试剂配制:准确称取3.15 g DNS溶于500.0 mL去离子水中,搅拌5 s,水浴至45℃。后加入100.0 mL 0.2 g/mL氢氧化钠溶液,期间不断搅拌,直到溶液清澈透明。再逐步添加91.0 g四水酒石酸钾钠、2.5 g苯酚和2.5 g无水亚硫酸钠,45℃水浴加热,补加300.0 mL去离子水,期间不断搅拌,直至加入的物质完全溶解,冷却至25℃后用去离子水定容至1.0 L,避光保存7 d后备用。
标准曲线制作:准确配制0.1、0.2、0.3、0.4、0.5、0.6、0.7 mg/mL葡萄糖标准液。分别吸取0.5 mL上述葡萄糖标准液于7支20 mL具塞刻度试管中,加入1.5 mL DNS试剂,摇匀沸水浴5 min,取出冷却后用去离子水定容至20.0 mL,充分混匀。于540 nm处测定吸光值,设3个重复,求平均值。以反应体系中葡萄糖标准品的浓度为横坐标、吸光值为纵坐标作标准曲线,计算回归方程为:y = 0.057 6x-0.013 6,相关系数R2 = 0.997 1。
滤纸纤维素酶活性测定:吸取0.5 mL酶液和1.5 mL 0.05 mol/L柠檬酸-磷酸盐缓冲液(pH 5.0)加入20 mL具塞刻度试管中,50℃水浴保温10 min后,加入50 mg滤纸条(约1 cm × 6 cm),50℃水浴保温1 h,取出立即加入1.5 mL DNS试剂,煮沸5 min后取出冷却,用去离子水稀释至20 mL。于540 nm处测定吸光值,设3个重复,求平均值。向已吸取酶液和缓冲液的试管中直接加入DNS试剂以钝化酶活,作为空白对照。定义1 min内催化产生1 μmol葡萄糖所需的酶量为1个酶活力单位(U)。
1.2.6 统计分析所得结果以平均值±标准差表示。利用SPSS 20.0软件对数据进行单因素方差分析(ANOVA)、t-检验和LSD检验。*P < 0.05和**P < 0.01分别为差异显著和差异极显著。
2 结果 2.1 竹生薄孔菌在液体培养条件下产纤维素酶活性的变化图 1显示了褐腐真菌竹生薄孔菌在液体培养条件下产纤维素酶的变化。可以看到,在培养的20 d内,竹生薄孔菌纤维素酶活性呈先上升后下降的趋势,前1-6 d内,酶活匀速上升,到第6天达到一个小的峰值(8.49 U/mL);随后略有下降,到第7天酶活为5.29 U/mL;从第7天开始,酶活直线上升,到第10天达到最大值,为18.70 U/mL;此后随着培养时间的延长,酶活逐步下降,其中以第14-15天内的下降幅度最大;到培养第20天时,测得的酶活仅为0.83 U/mL。由上述可知,若以得到纤维素酶为目的进行液体培养,则第10天是其最佳的酶液收获期;以下利用响应面设计优化竹生薄孔菌产纤维素酶液体培养基的试验均设在培养第10天取样。
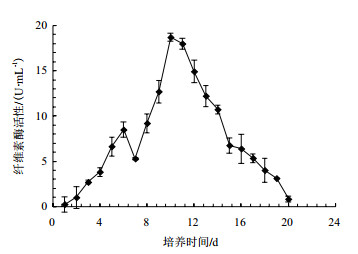
|
| 图 1 竹生薄孔菌液体培养过程中纤维素酶活性的变化 |
选取试验次数为12的Plackett-Burman设计考察葡萄糖(X1)、蛋白胨(X2)、麸皮(X3)、Tween 80(X4)、KH2PO4(X5)、MgSO4(X6)、CoCl2(X7)、FeSO4(X8)、CaCl2(X9)、ZnSO4(X10)和维生素B1(X11)这11个因子对竹生薄孔菌产纤维素酶能力的影响。各因子分别取高(+1)、低(-1)两个水平,以纤维素酶活性为响应值,见表 1和2。利用Design Expert 8.0.6软件得到单因子线性模型:
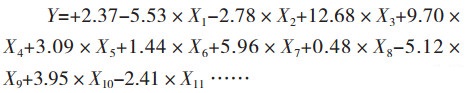 (4)
(4)
该模型P值为0.031 7,判定系数R2和校正判定系数R2分别为0.998 7和0.992 6,说明该线性模型与试验数据的拟合程度较高,应用于实际预测后误差也较小。此外,X1、X2、X3、X4、X6、X7和X11的P值均小于0.05,说明它们对竹生薄孔菌产纤维素酶能力的影响显著,根据线性模型系数大小可将因子的影响程度排序为X3 > X4 > X7 > X1 > X2 > X11 > X6,选其中的葡萄糖(X1)、麸皮(X3)、Tween 80(X4)和CoCl2(X7)作为最陡爬坡设计研究对象。在选取的4个因子中,因子葡萄糖(X1)的系数为负数,适当减少剂量可提高纤维素酶活性,为负效应因子;其他因子麸皮(X3)、Tween 80(X4)和CoCl2(X7)的系数为正数,适当增加剂量可提高纤维素酶活性,为正效应因子。
2.2.2 最陡爬坡设计由Plackett-Burman设计试验结果可知,葡萄糖(X1)、麸皮(X3)、Tween 80(X4)和CoCl2(X7)这4个因子对竹生薄孔菌产纤维素酶能力有显著的影响,且其中葡萄糖(X1)为负效应因子,麸皮(X3)、Tween 80(X4)和CoCl2(X7)为正效应因子,据此设定它们的变化方向及步长进行试验,设计及结果见表 3。可以看到,第4组试验的纤维素酶活性最高,即当葡萄糖(X1)、麸皮(X3)、Tween 80(X4)和CoCl2(X7)分别为7.0 g/L、35.0 g/L、10.0 mL/L和0.1 g/L时,竹生薄孔菌纤维素酶活性最高,为51.96 U/mL,故以第4组的条件作为Box-Behnken设计的中心点。
2.2.3 Box-Behnken设计Box-Behnken设计的编码变量及水平取值、Box-Behnken设计及结果列于表 4。可见,第9组试验的纤维素酶活性最高,即当葡萄糖(X1)、麸皮(X3)、Tween 80(X4)和CoCl2(X7)分别为5.0 g/L、35.0 g/L、9.0 mL/L和0.1 g/L时,竹生薄孔菌产纤维素酶的能力最强,酶活为132.44 U/mL。各因子经回归拟合后,得到对编码变量的二次多元回归方程:
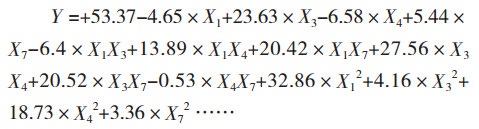 (5)
(5)
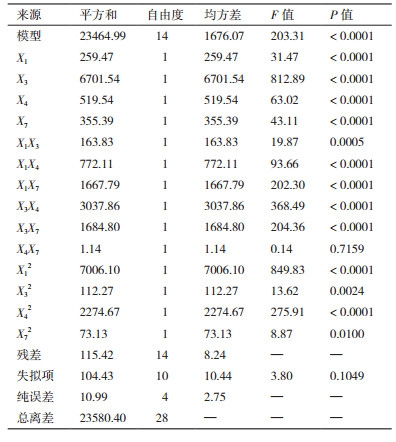
方差分析见表 5,F值为203.31,P < 0.000 1,表明该模型与试验数据的拟合度较高,可信度水平大于99.99%。判定系数R2为0.995 1,校正后的判定系数R2为0.990 2,说明只有0.98%的变化不能由该模型解释。失拟项的F值为3.80,P值为0.104 9,不显著,说明该模型不存在失拟因素。
如图 1,试验值与预测值分布的平价图呈现一条直线,表明该模型预测值与试验值的差异很小,因此在可变化的范围内可用该模型指导试验。
此外,利用回归方程分析结果绘制了竹生薄孔菌纤维素酶活性随各因子变化的等高线图和响应曲面图,见图 3。由表 5中的各参数P值可知,一次项X1、X3、X4和X7、交互项X1X3、X1X4、X1X7、X3X4和X3X7、二次项X12、X32、X42和X72对响应值Y的影响显著(P < 0.05),而交互项X4X7的影响不显著(P > 0.05)。
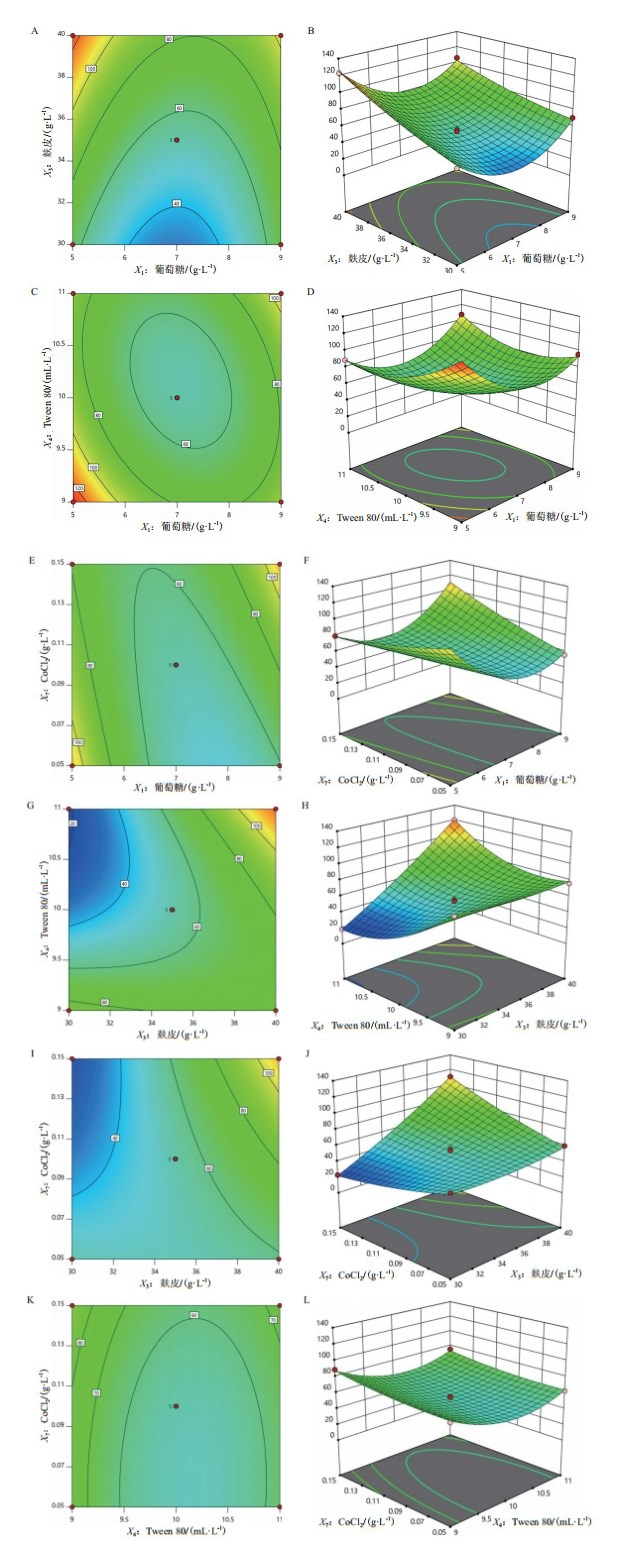
|
| 图 3 葡萄糖、麸皮、Tween80和CoCl2四因子的相互作用对竹生薄孔菌纤维素酶活性影响的等高线图和响应曲面图 |
由回归方程得到竹生薄孔菌产纤维素酶的优化液体培养基为:葡萄糖7.44 g/L、蛋白胨5.0 g/L、麸皮35.31 g/L、Tween 80 9.57 mL/L、KH2PO4 1.0 g/L、MgSO4·7H2O 0.5 g/L、CoCl2·6H2O 0.06 g/L、FeSO4·7H2O 0.03 g/L、CaCl2 0.5 g/L、ZnSO4·7H2O 0.05 g/L、维生素B1 0.01 g/L,此时纤维素酶活性为52.65 U/mL。按此条件进行验证试验,重复3次,得到平均纤维素酶活性为53.17 U/mL,与上述预测值相近,比优化前提高了2.84倍,表明回归方程能够真实地反映各因子对竹生薄孔菌纤维素酶活性的影响,证明应用响应面设计优化液体培养基以提高纤维素酶活性的方案切实可行,回归方程准确可靠。
3 讨论由于真菌所产纤维素酶多为胞外酶,种类丰富且易被提取、纯化,更方便应用于科学研究或商业化应用,因而已成为当前研究的热点[42-43]。研究发现,不同真菌合成的纤维素酶其降解能力有显著差异,且对环境的适应能力也各不相同。贾翠英等[44]通过单因素试验和正交设计试验优化了硬毛粗盖孔菌(Funalia trogii (Berk.) Bondartsev & Singer)的最适产纤维素酶条件:淀粉2%(质量体积比)、酵母粉0.4%(质量体积比)、干麸皮1.5%(质量体积比)、KH2PO4 2.0 g/L、MgSO4·7H2O 0.3 g/L、CaCl2 0.3 g/L、FeSO4·7H2O 0.006 g/L,此时酶活为56.62 U/mL。Wang等[45]利用响应面设计得到瘤盖拟层孔菌(Fomitopsis palustris (Berk. & M.A. Curtis) Gilb. & Ryvarden)产纤维素酶的最适培养基为麦麸2%(质量体积比)、(NH4)2SO4 0.5 g/L、KH2PO4 0.5 g/L、MgSO4·7H2O 0.5 g/L、尿素4.46 g/L、Tween 80 27.83 L/L,此时酶活可达到130.45 U/mL。李杏春等[46]通过测定野生大伏革菌(Phlebiopsis gigantea (Fr.) Jülich)菌株在最优条件下所产纤维素酶的活力高低来达到筛选防治针叶树根腐病高效菌株的目的,得到大伏革菌最适产纤维素酶条件为:葡萄糖30.0 g/L、蛋白胨5.0 g/L、KH2PO4 3.0 g/L、MgSO4·7H2O 1.5 g/L、装液量120 mL/250 mL、接种量5%(体积比)、初始pH 4.0。
响应面设计作为一种寻找多因子系统中最佳的数学统计方法,通过综合试验设计和分析数据,建立多元二次回归方程并拟合因素与响应值之间的关系,以最经济的方式、较少的试验次数和较短的时间对所选的试验参数进行全面研究,能够快速准确地确定多因子系统的最优条件,还可对实验中影响响应值的各因子及其交互作用进行评价[45, 47-48]。鉴于上述,响应面设计目前已被广泛应用于各种条件优化及模型的建立[49-52]。
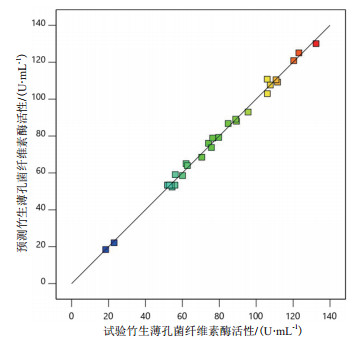
|
| 图 2 竹生薄孔菌纤维素酶活性试验值与预测值分布的平价图 |
本研究首先通过常规液体发酵培养确定了竹生薄孔菌纤维素酶的最佳收获期是培养第10天;其次利用Plackett-Burman设计从11个培养基组分中筛选出葡萄糖、麸皮、Tween 80和CoCl2含量4个显著因子,再进行最陡爬坡设计得到Box-Behnken设计的中心点;最后通过Box-Behnken设计进行试验,建立数学模型,优化了竹生薄孔菌产纤维素酶的液体培养基,将纤维素酶活性从最初的18.70 U/mL提高至53.17 U/mL,这些均为天然纤维素酶的进一步开发利用提供了理论依据,同时也说明响应面设计对于寻找优化方案确实是一种经济有效的数学统计方法,可用于确定试验因素及其交互作用在工艺过程中对目标响应值的影响,精确地表达因素和响应值之间的关系。其中,Plackett-Burman设计主要应用于成分优化及关键参数的筛选,通过试验设计和数据分析,能够从众多因素中快速有效地筛选出主效因子,从而减少优化过程中的考察因素数和试验次数。最陡爬坡设计主要是在Plackett-Burman设计试验结果的基础上进一步逼近最优区域,以结果最佳的组合确定Box-Behnken设计的中心点。而Box-Behnken设计则是在上述二者设计试验结果的基础上通过试验设计和数据分析,得到最优组合。
4 结论本研究通过响应面Plackett-Burman设计、最陡爬坡设计、Box-Behnken设计得到褐腐真菌竹生薄孔菌产纤维素酶的最适液体培养基为:葡萄糖7.44 g/L、蛋白胨5.0 g/L、麸皮35.31 g/L、Tween 80 9.57 mL/L、KH2PO4 1.0 g/L、MgSO4·7H2O 0.5 g/L、CoCl2·6H2O 0.06 g/L、FeSO4·7H2O 0.03 g/L、CaCl2 0.5 g/L、ZnSO4·7H2O 0.05 g/L、维生素B1 0.01 g/L,在此条件下得到纤维素酶活性为53.17 U/mL,与理论值52.65 U/mL相近,比优化前提高了2.84倍。
| [1] |
Ullah K, Sharma VK, Ahmad M, et al. The insight views of advanced technologies and its application in bio-origin fuel synthesis from lignocellulose biomasses waste, a review[J]. Renewable and Sustainable Energy Reviews, 2018, 82: 3992-4008. DOI:10.1016/j.rser.2017.10.074 |
| [2] |
Arevalo-Gallegos A, Ahmad Z, Asgher M, et al. Lignocellulose: a sustainable material to produce value-added products with a zero waste approach-a review[J]. Int J Biol Macromol, 2017, 99: 308-318. DOI:10.1016/j.ijbiomac.2017.02.097 |
| [3] |
Van Dyk JS, Gama R, Morrison D, et al. Food processing waste: problems, current management and prospects for utilisation of the lignocellulose component through enzyme synergistic degradation[J]. Renewable and Sustainable Energy Reviews, 2013, 26: 521-531. DOI:10.1016/j.rser.2013.06.016 |
| [4] |
Karimi K, Taherzadeh MJ. A critical review of analytical methods in pretreatment of lignocelluloses: composition, imaging, and crystallinity[J]. Bioresource Technology, 2016, 200: 1008-1018. DOI:10.1016/j.biortech.2015.11.022 |
| [5] |
Chen HY, Liu JB, Chang X, et al. A review on the pretreatment of lignocellulose for high-value chemicals[J]. Fuel Processing Technology, 2017, 160: 196-206. DOI:10.1016/j.fuproc.2016.12.007 |
| [6] |
Menon V, Rao M. Trends in bioconversion of lignocellulose: biofuels, platform chemicals & biorefinery concept[J]. Progress in Energy and Combustion Science, 2012, 38: 522-550. DOI:10.1016/j.pecs.2012.02.002 |
| [7] |
Chen HZ, Qiu WH. Key technologies for bioethanol production from lignocellulose[J]. Biotechnology Advances, 2010, 28: 556-562. DOI:10.1016/j.biotechadv.2010.05.005 |
| [8] |
Jönsson LJ, Martín C. Pretreatment of lignocellulose: formation of inhibitory by-products and strategies for minimizing their effects[J]. Bioresource Technology, 2016, 199: 103-112. DOI:10.1016/j.biortech.2015.10.009 |
| [9] |
Van Dyk JS, Pletschke BI. A review of lignocellulose bioconversion using enzymatic hydrolysis and synergistic cooperation between enzymes-factors affecting enzymes, conversion and synergy[J]. Biotechnology Advances, 2012, 30: 1458-1480. DOI:10.1016/j.biotechadv.2012.03.002 |
| [10] |
Ni J, Tokuda G. Lignocellulose-degrading enzymes from termites and their symbiotic microbiota[J]. Biotechnology Advances, 2013, 31: 838-850. DOI:10.1016/j.biotechadv.2013.04.005 |
| [11] |
Liang LY, Xue DS. Kinetics of cellulose hydrolysis by halostable cellulase from a marine Aspergillus niger at different salinities[J]. Process Biochemistry, 2017, 63: 163-168. DOI:10.1016/j.procbio.2017.09.004 |
| [12] |
Idris ASO, Pandey A, Rao SS, et al. Cellulase production through solid-state tray fermentation, and its use for bioethanol from sorghum stover[J]. Bioresource Technology, 2017, 242: 265-271. DOI:10.1016/j.biortech.2017.03.092 |
| [13] |
Li JG, Zhang SK, Li HL, et al. Cellulase pretreatment for enhancing cold caustic extraction-based separation of hemicelluloses and cellulose from cellulosic fibers[J]. Bioresource Technology, 2018, 251: 1-6. DOI:10.1016/j.biortech.2017.12.026 |
| [14] |
Prajapati BP, Suryawanshi RK, Agrawal S, et al. Characterization of cellulase from Aspergillus tubingensis NKBP-55 for generation of fermentable sugars from agricultural residues[J]. Bioresource Technology, 2018, 250: 733-740. DOI:10.1016/j.biortech.2017.11.099 |
| [15] |
Wilson DB. Cellulases and biofuels[J]. Current Opinion in Biotechnology, 2009, 20: 295-299. DOI:10.1016/j.copbio.2009.05.007 |
| [16] |
Behera BC, Sethi BK, Mishra RR, et al. Microbial cellulases-diversity & biotechnology with reference to mangrove environment: a review[J]. Journal of Genetic Engineering and Biotechnology, 2017, 15: 197-210. DOI:10.1016/j.jgeb.2016.12.001 |
| [17] |
Juturu V, Wu JC. Microbial cellulases: engineering, production and applications[J]. Renewable and Sustainable Energy Reviews, 2014, 33: 188-203. DOI:10.1016/j.rser.2014.01.077 |
| [18] |
Imran M, Anwar Z, Zafar M, et al. Hyper-productivity, characterization, and exploitation of a cellulase complex from a novel isolate of Aspergillus tubingenesis S2 using lignocellulose-based material[J]. BioResources, 2017, 12: 5649-5663. |
| [19] |
Dashtban M, Maki M, Leung KT, et al. Cellulase activities in biomass conversion: measurement methods and comparison[J]. Critical Reviews in Biotechnology, 2010, 30: 302-309. DOI:10.3109/07388551.2010.490938 |
| [20] |
Shajahan S, Moorthy IG, Sivakumar N, et al. Statistical modeling and optimization of cellulase production by Bacillus licheniformis NCIM 5556 isolated from the hot spring, Maharashtra, India[J]. Journal of King Saud University-Science, 2017, 29: 302-310. |
| [21] |
Zhang F, Zhao XQ, Bai FW. Improvement of cellulase production in Trichoderma reesei Rut-C30 by overexpression of a novel regulatory gene Trvib-1[J]. Bioresource Technology, 2018, 247: 676-683. DOI:10.1016/j.biortech.2017.09.126 |
| [22] |
贾博涵, 周伟, 赵罗迪, 等. 一株产纤维素酶细菌的分离鉴定及酶学特性研究[J]. 生物技术通报, 2014(11): 187-192. |
| [23] |
柴秀娟, 李曹龙, 孔德真, 等. 产纤维素酶菌株的筛选、鉴定及产酶条件的优化[J]. 生物技术通报, 2014(9): 164-170. |
| [24] |
李争明, 张娟, 邓中洋, 等. 纤维素酶产生菌的筛选、鉴定及发酵产酶条件优化[J]. 生物技术通报, 2015(5): 146-152. |
| [25] |
王垚, 韩燕峰, 虞泓, 等. 爱尔兰帚霉产低温纤维素酶的酶学性质和发酵工艺[J]. 菌物学报, 2017, 36(8): 1132-1140. |
| [26] |
于俊杰, 赫荣琳, 武改红, 等. 复合木质纤维素酶菌株筛选及其培养条件优化[J]. 生物技术通报, 2013(4): 101-109. |
| [27] |
李慧燕, 林良才, 肖冬光, 等. 粗糙脉孢菌C2H2转录因子家族基因敲除突变体产纤维素酶筛选分析[J]. 菌物学报, 2016, 35(2): 161-169. |
| [28] |
Okereke OE, Akanya HO, Egwim EC. Purification and characterization of an acidophilic cellulase from Pleurotus ostreatus and its potential for agrowastes valorization[J]. Biocatalysis and Agricultural Biotechnology, 2017, 12: 253-259. DOI:10.1016/j.bcab.2017.10.018 |
| [29] |
Albuquerque ED, Torres FAG, Fernandes AAR, et al. Combined effects of high hydrostatic pressure and specific fungal cellulase improve coconut husk hydrolysis[J]. Process Biochemistry, 2016, 51(11): 1767-1775. DOI:10.1016/j.procbio.2016.07.010 |
| [30] |
Metreveli E, Kachlishvili E, Singer SW, et al. Alteration of white-rot basidiomycetes cellulase and xylanase activities in the submerged co-cultivation and optimization of enzyme production by Irpex lacteus and Schizophyllum commune[J]. Bioresource Technology, 2017, 241: 652-660. DOI:10.1016/j.biortech.2017.05.148 |
| [31] |
Tamboli AS, Waghmare PR, Khandare RV, et al. Comparative analyses of enzymatic activity, structural study and docking of fungal cellulases[J]. Gene Reports, 2017, 9: 54-60. DOI:10.1016/j.genrep.2017.08.008 |
| [32] |
Srivastava N, Srivastava M, Mishra PK, et al. Applications of fungal cellulases in biofuel production: advances and limitations[J]. Renewable and Sustainable Energy Reviews, 2018, 82: 2379-2386. DOI:10.1016/j.rser.2017.08.074 |
| [33] |
Obeng EM, Budiman C, Ongkudon CM. Identifying additives for cellulase enhancement-a systematic approach[J]. Biocatalysis and Agricultural Biotechnology, 2017, 11: 67-74. DOI:10.1016/j.bcab.2017.06.006 |
| [34] |
Kuhad RC, Deswal D, Sharma S, et al. Revisiting cellulase production and redefining current strategies based on major challenges[J]. Renewable and Sustainable Energy Reviews, 2016, 55: 249-272. DOI:10.1016/j.rser.2015.10.132 |
| [35] |
Zhang YHP, Himmel ME, Mielenz JR. Outlook for cellulase improvement: screening and selection strategies[J]. Biotechnology Advances, 2006, 24: 452-481. DOI:10.1016/j.biotechadv.2006.03.003 |
| [36] |
戴玉成, 庄剑云. 中国菌物已知种数[J]. 菌物学报, 2010, 29(5): 625-628. |
| [37] |
Ma FY, Yang N, Xu CY, et al. Combination of biological pretreatment with mild acid pretreatment for enzymatic hydrolysis and ethanol production from water hyacinth[J]. Bioresource Technology, 2010, 101: 9600-9604. DOI:10.1016/j.biortech.2010.07.084 |
| [38] |
Sindhu R, Binod P, Pandey A. Biological pretreatment of lignocellulosic biomass-an overview[J]. Bioresource Technology, 2016, 199: 76-82. DOI:10.1016/j.biortech.2015.08.030 |
| [39] |
Shirkavand E, Baroutian S, Gapes DJ, et al. Combination of fungal and physicochemical processes for lignocellulosic biomass pretreatment-a review[J]. Renewable and Sustainable Energy Reviews, 2016, 54: 217-234. DOI:10.1016/j.rser.2015.10.003 |
| [40] |
Cui BK, Li HJ, Dai YC. Wood-rotting fungi in eastern China 6. Two new species of Antrodia (Basidiomycota) from Mt. Huangshan, Anhui Province[J]. Mycotaxon, 2011, 116: 13-20. DOI:10.5248/116.13 |
| [41] |
Ghose TK. Measurement of cellulase activities[J]. Pure and Applied Chemistry, 1987, 59: 257-268. DOI:10.1351/pac198759020257 |
| [42] |
Deswal D, Khasa YP, Kuhad RC. Optimization of cellulase production by a brown rot fungus Fomitopsis sp. RCK2010 under solid state fermentation[J]. Bioresource Technology, 2011, 102: 6065-6072. DOI:10.1016/j.biortech.2011.03.032 |
| [43] |
Jain L, Agrawal D. Performance evaluation of fungal cellulases with dilute acid pretreated sugarcane bagasse: a robust bioprospecting strategy for biofuel enzymes[J]. Renewable Energy, 2018, 115: 978-988. DOI:10.1016/j.renene.2017.09.021 |
| [44] |
贾翠英, 张玉辉, 王振河. 一株毛栓菌静置液体发酵产纤维素酶条件研究[J]. 菌物学报, 2012, 31(5): 736-744. |
| [45] |
Wang W, Yuan TQ, Wang K, et al. Statistical optimization of cellulase production by the brown rot fungi, Fomitopsis palustris, and its application in the enzymatic hydrolysis of LHW-pretreated woody biomass[J]. Process Biochemistry, 2012, 47: 2552-2556. DOI:10.1016/j.procbio.2012.08.004 |
| [46] |
李杏春, 何双辉, 戴玉成. 大伏革菌产纤维素酶条件优化及高效菌株筛选[J]. 生物技术通报, 2014(4): 152-158. |
| [47] |
Imran M, Anwar Z, Irshad M, et al. Optimization of cellulase production from a novel strain of Aspergillus tubingensis IMMIS2 through response surface methodology[J]. Biocatalysis and Agricultural Biotechnology, 2017, 12: 191-198. DOI:10.1016/j.bcab.2017.10.005 |
| [48] |
汪彬彬, 车振明. Plackett-Burman和Box-Benhnken Design实验设计法优化华根霉产糖化酶发酵培养基的研究[J]. 食品科技, 2011, 36(5): 41-45. |
| [49] |
崔秀秀, 韩梅, 李丽娜, 等. 利用响应面法优化耐冷纤维素降解菌产内切纤维素酶的发酵条件[J]. 华中农业大学学报, 2016, 35(4): 62-69. |
| [50] |
藏金萍, 韩志校, 姜军坡. 响应面法优化产纤维素酶菌株深层液体发酵的条件[J]. 江苏农业科学, 2016, 44(2): 368-370, 374. |
| [51] |
郑豪盈, 樊永欣, 张林, 等. 书虱伴生菌中纤维素酶产生菌的筛选、鉴定及最佳产酶发酵条件的优化[J]. 环境工程学报, 2015, 9(8): 4090-4096. DOI:10.12030/j.cjee.20150880 |
| [52] |
张辉, 桑青. 响应面法优化黑曲霉HQ-1产纤维素酶固体发酵条件[J]. 中国酿造, 2011(7): 17-21. |