植物细胞壁是获得生物燃料最丰富的可再生原料来源[1]。果胶是以α-1,4-糖苷键联接而成的聚半乳糖醛酸天然多糖,还含鼠李糖、阿拉伯糖和半乳糖等中性糖,产生于高等植物初级细胞壁和细胞间质内[2]。果胶作为细胞壁最复杂成分,赋予植物初生壁灵活性[3]。植物初生细胞壁中纤维素、半纤维素和果胶分别占30%、30%和35%左右[4],果胶占植物细胞壁比重较大,但目前国内外对果胶酶的研究较纤维素酶等少。微生物果胶酶现已广泛应用于食品工业、医药工业、纺织工业、造纸工业、环境领域、生物技术领域及饲料工业等领域[5]。果胶酶是真菌侵染植物细胞壁时最早分泌的酶类物质,对微生物利用天然纤维质材料起关键作用[6]。果胶酶主要作用于果胶类物质,降解植物细胞壁中的果胶成分,导致组织解体从而引起腐烂、枯萎等症状,在致病过程中有重要意义[7]。Morten等[8]研究发现一种切叶蚁分泌的果胶酶成分作用于植物细胞壁,能加速真菌对植物组织的消化。同时,果胶酶能提高纤维素酶水解的效率,可以降解细胞间质的果胶成分,而改变植物细胞壁的结构,增加纤维素酶和半纤维素酶在底物上的结合位点,进而提高酶解效率[9]。为更好地满足工业对高效植物细胞壁降解酶的需求,对果胶酶的研究有重要意义[10]。镰刀菌是广泛分布在土壤和有机体内的真菌,营寄生或腐生生活,常诱发萎蔫、根腐及各型腐烂,造成农作物减产[11]。作为世界四大酶制剂之一,目前我国大部分果胶酶制剂仍需从国外进口,主要问题在于分离纯化的果胶酶活性较低[12]。1999年诺维信公司便推出了用基因工程改造菌种生产的“BiPoerp”碱性果胶酶,垄断了中国碱性果胶酶市场[13],由于国外果胶酶研究较早、技术较成熟,我国更须加快研究进展以获取高活力的果胶酶应用于工业化商业化等领域。田飞等[14]从青藏高原通过筛选,分离鉴定了一株能高效降解植物细胞壁的镰刀菌菌株Q7-31T,其粗酶液降解植物细胞壁的效率明显高于其他菌株,经蛋白组学分析发现其含有16种糖苷水解酶类,其中1种为果胶酶,但果胶酶性质及结构等尚不清楚。为进一步研究镰刀菌侵染植物细胞壁的作用机理,本研究以镰刀菌Q7-31T为实验材料,研究果胶酶PGL1以期对研究植物细胞壁降解机制及分离纯化高活力果胶酶具有一定指导。
1 材料与方法 1.1 材料 1.1.1 菌种镰刀菌Q7-31T由青海大学微生物实验室于2007年5月从青海湖地区分离得到现保藏在中国普通微生物菌种保藏管理中心。
1.1.2 试剂果胶(Pectin)、蛋白胨、十二烷基硫酸钠(SDS)购自Sigma公司,三羟甲基氨基甲烷(Tris)、低分子量蛋白Marker购自北京索莱宝科技有限公司,Sephacry S-100、DEAE-Sepharose Fast Flow购自Pharmacia公司。
1.1.3 仪器设备数显恒温水浴锅HH-M8(上海赫田科学仪器有限公司);TS-200恒温培养摇床(上海仪纯实业有限公司);250D光照培养箱(吉特实验仪器厂);LDZX-40Ⅱ立式自动压力蒸汽灭菌器(上海申安医疗器械厂);UGP-VCY-1000-WxDxH生物超净工作台(统钜净化设备上海有限公司);Adventurer TM天平(奥豪斯国际贸易(上海)有限公司);UV-9200紫外可见分光光度计(北京瑞利分析仪器公司);TDL-40B型台式低速离心机(上海安亭仪器厂);MD44透析袋(北京索莱宝科技有限公司)。
1.2 方法 1.2.1 菌株从液体石蜡封存的菌种中挑取直径0.5 cm的琼脂块,接种到PDA平板培养基(马铃薯200 g,蔗糖20 g,琼脂15-20 g,蒸馏水1 000 mL)中心,25℃恒温培养箱中活化5 d;从PDA固体培养基上取1.0 cm的菌块转接至液体种子培养基(葡萄糖20 g/L,蛋白胨3 g/L,Mendels营养盐:(NH4)2SO4 1.4 g/L、KH2PO4 2.0 g/L、尿素0.3 g/L、CaCl2 0.3 g/L、MgSO4 0.3 g/L、FeSO4 5.0 mg/L、MnSO4 1.6 mg/L、ZnSO4 1.4mg/L、CoCl2 2.0 mg/L,pH自然,装液量为60 mL/150 mL),60 r/min室温振荡培养;将液体种子按10%接种量转接至产酶液体发酵培养基(燕麦秸秆5 g/L,蛋白胨8 g/L,Mendels营养盐,pH自然,装液量100 mL/250 mL)20℃、120 r/min振荡培养。发酵液经8层无菌纱布过滤得粗酶液,于4℃保存。
1.2.2 果胶酶活力的测定果胶裂解酶作用于果胶的消除反应使底物生成Δ-4,5-不饱和半乳糖醛酸基,酶活性测定:取适量底物溶液和酶液迅速混合,在一定条件下反应一定时间,于235 nm下检测吸收值,根据朗伯-比尔定律得出酶活[15]。内切、外切纤维素酶活力测定方法参见田飞、常鑫园等[14, 16]研究方法。
1.2.3 蛋白含量的测定蛋白含量的测定采用Bradford法[17],以牛血清白蛋白(BSA)为标准绘制标准曲线。在2 mL离心管中加入适当稀释的蛋白样品200 μL和1 mL考马斯亮蓝G-250染液,在595 nm下测定光吸收值。
1.2.4 硫酸铵分级沉淀发酵液经8层无菌纱布过滤,得到粗酶液。将固体硫酸铵研磨细,缓慢加入粗酶液中,调节饱和度到50%-90%,充分混匀后,4℃静置2 h,随后4℃、12 000×g离心20 min收集沉淀;沉淀以pH 7-7.5的Tris-HCl缓冲液重悬,重悬液经透析袋(MD透析袋,北京索莱宝科技有限公司)脱盐,4℃保存备用。
1.2.5 Sephacry S-100葡聚糖凝胶层析硫酸铵分级沉淀后的样品加入到Sephacry S-100葡聚糖凝胶层析柱(26 mm×700 mm),以Tris-HCl缓冲液(0.02 mol/L、pH7.0)洗脱、流速为1.5 mL/min,用核酸蛋白检测仪检测蛋白的分布,DNS法测定酶的分布(方法如前文所描述)。
1.2.6 DEAE弱阴离子交换层析取凝胶层析柱收集的果胶酶活性部分,吸附到pH7.0 DEAE琼脂糖凝胶弱阴离子交换柱(15 mm×50 mm)上,用含有NaCl(0.02-1.00 mol/L,pH7.0)的缓冲液进行线性梯度洗脱(1.8 mL/min),用核酸蛋白检测仪检测蛋白分布;样品经MD44透析袋透析脱盐后,检测内切葡聚糖酶的分布(方法如前所述)。
1.2.7 SDS-PAGE用SDS-PAGE法检测每一步实验蛋白质分子量和分离纯化过程中蛋白纯度[18]。分离胶的浓度12.5%,电泳电压为120 V;浓缩胶的浓度3%,电泳电压为80 V[19]。
1.2.8 最适温度、pH及稳定性测定最适温度测定:将底物处于pH7.0的缓冲溶液中,在20-70℃进行酶促反应,之后测定果胶酶的最适反应温度;在同上温度水浴30 min后测定酶的稳定性;最适pH测定:将底物处于不同pH的缓冲液pH1-14在40℃条件下反应,然后测定果胶酶活力,确定最适反应pH;将样品分别在上述pH处理30 min,40℃,测定酶的pH稳定性。
1.2.9 底物特异性测定分别采取浓度均为0.5%的果胶、羧甲基纤维素钠、桦木木聚糖、可溶性淀粉、燕麦秸秆粉、麸皮和滤纸作为底物,酶活力测定方法参考上述内容。
1.2.10 米氏常数测定配制不同质量浓度[S]的果胶底物溶液:10、8、6、4、2、1.5 mg/mL,加入酶液后40℃条件下反应30 min。测定外切酶活。以1/V为纵坐标,1/[S]为横坐标作图,1/V与1/[S]成直线关系,从而求出Km及Vmax,Vmax=1/b,Km=a/b。
1.2.11 金属离子对酶活力的影响酶液中分别加入各种金属离子化合物:Na+、K+、Mg2+、Zn2+、Ca2+、Fe2+ 等,各种金属离子在反应体系中的终浓度达到4 mmol/L。40℃作用30 min后测定果胶酶活力。没有加入金属离子的组酶活力作为100%。
1.2.12 蛋白鉴定蛋白鉴定由北京华大基因完成,具体实验方法参照田飞、Terova等[20, 21]的研究。
1.2.13 数据处理与误差分析采用SPSS 19.0数据处理软件进行误差分析:采用两样本(测定管与对照管)平均数t检验法进行显著性检验(P < 0.01,线性相关性极显著;0.05 > P > 0.01,线性相关性显著;P > 0.05,线性相关性不显著)[22]。
1.2.14 生物信息学分析二级结构预测:应用SOPMA在线工具对果胶酶进行蛋白质二级结构的预测;疏水性/亲水性分析:应用ProtScale Server对蛋白氨基酸序列的疏水性/亲水性进行预测;磷酸化位点预测:利用在线工具NetPhos 3.1 Server(http://www.cbs.dtu.dk/services/NetPhos/)对蛋白质氨基酸序列进行潜在酸化位点分析;糖基化位点预测:将蛋白质氨基酸序列在丹麦技术大学生物序列分析中心的网站,使用在线预测工具NetNGlyc 1.0(http://www.cbs.dtu.dk/services/NetNGlyc/)进行预测;跨膜结构域预测:应用TMHMM Server v. 2.0(http://www.cbs.dtu.dk/services/TMHMM/),对果胶酶氨基酸序列的跨膜结构域进行预测;功能结构域分析:InterProScan(http://www.ebi.ac.uk/interpro/)把SWISS-PROT、EMBL、PROTSITE、PRINTS、PFAM、ProDom等数据库提供的蛋白质序列中的结构域、motif等信息统一起来;三级结构域预测:通过Swiss-Model程序,运用同源建模法对果胶酶三级结构进行预测。
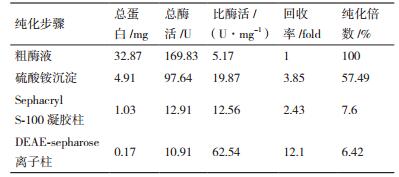
2 结果 2.1 果胶酶分离纯化果胶酶活性的蛋白样品在硫酸铵为50%-90%的饱和度条件下沉淀下来,蛋白样品在Tris-HCl(0.02 mol/L,pH7.5)缓冲液中重悬,脱盐,取20 mL重悬液加入到SephacryS-100葡聚糖凝胶柱进行分级分离,如图 1所示,有P1、P2、P3三个蛋白峰。
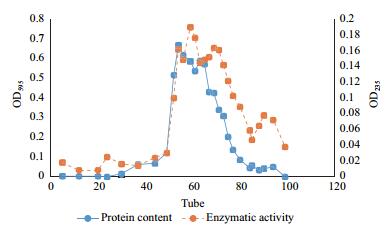
|
| 图 1 Sephacryl S-100凝胶过滤洗脱图谱 |
对具有果胶酶酶活的蛋白样品收集起来浓缩后上样到DEAE琼脂糖凝胶弱阴离子交换柱上进行二次分离纯化,洗脱图谱如图 2所示,有一个穿透峰F1和一个洗脱峰F2,酶活力检测发现有明显果胶酶活力的酶样品主要集中在F1处,F2处蛋白样品无明显酶活力。
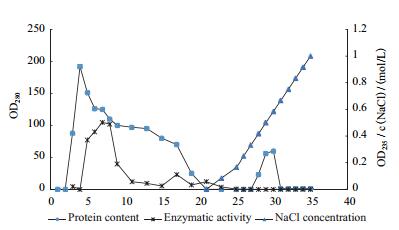
|
| 图 2 DEAE-Sepharose Fast Flow阴离子交换洗脱图谱 |
从图 2可以看出,存在两个洗脱峰(F1,F2),经酶活测定发现,果胶酶活主要集中在穿透峰F1处,而F2处的蛋白样品无果胶酶活力。分别对粗酶液、硫酸铵沉淀后重悬液、经过葡聚糖凝胶柱后及DEAE琼脂糖凝胶弱阴离子交换柱的蛋白进行SDS-PAGE检测。最终得到单一条带,经凝胶成像系统分析,分子量为36.21 kD左右(图 3)。以800 mL发酵液开始,果胶酶PGL1从粗酶液到最后的纯化产物共纯化了6.24倍,回收率为12.1 %,酶纯化后的比活力为62.54 U/mg,分离纯化结果如表 1所示。
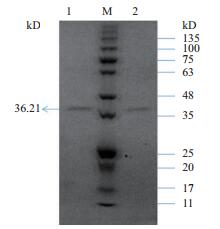
|
| 图 3 分离纯化蛋白的SDS-PAGE电泳图 1、2:分离纯化后样品;M:蛋白Marker |
及PGL1最适反应温度、pH及其耐受情况如图 4-A、4-B所示,最适反应温度为40℃,最适反应pH为8;纯化的酶在40℃下恒温水浴1 h仍能维持原有酶活力的90%以上,温度超过40℃后,酶活力开始降低,60℃水浴1 h后酶活力仅能维持原来的45%;pH稳定性检测发现该酶在pH8. 0-10. 0范围内可以保持60%以上。
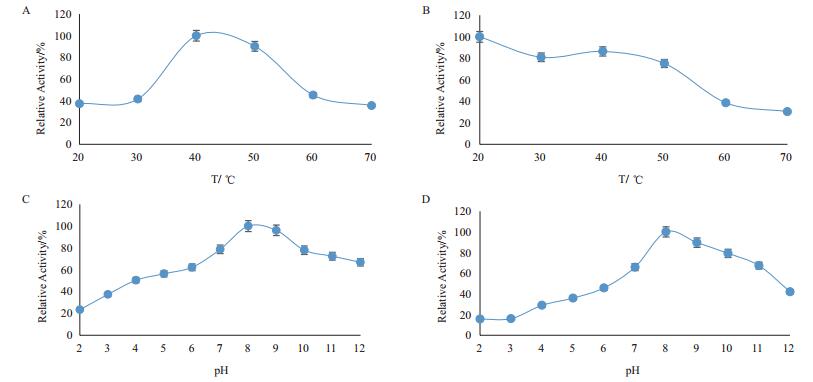
|
| 图 4 PGL1最适温度、最适pH和耐受性检测 A:不同温度下酶活力;B:温度稳定性检测;C:不同pH下酶活力;D:pH稳定性检测 |
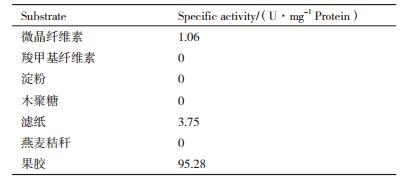
该酶对果胶具有较高酶活力,对于微晶纤维素和植物细胞壁具有不明显酶活力,对于羧甲基纤维素、淀粉、木聚糖和燕麦秸秆没有降解活力(表 2)。
经过Lineweaver-Burk双倒数法,由图 5计算知PGL1降解果胶的Km值为22.35 mg/mL,Vmax为23.53 μg/(mL·min)。
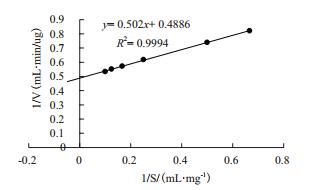
|
| 图 5 PGL1最适温度、最适pH和耐受性检测 |
金属离子检测结果(图 6)表明,Na+、K+、Mg2+、Zn2+、Ca2+、Fe2+等对PGL1均有抑制作用,其中Na+、Zn2+有很明显的抑制作用。
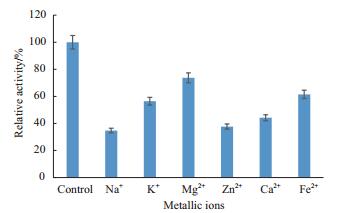
|
| 图 6 金属离子对PGL1的果胶酶活力的影响 |
首先对PGL1的蛋白样品进行双向电泳检测结果显示,PGL1的分子量为36.21 kD、等电点为8.13;进行串联质谱分析先通过一级质谱获得蛋白的指纹图谱(PMF),再选取有代表性的肽段,进一步破碎后进行二级质谱检测,质谱图如图 7所示。二级质谱结果结合母离子信息和PMF的结果进行搜库(NCBI)比较,经Mascot搜索引擎检索,检索得分66分(75分为阳性),NCBI注册号为gi︱342877242(分子量为34.31 kD,等电点为8.33),它们的序列相似但并不完全相同,分子量和等电点与质谱结果对应的蛋白存在差异,酶学特性显示为果胶酶。综合上述研究结果,将PGL1鉴定为一种新型的PL-1家族的果胶酶。
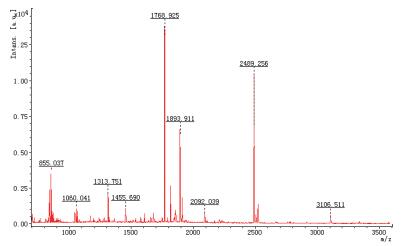
|
| 图 7 果胶酶PGL1质谱鉴定图谱 |
通过对Q7-31T菌株的PGL1果胶酶进行二级结构预测、潜在磷酸化位点分析、糖基化位点预测、跨膜结构域预测、疏水性/亲水性分析和三级结构的预测一系列生物信息学分析,以探究果胶酶降解相关酶系的特点和对纤维素降解机制的作用。
2.4.1 果胶酶PGL1的二级结构预测通过SOPMA在线工具对果胶酶PGL1进行蛋白质二级结构的预测,结果如图 8。对二级结构元件进行统计分析可知:果胶酶PGL1的二级结构构成元件均为α-螺旋、延伸链、β-转角和无规则卷曲四种,其中无规则卷曲和延伸链比例较高,分别占37.96%和31.17%;α-螺旋占20.06%;而β-转角最少,仅占10.80%。
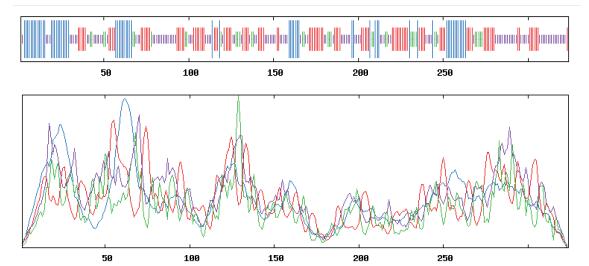
|
| 图 8 Q7-31T果胶酶PGL1二级结构预测结果图 注:竖线从长到短依次为:α-螺旋(蓝色)、延伸链(红色)、β-转角(绿色)和无规则卷曲(紫色) |
对PGL1氨基酸序列的疏水性/亲水性进行预测,负值越小表示亲水性越强,正值越大表示疏水性越强,介于-0.5到+0.5之间的为两性氨基酸。由图 9可看出PGL1的N端和C端亲水性较强,而肽链中间部分主要表现为疏水性,可能与其空间结构与对环境的适应有关。
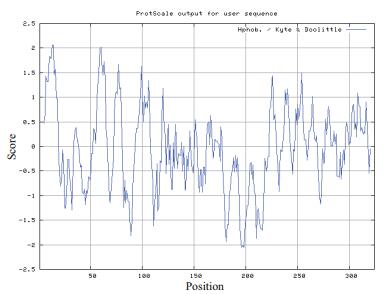
|
| 图 9 Q7-31T菌株果胶酶PGL1疏水性/亲水性分析结果图 |
利用NetPhos 3.1 Server对果胶酶PGL1氨基酸序列进行潜在酸化位点分析。依据分值大于0.5的氨基酸位点都是酸化位点,发现该酶在丝氨酸(Ser)、苏氨酸(Thr)和酪氨酸(Tyr)三个氨基酸上都具有潜在的酸化位点,结果如图 10所示。可以发现丝氨酸上的潜在酸化位点数明显比苏氨酸和酪氨酸上的磷酸化位点数高。果胶酶的磷酸位点共13个(Ser:9;Thr:3;Tyr:1),相比于Q7-31T所产的纤维素酶GH5-281和CBH-C的磷酸化位点数(45个;38个)较少[23],说明果胶酶可能本身降解纤维素能力相对较弱,但能辅助纤维素酶更高效地降解植物细胞壁。果胶酶在植物纤维加工等方面很有应用前景[24]。
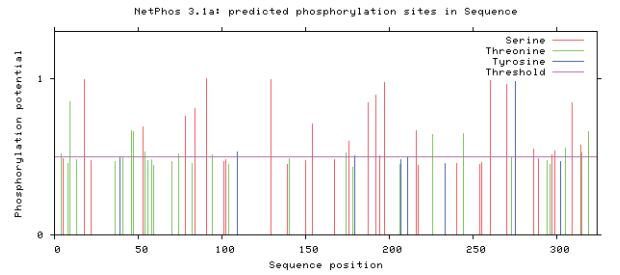
|
| 图 10 Q7-31T果胶酶PGL1潜在磷酸化位点分析结果图 |
使用在线预测工具NetNGlyc 1.0对PGL1的氨基酸序列进行糖基化位点预测,参数默认,得分大于0.5即认为为糖基化位点[25]。糖基化能增强酶蛋白对蛋白水解酶的抗性。N-糖基化能促进蛋白质的正确折叠、稳定蛋白质构象,对治疗性蛋白也具有重要的药理学意义[26]。预测结果分析可知,果胶酶PGL1存在2个潜在的N-糖基化位点,推测与其诱导合成存在很大的联系。可能由于PGL1属于诱导酶,其合成和加工受到镰刀菌Q7-31T的严格调控等有关。
2.4.5 果胶酶PGL1的跨膜结构域预测通过TMHMM Server v. 2.0,对Q7-31T果胶酶氨基酸序列的跨膜结构域进行预测,由预测结果可知,果胶酶PGL1不属于跨膜蛋白,即为分泌酶,这也与PGL1分离得到的过程相一致。
2.4.6 果胶酶PGL1的功能结构域分析应用InterProScan软件对PGL1进行功能结构域分析结果如图 11。虽然PL-1与PL-2、PL-10家族的果胶酸裂解酶采用不同的折叠方式,但它们的催化位点呈现相似的拓扑结构、保守功能性残基有相同的排列方式[27]。图 11预测了PGL1的功能,揭示了PGL1是降解植物细胞壁果胶层过程中的毒力因子。
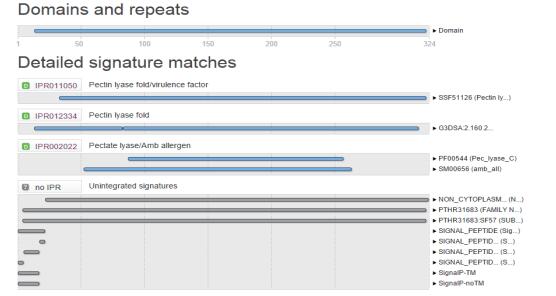
|
| 图 11 PGL1功能结构域分析结果图 |
通过Swiss-Model程序,运用同源建模法对Q7-31T果胶酶的三级结构进行预测,如图 12所示,PGL1预测的空间构型呈中空桶状结构,酶的比表面积大,能与底物更大程度地接触,这可能与酶的高效降解活性存在很大的联系。由PGL1结构与其他PL-1家族的果胶酶相比较,发现PL-1家族的果胶酶有广泛的结构相似性,这也表明它们由相同的祖先演化而来[28]。
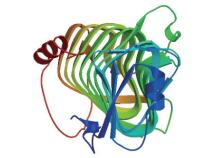
|
| 图 12 Q7-31T果胶酶PGL1三级结构预测结果图 |
研究发现,PGL1是Q7-31T诱导降解植物细胞壁最早分泌的酶,对纤维素酶降解能力有促进作用,由图 13可知,镰刀菌Q7-31T降解植物细胞壁时存在“顺序作用模型”。由蛋白组学分析[14](表未给出)可知,果胶酶PGL1灰度覆盖仅占糖苷水解酶的4.48%,但能在短时间内降解植物细胞壁中含量较多的果胶等物质,显示出PGL1高效降解底物的能力。
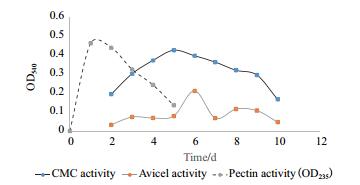
|
| 图 13 不同发酵时间下果胶酶和内、外切酶活变化情况 镰刀菌Q7-31T菌株在发酵产酶时分泌不同胞外酶存在时间上的先后顺序,果胶酶PGL1最早分泌,随后内、外切酶先后分泌,且在降解植物细胞壁时也存在作用顺序 |
近年来国内外许多学者对微生物果胶酶生产菌株的筛选、育种、鉴定、发酵、酶的分离纯化与酶学性质及酶的分子生物学特性与应用等方面进行了大量的研究[5],根据2001年Kashyap等[29]研究报道,果胶酶占世界食品酶中25%的销售额,而目前我国由于对果胶酶研究较晚、实验技术较落后和酶系信息的不完善等导致果胶酶制剂大多依赖于国外进口。本研究分离纯化了具有高活力的果胶酶PGL1,得到了Q7-31T降解植物细胞壁的“顺序作用模型”,并对PGL1进行了生物信息学分析。这对植物细胞壁降解机制提供一定的理论支持,且能提供分离纯化高活力果胶酶的实验方法,进一步完善镰刀菌属酶系信息。本实验分离纯化得到果胶酶PGL1,从粗酶液到最后的纯化产物共纯化了6.24倍,回收率为12.1%,比徐伟等[30]纯化重组菌Escherichia coli BL21/pET-pel表达的果胶酶纯化倍数(1.71)更高。许多果胶酶均为Ca2+依赖型,而本研究发现Ca2+对PGL1有抑制作用。有研究表明PL-1家族酶的结构不需要金属阳离子,因为其底物中大多羧酸基团都是甲基酯化的[31]。PGL1的β-转角最少,仅占10.80%,而Kamen等[32]研究的pelC二级结构中β-转角占最大比例(30%)。
果胶酶是参与植物细胞壁降解的重要的酶,利用β-消除机制降解果胶,能造成植物组织的断离[20]。果胶酶是植物病原菌所必不可少的,而从青藏高原分离得到的植物病原菌Q7-31T对植物细胞壁降解具有高效性。致病菌分泌的果胶酶降解植物细胞壁是使植物通过各种诱导因子的检测来识别病原体的攻击,果胶裂解酶还可以通过释放引起植物先天免疫的寡聚物间接地激活植物的防御系统[33]。图 11也预测了PGL1的功能,揭示了PGL1是降解植物细胞壁果胶层过程中的毒力因子,这与Mayans O.等人从Aspergillus得到的果胶酶A具有相似功能[34],当果胶主链被甲基化时,通过果胶裂解酶(微生物毒力因子)来切割。果胶裂解酶具有裂解果胶并在其非还原末端通过4-脱氧-α-D-甘露糖-4-烯醛酸糖基产生低聚糖的作用。本实验从镰刀菌Q7-31T中分离纯化得到一种具有高活力高回收率的新型PL-1家族果胶酶PGL1并对其进行了生物信息学分析,完善了镰刀菌属Q7-31T酶系信息、进一步提供了对植物细胞壁降解的作用机制信息,本研究的方法等也能为我国酶制剂的高活力生产提供一定的理论支持。
4 结论本研究以0.8 %蛋白胨为氮源,0.5%燕麦秸秆为碳源诱导发酵培养镰刀菌Q7-31T;采用Sephacry S-100凝胶柱层析和DEAE琼脂糖弱阴离子交换柱层析对粗酶液进行分离纯化得到果胶酶PGL1;对其进行了酶学性质分析和串联质谱鉴定,结果表明:PGL1分子量为36.21 kD,等电点为8.13;PGL1最适反应温度为40℃,最适反应pH为8;在20℃到50℃和pH 8.0到10.0能保持较高稳定性;Na+、K+、Mg2+、Zn2+、Ca2+ 和Fe2+对PGL1有抑制作用;串联质谱鉴定表明该蛋白为一种新型PL-1家族果胶酶;最后通过生物信息学分析结构及预测功能:PGL1二级结构构成元件中主要为无规则卷曲和延伸链,分别占37.96%和31.17%;PGL1两端亲水性强而中部疏水性强;磷酸化位点共13个(Ser:9;Thr:3;Tyr:1);PGL1存在2个潜在的N-糖基化位点;跨膜结构域预测显示其为分泌酶;功能结构域预测出其为植物细胞壁降解的毒力因子。
| [1] |
Marriott PE, Gómez LD, Mcqueenmason SJ. Unlocking the potential of lignocellulosic biomass through plant science[J]. New Phytologist, 2016, 209(4): 1366-1381. DOI:10.1111/nph.13684 |
| [2] |
魏海香. 甘薯果胶的制备及其理化性质研究[D]. 雅安: 四川农业大学, 2006. http://cdmd.cnki.com.cn/Article/CDMD-10626-2006160483.htm
|
| [3] |
Dick-Pérez M, Zhang Y, Hayes J, et al. Structure and interactions of plant cell-wall polysaccharides by two-and three-dimensional magic-angle-spinning solid-state NMR[J]. Biochemistry, 2011, 50(6): 989. DOI:10.1021/bi101795q |
| [4] |
赵庆新, 袁生. 植物细胞壁研究进展[J]. 生物学通报, 2007, 42(7): 8-9. |
| [5] |
李祖明, 张洪勋, 白志辉, 等. 微生物果胶酶研究进展[J]. 生物技术通报, 2010(3): 42-49. |
| [6] |
Lee SJ, Saravanan RS, Damasceno CMB, et al. Digging deeper into the plant cell wall proteome[J]. Plant Physiology & Biochemistry, 2004, 42(12): 979. |
| [7] |
母玉翠, 蒋冰心, 李萍, 等. 油菜菌核病菌致病力与多聚半乳糖醛酸酶活性的关系[C]//中国植物病理学会2012年学术年会论文集, 2012. http://www.wanfangdata.com.cn/details/detail.do?_type=conference&id=9040635
|
| [8] |
Schiøtt M, Rogowska-Wrzesinska A, Roepstorff P, et al. Leaf-cutt-ing ant fungi produce cell wall degrading pectinase complexes remi-niscent of phytopathogenic fungi[J]. BMC Biology, 2010, 8(1): 156. DOI:10.1186/1741-7007-8-156 |
| [9] |
张涛, 王联结. 纤维素酶协同作用蛋白质的研究进展[J]. 现代化工, 2012, 32(8): 29-32. |
| [10] |
Ravalason H, Grisel S, Chevret D, et al. Fusarium verticillioides secretome as a source of auxiliary enzymes to enhance saccharification of wheat straw[J]. Bioresour Technol, 2012, 114(2): 589-596. |
| [11] |
俞大绂. 镰刀菌分类学的意义[J]. 微生物学报, 1977(2): 129-134. |
| [12] |
裘纪莹, 王未名, 陈建爱, 等. 微生物果胶酶的研究进展[J]. 中国食品添加剂, 2010(4): 238-241. |
| [13] |
疏秀林, 施庆珊, 欧阳友生, 等. 微生物发酵生产果胶酶的研究概述[J]. 发酵科技通讯, 2010, 39(1): 25-27. |
| [14] |
田飞. 镰刀菌Q7-31T植物细胞壁降解酶协同研究[D]. 西宁: 青海大学, 2015.
|
| [15] |
丁凤平, 张秀之. 碱性果胶酶PATE及其测定方法[J]. 中国生化药物杂志, 1993, 7(64): 42-43. |
| [16] |
常鑫园, 谢占玲, 张凤梅, 等. 镰刀菌Q7-31T内切葡聚糖酶Egn21的分离纯化及酶学性质[J]. 微生物学报, 2017, 57(1): 33-42. |
| [17] |
Kruger NJ. The bradford method for protein quantitation[J]. Methods in Molecular Biology, 1994, 32: 9-15. |
| [18] |
Yang XZ, Shi PJ, Huang HQ, et al. Two xylose-tolerant GH43 bifunctional β-xylosidase/α-arabionosidases and one GH11 xylanase from Humicola insolens and their synergy in the degradation of xylan[J]. Food Chemistry, 2014, 148: 381-387. DOI:10.1016/j.foodchem.2013.10.062 |
| [19] |
Hu JG, Arantes V, Pribowo A, Saddle JN. The synergistic action of accessory enzymes enhances the hydrolytic potential of a "cellulase mixture" but is highly substrate specific[J]. Biotechnology for Biofuels, 2013, 6: 112. DOI:10.1186/1754-6834-6-112 |
| [20] |
田飞, 谢占玲, 郭璟, 等. 镰刀菌Q7-31T内切葡聚糖酶Egn20的分离纯化鉴定及酶学特性[J]. 微生物学报, 2015, 55(8): 1042-1049. |
| [21] |
Terova G, Pisanu S, Roggio T, et al. Proteomic profiling of sea bass muscle by two-dimensional gel electrophoresis and tandem mass spectrometry[J]. Fish Physiology and Biochemistry, 2014, 40(1): 311-322. DOI:10.1007/s10695-013-9855-x |
| [22] |
Brown JR, Dinu V. High performance computing methods for the integration and analysis of biomedical data using SAS[J]. Computer Methods and Programs in Biomedicine, 2013, 112(3): 553-562. DOI:10.1016/j.cmpb.2013.07.003 |
| [23] |
赵联正, 谢占玲, 贾贤卿. 镰刀菌Q7-31T菌株植物细胞壁降解酶系的结构[J]. 贵州农业科学, 2015(5): 138-145. |
| [24] |
Zhou Z, Liu Y, Chang Z, et al. Structure-based engineering of a pectate lyase with improved specific activity for ramie degumming[J]. Applied Microbiology & Biotechnology, 2017, 101(7): 1-11. |
| [25] |
Caragea C, Sinapov J, Silvescu A, et al. Glycosylation site prediction using ensembles of Support Vector Machine classifiers[J]. BMC Bioinformatics, 2007, 8: 438. DOI:10.1186/1471-2105-8-438 |
| [26] |
王家红, 童玥, 朱玥, 等. 蛋白质糖基化的研究进展[J]. 药物生物技术, 2011, 18(1): 77-80. |
| [27] |
Charnock SJ, Brown IE, Turkenburg JP, et al. Convergent Evolution Sheds Light on the Anti-尾-Elimination Mechanism Common to Family 1 and 10 Polysaccharide Lyases[J]. Proc Natl Acad Sci USA, 2002, 99(19): 12067-12072. DOI:10.1073/pnas.182431199 |
| [28] |
Jenkins J, Mayans O, Pickersgill R. Structure and evolution of parallel beta-helix proteins[J]. J Struct Biol, 1998, 122: 236-246. DOI:10.1006/jsbi.1998.3985 |
| [29] |
Kashyap DR, Vohra PK, Chopra S, et al. Applications of pectinases in the commercial sector: a review[J]. Bioresource Technology, 2001, 77(3): 215. DOI:10.1016/S0960-8524(00)00118-8 |
| [30] |
徐伟, 姚晓静, 付大伟. 基因工程菌Escherichia coli BL21/pET-pel重组果胶酶的纯化及酶学性质[J]. 食品科学, 2014, 35(23): 245-248. DOI:10.7506/spkx1002-6630-201423047 |
| [31] |
Hugouvieux-Cotte-Pattat N, Condemine G, Shevchik VE. Bacterial pectate lyases, structural and, functional diversity[J]. Environmental Microbiology Reports, 2014, 6(5): 427. DOI:10.1111/emi4.2014.6.issue-5 |
| [32] |
Kamen DE, Woody RW. Folding kinetics of the protein pectate lyase C reveal fast-forming intermediates and slow proline isomerization[J]. Biochemistry, 2002, 41(14): 4713-4723. DOI:10.1021/bi0115129 |
| [33] |
Marínrodríguez MC, Orchard J, Seymour GB. Pectate lyases, cell wall degradation and fruit softening[J]. Journal of Experimental Botany, 2002, 53(377): 2115-2119. DOI:10.1093/jxb/erf089 |
| [34] |
Mayans O, Scott M, Connerton I, et al. Two crystal structures of pectin lyase A from Aspergillus, reveal a pH driven conformational change and striking divergence in the substrate-binding clefts of pectin and pectate lyases[J]. Structure, 1997, 5(5): 677. DOI:10.1016/S0969-2126(97)00222-0 |