酿酒酵母(Saccharomyces cerevisiae)作为研究衰老机制的经典模式生物,既具有原核生物生长快、基因操作简单的特点,又具有真核生物翻译后加工修饰的功能特点[1]。研究酵母发现其具有多条比较保守的参与寿命调控的通路(如Sch9p、Tor1p和Ras/PKA),且这些通路与哺乳动物的信号通路都很相似。因此,通过研究酵母中人类的同源基因,可以获得功能未知人类新基因的相关信息,为进一步研究高等生物细胞寿命的具体机制提供理论依据。
酿酒酵母蛋白质O-甘露糖转移酶(Protein O-mannosyltransferase,PMT)家族是一类进化上保守的酶类,共有7个成员(Pmt1-7p),分为PMT1、PMT2和PMT4三个亚家族,具有50%-80%同源性,彼此之间有信息互补的特点。酵母PMT家族甘露糖基化修饰未折叠或错误折叠蛋白质,参与调控内质网蛋白质翻译后的加工成熟,进而影响细胞内蛋白质的动态平衡[2-4]。本课题组前期实验结果提示酵母PMT家族中PMT1基因缺失延长细胞的复制性寿命,且与内质网应激反应通路的活性密切相关[5]。本文主要研究PMT3单基因缺失,以及PMT1与PMT3双基因缺失对酵母细胞寿命的影响,旨为进一步探讨PMT家族其它成员在酵母寿命调节机制中的作用,以及为甘露糖基化修饰反应与人类相关疾病的研究提供借鉴。
1 材料与方法 1.1 材料 1.1.1 试剂Easy Taq DNA聚合酶(货号:AP111)、HiFi DNA聚合酶(货号:AP131)和DNA分子量标准购自北京全式金生物技术有限公司;实验所用的限制性内切酶MluⅠ购自TaKaRa公司;酵母提取物(货号:LP0021)和胰蛋白胨(货号:LP0042)购自生工生物工程(上海)股份有限公司;SD培养基(货号:630411和630412)和限制性氨基酸(货号:630414)购自Clontech公司。
1.1.2 材料酿酒酵母BY4742(MATα his3Δ1 leu2Δ0 lys2Δ0 ura3Δ0)、PMT1基因缺失菌株(pmt1Δ)、感受态大肠杆菌(DH5α)、载体pRS306和重组载体pRS305-pmt3-ko为实验室保存。载体为低拷贝穿梭载体,在大肠杆菌中具有氨苄青霉素抗性,分别带有Ura和Leu营养筛选标签。引物由上海英骏生物技术有限公司合成,如表 1。
构建方法参照文献进行[6-7]。以载体pRS306为模板,PMT3-F和PMT3-R为引物,PCR扩增PMT3基因破坏元件。采用醋酸锂方法将破坏元件转化进野生型酵母细胞(BY4742),经Ura营养型缺陷培养基筛选。PCR鉴定阳性克隆基因组DNA,鉴定引物分别为PMT3基因开放阅读框外侧引物(V-Pmt3-F/V-Pmt3-R),营养筛选标签URA3基因内部引物和PMT3基因开放阅读框外侧引物(URA-int-F/V-Pmt3-R),采用Easy Taq DNA聚合酶,反应体系和反应条件参照说明书进行。
1.2.2 PMT1与PMT3双基因缺失菌株的构建和验证构建方法参照文献进行[5, 8-9]。限制性内切酶MluⅠ酶切重组载体pRS305-pmt3-ko,采用醋酸锂方法将线性化重组载体转化pmt1Δ酵母细胞。经过Leu和Ura营养缺陷型培养基筛选,PCR鉴定阳性克隆基因组DNA,鉴定引物分别为营养筛选标签LEU2基因内部引物(Leu-int-F/Leu-int-R),PMT3基因开放阅读框外侧引物(Pmt3-N-F/Pmt3-C-R),PMT3基因开放阅读框外侧引物与营养筛选标签LEU2基因内部引物(V-Pmt3-F/Leu-int-R),PMT3基因开放阅读框内部引物(Pmt3-int-F/Pmt3-int-R),及鉴定pmt1Δ菌株所需的引物(V-Pmt1-F/V-Pmt1-R;URA-int-F/ V-Pmt1-R)。采用TransTaq HiFi DNA聚合酶,反应体系和反应条件参照说明书进行。
1.2.3 复制性寿命检测酿酒酵母复制性寿命的检测方法参照文献进行[10-11]。在光学显微镜下利用纤维操作系统分离、统计酵母母细胞产生子细胞的数目。采用SPSS 19.0统计软件。寿命两组间的分析采用Wilcoxon秩和检验,复制性寿命用平均数表示。P < 0.05为差异有统计学意义。
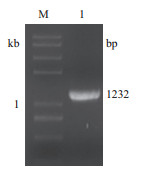
2 结果 2.1 PMT3基因破坏元件扩增结果PMT3基因破坏元件(含URA3基因编码序列)全长1 232 bp,PCR扩增产物经过1%琼脂糖凝胶电泳确认正确(图 1)。
|
| 图 1 PMT3基因破坏元件扩增结果 1:PMT3基因破坏元件;M:DNA marker |
破坏元件转化野生型酵母细胞(BY4742),PCR验证阳性克隆和野生型酵母细胞基因组DNA,结果(图 2)显示,引物V-Pmt3-F和V-Pmt3-R所扩增片段大小分别为1 619 bp和2 734 bp;引物URA-int-F和V-Pmt3-R所扩增片段分别为710 bp和0 bp,结果与预期大小一致。实验结果表明,PMT3基因缺失菌株(pmt3Δ)构建成功。

|
| 图 2 PMT3基因缺失酵母菌株的验证图 1-2:以阳性克隆基因组DNA为模板的PCR产物片段,3-4:以野生型酵母细胞基因组DNA为模板的PCR产物片段;M:DNA marker |
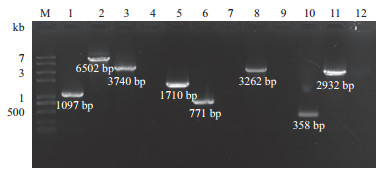
基于基因同源重组的原理,构建PMT1与PMT3双基因缺失菌株。PCR方法验证阳性克隆和野生型酵母细胞基因组DNA,结果(图 3)显示,引物Leu-int-F和Leu-int-R所扩增片段大小分别为1 097 bp和0 bp;引物Pmt3-N -F和Pmt3-C-R所扩增片段大小分别为6 502 bp和3 262 bp;引物V-Pmt3-F和Leu-int-R所扩增片段大小分别为3 740 bp和0 bp;引物Pmt3-int-F和Pmt3-int-R所扩增片段大小分别为0 bp和358 bp;引物V-Pmt1-F和V-Pmt1-R所扩增片段大小分别为1 710 bp和2 932 bp;引物URA-int-F和V-Pmt1-R所扩增片段大小分别为771 bp和0 bp,结果与预期大小一致。实验结果表明,PMT1与PMT3双基因缺失菌株(pmt1Δpmt3Δ)构建成功。
|
| 图 3 PMT1与PMT3双基因缺失酵母菌株的验证图 1-6:以阳性克隆基因组DNA为模板的PCR产物片段;7-12:以野生型酵母细胞基因组DNA为模板的PCR产物片段;M:DNA marker |
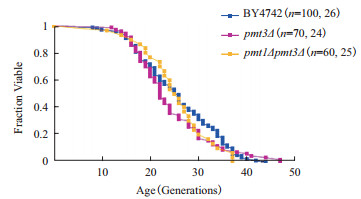
选取BY4742、PMT3基因缺失菌株(pmt3Δ)和双基因缺失菌株(pmt1Δpmt3Δ),检测酵母细胞的复制性寿命。结果(图 4)显示,与BY4742酵母细胞的寿命(平均26代)比较,pmt3Δ菌株的寿命无明显变化,平均寿命为24代(P > 0.05),pmt1Δpmt3Δ菌株的寿命也无明显变化,平均寿命为25代(P > 0.05)。实验结果表明,PMT3基因缺失不影响酵母细胞的复制性寿命,PMT1和PMT3双基因缺失也不影响酵母细胞的复制性寿命。
|
| 图 4 BY4742、pmt3Δ菌株和pmt1Δpmt3Δ菌株复制性寿命曲线图 BY4742:野生型酵母菌株,圆括号内数字代表实验细胞数量和平均复制性寿命 |
酿酒酵母PMT家族参与调控蛋白质翻译后的糖基化修饰反应,这一糖基化修饰反应在多种细胞生物学功能中起着非常重要作用[12-15]。酵母PMT家族成员之间易形成聚合体,且主要以Pmt1p-Pmt2p、Pmt3p-Pmt5p和Pmt4p-Pmt4p几种聚合体形式存在,并最大发挥其生物学功能。在敲除一个基因的前提下,Pmt1p-Pmt3p或Pmt2p-Pmt5p也可形成聚合体,这种协同补偿作用能维持酵母细胞内甘露糖转移酶的活性,进而调控细胞的生长与增殖[16]。
酿酒酵母是以非对称性分裂的方式出芽,每次都能分裂出一个比自己小的子细胞,通过研究酵母母细胞产生子细胞的数量,反应酵母细胞的分裂增殖能力[17]。本课题组前期研究结果提示PMT家族中PMT1基因缺失延长酵母细胞的复制性寿命,PMT2基因缺失对酵母细胞的寿命无明显影响[5, 18]。为探讨PMT家族其它成员基因缺失与细胞分裂增殖之间的关系,本文研究PMT3基因缺失对酵母细胞复制性寿命的影响,基于基因同源重组的原理[5, 9],构建了PMT3单基因缺失酵母菌株,重组发生在染色体水平上,表型稳定遗传。结果发现,缺失PMT3基因没有影响酵母细胞的复制性寿命,提示酵母PMT家族单基因对细胞的正常分裂增殖来说是非必需的。
研究已报道PMT家族中双基因缺失的酵母细胞表现为甘露糖转移酶活性降低,细胞形态变化,生长缺陷等特征。三基因缺失的酵母细胞仅在渗透压稳定环境中存活[19]。如PMT1和PMT2双基因缺失的酵母细胞甘露糖转移酶活性约为野生型酵母细胞的20%,双基因或三基因缺失的酵母细胞内可见多个核仁,细胞生长较慢等表型特征,推测这可能与细胞内甘露糖转移酶活性过低相关[20]。为进一步探讨双基因缺失对酵母细胞分裂增殖的影响,本文研究PMT1与PMT3双基因缺失菌株的复制性寿命,结果发现,PMT1与PMT3双基因缺失对酵母细胞的寿命无明显影响。推测此双基因缺失影响了Pmt1p-Pmt3p聚合体的形成,但并不影响Pmt2p-Pmt5p等其它聚合体的生物学功能,提示PMT家族其它成员可能补偿了双基因缺失在细胞内的生理功能,但具体的机制还不清楚,有待进一步研究。
4 结论基于基因同源重组的原理,本文成功构建酿酒酵母PMT3单基因缺失菌株和PMT1与PMT3双基因缺失菌株。通过酵母光学显微镜检测野生型酵母菌株和基因缺失酵母菌株的复制性寿命,结果显示缺失PMT3基因不影响酵母细胞的复制性寿命,且PMT1和PMT3双基因缺失菌株的寿命亦无明显变化。
致谢 感谢美国华盛顿大学Matt Keaberlein教授和美国BUCK衰老研究所Brian K Kennedy博士赠送的BY4742菌株和质粒,以及在酵母实验技术方面给予的精心指导。| [1] |
Steinkraus KA, Kaeberlein M, Kennedy BK. Replicative aging in yeast: the means to the end[J]. Annual Review of Cell and Developmental Biology l, 2008, 24: 29-54. DOI:10.1146/annurev.cellbio.23.090506.123509 |
| [2] |
Goder V, Melero A. Protein O-mannosyltransferases participate in ER protein quality control[J]. Journal of Cell Science, 2011, 124: 144-153. DOI:10.1242/jcs.072181 |
| [3] |
Xu C, Wang S, Thibault G, et al. Futile protein folding cycles in the ER are terminated by the unfolded protein O-mannosylation pathway[J]. Science, 2013, 340(6135): 978-981. DOI:10.1126/science.1234055 |
| [4] |
Gentzsch M, Immervoll T, Tanner W. Protein O-glycosylation in Saccharomyces cerevisiae : the protein O-mannosyltransferases Pmt1p and Pmt2p function as heterodimer[J]. FEBS Letters, 1995, 377(2): 128-130. DOI:10.1016/0014-5793(95)01324-5 |
| [5] |
Cui HJ, Liu XG, McCormick M, et al. PMT1 deficiency enhances basal UPR activity and extends replicative lifespan of Saccharomyces cerevisiae[J]. Age(Dordr), 2015, 37(3): 9788. |
| [6] |
方炳雄, 赵炜, 崔红晶, 等. 酿酒酵母寿命的研究方法及进展[J]. 国际老年医学杂志, 2013, 34(1): 28-34. |
| [7] |
Zhao W, Zheng HZ, Zhou T, et al. CTT1 overexpression increases the replicative lifespan of MMS-sensitive Saccharomyces cerevisiae deficient in KSP1[J]. Mechanisms of Ageing and Development, 2017, 164: 27-36. DOI:10.1016/j.mad.2017.03.008 |
| [8] |
Kaeberlein M, McVey M, Guarente L. The SIR2/3/4 complex and SIR2 alone promote longevity in Saccharomyces cerevisiae by two different mechanisms[J]. Genes and Development, 1999, 13(19): 2570-2580. DOI:10.1101/gad.13.19.2570 |
| [9] |
Zhao W, Fang BX, Niu YJ, et al. Nar1 deficiency results in shortened lifespan and sensitivity to paraquat that is rescued by increased expression of mitochondrial superoxide dismutase[J]. Mechanisms of Ageing and Development, 2014, 138: 53-58. DOI:10.1016/j.mad.2014.01.007 |
| [10] |
Steffen KK, Kennedy BK, Kaeberlein M. Measuring replicative life span in the budding yeast[J]. Jove-Journal of Visualized Experiment, 2009(28): pii: 1209. DOI:10.3791/1209 |
| [11] |
Chen YQ, Liu XG, Zhao W, et al. MET18 Deficiency increases the sensitivity of yeast to oxidative stress and shortens replicative lifespan by inhibiting catalase activity[J]. Biomed Research International, 2017, 2017: 7587395. |
| [12] |
Loibl M and Strahl S. Protein O-mannosylation: what we have learned from baker's yeast[J]. Biochim Biophys Acta, 2013, 1833(11): 2438-2446. DOI:10.1016/j.bbamcr.2013.02.008 |
| [13] |
Chugh S, Meza J, Sheinin YM, et al. Loss of N-acetylgalactosaminyltransferase 3 in poorly differentiated pancreatic cancer: augmented aggressiveness and aberrant ErbB family glycosylation[J]. British Journal Cancer, 2016, 114(12): 1376-1386. DOI:10.1038/bjc.2016.116 |
| [14] |
He Z, Luo L, Keyhani NO, et al. The C-terminal MIR-containing region in the Pmt1 O-mannosyltransferase restrains sporulation and is dispensable for virulence in Beauveria bassiana[J]. Appl Microbiol Biotechnol, 2017, 101(3): 1143-1161. DOI:10.1007/s00253-016-7894-9 |
| [15] |
Guo M, Tan L, Nie X, et al. The Pmt2p-Mediated Protein O-Mannosylation is required for morphogenesis, adhesive properties, cell wall integrity and full virulence of Magnaporthe oryzae[J]. Frontiers in Microbiology, 2016, 7: 630. |
| [16] |
Willer T, Brandl M, Sipiczki M, et al. Protein O-mannosylation is crucial for cell wall integrity, septation and viability in fission yeast[J]. Mol Microbiol, 2005, 57(1): 156-170. DOI:10.1111/j.1365-2958.2005.04692.x |
| [17] |
Okada M, Kusunoki S, Ishibashi Y, et al. Proteomics analysis for asymmetric inheritance of preexisting proteins between mother and daughter cells in budding yeast[J]. Genes to Cells, 2017, 22(6): 591-601. DOI:10.1111/gtc.2017.22.issue-6 |
| [18] |
崔红晶, 何欣, 袁源, 等. 酵母PMT2基因菌株构建及其对复制性寿命的影响[J]. 广东医学院学报, 2015, 33(5): 506-511. |
| [19] |
Gentzsch M, Tanner W. The PMT gene family: protein O-glycosylation in Saccharomyces cerevisiae is vital[J]. EMBO Jouranl, 1996, 15(21): 5752-5759. |
| [20] |
Argyros R, Nelson S, Kull A, et al. A phenylalanine to serine substitution within an O-protein mannosyltransferase led to strong resistance to PMT-inhibitors in Pichia pastoris[J]. PLoS One, 2013, 8(5): e62229. DOI:10.1371/journal.pone.0062229 |





