2. 农业部都市农业(北方)重点实验室,北京 100097;
3. 北京农学院资源与环境系,北京 102206;
4. 鲁东大学食品工程学院,烟台 264025
2. Key Laboratory of Urban Agriculture(North), Ministry of Agriculture, Beijing 100097;
3. Department of Resources and Environment, Beijing Agricultural College, Beijing 102206;
4. College of Food Engineering, Ludong University, Yantai 264025
榆黄菇(Pleurotus citrinopileatus)又名金顶侧耳、金顶蘑,属担子菌门(Basidiomycota),层菌纲(Hymenomycetes),伞菌目(Agaricales),口蘑科(Tricholomataceae),侧耳属(Pleurotus(Fr.)Quel.)[1]。其味道鲜美,营养丰富,含蛋白质、维生素和矿物质等多种营养成分,其中氨基酸含量尤为丰富,且必需氨基酸含量高[2]。榆黄菇属高营养、低热量食品,长期食用,有降低血压、降低胆固醇含量的功能,是老年人心血管疾病患者和肥胖症患者的理想保健食品[3]。
榆黄菇的人工栽培开始于20世纪70年代,现在已经能够进行周年栽培。但是到目前为止,在栽培上大都将采集的野生菌株直接用于生产,或者经过筛选驯化后作生产用菌种,新品种选育的报道较为少见[4]。食用菌种质资源是优良品种育种的基础材料,育种成效除了取决于掌握种质资源的数量,还很大程度取决于对这些种质资源多样性的遗传特性掌握。分子标记作为食用菌遗传多样性研究中的一个重要手段,其应用越来越广泛[5]。
ISSR是由Zietkeiwitcz等[6]1994年创建的一种简单重复序列间扩增多态性分子标记,结合了RAPD和SSR方法的优点,是一种具有稳定性和高重复性的分子标记方法,被广泛应用于食用菌方向[7-8]。SRAP是一种由美国加州大学Li等[9]开发的分子标记技术,通过独特的双引物设计,利用上游引物针对外显子区域,下游引物对内含子区域和启动子区域进行特异性扩增,上游引物和下游引物自由结合,配对出不同组合进行反应。由于物种及个体间的差异导致内含子、启动子和间隔序列不同进而产生多态性。
由于每一个方法的开发原理不同,聚类分析结果通常会存在较大的差异,将多个分子标记综合运用能最大优化聚类分析结果。目前,榆黄菇还没有系统的遗传多样性分析,鉴于此,本研究采用ISSR和SRAP两种分子标记的方法,对收集自全国19个榆黄菇菌株进行遗传多样性研究,旨为榆黄菇的分类鉴定、遗传育种和种质资源保护提供科学依据。
1 材料与方法 1.1 材料 1.1.1 实验材料本研究所用菌株的名称及来源参见表 1。
马铃薯葡萄糖琼脂粉(BD Difco),蛋白胨(OXIOD),分析纯磷酸二氢钾、硫酸镁等化学试剂,均购自国药集团北京化学试剂公司;2×Taq PCR MasterMix购自北京艾德莱生物科技有限公司;琼脂糖购自西班牙Biowest公司;所有引物由上海生工生物公司(Sangon)合成。PDA综合培养基:马铃薯葡萄糖琼脂粉40 g、硫酸镁1.5 g、磷酸二氢钾3 g、VB1 10 mg、水1 000 mL。
1.1.3 仪器PCR仪(Eppendorf Mastercycler Gradi-ent)、凝胶成像系统(Bio-Rad)、紫外可见分光光度计(Labtech UV9100)、电泳仪(北京六一DYY-Ⅲ-12B)、电子分析天平(奥豪斯)、台式高速冷冻离心机(Eppendorf 5415R)、移液器(Eppendorf)等。
1.2 方法 1.2.1 PCR扩增取在PDA平板上活化7-10 d的榆黄菇菌种,刮取少量菌丝作为实验材料,DNA的提取参照许峰等[10]方法。ISSR扩增反应体系及扩增程序参照冯伟林等[11]方法,ISSR-PCR反应的总体积为25 μL,体系为:12.5 μL 2×Taq PCR Master Mix,0.4 μmol/L引物(1 μL),20-50 ng DNA(1 μL),加灭菌双蒸水(10.5 μL)至25 μL。将反应液按上述体积加入0.2 mL离心管中,混匀,放入PCR仪中,进行PCR扩增。
ISSR-PCR程序设定:94℃预变性4 min,然后94℃变性30 s,以低于引物Tm值温度5℃复性45 s,72℃延伸2 min,共35个循环;72℃延伸7 min,4℃保存。
SRAP扩增反应体系及扩增程序参照边银丙等的方法[12],SRAP-PCR反应的总体积为25 μL,体系为:12.5 μL 2×Taq PCR Master Mix,0.4 μmol/L引物R(1 μL)、引物F(1 μL),20-50 ng DNA(1 μL),加灭菌双蒸水(9.5 μL)至25 μL。将反应液按上述体积加入0.2 mL离心管中,混匀,放入PCR仪中,进行PCR扩增。
SRAP-PCR程序设定:94℃预变性7 min,94℃变性1 min,35℃退火1 min,72℃延伸2 min,5个循环;94℃变性1 min,50℃退火1 min,72℃延伸90 s,共38个循环;最后72℃延伸10 min,4℃保存。
取8 μL PCR扩增产物,进行琼脂糖凝胶电泳(2%),置于紫外凝胶成像系统上观察并拍照记录。(所用引物参见表 2-3)
上述电泳图谱中同一水平上将扩增出的强带或分辨性好的弱带,均视为扩增阳性,并赋值“1”,未扩增出条带视为扩增阴性,赋值“0”,用NTSYSpc 2.10软件进行聚类分析。
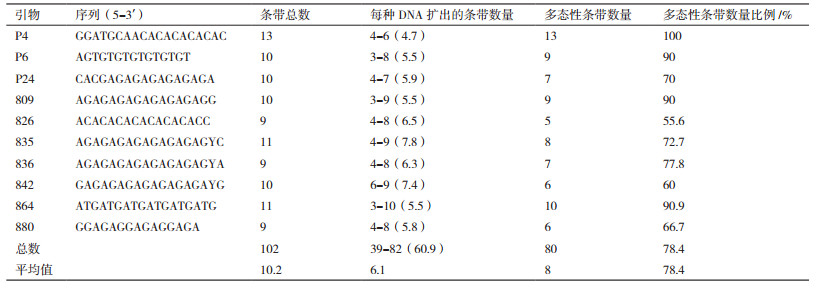
2 结果 2.1 供试菌株ISSR分析结果以提取的19个供试榆黄菇菌株的基因组DNA为模板,通过对不同引物的PCR扩增筛选,选出10条引物(表 2)在供试菌株上扩增效果良好,每个菌株都可以通过PCR扩增出DNA条带,且条带稳定、清晰、重复性好、分布合理。10条引物共扩增出102条DNA片段,其中多态性条带为80个,多态性为78.4%(表 4)。
不同引物扩增出的DNA条带数不等,其序列长度大多为200-2 000 bp,其中包含了丰富的DNA多态信息(图 1-图 2)。

|
| 图 1 引物P4对19株榆黄菇菌株的扩增结果 |

|
| 图 2 引物826对19株榆黄菇菌株的扩增结果 |
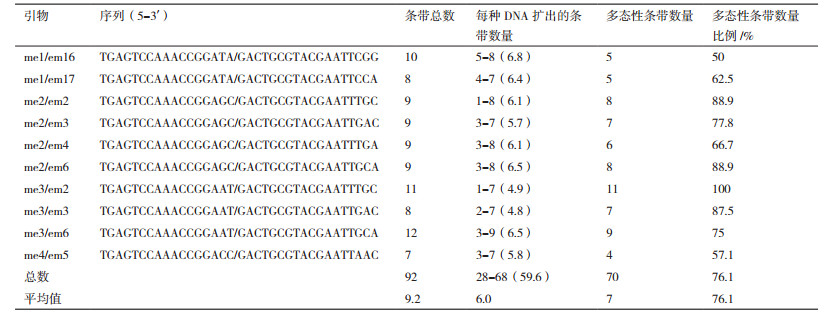
以提取的19个供试榆黄菇菌株的基因组DNA为模板,通过对不同引物的PCR扩增筛选,获得的10对引物(表 3)在供试菌株上扩增效果良好,每个菌株都可以通过PCR扩增出DNA条带,且条带稳定、清晰、重复性好、分布合理。10对引物共扩增出92条DNA片段,其中多态性条带为70条,多态性为76.1%(表 5)。
不同引物扩增出的DNA条带数不等,其序列长度大多为200-2 000 bp,其中包含了丰富的DNA多态信息(图 3-图 4)。

|
| 图 3 引物me1/em16对19株榆黄菇菌株的扩增结果 |
|
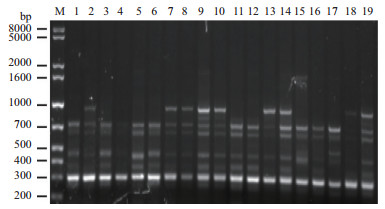
| 图 4 引物me2/em2对19株榆黄菇菌株的扩增结果 |
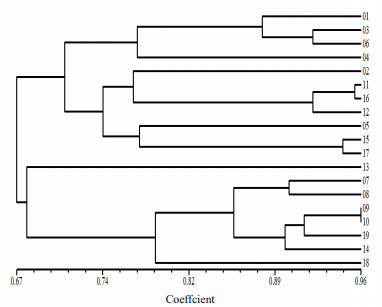
综合20条引物扩增出的194条DNA片段,其中多态性条带150条,多态性达77.3%。用NTSYSpc 2.10软件对19个榆黄菇供试菌株进行聚类分析,获得各菌株间的聚类图(图 5)。
|
| 图 5 19个榆黄菇菌株的聚类分析图 |
根据聚类分析结果可知,在相似系数为0.70水平上,可将19个供试榆黄菇菌株分为4大类,表明19个供试榆黄菇菌株间具有较大的遗传差异,第一类包括01号、03号、04号、06号,第二类包括02号、05号、11号、12号、15号、16号、17号,第三类为13号,第四类包括07号、08号、09号、10号、14号、18号、19号;而在0.96的相似值水平上,除09号和10号两个菌株未区分开外,其余17个菌株均可单独分开。
3 讨论食用菌菌种的鉴定和保护是食用菌产业的重要问题,市场上常出现“同名异物“或”同物异名”的现象,不仅不利于育种者的积极性也给生产者带了巨大的经济损失[4]。传统的菌种鉴别是在子实体层次对子实体的颜色、大小、朵型等形态特征及其生理生化反应特征、栽培特性等为依据,易受生长环境的影响,一般只能鉴定到属内种间这一水平,而对于种内菌株却难以鉴别,且整个鉴定流程时间长、准确率低[3]。
近年来,随着分子生物学技术的发展,已有多种分子标记技术(RAPD,ISSR和SRAP)应用于食用菌的分类鉴定[13]。周洁等[8]利用11个ISSR引物对40个姬菇杂交菌株及2个亲本菌株进行分析,分析结果表明,42个供试菌株遗传相似系数变异范围为0.428 6-0.853 7,在相似系数0.668 0时,42个菌株聚为7类,两亲本各具一类,杂交菌株与亲本菌株遗传差异较大。许峰等[10]利用9对SRAP引物对22个白色金针菇进行聚类分析,在相似系数0.820水平时,将供试菌株分为5大类,技术揭示了北京地区金针菇菌株的遗传多样性。张静等[14]运用SRAP,ISSR和RAPD技术对40个双胞蘑菇进行分析,其中SRAP平均多态率为70.21%;ISSR平均多态率为78.33%;RAPD平均多态率为61.94%,根据距离值不同,将40个双孢蘑菇分为6个或9个类群,更好的解释供试菌株间的亲缘关系。目前国内的分子标记技术在香菇[15]、木耳[16]等食用菌方面应用较广泛,但榆黄菇的报道较少,张秋胜等[17]和崔丹等[18]仅运用ISSR技术分析榆黄菇的遗传多样性。由于每一个方法的开发原理不同,聚类分析结果通常会存在比较大的差异,将多个分子标记综合运用能最大优化聚类分析结果[19]。
本实验共收集了不同地区的19个榆黄菇品种,通过ISSR和SRAP两种分子标记技术,利用筛选的引物以19个供试榆黄菇菌株的基因组DNA为模板进行PCR扩增,一共扩增出194条DNA片段,其中,多态性条带有150个,多态性比例为50%-100%不等,平均为77.3%,说明19种榆黄菇遗传多样性比较丰富,可以利用和开发的菌种资源较多;不同引物扩增出每种DNA的条带数量不等,为3-13条,扩增出的DNA片段的分子量大多为200-2 000 bp,包含了丰富的多态性。聚类分析结果表明:19株榆黄菇菌株间的遗传相似系数范围为0.67-0.95,在相似系数为0.70水平上,可将19个供试榆黄菇菌株分为4大类。7号和8号菌株同是引自山东莒县的菌株,聚类分析结果中聚为一簇,说明榆黄菇品种存在一定的地域性;11号和16号是来自不同地区的品种,亲缘关系虽然较近,但是仍然存在一定的差异,说明不是一个品种或同一品种通过不断的传代已经发生遗传变异;通过目前的手段没有能够区分9号和10号菌株,说明二者的亲缘关系很近,且两种菌株都是来自于江苏高邮,可能是同物异名。
本研究应用分子标记技术从DNA水平上系统分析评估榆黄菇种质资源的遗传多样性,为榆黄菇种质资源的鉴定、保护和育种提供了理论支持。
4 结论本实验筛选出适合供试榆黄菇菌株的10条ISSR引物和10对SRAP引物,利用筛选的引物以19个供试榆黄菇菌株的基因组DNA为模板进行PCR扩增,一共扩增出194条DNA片段,其中,多态性条带有150个,多态性比例为50%-100%不等,平均为77.3%,说明19个榆黄菇遗传多样性比较丰富;不同引物扩增出每种DNA的条带数量不等,为3-13条,扩增出的DNA片段的分子量大多为200-2 000 bp,包含了丰富的多态性。聚类分析结果表明:在相似系数为0.70水平上,可将19个供试榆黄菇菌株分为4大类,表明19个供试榆黄菇菌株间具有较大的遗传差异。
| [1] |
熊芳, 郑闽江, 刘新锐, 等. SCAR标记技术鉴别榆黄蘑品种[J]. 基因组学与应用生物学, 2010(3): 593-597. |
| [2] |
王玥玮, 王麒琳, 张立娟. 榆黄蘑营养成分及其生物活性的研究进展[J]. 食品研究与开发, 2017, 38(4): 201-203. |
| [3] |
潘春磊, 王延锋, 黄文, 等. 榆黄蘑10个菌株的农艺性状筛选及评价[J]. 中国食用菌, 2015(6): 13-16. |
| [4] |
张玉铎. 榆黄蘑单孢杂交及后代筛选[D]. 保定: 河北农业大学, 2010.
|
| [5] |
吴学谦, 李海波, 魏海龙, 等. SCAR分子标记技术在香菇菌株鉴定上的应用研究[J]. 菌物学报, 2005, 24(2): 259-266. |
| [6] |
Zietkiewicz E, Rafalski A, Labuda D. Genome fingerprinting by simple sequence repeat(SSR)-anchored polymerase chain reaction amplification[J]. Genomics, 1994, 20(2): 176-183. DOI:10.1006/geno.1994.1151 |
| [7] |
Liu J, Wang ZR, Li C, Bian YB, Xiao Y. Evaluating genetic diversity and constructing core collections of Chinese Lentinula edodes cultivars using ISSR and SRAP markers[J]. Journal of Basic Microbiology, 2015, 55(6): 749-760. DOI:10.1002/jobm.v55.6 |
| [8] |
周洁. ISSR分子标记在姬菇杂交菌株鉴定中的应用[D]. 雅安: 四川农业大学, 2010.
|
| [9] |
Li G, Quiros CF. Sequence -related amplified polymorphism(SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics, 2001, 103: 455-461. DOI:10.1007/s001220100570 |
| [10] |
许峰, 刘宇, 王守现, 等. 北京地区白色金针菇菌株的SRAP分析[J]. 中国农学通报, 2010, 26(10): 55-59. |
| [11] |
冯伟林, 蔡为明, 金群力, 等. ISSR分子标记分析杏鲍菇菌株遗传差异研究[J]. 中国食用菌, 2009, 28(1): 47-49. |
| [12] |
边银丙, 宋小亚. 几种新型DNA分子标记及其在食用菌研究中的应用[J]. 食用菌学报, 2006, 13(1): 78-81. |
| [13] |
张瑞颖, 胡丹丹, 左雪梅, 等. 分子标记技术在食用菌遗传育种中的应用[J]. 中国食用菌, 2011(1): 3-7. |
| [14] |
张静, 陈文炳, 邵碧英, 等. 双孢蘑菇SRAP、ISSR、RAPD标记遗传多样性和菌群分类研究[J]. 中国食品学报, 2010, 10(6): 7-13. |
| [15] |
贾定洪, 王波, 郑林用, 等. 应用拮抗及ISSR方法鉴定袋料香菇菌株[J]. 西南农业学报, 2013, 26(2): 832-834. |
| [16] |
刘华晶. 基于不同分子标记的黑龙江省野生黑木耳遗传多样性分析[D]. 哈尔滨: 东北林业大学, 2011.
|
| [17] |
张秋胜, 徐丙莲, 刘林德, 等. 中国金顶侧耳菌株遗传多态性的ISSR分析[J]. 食品科学, 2011, 32(05): 172-175. |
| [18] |
崔丹. 金顶侧耳种质资源多样性的研究[D]. 吉林: 吉林农业大学, 2012.
|
| [19] |
曲绍轩, 高山, 黄晨阳. SRAP、ISSR和RAPD分子标记技术在银耳菌株鉴别上的应用[J]. 食用菌学报, 2007, 14(3): 1-5. |