氧化葡萄糖酸杆菌是一种棒状革兰氏阴性、好氧嗜酸性α-谷氨酸杆菌,属于醋酸杆菌科[1]。由于其独特的广泛氧化碳水化合物的不完全氧化能力,使得氧化葡萄糖酸杆菌能够非常好地适应高糖和酸性环境。同时,高氧化速率与低生物质产量使得氧化葡萄糖酸杆菌有很高的工业应用价值[2]。氧化葡萄糖酸杆菌的产物谱包括维生素C前体L-山梨醇[3],饮料成分(如Bionade),以及产生鞣剂二羟基丙酮的甘油[4]等。氧化葡萄糖酸杆菌基因组序列的测定为研究该菌株基因组水平上的特征提供了强有力的方法[5-6]。
为了探究菌株的基因功能及其工业应用,需要建立有效的抑制或激活基因表达的方法。在氧化葡萄糖酸杆菌中,传统的抑制基因表达的方法是三亲本结合后通过同源重组进行基因敲除[7]。在很多情况下,这些过程耗时费力并且效率低下。
通常认为RNA分子是基因和它们编码的蛋白质之间的信息传递者[8]。近年来,非编码小RNA(small RNA,sRNA)成为生物研究领域的前沿和热点。已有研究表明sRNA参与生物体的各种生命活动,包括蛋白质合成、RNA的剪切和编辑、rRNA的修饰[8-9]以及生化反应的催化[9-11]。真核生物中sRNA主要分为两种,微小RNA(MicroRNA,miRNA)和短干扰RNA(Small interfering RNA,siRNA)。miRNA与核糖核蛋白复合物相关联,主要负责对调控一系列生命活动的目标基因进行负向调控,包括细胞命运的时间安排、干细胞维持、细胞凋亡和细胞极性[12-13]。siRNA则与RNA干扰的转录后形式和涉及染色质修饰的转录沉默有关[14]。
在大肠杆菌中,通过对非编码sRNA的分析得到了17种不同的sRNA,其中很多sRNA都与Hfq结合[15]。大肠杆菌中的sRNA也在很多生命活动承担各种作用:调节RNA聚合酶(6S RNA)的活性,标记降解蛋白质(tmRNA),以及调节翻译[16]。细菌中的sRNA转录后调节被认为是一种重要的调节机制,并且这种机制已经在分子水平上得到了证实。研究表明,大多数的sRNA直接与RBS序列或起始密码子区域结合,以抑制翻译的起始或影响mRNA的稳定性,从而抑制目标基因的翻译[17]。
在本研究通过设计一系列与RBS序列互补的sRNA来有效调控氧化葡萄糖酸杆菌中的基因表达,研究结果为氧化葡萄糖酸杆菌这一具有工业应用价值的菌株提供了另外一种调节基因表达、研究基因功能的方法。
1 材料与方法 1.1 材料菌株和质粒:大肠杆菌DH5α及BL21(DE3)购买自北京全式金生物技术有限公司。其中,DH5α主要用于重组质粒的扩增,BL21(DE3)主要用于重组蛋白质EI的表达。氧化葡萄糖酸杆菌DSM2003菌株为本实验室保存。质粒pET-28a(Novagen)、pBBR1MCS-5[18](庆大霉素抗性)为本实验室保存。
LB培养基:酵母粉5 g/L,胰蛋白胨10 g/L,NaCl 10 g/L,用于大肠杆菌的培养;山梨醇培养基:山梨醇80 g/L,酵母粉20 g/L,磷酸二氢钾1 g/L,硫酸镁0.3 g/L,谷氨酰胺0.1 g/L,用于氧化葡萄糖酸杆菌的培养。
抗生素:头孢霉素、庆大霉素和卡那霉素均购买自Biosharp。头孢霉素和庆大霉素工作液浓度为25 mg/mL;卡那霉素工作液浓度为50 mg/mL。
质粒抽提试剂盒购买自Axygen;Cycle-pure Kit、Gel Extraction Kit购买自Omega;各种限制性内切酶、T4连接酶购买自Thermofisher Scientific;PrimerSTAR MAX、DNA marker DL5000购买自TAKARA。
1.2 方法 1.2.1 氧化葡萄糖酸杆菌感受态的制备挑取新鲜活化的氧化葡萄糖酸杆菌单菌落,接种至5 mL山梨醇液体培养基中,30℃、200 r/min条件下培养22 h;按5:100的比例将试管内的菌液接种至100 mL山梨醇液体培养基(500 mL三角瓶)中,同样条件下培养至OD600约为0.5-0.6;将菌液冰浴20 min后,分装至预冷的50 mL离心管中,4℃、8 000 r/min离心15 min,弃上清收集菌体;用30 mL预冷的10%甘油重悬菌体沉淀,同样条件下离心15 min洗涤菌体,弃上清收集沉淀;重复上述步骤两次;最后用20 mL 10%甘油重悬菌体后,将菌液分装至1.5 mL离心管中,-80℃保存备用。
1.2.2 引物设计所有引物(表 1)均用APE软件设计,由上海捷瑞生物工程有限公司合成。质粒测序由上海睿迪生物科技有限公司完成。
以氧化葡萄糖酸杆菌DSM2003菌株的全基因组为模板,tufB-F和tufB-R为引物,通过PCR得到延伸因子TU启动子(tufB启动子)片段[19]。将PCR产物和质粒pBBR1MCS-5用限制性内切酶Sac Ⅰ和Xba Ⅰ进行酶切,再通过T4连接酶连接得到重组质粒pBBR-tufB。GFP片段为本实验室保存,以GFP-F和GFP-R为引物通过PCR扩增。扩增的GFP和pBBR-tufB用限制性内切酶Xba Ⅰ和BamH Ⅰ酶切,再通过T4连接酶连接得到重组质粒pBBR-tufB-GFP。
1.2.4 顺式阻遏(cis repressor,cR)系统的设计与构建将含有RBS互补序列的不同长度的短核苷酸序列cR(cR1、cR2、cR3和cR4)直接插入到RBS上游。在转录后期,互补的核苷酸序列在mRNA序列的5'-非翻译区(UTR)自然折叠形成包含RBS的茎-环结构,该结构阻断了核糖体30S亚基对RBS的识别,最终抑制GFP的表达。cR序列由环和茎两部分组成:其中,各cR序列的环序列完全相同,而cR1、cR2、cR3和cR4的茎序列长度分别为32 nt、38 nt、44 nt和50 nt(图 1A)。cR1、cR2、cR3和cR4均以tufB-GFP为模板,分别以引物cR1-F、cR1-R-1、cR1-F-1、cR1-F-2和cR1-R,cR2-F、cR2-R-1、cR2-F-1和cR2-R,cR3-F、cR3-R-1、cR3-F-1和cR3-R,cR4-F、cR4-R-1、cR4-F-1和cR4-R(表 1),通过重叠PCR扩增出含有cR序列的tufB-GFP。再通过Sac Ⅰ和BamH Ⅰ酶切、T4连接酶连接构建顺式阻遏重组质粒。
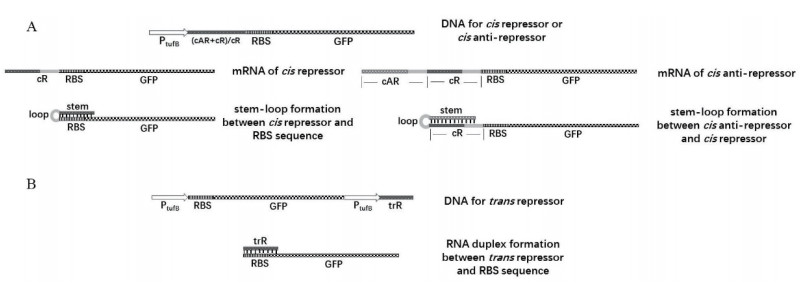
|
| 图 1 氧化葡萄糖酸杆菌中翻译水平上控制基因表达的小RNA序列的设计与构建 A:在顺式阻遏构型中,在PtufB启动子和RBS之间插入了一段与RBS互补的顺式阻遏序列。mRNA转录后在其5'末端形成的茎-环结构能够阻碍核糖体的结合,从而抑制蛋白质翻译过程。在顺式抗阻遏构型中,在启动子和顺式阻遏序列之间插入了一段与顺式阻遏序列互补的顺式抗阻遏序列。转录后,在顺式抗阻遏序列和顺式阻遏序列之间优先形成茎-环结构,它能妨碍由顺式阻遏序列和RBS形成茎-环结构,从而使得核糖体能与暴露的RBS结合并启动蛋白质翻译;B:在反式构型中,与RBS互补的反式阻遏序列在独立的启动子控制下转录。这一反式阻遏能与目标基因的RBS形成RNA双链,从而阻碍核糖体的结合及蛋白质翻译 |
将一段含有cR2互补序列的DNA序列cAR插入顺式阻遏序列cR的上游,其中cAR序列与cR2序列的互补区长度为44 nt。由于两者之间互补区更长,其转录后能与cR序列形成稳定的茎环结构,从而使RBS暴露,便于核糖体结合,开启蛋白质翻译过程(图 1-A)。以cAR-F、cAR-R-1、cAR-R-2、cAR-F-1和cAR-R(表 1)为引物,顺式阻遏系统重组质粒为模板,通过重叠PCR,将cAR序列插入至cR序列上游。
1.2.6 反式阻遏(trans repressor,trR)系统的设计与构建trR序列的转录受独立的启动子控制。其转录后形成游离的sRNA,通过互补与目标mRNA的RBS区结合,抑制基因的表达(图 1-B)。trR序列与cR2的茎序列完全相同。将受独立的tufB启动子控制的trR序列插入至pBBR-tufB-GFP下游。PtufB-trR片段以tufB序列为模板,PtufB-trR-F、PtufB-trR-R-1和PtufB-trR-R-2(表 1)为引物,PCR扩增后,与pBBR-PtufB-GFP经EcoR Ⅰ和Cla Ⅰ酶切、T4连接酶连接,得到反式阻遏重组质粒。在对照质粒中,引入与trR长度相当的任意序列插入至trR相同位置。
1.2.7 抑制氧化葡萄糖酸杆菌内源基因EI表达的sRNA设计根据“五密码子窗口”(Five codon window)理论[17],设计了4种长度均为30 nt的sRNA(30-sRNA),它们分别与细菌中磷酸烯醇丙酮酸:糖磷酸转移酶系统(PST)中EI酶的开放读框(Open reading frame,ORF)的不同区域互补。其中,EI-sRNA-1与EI开放读框的前10个核苷酸以及起始密码子上游的20个核苷酸互补;EI-sRNA-2与EI开放读框的前15个核苷酸以及起始密码子上游的15个核苷酸互补;EI-sRNA-3与EI开放读框的前20个核苷酸以及起始密码子上游的10个核苷酸互补;EI-sRNA-4与EI开放读框的前25个核苷酸以及起始密码子上游的5个核苷酸互补。EI-sRNA-1、EI-sRNA-2、EI-sRNA-3和EI-sRNA-4以氧化葡萄糖酸杆菌基因组为模板,分别以EI-sRNA-1-F、EI-sRNA-1-R-1和EI-sRNA-1-R-2,EI-sRNA-2-F,EI-sRNA-2-R-1和EI-sRNA-2-R-1,EI-sRNA-3-F、EI-sRNA-3-R-1和EI-sRNA-3-R-2,EI-sRNA-4-F、EI-sRNA-4-R-1和EI-sRNA-4-R-2为引物,通过重叠PCR扩增。得到的PCR产物经酶切连接后插入pBBR1MCS-5质粒的Sac Ⅰ和Xba Ⅰ位点之间。
1.2.8 电转化所有调控系统的重组质粒均通过电转化(2 000 V,200 Ω,25 μF)导入氧化葡萄糖酸杆菌感受态细胞中。
1.2.9 报告基因GFP的检测含有以GFP作为报告基因的顺式阻遏系统、顺式抗阻遏系统和反式阻遏系统重组质粒的氧化葡萄糖酸杆菌DSM2003菌株均在山梨醇培养基中培养至OD600约为0.6。取1.5 mL菌液,4℃、8 000 r/min离心2 min,再用PBS(pH7.4)洗3次,最后取700 μL PBS重悬菌体。各菌体重悬液的荧光值通过SpectraMax酶标仪检测,其中激发波长485 nm,发射波长535 nm。所得数据用Soft Max Prov5.0.1.分析。每个样品检测3个平行样品。
1.2.10 EI蛋白及其抗体的制备以氧化葡萄糖酸杆菌DSM2003基因组为模板,EI-F和EI-R(表 1)为引物,通过PCR对细菌中PST中的EI酶编码序列进行扩增。扩增产物和pET28经过Sac Ⅰ和Sal Ⅰ酶切后,以T4连接酶连接得到重组质粒pET28a-EI,再将重组质粒转化至E. coli BL21(DE3)感受态细胞中,37℃培养至OD600达到0.6。加入0.5 mmol/L IPTG,18℃诱导12 h。EI蛋白通过5 mL Ni-NTA层析柱(GE Healthcare Life Science,美国)纯化。纯化后的EI蛋白纯度由SDS-PAGE测定,浓度用Bradford法测定。纯化的EI蛋白作为抗原,委托苏州新赛美生物技术有限公司制备兔抗EI血清,并将其作为Western blot检测的一抗。
1.2.11 Western Blot检测将含有pBBR-tufB-EI-sRNA系列质粒的氧化葡萄糖酸杆菌培养至OD600达到0.6。取50 mL菌液8 000 r/min离心2 min,得到的菌体沉淀用10 mL PBS(pH7.4)重悬。超声破碎(超声3 s,停止7 s,99个循环)后,8 000 r/min离心30 min,所得的上清即为全细胞蛋白。将所有样品的上清液浓度调至0.5 mg/mL,根据文献[20]中的操作步骤用一抗对各样品进行Western blot检测。通过ECLTM-Prime(GE Healthcare Life Science)进行显色。
2 结果 2.1 顺式阻遏系统对基因表达的影响在顺式阻遏系统中,插入报告基因上游的DNA片段在转录后期与RBS形成茎-环结构,这样的茎-环结构理论上能够干扰核糖体的结合并最终抑制GFP的表达。细胞荧光信号分析结果(图 2)显示,与阳性对照组相比,含有cR1(环长6 nt,茎长32 nt)的菌株荧光信号明显降低。但与作为阴性对照的野生型菌株相比,荧光信号仍然很高,说明cR1只实现了部分抑制。
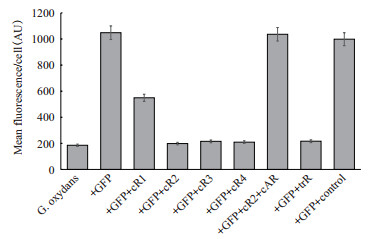
|
| 图 2 氧化葡萄糖酸杆菌中小RNA在翻译水平上对基因表达的调节 |
为了检测茎-环结构中互补序列长度的增加,是否会加强顺式阻遏系统的阻遏效果,我们又设计了一系列互补序列长度不同的顺式阻遏序列。如图 2所示,含有cR2(茎互补序列长度为38 nt)的菌株与不含有报告基因的阴性对照菌株相比,荧光量已经下降到同一水平,这说明GFP的表达已被完全抑制。继续延长茎的互补序列长度,对报告基因表达的抑制效果没有显著变化。在PtufB启动子控制下,报告基因gfp能在氧化葡萄糖酸杆菌中顺利表达(+GFP),而插入顺式阻遏序列cR1能显著降低细胞荧光量(+GFP+cR1)。随着顺式阻遏序列与RBS之间所形成的茎区域从32 bp延伸到38 bp(+GFP+cR2),可以检测到进一步的抑制效果。茎区域的进一步延伸(+GFP+cR3和+GFP+cR4)对阻遏作用没有影响。由于顺式抗阻遏序列与阻遏序列之间更易于形成茎-环结构,在启动子和顺式阻遏序列之间引入顺式抗阻遏序列能够通过阻碍RBS和顺式阻遏序列之间茎-环结构的形成而抑制翻译水平上的阻遏作用(+GFP+cR2+cAR)。与RBS互补的反式阻遏序列由独立启动子控制转录后,能显著抑制GFP的翻译(+GFP+trR)。而非互补的对照序列对蛋白质的表达没有影响(+GFP+control)。
2.2 顺式抗阻遏系统对基因表达的影响为了验证报告基因GFP表达的抑制是否由所形成的茎环结构引起的,我们设计了顺式抗阻遏系统。将cAR序列插入在cR2序列的上游,由于它能够与顺式阻遏序列形成更为稳定的二级结构,从而阻碍后者与RBS形成茎环结构。荧光检测结果显示(图 2),与细胞荧光值被cR2完全抑制的菌株相比,引入cAR序列导致菌株的荧光值恢复。
2.3 反式阻遏系统对基因表达的影响不同于顺式阻遏系统,在反式阻遏系统中,游离的非编码sRNA与报告基因mRNA的RBS结合,从而形成RNA二聚体抑制蛋白质翻译。如图 2所示,将trR序列导入正常表达GFP的菌株,会导致细胞的荧光值下降到与不含有gfp基因的野生型菌株相当的水平;而导入对照序列(不与RBS互补)则不会对细胞的荧光值产生明显影响。这一结果表明,所设计的反式阻遏系统是序列特异性的。
2.4 sRNA对EI表达的影响为了检验sRNA对氧化葡萄糖酸杆菌内源性基因的抑制效果,我们基于反式阻遏系统的设计原理,构建了一系列与EI起始密码子周围不同区域互补的sRNA。Western blot结果(图 3)显示,当EI-sRNA-1和EI-sRNA-2存在时,EI的表达量显著降低。而EI-sRNA-1和EI-sRNA-2所编码的sRNA分别与EI开放阅读框的前10个核苷酸和前15个核苷酸互补。如果sRNA与开放阅读框的互补序列进一步移向mRNA的3'端,无法观察到有效的抑制效果。

|
| 图 3 氧化葡萄糖酸杆菌中小RNA在翻译水平上对内源基因pstI表达调节的Western杂交分析 携带各种pBBR-tufB-EI-sRNAs的氧化葡萄糖酸杆菌DSM2003菌株经细胞破碎后,将上清液的总蛋白浓度调节为0.5 mg/mL,以抗EI血清作为一抗进行Western杂交分析。其中,以纯化后的EI作为阳性对照(泳道1)。与野生型菌株(泳道2)相比,在含有EI-sRNA-1(泳道3)和EI-sRNA-2(泳道4)的样品中,目标基因的表达受到了明显抑制,而在含有EI-sRNA-3(泳道5)或EI-sRNA-4(泳道6)的样品中,没有发现阻遏效应 |
虽然氧化葡萄糖酸杆菌作为一种工业菌株已经得到了广泛的应用,但对其基因表达进行调控的方法却很有限。在氧化葡萄糖酸杆菌中实现特异性基因表达抑制的传统方法是基因敲除。该方法通过三亲本接合及细胞内同源重组得以实现。这种基因敲除的方法在很多时候是耗时、费力和低效的。例如,我们曾尝试用这种方法在氧化葡萄糖杆菌中构建pstI(编码PTS的EI蛋白)缺失菌株,但经过长时间的筛选最终未能获得突变菌株(实验结果未展示)。
由于sRNA在某些细菌中已经成功地用于在转录后水平上有效调控特定基因的表达,在本研究中我们尝试构建了3种类型的人工sRNA,检测类似的方法能否在氧化葡萄糖酸杆菌中实现对特定基因的表达抑制。
正如含有顺势阻遏子的菌株荧光值显著降低所显示的,在我们所构建的顺式调控系统中,位于报告基因上游的茎环结构覆盖了RBS区,从而抑制GFP的产生。而且,这样的抑制效果取决于与RBS互补的茎序列的长度。在茎序列长度为32 nt的cR1序列中,与RBS互补的区域延伸至RBS下游6 nt处,这一序列只能实现对细胞荧光值的部分抑制,说明细胞中仍能翻译生成部分报告基因。而在cR2序列中,其与RBS互补的区域延伸至RBS下游12 nt处,荧光检测结果显示,报告基因的表达基本被完全抑制。
在顺式阻遏系统中引入抗阻遏cAR序列的情况下,由于cR序列能与cAR序列形成更稳定的二级结构(cR2-RBS所形成的茎环结构中,互补区形成的茎长度为38 bp,而cR-cAR形成的茎环结构中茎长度为44 bp),导致RBS暴露,无法抑制报告基因的表达。
这样的结果清楚地表明,sRNA与RBS结合的机制同样能够影响氧化葡萄糖酸杆菌中基因的表达,这为在氧化葡萄糖酸杆菌中特异性抑制基因表达提供了一种不同于基因敲除的有效方法。
顺式阻遏系统需要在目的基因上游引入一段RBS互补序列,但目前还缺乏在氧化葡萄糖酸杆菌染色体上进行高效基因操作的方法。为此,我们构建了反式阻遏系统以检测游离的非编码sRNA能否与RBS结合,从而抑制蛋白质的翻译。结果表明,由独立启动子引导的非编码sRNA能够以碱基互补配对的原则与RBS结合并特异性地抑制报告基因的表达。不同于顺式调控系统中的阻遏序列,反式调控系统中的阻遏序列能够通过在氧化葡萄糖酸杆菌中引入携带相应序列的重组质粒产生,而无需在细菌染色体上进行基因操作,为氧化葡萄糖酸杆菌中特异性抑制基因的表达提供了更为灵活的选择。
基于以GFP为目的基因得到的数据,本研究又在氧化葡萄糖酸杆菌中构建了一系列针对pstI基因(编码EI)的反式阻遏sRNA,以检测这种机制能否有效抑制内源性基因的表达。正如western blot实验结果显示,我们所构建的调控序列能够以序列依赖的方式显著抑制氧化葡萄糖酸杆菌内源基因pstI的表达。其中,开放阅读框5'端的15-20个核苷酸以及起始密码子上游15-20个核苷酸可能对抑制蛋白质的翻译具有重要作用。这些发现有助于促进对氧化葡萄糖酸杆菌这一重要工业微生物的基因功能分析。这也表明人工设计sRNA能够与氧化葡萄糖酸杆菌中的一系列翻译调控组件相容,扩大了合成基因网络的调控潜力[21]。本研究中设计的sRNA可应用于氧化葡萄糖酸杆菌基因回路的构建。
此外,在很多细菌中,sRNA对靶mRNA的翻译抑制通常会导致mRNA的快速降解[22-25]。在本研究所构建的顺式阻遏调控系统和反式阻遏调控系统中,阻遏序列与靶mRNA的结合对mRNA的稳定性造成了怎样的影响,仍需要进一步的研究。
4 结论本研究在氧化葡萄糖酸杆菌中构建了顺式和反式阻遏sRNA,这些sRNA能够以碱基互补的方式抑制报告基因的表达。在此基础上,本研究提供了一种在氧化葡萄糖酸杆菌中不同于传统基因敲除的特异性抑制内源基因表达的方法。
| [1] |
Gupta A, Singh VK, Qazi GN, et al. Gluconobacter oxydans:its biotechnological applications[J]. Journal of Molecular Microbiology & Biotechnology, 2001, 3(3): 445. |
| [2] |
Deppenmeier U, Hoffmeister M, Prust C. Biochemistry and biotechnological applications of Gluconobacter strains[J]. Applied Microbiology & Biotechnology, 2002, 60(3): 233-242. |
| [3] |
Robert DH. Recent patents on vitamin C:opportunities for crop improvement and single-step biological manufacture[J]. Recent Patents on Food, Nutrition & Agriculture, 2009, 1(1): 39-49. |
| [4] |
De Muynck C, Pereira C, Naessens M, et al. The genus Gluconobacter oxydans:comprehensive overview of biochemistry and biotechnological applications[J]. Critical Reviews in Biotechnology, 2007, 27(3): 147-171. DOI:10.1080/07388550701503584 |
| [5] |
Prust C, Hoffmeister M, Liesegang H, et al. Complete genome sequence of the acetic acid bacterium Gluconobacter oxydans[J]. Nature Biotechnology, 2005, 23: 195-200. DOI:10.1038/nbt1062 |
| [6] |
Deppenmeier U, Ehrenreich A. Physiology of acetic acid bacteria in light of the genome sequence of Gluconobacter oxydans[J]. Journal of Molecular Microbiology Biotechnology, 2009, 16: 69-80. DOI:10.1159/000142895 |
| [7] |
Hölscher T, Görisch H. Knockout and overexpression of pyrroloquinoline quinone biosynthetic genes in Gluconobacter oxydans 621H[J]. J Bacteriol, 2006, 188: 7668-7676. DOI:10.1128/JB.01009-06 |
| [8] |
Joyce GF. The antiquity of RNA-based evolution[J]. Nature, 2002, 418: 214-221. DOI:10.1038/418214a |
| [9] |
Isaacs FJ, Dwyer DJ, Ding C, et al. Engineered riboregulators enable post-transcriptional control of gene expression[J]. Nature Biotechnology, 2004, 22: 841-847. DOI:10.1038/nbt986 |
| [10] |
Kruger K, Grabowski PJ, Zaug AJ, et al. Self-splicing RNA:Autoexcision and autocyclization of the ribosomal RNA intervening sequence of tetrahymena[J]. Cell, 1982, 31: 147-157. DOI:10.1016/0092-8674(82)90414-7 |
| [11] |
Doudna JA, Cech TR. The chemical repertoire of natural ribozymes[J]. Nature, 2002, 418: 222-228. DOI:10.1038/418222a |
| [12] |
Ambros V. MicroRNA pathways in flies and worms:Growth, death, fat, stress, and timing[J]. Cell, 2003, 113: 673-676. DOI:10.1016/S0092-8674(03)00428-8 |
| [13] |
Carrington JC, Ambros V. Role of microRNAs in plant and animal development[J]. Science, 2003, 301: 336. DOI:10.1126/science.1085242 |
| [14] |
Finnegan EJ, Matzke MA. The small RNA world[J]. Journal of Cell Science, 2003, 116: 4689-4693. DOI:10.1242/jcs.00838 |
| [15] |
Massé E, Gottesman S. A small RNA regulates the expression of genes involved in iron metabolism in Escherichia coli[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(7): 4620-4625. DOI:10.1073/pnas.032066599 |
| [16] |
Wassarman KM, Repoila F, Rosenow C, et al. Identification of novel small RNAs using comparative genomics and microarrays[J]. Genes & Development, 2001, 15(13): 1637-1651. |
| [17] |
Desnoyers G, Bouchard MP, Massé E. New insights into small RNA-dependent translational regulation in prokaryotes[J]. Trends in Genetics, 2013, 29(2): 92. DOI:10.1016/j.tig.2012.10.004 |
| [18] |
Kovach ME, Elzer PH, Hill DS, et al. Four new derivatives of the broad-host-range cloning vector pBBR1MCS, carrying different antibiotic-resistance cassettes[J]. Gene, 1995, 166(1): 175-176. DOI:10.1016/0378-1119(95)00584-1 |
| [19] |
Merfort M, Herrmann U, Bringer-Meyer S, Sahm H. High-yield 5-keto-D-gluconic acid formation is mediated by soluble and membrane-bound gluconate-5-dehydrogenases of Gluconobacter oxydans[J]. Appl Microbiol Biotechnol, 2006, 73(2): 443-451. DOI:10.1007/s00253-006-0467-6 |
| [20] |
Rudzinska M, Kowalewska B, Sikorska K. Clinical usefulness of Western blotting and ELISA avidity for the diagnosis of human toxocariasis[J]. Parasite Immunology, 2017, 39(1): e12400. DOI:10.1111/pim.2017.39.issue-1 |
| [21] |
Massé E, Escorcia FE, Gottesman S. Coupled degradation of a small regulatory RNA and its mRNA targets in Escherichia coli[J]. Genes & Development, 2003, 17(19): 2374-2383. |
| [22] |
Hasty J, McMillenD, Collins JJ. Engineered gene circuits[J]. Nature, 2002, 420: 224-230. DOI:10.1038/nature01257 |
| [23] |
Prévost K, Desnoyers G, Jacques JF, et al. Small RNA-induced mRNA degradation achieved through both translation block and activated cleavage[J]. Genes & Development, 2011, 25: 385-396. |
| [24] |
Morita T, Maki K, Aiba H. RNase E-based ribonucleoprotein complexes:mechanical basis of mRNA destabilization mediated by bacterial noncoding RNAs[J]. Genes & Development, 2005, 19: 2176-2186. |
| [25] |
Ikeda Y, Yagi M, Morita T, et al. Hfq binding at RhlB-recognition region of RNase E is crucial for the rapid degradation of target mRNAs mediated by sRNAs in Escherichia coli[J]. Molecular Microbiology, 2011, 79(2): 419-432. DOI:10.1111/mmi.2011.79.issue-2 |





