2. 中国科学院微生物研究所,北京 100080
2. Institute of Microbiology, Chinese Academy of Sciences, Beijing 100080
金针菇(Flammulina velutipes)又被称为金钱菇、小火菇,是目前食用菌消费市场上常见的品种之一,其农艺性状具有菌柄细长,菌盖小的特点[1]。金针菇子实体富含各类氨基酸,其中尤以精氨酸含量显著高于其他食用菌[2]。此外,在金针菇的子实体中,菌柄部位为主要供食用的部分。因此在实际生产实践中,菌柄长度往往决定了金针菇商品性状优良与否。目前在子实体菌柄伸长机制的研究中依然以传统的研究方式居多,但随着金针菇基因组破译和不断丰富的分子生物学操作技术,为揭示金针菇子实体菌柄伸长发育的分子机理研究提供数据支撑[3]。
腺苷酸合成酶系(Adenylate-forming enzymes)是一类广泛分布在生物界当中的一种酶系,其在原核与真核生物的脂肪酸代谢、能量代谢等生理生化过程中扮演了重要角色[4]。腺苷酸合成酶的作用机制在ATP、Mg2+等辅因子的帮助下催化弱亲核性的羧酸基团与弱亲电性的磷酸基团间的聚合反应[5-6]。根据结构特点,该酶系分为3大家族,分别为ClassⅠ、ClassⅡ和ClassⅢ。其中,ClassⅠ又分为3个亚族,分别为ClassⅠa:NRPS(Adenylation domains of nonribosomal peptide synthetases)家族、ClassⅠb:乙酰辅酶A合成酶和脂酰辅酶A合成酶家族、ClassⅠc:荧光素酶家族[7-9]。有研究发现,腺苷酸合成酶系中荧光素酶能够催化分子氧氧化荧光素而发光的过程,其作用的实质是该酶能够在ATP、Mg2+辅因子的帮助下催化脂肪酸生成脂酰辅酶A,从而催化该反应的进行[10]。另外,该酶系在生物体内各不同的生物过程中也起到了重要作用,如蛋白质合成、DNA合成及次生代谢产物合成等[11-18]。
本研究以本实验室前期筛选得到金针菇编码腺苷酸合成酶基因Fv-Afe1作为对象基因,结合生物信息学、RNAseq等实验技术手段,分析了Fv-Afe1基因及其产物的基本结构,并通过荧光定量手段探究其在金针菇子实体菌柄伸长及在子实体发育过程中的作用。
1 材料与方法 1.1 材料金针菇双核菌株FL19和金针菇单核菌株L11,均由福建省食用菌种质资源保藏与管理中心提供。
1.2 方法 1.2.1 样品收集金针菇双核菌株FL19出菇参照吕作舟等的方法[19],采集菌丝并在出菇后采集子实体原基、伸长期菌柄、伸长期菌盖、成熟期菌柄和成熟期菌盖存放于-80℃,用于RNA的提取。
1.2.2 参考基因组和转录组金针菇单核菌株L11基因组数据和双核菌株H1123转录组及表达谱数据均由福建农林大学菌物研究中心提供,L11基因组登录号为APIA00000000(BioProject:191865)。L11基因组数据及H1123转录组均由深圳华大基因研究院进行测序。
1.2.3 RNA的提取及第一链cDNA的合成采用OMEGA E.Z.N.A.Plant RNA Kit(R6827-01)试剂盒(Omega Bio-Tek,美国)提取双核菌丝体总RNA,然后使用TransScript All-in-One First-Strand cDNA Sythesis Super Mix for qPCR试剂盒(全式金生物技术有限公司,北京)反转录合成cDNA,操作参照试剂盒说明书。
1.2.4 Fv-Afe1基因的确定及生物信息学分析将金针菇H1123的子实体原基、伸长期菌柄、伸长期菌盖、成熟期菌柄和成熟期菌盖的转录组数据进行筛选、分析并获取基因编号,以L11基因组数据、H1123转录组数据作为参考,获取基因DNA和氨基酸序列。采用SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP)进行蛋白质信号肽预测与分析;采用TargetP程序(http://www.cbs.dtu.dk/services/TargetP)对蛋白质亚细胞进行定位分析;使用TMHMM Server v. 2.0在线跨膜结构域预测工具(http://www.cbs.dtu.dk/services/TMHMM/)对金针菇类信息素受体家族蛋白进行跨膜结构域预测。
采用Phyre2程序(http://www.sbg.bio.ic.ac.uk/phyre2)分析蛋白质二级结构和三级结构;将验证后的Fv-Afe1序列提交到EBI的Interpro数据库上预测分析其保守结构域;并从NCBI数据库中下载部分腺苷酸合成酶氨基酸序列,应用MEGA6.0软件采用邻位相连法(Neighbor-Joining)进行系统发育树的构建,Bootstrap value设为1 000[20]。参照严俊杰等[21]的方法,以Fv-Afe1基因序列分别向两侧延长1.5 kb作为参考序列,采用ZOOM软件将转录组测序读段定位至参考序列上[22],参数设置为双末端模式,允许碱基错配数设置为38 bp。对定位结果进行分析,确定基因内含子、外显子。
1.2.5 Fv-Afe1基因表达量分析以Fv-Afe1全长基因序列为参照序列(Reference sequence),通过ZOOM软件将金针菇6个生长时期的表达标签库(Tag library)进行定位(Mapping),统计在Fv-Afe1基因上具有唯一定位的标签种类及其数量并且进行标准化处理,最终数据输出用TPM(Transcript per million clean tags)表示。
TransStart TOP Green qPCR(全式金生物技术有限公司,北京)作为实时荧光定量PCR(qRT-PCR)的定量试剂,使用CFX96实时荧光定量PCR仪(Bio-Rad,美国)进行PCR反应,采用Primer Premier 5进行实时荧光定量PCR引物设计,以GAPDH基因作为内参基因进行表达量检测,引物序列见表 1。反应体系及反应程序参照实时荧光定量PCR试剂盒说明书。相对表达量采用2-△△Ct法处理。
Fv-Afe1在金针菇L11基因组中的ID号为gene9981,通过Blastp的结果显示该基因含有腺苷酸合成酶的保守结构域,属于腺苷酸合成酶超家族成员基因,将其命名为Fv-Afe1。基因全长为967bp,通过转录组reads定位获得该基因的转录本全长为795 bp,含有4个外显子,3个内含子(图 1)。
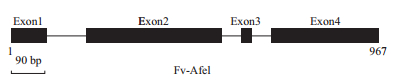
|
| 图 1 Fv-Afe1基因的结构 |
将Fv-Afe1的氨基酸序列提交到EBI蛋白数据库中进行结构域分析,InterPro结果显示金针菇Fv-Afe1氨基酸序列含有AMP-dependent synthetase/ligase(IPR000873)结构域和AMP-binding enzyme,C-terminal domain(IPR025110)结构域,这两个结构域与腺苷酸合成酶氨基酸的典型结构域保持一致,用Phyre2在线预测Fv-Afe1三级结构发现,Fv-Afe1三级结构被分为两个Domain,它们之间由铰链结构(Hinge)连接,Fv-Afe1的N端Domain结构包括α螺旋,β桶(图 2)。从Interpro分析结果和Phyre2蛋白质三级结构预测,Fv-Afe1从蛋白结构上来看具有腺苷酸合成酶的催化活性。对该氨基酸序列进行跨膜结构、信号肽和亚细胞定位预测,结果显示,Fv-Afe1基因产物不具备跨膜位点,无信号肽存在,亚细胞定位显示其定位于胞内,说明该蛋白属于胞内酶,作用于细胞内。

|
| 图 2 Fv-Afe1蛋白质三维结构图 |
从NCBI下载部分已明确作用、且结构典型的腺苷酸合成酶家族氨基酸序列与Fv-Afe1进行系统发育分析(图 3)。从系统发育树可以看到,Fv-Afe1与真菌腺苷酸酶基因聚在一支,其他两支分别为以源氏萤(Luciola cruciata)为代表荧光素酶家族和以肺炎双球菌(Streptococcus pneumoniae)为代表的腺苷酸激酶家族。通过Fv-Afe1基因与糙皮侧耳(Pleurotus osteatus)、双色蜡蘑(Laccaria bicolor)、紫蜡蘑(Laccaria amethystina)和云芝(Trametes versicolor)的腺苷酸合成酶基因进行系统发育分析,结果显示它们之间的同源性极高,置信度达到100%。

|
| 图 3 Fv-Afe1系统进化分析 |
金针菇的转录组和表达谱数据分析中可以得出Fv-Afe1基因在各个时期均表达,其中在伸长期菌柄表达量显著高于其它时期,该基因在金针菇各个发育时期不同部位的荧光定量结果也符合这一规律(图 4),说明这一基因可能与金针菇菌柄伸长有较为密切的关系。
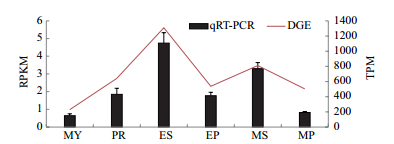
|
| 图 4 Fv-Afe1基因在金针菇的6个时期的表达谱及qRT-PCR的表达分析 MY:菌丝;PR:原基;ES:伸长期菌柄;EP:伸长期菌盖;MS:成熟期菌柄;MP:成熟期菌盖 |
有研究发现金针菇的菌盖和菌柄的交界处下1-2 mm处是伸长的主要区域[23]。另外,有发现认为在真菌中菌柄的伸长生长是通过吸收水分以及细胞的表面积扩张实现的,细胞壁的松弛也使得新的物质掺入从而适应细胞的伸长[24]。也有研究发现一些酶系和转录因子对细胞形态建成的变化具有作用[25]。因此,菌柄伸长机制是一个复杂的过程,涉及到多种酶系的调控网络[26]。本研究基于基因组数据,在金针菇中鉴定获得一个腺苷酸合成酶基因并对其基因结构及不同发育时期的表达模式进行了详细的分析,这对于其他担子菌的相关研究具有一定的参考价值。
有研究认为金针菇菌柄伸长是受到菌褶中激素的调控,类似于植物中关于生长素的“酸生长学说”[27]。该研究认为生长素通过结合并活化存在于质膜上质子泵(H+-ATP酶),转运细胞内的氢离子到细胞壁上,降低pH值,使基质环境处于酸性,进而细胞壁中的一些氢键断裂,同时活化了细胞壁上的多糖水解酶,催化断裂纤维素微纤丝和葡聚糖之间连接的键,最终致使细胞壁变得松弛。
腺苷酸合成酶超家族广泛存在于生物中,该家族涉及生化过程中重要的中间代谢物脂酰辅酶A和乙酰辅酶A的合成与分解,使得该家族基因能够参与到蛋白质合成、脂质分解代谢及能量代谢等生化过程中[28-29]。而科研人员在肺炎双球菌(Streptococcus pneumoniae)中发现,腺苷酸激酶的表达与其生长速率存在关联,作用机制是通过增加ATP在机体的含量从而影响肺炎双球菌的生长[30]。本研究通过转录组和表达谱数据显示,Fv-Afe1在金针菇伸长菌柄时期具有最高表达,在成熟期菌柄具有次高表达,这说明该基因可能参与到了菌柄形态建成的基因调控,特别是对于菌柄的伸长过程可能起到了重要作用。但是关于该基因具体以何种方式参与到金针菇菌柄伸长过程,以及该基因位于基因调控网络何种位置还需更加深入的研究。
4 结论氨基酸三级结构预测、生物信息学分析及系统发育显示Fv-Afe1属于腺苷酸合成酶超家族成员,荧光定量实验和转录组表达谱分析显示该基因在菌柄伸长期菌柄部位具有最高表达,成熟期菌柄具有次高表达,这说明该基因与菌柄伸长机制有关。
| [1] |
毕志树, 郑国扬, 李泰辉. 广东大型真菌志[M]. 广州: 广东科技出版社, 1994.
|
| [2] |
魏华, 谢俊杰. 金针菇营养保健作用[J]. 天然产物研究与开发, 1997, 9(2): 92-97. |
| [3] |
Lu YP, Chen RL, Long Y, et al. A jacalin-related lectin regulated the formation of aerial mycelium and fruiting body in Flammulina velutipes[J]. Int J Mol Sci, 2016, 17(12): 1884. DOI:10.3390/ijms17121884 |
| [4] |
Zhang Z, Zhou R, Sauder JM, et al. Structural and functional studies of fatty Acyl adenylate ligases from E. coli and L. pneumophila[J]. J Mol Biol, 2011, 406: 313-324. DOI:10.1016/j.jmb.2010.12.011 |
| [5] |
Bronstein I, Fortin J, Stanley PE, et al. Chemiluminescent and bioluminescent reporter gene assays[J]. Anal Biochem, 1994, 219: 169-181. DOI:10.1006/abio.1994.1254 |
| [6] |
Greer LF Ⅲ, Szalay AA. Imaging of light emission from the expression of luciferases in living cells and organisms: a review[J]. Luminescence, 2002, 17: 43-73. DOI:10.1002/(ISSN)1522-7243 |
| [7] |
Schmelz S, Naismith JH. Adenylate-forming enzymes[J]. Current Opinion in Structural Biology, 2009, 19: 666-671. DOI:10.1016/j.sbi.2009.09.004 |
| [8] |
Wu R, Reger AS, Lu X, et al. The mechanism of domain alternation in the acyl-adenylate forming ligase superfamily member 4-chlorobenzoate: coenzyme A ligase[J]. Biochemistry, 2009, 48: 4115-4125. DOI:10.1021/bi9002327 |
| [9] |
Weimer KM, Shane BL, Brunetto M, et al. Evolutionary basis for the coupled-domain motions in Thermus thermophilus leucyl-tRNA synthetase[J]. J Biol Chem, 2009, 284(15): 10088-10099. DOI:10.1074/jbc.M807361200 |
| [10] |
Perona JJ, Hou YM. Indirect readout of tRNA for aminoacylation[J]. Biochemistry, 2007, 46: 10419-10432. DOI:10.1021/bi7014647 |
| [11] |
Inouye S. Firefly luciferase: an adenylate-forming enzyme for multicatalytic functions[J]. Cell Mol Life Sci, 2010, 67: 387-404. DOI:10.1007/s00018-009-0170-8 |
| [12] |
Schulman BA, Harper JW. Ubiquitin-like protein activation by E1 enzymes: the apex for downstream signalling pathways[J]. Nat Rev Mol Cell Biol, 2009, 10: 319-331. |
| [13] |
Shuman S. DNA ligases: progress and prospects[J]. J Biol Chem, 2009, 284: 17365-17369. DOI:10.1074/jbc.R900017200 |
| [14] |
Chapman-Smith A, Cronan JJE. The enzymatic biotinylation of proteins: a post-translational modification of exceptional specificity[J]. Trends Biochem Sci, 1999, 24: 359-363. DOI:10.1016/S0968-0004(99)01438-3 |
| [15] |
Chen L, Petrelli R, Felczak K, et al. Pankiewicz, Nicotinamide adenine dinucleotide based therapeutics[J]. Curr Med Chem, 2008, 15: 650-670. DOI:10.2174/092986708783885282 |
| [16] |
Watkins PA. Fatty acid activation, Prog[J]. Lipid Res, 1997, 36: 55-83. DOI:10.1016/S0163-7827(97)00004-0 |
| [17] |
Sieber SA, Marahiel MA. Molecular mechanisms underlying nonribosomal peptide synthesis: approaches to new antibiotics[J]. Chem Rev, 2005, 105: 715-738. DOI:10.1021/cr0301191 |
| [18] |
Gulick AM. Conformational dynamics in the acyl-CoA synthetases. Adenylation domains of non-ribosomal peptide synthetases, and firefly luciferase[J]. ACS Chem Biol, 2009, 4: 811-827. DOI:10.1021/cb900156h |
| [19] |
吕作舟, 张引芳, 等. 食用菌关键技术问答:金针菇真姬菇杏鲍菇杨树菇[M]. 北京: 化学工业出版社, 2010, 42-64.
|
| [20] |
Tamura K, Stecher G, Peterson D, et al. MEGA6: molecular evolutionary genetics analysis version 6. 0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [21] |
严俊杰, 郭丽羡, 赵静静, 等. 草菇谷胱甘肽S-转移酶编码基因(vv-gto1)的序列分析及其差异表达[J]. 微生物学报, 2014, 10: 1171-1177. |
| [22] |
Zhang ZF, Lin H, Ma B. ZOOM Lite: next-generation sequencing data mapping and visualization software[J]. Nucleic Acids Research, 2010, 38: 743-748. |
| [23] |
李继影. 毛柄金线菌子实体菌柄细胞壁伸长生长的研究[D]. 南京: 南京师范大学, 2005. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y802017
|
| [24] |
Jacobson K, Mouritsen OG, Anderson RGW. Lipid rafts: at a crossroad between cell biology and physics[J]. Nat Cell Biol, 2007, 9(1): 7-14. DOI:10.1038/ncb0107-7 |
| [25] |
Guan XL, Souza CM, Pichler H, et al. Functional interactions between sphingolipids and sterols in biological membranes regulating cell physiology[J]. Mol Biol Cell, 2009, 20(7): 2083-2095. DOI:10.1091/mbc.E08-11-1126 |
| [26] |
Tyler KM, Fridberg A, Toriello KM, et al. Flagellar membrane localization via association with lipid rafts[J]. J Cell Sci, 2009, 122(6): 859-866. DOI:10.1242/jcs.037721 |
| [27] |
Lingwood D, Kai S. Lipid rafts as a membrane-organizing principle[J]. Science, 2010, 327(5961): 46-51. DOI:10.1126/science.1174621 |
| [28] |
Rayle DL, Cleland R. Enhancement of wall loosening and elongation by acid solutions[J]. Plant Physiol, 1970, 46(2): 250-253. DOI:10.1104/pp.46.2.250 |
| [29] |
Robertson NF. Experimental control of hyphal branching and branch form in hyphomycetous fungi[J]. Journal of the Linnean Society of London, Botany, 1959, 56(366): 207-211. DOI:10.1111/j.1095-8339.1959.tb02494.x |
| [30] |
Thach TT, Luong TT, Lee S, et al. Adenylate kinase from Streptococcus pneumoniae is essential for growth through its catalytic activity[J]. FEBS Open Bio, 2014, 4: 672-682. DOI:10.1016/j.fob.2014.07.002 |





