植酸酶又称肌醇六磷酸酶,是能够将植酸分解为肌醇和无机磷的一类磷酸酯酶,广泛地存在于动物、植物、微生物中[1-3]。植酸酶在饲料工业具有广阔的应用前景和巨大的商业价值。植酸酶作为饲料添加剂能够提高磷的利用率,减轻植酸磷对环境的污染;解除饲料中植酸的抗营养作用,提高机体对多种微量元素及蛋白质的利用率、促进动物生长发育和提高其生产性能、减少动物粪便中磷的排放[1-3]。目前,约75%的单胃动物饲料中含有植酸酶,植酸酶的全球市场总销售额高达3.5亿美元,并以每年10%的速度增长[4]。
植酸酶作为一种单胃动物饲料添加剂,其饲喂效果已经在世界范围内得到确认[5]。但是,目前我国植酸酶在饲料中的推广和应用还相当有限,究其原因主要是:天然材料中植酸酶含量太低导致植酸酶生产成本过高;当前商业化植酸酶的热稳定性、活性、pH作用范围以及蛋白酶抗性等酶学性质难以完全满足饲料工业的要求[6-7]。近年来利用基因工程技术提高植酸酶的表达量,以及随着对植酸酶晶体结构的揭示,利用基因工程技术和蛋白质工程技术在分子水平上改造植酸酶基因,从而改变植酸酶的酶学性质,提高其在饲料中使用的有效性已成为国内外学者研究的热点[8-9]。
耶尔森氏菌属Yersinia来源的植酸酶具有酸性条件下的高稳定性和高酶活、较强的蛋白酶抗性和热稳定性等突出性质[10-15]。因此,来源于Yersinia的植酸酶在饲料工业具有巨大的应用潜力,目前德国BASF公司正在对来源于Yersinia的植酸酶进行商业化生产[5]。来源于Yersinia intermedia的植酸酶YiAPPA是目前已知的植酸酶活性最高的植酸酶,其酶活高达3 960 U/mg(37℃、pH4.5),是目前应用最广泛的植酸酶Aspergillus niger PhyA酶活的40倍[10]。YiAPPA属于组氨酸酸性磷酸酶(Histidine acid phosphatase,HAP,EC 3.1.3.2),具有HAP分子结构和催化机制上的共同特征[10]。其最适反应条件为55℃、pH4.5;于pH2.0-5.5条件下具有50%以上的相对酶活;于pH1.0-8.0条件下具有80%以上的稳定性;于80℃保温15 min后,YiAPPA的剩余酶活约为54%;对于胃蛋白酶和胰蛋白酶均具有较强的抗性。YiAPPA的酶学性质使其在饲料工业中具有巨大的应用潜力,但是YiAPPA的酶学性质(特别是热稳定性)难以完全满足饲料工业的要求。以YiAPPA的热稳定性为例,饲用酶在饲料的造粒过程中一般需要经过一个75-93℃的短暂高温,而YiAPPA于80℃的半衰期仅为15 min,尚难以达到饲料工业的要求。本研究拟将YiAPPA与来源于嗜热菌Geobacillus thermoleovorans的嗜热酸性α-淀粉酶GTamy的生淀粉结合域SBD进行融合,获得酶学性质改良的融合酶,从而提高植酸酶YiAPPA在饲料工业中的应用价值。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒大肠杆菌Escherichia coli JM109、枯草芽孢杆菌Bacillus subtilis WB600、枯草芽孢杆菌表达载体pSTOP1622、重组质粒pSTOP1622-AbnA-gtamyhds均由本实验室保存。
1.1.2 培养基大肠杆菌E. coli JM109和枯草芽孢杆菌B. subtilis WB600的培养均采用LB培养基。
1.1.3 试剂KOD-Plus-neo DNA聚合酶购自日本Toyobo公司;DNA限制性内切酶、T4 DNA连接酶、DNA Marker、蛋白质Marker购自美国Fermentase公司;DNA胶回收试剂盒、质粒抽提试剂盒E.Z.N.A.购自美国Omega Bio-tek公司;Chelating SepharoseTM Fast Flow购自美国GE Healthcare公司;Bradford法蛋白浓度测定试剂盒购自上海生工生物工程股份有限公司;其他化学试剂均为国产或进口分析纯;基因合成由上海博益生物科技有限公司完成,聚合酶链式反应(Polymerase chain reaction,PCR)引物合成和测序由上海生工生物工程股份有限公司完成。
1.2 方法 1.2.1 分子克隆技术和表达产物的聚丙烯酰胺凝胶电泳(SDS-PAGE)分析分子克隆技术和表达产物的SDS-PAGE分析参照文献[16]进行。
1.2.2 重组质粒pSTOP1622-yiappah和pSTOP1622-yiappa-sbdh的构建及鉴定由上海博益生物科技有限公司合成包含编码来源于Bacillus subtilis US417的植酸酶的信号肽[17]、不含信号肽的YiAPPA以及连接肽(G4S)2的核苷酸序列。根据人工合成的基因序列设计PCR引物P1、P2及P3,如表 1所示。以合成基因为模板,以P1、P2为引物,进行PCR扩增。PCR扩增条件为:98℃ 5 min;98℃ 20 s,60℃ 20 s,74℃ 2 min,30个循环;74℃,10 min。扩增产物经Spe Ⅰ和Sac Ⅰ双酶切,连接至载体pSTOP1622,构建重组质粒pSTOP1622-yiappa。以合成基因为模板,以P1、P3为引物,进行PCR扩增,PCR扩增条件同上。扩增产物经Spe Ⅰ和Sac Ⅰ双酶切,连接至载体pSTOP1622,构建重组质粒pSTOP1622-yiappah。采用Spe Ⅰ和Sac Ⅰ双酶切重组质粒鉴定是否有外源基因的插入。根据嗜热酸性α-淀粉酶GTamy的基因序列设计PCR扩增其淀粉结合域SBD所需的引物P4和P5(表 1)。以重组质粒pSTOP1622-AbnA-gtamyhds为模板,以P4、P5为引物,进行PCR扩增。PCR扩增条件为:98℃ 5 min;98℃ 20 s,60℃ 20 s,74℃ 30 s,30个循环;74℃,10 min。扩增产物经Sac Ⅰ和Kpn Ⅰ双酶切,连接至经同样双酶切处理后的重组质粒pSTOP1622-yiappa,构建重组质粒pSTOP1622-yiappa-sbdh。采用Spe Ⅰ和Kpn Ⅰ双酶切重组质粒鉴定是否有外源基因的插入。
枯草芽孢杆菌B. subtilis WB600感受态细胞的制备和转化采用改进的Spizizen法[18]进行。
1.2.4 重组酶的诱导表达和纯化接种重组枯草芽孢杆菌单克隆到LB液体培养基中,用250 mL三角瓶进行培养,其中培养基的装液量为20 mL,培养温度为37℃,转速为200 r/min,培养时间为10 h。然后转接该培养物于新鲜LB液体培养基中,用250 mL三角瓶进行培养,其中培养基装液量为25 mL,接种量为3%,培养温度为37℃,转速为200 r/min。当培养至菌体OD600nm达到1时,添加终浓度为0.5%的木糖,诱导细胞表达重组蛋白质,诱导时间为36 h。
采用Ni2+亲和层析柱对发酵上清液中目的蛋白质进行纯化,用200 mmol/L咪唑洗脱缓冲液洗脱,即得到纯化后的重组酶。利用SDS-PAGE检测重组蛋白质的纯度,并采用Bradford法[19]测定重组蛋白质的浓度。
1.2.5 植酸酶的酶活力测定取250 μL酶液于离心管中,加入750 μL 0.25 mol/L醋酸钠缓冲液(pH4.5),混匀。向实验组管中加入2 mL 1.5 mmol/L植酸钠溶液(0.25 mol/L醋酸钠缓冲液,pH4.5),对照组中加入2 mL颜色/终点混合液(钼酸铵/钒酸铵/硝酸),摇匀。于37℃反应30 min后,立即向实验组中加入2 mL颜色/终点混合液,对照组中加入2 mL 1.5 mmol/L植酸钠溶液,并混匀,于415 nm处测定光吸收值。植酸酶活力单位(U)定义为:在37℃、pH4.5的条件下,每分钟从1.5 mmol/L植酸钠溶液中释放出1 μmol/L无机磷所需要的植酸酶量为一个酶活力单位(U)。
1.2.6 植酸酶的酶学性质植酸酶的最适反应pH:将酶液于不同pH(1.0-8.0)条件下测定植酸酶活性。所使用的缓冲液如下:0.25 mol/L甘氨酸-盐酸缓冲液,pH1.0-3.5;0.25 mol/L醋酸钠-醋酸缓冲液,pH3.5-6.0;0.25 mol/L Tris-盐酸缓冲液,pH6.0-8.5。
植酸酶的pH稳定性:将酶液用不同pH(1.0-12.0)缓冲液稀释,使其在不同pH条件下于37℃处理2 h,然后用最适pH缓冲液稀释,并根据如上反应体系测定样品的酶活力。所使用的缓冲液如下:0.25 mol/L甘氨酸-盐酸缓冲液,pH1.0-3.5;0.25 mol/L醋酸钠-醋酸缓冲液,pH3.5-6.0;0.25 mol/L Tris-盐酸缓冲液,pH6.0-8.5;0.25 mol/L甘氨酸-氢氧化钠缓冲液,pH8.5-12.0。
植酸酶的最适反应温度:按照如上反应体系测定样品的酶活力,分别于30-90℃反应30 min,测定不同温度条件下样品的酶活力,并以相对酶活对温度作图,确定其最适反应温度。
植酸酶的动力学参数:用0.25 mol/L醋酸钠缓冲液(pH4.5)配制不同浓度的植酸钠溶液(0.0625%、0.1%、0.125%、0.2%、0.25%、0.5%、1.0%和1.5%),分别向植酸钠溶液中加入等量的酶液,按照1.2.5方法测定酶活。根据双倒数作图法以底物浓度的倒数为横坐标,以比酶活力的倒数为纵坐标作图,直线的斜率为Km/Vmax,截距为1/Vmax,计算以醋酸钠为底物时的米氏常数Km、最大反应速度Vmax和反应常数kcat。
植酸酶的热稳定性:取250 μL酶液于离心管中,加入750 μL 0.25 mol/L醋酸钠缓冲液(pH4.5),混匀。酶液于80℃保温0-40 min后,根据如上反应体系测定样品的酶活力。
植酸酶的蛋白酶抗性:分别用0.25 mol/L Gly-HCl缓冲液(pH 2.0)和0.25 mol/L Tris-HCl缓冲液(pH 7.0)配制0.1 mg/mL的胃蛋白酶和胰蛋白酶。按照蛋白酶与植酸酶质量比为1:10的比例在植酸酶中分别加入胃蛋白酶和胰蛋白酶,于37℃分别保温2 h后,向其中加入蛋白酶抑制剂终止反应,然后对蛋白酶处理的样品用最适pH缓冲液(0.25 mol/L醋酸钠缓冲液,pH 4.5)进行100倍稀释,再根据如上反应体系测定样品的酶活力。
1.2.7 重组酶的生玉米淀粉结合率向0.1 mg/mL酶液中加入生玉米淀粉至其终浓度为0-10%,混合体系在20℃的恒温振荡培养箱孵育3 h,振荡速率为180 r/min。反应结束后,测定上清中的残余植酸酶活性。结合率AR计算公式如下:AR%=(O-R)/O×100。R:上清残余酶活力;O:初始酶活力。
1.2.8 数据统计分析以上酶学性质研究实验中,每个实验做3个平行。运用软件SigmaPlot 11.0对试验数据进行统计分析并作图,数据均以x±s表示。
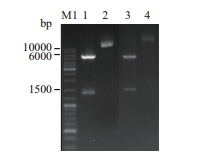
2 结果 2.1 重组质粒的酶切鉴定采用限制性内切酶Spe Ⅰ和Sac Ⅰ酶切质粒pSTOP1622-yiappah,采用限制性内切酶Spe Ⅰ和Kpn Ⅰ酶切质粒pSTOP1622-yiappa-sbdh,得到大小分别约为1.3 kb和1.6 kb的两个片段(图 1),大小与理论值相符。同时将以上重组质粒送至上海生工生物工程股份有限公司进行插入片段测序,并将测序结果(结果未显示)与对应的基因序列进行了比对确认,证实重组质粒构建成功。
|
| 图 1 重组质粒的酶切鉴定 M:1 kb ladder;1:质粒pSTOP1622-yiappah经Spe Ⅰ和Sac Ⅰ双酶切的产物;2:质粒pSTOP1622-yiappah;3:质粒pSTOP1622-yiappa-sbdh经Spe Ⅰ和Kpn Ⅰ双酶切的产物;4:质粒pSTOP1622-yiappa-sbdh |
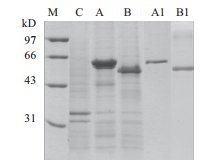
将阴性对照pSTOP1622和重组质粒pSTOP1622-yiappa-sbdh、pSTOP1622-yiappah分别转化枯草芽孢杆菌B. subtilis WB600,获得重组枯草芽孢杆菌并进行诱导表达。待表达完成后,收集发酵上清液,采用SDS-PAGE分析发酵上清液中重组植酸酶的表达情况。如图 2所示,目的蛋白质YiAPPA-SBD和YiAPPA均得到成功表达和纯化,YiAPPA-SBD的表观分子质量约为58 kD,YiAPPA的表观分子质量约为46 kD,两者的大小均与理论值相符。
|
| 图 2 重组植酸酶的SDS-PAGE检测结果 C:含有质粒pSTOP1622的重组枯草芽孢杆菌的发酵上清液样品;A:含有重组质粒pSTOP1622-yiappa-sbdh的重组枯草芽孢杆菌的发酵上清液样品;B:含有重组质粒pSTOP1622-yiappah的重组枯草芽孢杆菌的发酵上清液样品;A1:采用Ni2+亲和层析获得的YiAPPA-SBD纯化样品;B1:采用Ni2+亲和层析获得的YiAPPA纯化样品 |
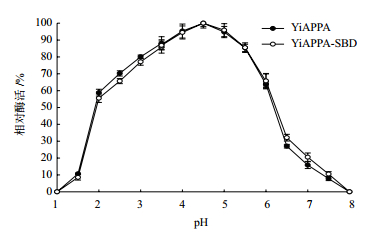
将酶液于不同pH(1.0-8.0)条件下测定重组植酸酶的相对酶活,结果如图 3所示。重组植酸酶YiAPPA和YiAPPA-SBD的最适反应pH值均为4.5,并且重组植酸酶YiAPPA和YiAPPA-SBD在pH1.0-8.0范围内具有相似的相对酶活。
|
| 图 3 pH对酶活的影响 |
由图 4可知,重组植酸酶YiAPPA和YiAPPA-SBD在pH1.0-12.0范围内具有相似的稳定性。其中,在pH1.0-3.0范围内,重组植酸酶具有80%以上的相对酶活;在pH4.0-10.0范围内,重组植酸酶具有90%以上的相对酶活;在pH11.0-12.0范围内,重组植酸酶均不稳定,很快丧失活性。
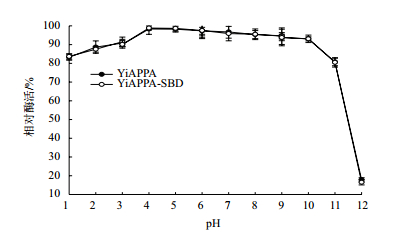
|
| 图 4 pH对稳定性的影响 |
按照植酸酶的酶活力测定反应体系,于30-90℃下测定重组植酸酶的酶活力,以样品的相对酶活对温度作图的结果如图 5所示。重组植酸酶YiAPPA和YiAPPA-SBD的最适反应温度均为55℃。在30-55℃范围内,两者具有相似的相对酶活;在55-90℃范围内,YiAPPA-SBD的相对酶活均高于YiAPPA的相对酶活。以上结果表明,融合酶YiAPPA-SBD的高温活性得到提高。

|
| 图 5 温度对酶活的影响 |
重组植酸酶以植酸钠为底物时的比活力、米氏常数Km和反应常数kcat如表 2所示。与YiAPPA相比,融合酶YiAPPA-SBD的比活力、Km及kcat值均未明显改变。即SBD在YiAPPA的C末端的融合表达未影响YiAPPA的底物结合能力和底物催化能力。
将重组植酸酶YiAPPA和YiAPPA-SBD于80℃分别保温不同时间,测定其相对酶活,比较两者的热稳定性,结果如图 6所示。在80℃下,融合酶YiAPPA-SBD的热稳定性明显高于YiAPPA的热稳定性。其中YiAPPA于80℃的半衰期约为15 min,YiAPPA-SBD于80℃的半衰期约为30 min,提高了2倍。
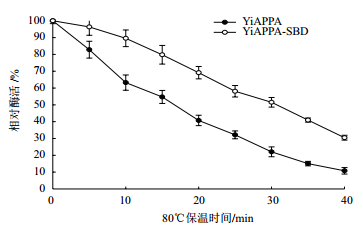
|
| 图 6 80℃重组植酸酶的热稳定性 |
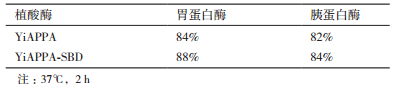
向重组植酸酶样品中分别加入胃蛋白酶和胰蛋白酶,于37℃分别保温2 h后,测定样品的剩余酶活力。表 3中结果显示,重组植酸酶YiAPPA和YiAPPA-SBD对蛋白酶具有相近的抗性。重组植酸酶经胃蛋白酶处理后仍保留有84%以上的相对酶活;重组植酸酶经胰蛋白酶处理后仍保留有82%以上的相对酶活。
向重组植酸酶样品中加入不同浓度的生玉米淀粉,待孵育结束后测定重组植酸酶对生玉米淀粉的结合率,结果如图 7所示。在生玉米淀粉浓度为0-10%的条件下,重组植酸酶YiAPPA对生玉米淀粉的结合率为0;融合酶YiAPPA-SBD对生玉米淀粉的结合率随着生玉米淀粉浓度的增加而提高。当生玉米淀粉浓度大于8%时,YiAPPA-SBD对其结合率达到80%以上。
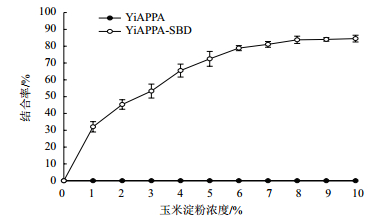
|
| 图 7 重组植酸酶对生玉米淀粉的结合率 |
向0.1 mg/mL重组植酸酶中加入生玉米淀粉至其浓度为0、15%、30%、45%和60%,混合体系在20℃的恒温振荡培养箱孵育3 h,然后取1 mL酶液样品直接加入底物进行植酸酶的酶活力测定,以样品的相对酶活对反应体系中生玉米淀粉终浓度作图的结果如图 8所示。在生玉米淀粉存在的条件下,未结合生玉米淀粉的YiAPPA与结合了生玉米淀粉的YiAPPA-SBD的相对酶活基本一致,这表明生玉米淀粉的结合不影响融合酶的植酸酶活性。
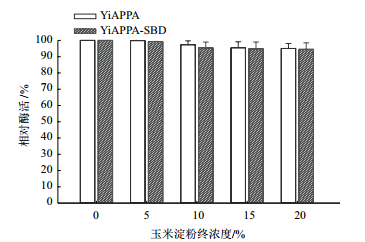
|
| 图 8 生玉米淀粉对重组植酸酶相对酶活的影响 |
目前商业化植酸酶主要是来源于真菌和细菌,如来源于黑曲霉A. niger的植酸酶PhyA、来源于大肠杆菌Escherichia coli的植酸酶AppA和AppA2、来源于Citrobacter braakii的植酸酶CbAppA[5]。但是这些商业化植酸酶具有热稳定性差、酶活低、pH作用范围窄等缺点。例如,饲用酶在饲料的造粒过程中一般需要经过一个75-93℃的短暂高温。A. niger PhyA经70℃处理20 min后仅残留30%的活性,在此温度范围内易失活,酶活性大大降低;添加在饲料中的植酸酶,最终的作用场所是动物的胃肠道,主要场所是胃,降解时的pH为1.8-3.5。A. niger PhyA的最适pH值分别是pH 2.5和5.5,在两个峰值之间的酶活相对偏低。商业化植酸酶的这些缺点不利于植酸酶在饲料工业的推广与应用。研究者已致力于采用分子生物学手段改造这些植酸酶的酶学性质,并取得了较好的研究结果。Zhang等[20]对比分析了A. niger PhyA和具有优良热稳定性的A. fumigatus Afp的三级结构,采用定点突变向PhyA中引入Afp中对应的氨基酸残基,获得了变性温度提高7℃的突变体A58E/P65S/Q191R/T271R。Kim等[21]采用易错PCR来提高E.Coli AppA2的热稳定性,获得了热变性温度提高6-7℃的两个突变体K64E和K65E/K97M/S209G。Sanchez-Romero等向来源于Citrobacter braakii的植酸酶CbAppA中引入3对二硫键,使其变性温度提高了12.1℃[22]。Tian等[23]对比了多种不同植酸酶的氨基酸序列,发现第53位和第91位氨基酸残基具有较强可变性。来源于A. niger 113的植酸酶PhyI 1s与来源于A. niger NRRL 3135的植酸酶PhyA的氨基酸序列的相似性高达98%,但PhyI 1s的活性仅为PhyA的1/10。他们将PhyI 1s中第53位和第91位氨基酸残基突变为PhyA中对应的氨基酸残基,获得了催化效率(kcat/Km)分别提高了4.08倍的突变体Q53R和2.84倍的突变体K91R。Ushasree等[24]对来源于A. niger NII 08121的植酸酶进行定点突变,获得了最适pH为3.2的突变体P212H。
嗜热酸性生淀粉α-淀粉酶GTamy具有优良的高温活性和热稳定性,其最适反应温度为80℃,于80℃的半衰期为184 min,其活性和热稳定性均不依赖于Ca2+[25]。GTamy属于糖苷水解酶类的第13家族,具有第13家族酶分子结构和催化机制上的共同特征,如保守区域、保守的催化活性中心、金属离子结合位点和3个结构域等[25]。GTamy的结构域C作为生淀粉结合域SBD,是其直接水解未经糊化处理的玉米淀粉的关键[25]。SBD作为淀粉酶的天然部分主要有以下作用:使淀粉酶分子可以在溶液中与不溶性底物(淀粉颗粒)结合,将底物运送到催化结构域的活性位点以及使淀粉颗粒表面破裂[26-29]。除此之外,SBD还具有一些特殊功能。例如,Gt-amy Ⅱ的SBD还与其热稳定性相关[30];AmyP的SBD与其可溶性淀粉酶活相关[31]。已有研究表明SBD与异源蛋白质的融合表达会对异源蛋白质的酶学性质产生影响。例如,Hua等[32]将来源于Bacillus stearothermophilus的亮氨酸氨肽酶LAPII与来源于Bacillus sp. TS-23的α-淀粉酶的生淀粉结合域SBD进行融合表达,获得了高温活性和热稳定性均得到提高的融合酶LAPsbd。
本研究通过在植酸酶YiAPPA的C末端融合嗜热酸性α-淀粉酶GTamy的生淀粉结合域SBD,获得了热稳定性和高温活性均得到提高的融合酶YiAPPA-SBD。这表明将GTamy-SBD与YiAPPA进行融合表达,有利于维持YiAPPA高温条件下的结构稳定性。并且融合酶YiAPPA-SBD仍保留YiAPPA的其它优良酶学性质,如其最适反应条件为55℃、pH4.5,37℃的绝对酶活约为3 900 U/mg,并具有优良的pH稳定性和蛋白酶抗性。热稳定性和高温活性均得到提高的融合酶YiAPPA-SBD较YiAPPA更适于饲料工业的应用。此外,融合酶YiAPPA-SBD具有玉米淀粉结合能力,而且生玉米淀粉的结合不影响融合酶的植酸酶活性,这表明植酸酶YiAPPA和生淀粉结合域SBD均能独立发挥功能。接下来我们将根据这一特点,探索利用玉米淀粉吸附法一步纯化发酵上清液中融合酶YiAPPA-SBD的条件,从而简化融合酶YiAPPA-SBD的生产工艺。
4 结论本研究获得了高温活性和热稳定性均得到提高的融合酶YiAPPA-SBD,其于55-90℃范围内的相对酶活均高于YiAPPA的相对酶活,于80℃的半衰期提高约2倍。并且融合酶YiAPPA-SBD获得了对生玉米淀粉的结合能力,在生玉米淀粉浓度>8%的条件下,YiAPPA-SBD对其结合率达到80%以上。此外,融合酶YiAPPA-SBD保留有YiAPPA的其他优良酶学性质,最适反应pH为4.5,37℃的绝对酶活高达3 900 U/mg,并具有优良的pH稳定性和蛋白酶抗性。
| [1] |
Rao D, Rao KV, Reddy TP, et al. Molecular characterization, physic-ochemical properties, known and potential applications of phytases: an overview[J]. Crit Rev Biotechnol, 2009, 2: 182-198. |
| [2] |
Bhavsar K. Current research and future perspectives of phytase bioprocessing[J]. RSC Advances, 2014, 4(51): 26677-26691. DOI:10.1039/C4RA03445G |
| [3] |
Yao MZ, Zhang YH, Lu WL, et al. Phytases: crystal structures, protein engineering and potential biotechnological applications[J]. Journal of Applied Microbiology, 2012, 112(1): 1-14. DOI:10.1111/jam.2011.112.issue-1 |
| [4] |
Chen CC, Cheng KJ, Ko TP, et al. Current progresses in phytase research: three-dimensional structure and protein engineering[J]. ChemBioEng Reviews, 2015, 2(2): 76-86. DOI:10.1002/cben.v2.2 |
| [5] |
Greiner R, Konietzny U. Update on characteristics of commercial phytases[C]. Rome: International Phytase Symposium, 2012. https://www.researchgate.net/publication/322301882_Update_on_Characteristics_of_Commercial_Phytases
|
| [6] |
Jain J, Singh B. Characteristics and biotechnological applications of bacterial phytases[J]. Process Biochemistry, 2016, 51(2): 159-169. DOI:10.1016/j.procbio.2015.12.004 |
| [7] |
Singh B, Kunze G, Satyanarayana T. Developments in biochemical aspects and biotechnological applications of microbial phytases[J]. Biotechnol Mol Biol Rev, 2011, 6(3): 69-87. |
| [8] |
Shivange A V, Schwaneberg U. Recent advances in directed phytase evolution and rational phytase engineering[M]//Directed Enzyme Evolution: Advances and Applications. Springer International Publishing, 2017: 145-172.
|
| [9] |
Ushasree M V, Shyam K, Vidya J, et al. Microbial phytase: impact of advances in genetic engineering in revolutionizing its properties and applications[J]. Bioresour Technol, 2017, 245(ptB): 1790-1799. |
| [10] |
Huang H, Luo H, Yang P, et al. A novel phytase with preferable characteristics from Yersinia intermedia[J]. Biochemical and Biophysical Research Communications, 2006, 350(4): 884-889. DOI:10.1016/j.bbrc.2006.09.118 |
| [11] |
Huang H, Luo H, Wang Y, et al. A novel phytase from Yersinia rohdei with high phytate hydrolysis activity under low pH and strong pepsin conditions[J]. Appl Microbiol Biotechnol, 2008, 80(3): 417-426. DOI:10.1007/s00253-008-1556-5 |
| [12] |
Shivange AV, Serwe A, Dennig A, et al. Directed evolution of a highly active Yersinia mollaretii phytase[J]. Appl Microbiol Biotechnol, 2012, 95(2): 405-418. DOI:10.1007/s00253-011-3756-7 |
| [13] |
Fu D, et al. A highly pH-stable phytase from Yersinia kristeensenii: cloning, expression, and characterization[J]. Enzyme Microb Technol, 2008, 42(6): 499-505. DOI:10.1016/j.enzmictec.2008.01.014 |
| [14] |
Fu D, Li Z, Huang H, et al. Catalytic efficiency of HAP phytases is determined by a key residue in close proximity to the active site[J]. Appl Microbiol Biotechnol, 2011, 90(4): 1295-1302. DOI:10.1007/s00253-011-3171-0 |
| [15] |
Ariza A, Moroz OV, Blagova EV, et al. Degradation of phytate by the 6-phytase from Hafnia alvei: a combined structural and solution study[J]. PLoS One, 2013, 8(5): e65062. DOI:10.1371/journal.pone.0065062 |
| [16] |
Green MR, Sambrook J. Molecular cloning: a laboratory manual[M]. New York: Cold Spring Harbor Laboratory Press, 2012.
|
| [17] |
Farhat A, Chouayekh H, Farhat MB, et al. Gene cloning and characterization of a thermostable phytase from Bacillus subtilis US417 and assessment of its potential as a feed additive in comparison with a commercial enzyme[J]. Molecular Biotechnology, 2008, 40(2): 127. DOI:10.1007/s12033-008-9068-1 |
| [18] |
Anagnostopoulos C, Spizizen J. Requirements for transformation in Bacillus subtilis[J]. J Bacteriol, 1961, 81(5): 741. |
| [19] |
Laemmli UK. Cleavage of structural proteins during the assembly of the head of bacteriophage T4[J]. Nature, 1970, 227: 680-685. DOI:10.1038/227680a0 |
| [20] |
Zhang W, et al. Adopting selected hydrogen bonding and ionic interactions from Aspergillus fumigatus phytase structure improves the thermostability of Aspergillus niger PhyA phytase[J]. Appl Environ Microbiol, 2007, 9: 3069-3076. |
| [21] |
Kim MS, Lei XG. Enhancing thermostability of Escherichia coli phytase AppA2 by error-prone PCR[J]. Appl Microbiol Biotechnol, 2008, 79(1): 69-75. DOI:10.1007/s00253-008-1412-7 |
| [22] |
Sanchez-Romero I, Ariza A, Wilson K S, et al. Mechanism of protein kinetic stabilization by engineered disulfide crosslinks[J]. PLoS One, 2013, 8(7): e70013. DOI:10.1371/journal.pone.0070013 |
| [23] |
Tian YS, Peng RH, Xu J, et al. Semi-rational site-directed mutagenesis of phyI1s from Aspergillus niger 113 at two residue to improve its phytase activity[J]. Molecular Biology Reports, 2011, 38(2): 977-982. DOI:10.1007/s11033-010-0192-1 |
| [24] |
Ushasree MV, Vidya J, Pandey A. Replacement P212H altered the pH-temperature profile of phytase from Aspergillus niger NII 08121[J]. App Biochem Biotechnol, 2015, 175(6): 3084-3092. DOI:10.1007/s12010-015-1485-6 |
| [25] |
Mehta D, Satyanarayana T. Biochemical and molecular characteriz-ation of recombinant acidic and thermostable raw-starch hydrolys-ing α-amylase from an extreme thermophile Geobacillus thermoleov-orans[J]. J Mol Catal B Enzym, 2013, 85: 229-238. |
| [26] |
Boraston AB, Bolam DN, Gilbert HJ, et al. Carbohydrate-binding modules: fine-tuning polysaccharide recognition[J]. Biochemical Journal, 2004, 382(3): 769-781. DOI:10.1042/BJ20040892 |
| [27] |
Božić N, Lončar N, Slavić MŠ, et al. Raw starch degrading α-amylases: an unsolved riddle[J]. Amylase, 2017, 1: 12-25. |
| [28] |
Møller MS, Svensson B. Structural biology of starch-degrading enzymes and their regulation[J]. Curr Opin Structural Biol, 2016, 40: 33-42. DOI:10.1016/j.sbi.2016.07.006 |
| [29] |
Cockburn D, Svensson B. Structure and functional roles of surface binding sites in amylolytic enzymes[M]. Understanding Enzymes: Function, Design, Engineering, and Analysis, Pan Stanford Publishing, 2016, 267-296.
|
| [30] |
Mehta D, Satyanarayana T. Domain C of thermostable α-amylase of Geobacillus thermoleovorans mediates raw starch adsorption[J]. Appl Microbiol Biotechnol, 2014, 98(10): 4503-4519. DOI:10.1007/s00253-013-5459-8 |
| [31] |
Peng H, Zheng Y, Chen M, et al. A starch-binding domain identified in α-amylase(AmyP)represents a new family of carbohydrate-binding modules that contribute to enzymatic hydrolysis of soluble starch[J]. FEBS Letters, 2014, 588(7): 1161-1167. DOI:10.1016/j.febslet.2014.02.050 |
| [32] |
Hua YW, Chi MC, Lo HF, et al. Fusion of Bacillus stearothermop-hilus leucine aminopeptidase Ⅱ with the raw-starch-binding domain of Bacillus sp. strain TS-23 α-amylase generates a chimeric enzyme with enhanced thermostability and catalytic activity[J]. J Ind Microbiol Biotechnol, 2004, 31(6): 273-277. DOI:10.1007/s10295-004-0146-5 |