沙门氏菌是一种常见的食源性致病菌[1]。在中国境内,食用被沙门氏菌污染的肉制品将会导致食源性疾病的爆发,导致伤寒、肠胃炎和急性败血性传染病[2]。在美国,沙门氏菌已经造成大约100万例食源性疾病,每年大概有378人死亡[3],世界卫生组织已经把沙门氏菌归类于人畜共患病病原菌。近年来,由于抗生素的不规范使用,导致沙门氏菌的耐药性日益严重。尤其是在畜牧养殖这一行业,越来越多的养殖者在经济利益的驱使下,肆无忌惮的将各种抗生素添加到饲料中,从而使得这一状况变得愈加顽劣[4]。根据近年来的相关研究发现,沙门氏菌对磺胺类、四环素类、喹诺酮类、氨基糖苷类和氯霉素类的耐药性呈大幅度的增加的趋势[5-7],这一现象不仅对动物,对人类的生命健康也造成了巨大的危害,值得社会的广泛关注[8]。
本实验从江苏省徐州市5个区3个县的各大市场采购猪肉500份,并从中分离出60株沙门氏菌样本进行药敏试验、血清型和生化试验鉴定,并根据喹诺酮、四环素类、磺胺类、氨基糖苷类和氯霉素类的耐药基因保守区设计12对引物进行PCR检测,期望得知耐药基因和耐药菌株的关系,旨在为今后耐药机制的研讨奠定实践基础。
1 材料与方法 1.1 材料 1.1.1 菌株来源2014.9-2016.6年间,自江苏省徐州市5个区3个县的各大市场以及超市采集猪肉样品500份用于该实验。沙门氏菌标准株C79-13购自中国兽医药品监察所。
1.1.2 试剂与试药引物由生工生物工程技术股份有限公司合成,PCR所需的试剂均购置于北京百泰克生物技术有限公司,药敏纸片购买于北京兰伯瑞生物技术有限公司。10种抗生素的药敏纸片分别为:复方新诺明(Sulfamethoxazole trimethoprim,SXT,25 μg)、四环素(tetracyclines,TE,30 μg)、氯霉素(Chloramphenicol,C,30 μg)、环丙沙星(Ciprofloxacin,CIP,5 μg)、诺氟沙星(Norfloxacin,NOR,5 μg)、萘啶酸(Nalidixic acid,NAL,30 μg)、阿米卡星(Amikacin,AK,30 μg)、链霉素(Streptomycin,S,10 μg)、庆大霉素(Gentamicin,GEN,10 μg)、壮观霉素(Spectinomycin,SPT,10 μg)。缓冲蛋白胨水、改良Msrv和XLT4琼脂培养基购置于北京兰伯瑞生物技术有限公司。
1.1.3 仪器恒温培养摇床THZ-100B购于昆山一恒仪器有限公司;DHP-9052恒温培养箱购买于上海一恒科学仪器有限公司;台式离心机TGL-16G购买上海安亭科技仪器厂;DYY-70型电泳仪购买于艾科公司;TC-25H PCR基因扩增仪购买于杭州博日科技有限公司,其他仪器均为实验室常规仪器。
1.2 方法 1.2.1 采样将采集到的猪肉样本放入灭菌袋,并加入运送培养基用来保存,保存时限不超过12 h,共采集猪肉样品500份。
1.2.2 细菌的分离与鉴定将采购的样本放入装有BPW培养液的无菌密封袋,37℃摇床过夜。次日吸取1 mL BPW前增菌液至msrv改良肉汤半固体培养基上,置于42℃培养24 h。挑取可疑白色菌落,三区划线于XLT4平板,37℃培养24 h[9]。对疑似沙门氏菌进行针对性的PCR鉴定,沙门氏菌侵染宿主细胞与肠毒素基因Stn有着密不可分的联系,因而可以使用Stn基因来检测与鉴定沙门菌[10]。针对Stn的核苷酸序列设引物[11]。上游序列:CCCTTTCCCGCTATCGGTAA,下游序列:CATGAACTGGCGCAGGTGAT目的片段为260 bp。PCR反应条件:94℃预变性5 min;94℃预变性45 s,55℃退火45 s,72℃延伸30 s,30个循环;72℃延伸10 min。对PCR产物进行琼脂糖凝胶电泳检测并进行DNA胶回收,将回收的产物送去生工生物工程技术股份有限公司测序。
1.2.3 生化试验生化试验按照郭万柱等[12]的介绍方法将分离株分别接种于尿素、赖氨酸、鸟氨酸脱羧酶、靛基质、葡萄糖、乳糖和甘露醇等生化发酵管,观察并记录特点。
1.2.4 血清型鉴定血清学鉴定采用玻片凝集法,同时用生理盐水对照。按照沙门菌属诊断血清的说明书确定抗原,查阅《沙门菌属血清型诊断》,根据测定的抗原结果确定分离株的血清型。
1.2.5 药敏实验用K-B法和CLSI推荐的抗微生物药物敏感实验执行标准进行药敏实验。用镊子将药敏纸片贴入涂有菌液的平板上,于37℃过夜培养,次日测量药敏纸片抑菌圈的直径,同种方法做沙门氏菌标准株C79-13的质量控制,观察结果[13-15]。
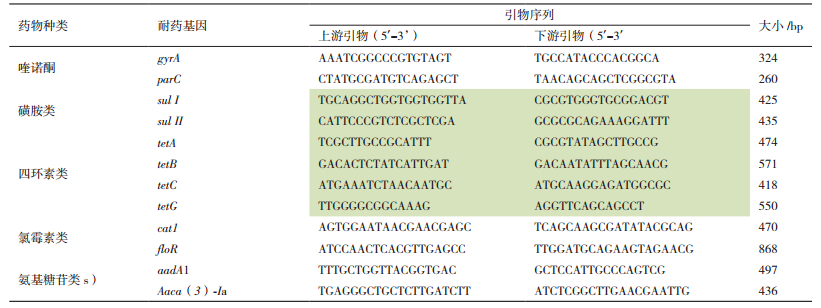
1.2.6 耐药基因引物的设计依照参考文献[16-19]和GenBank中公布的ORIGIN,12对特异性引物利用DNAMAN等软件被设计出来,并由生工生物工程技术股份有限公司合成,表 1为各引物的序列。
模板DNA由DNA提取试剂盒获得。PCR反应:模板DNA 2.5 μL、2×Taq Master Mix 12.5 μL、上游引物(20-25 μmol/L)1 μL、下游引物(20-25 μmol/L)1 μL、ddH2O 8 μL,总体积25 μL。
PCR扩增:94℃预变性4 min,94℃变性30 s,退火温度:aadA1 54℃、Aaca(3)-Ia 55℃、sulⅠ 51℃、sulⅡ 52℃、gyrA 55℃、parC 62℃、tetA 55℃、tetB 55℃、tetC 54℃、tetG 55℃、floR 55℃、cat1 55℃ 30 s,72℃延伸30 s,共30个循环,最后72℃延伸7 min。通过AGE对基因片段的大小进行检测,电泳条件:电压65-75 V,电流45-60 mA,时间25-30 min。
2 结果 2.1 沙门氏菌的初步鉴定结果将采集的500份猪肉样品通过msrv改良肉汤半固体培养基进行初筛,再经过XLT4培养基进行复筛,可见黑色中心,周围有透明晕环菌落,初步分离鉴定疑似沙门氏菌65株,再经过Stn基因的分离检测,电泳产物回收测序,确定沙门氏菌60株。猪源沙门氏菌的分离率达12%。
2.2 沙门菌的生化试验生化试验结果显示该沙门菌对乳糖不发酵,对葡萄糖和甘露醇均能发酵产酸产气,尿素、靛基质呈阴性,鸟氨酸脱酸酶、赖氨酸呈阳性,具体见表 2。
60株分离菌共鉴定出11种血清型。其中,德尔卑血清型在徐州市肉制品中的分离率最高,为28.3%,其他优势血清型为:肠炎沙门菌、里森沙门菌及鼠伤寒沙门菌,检出率分别为23.3%、10%和10%,具体结果见表 3。
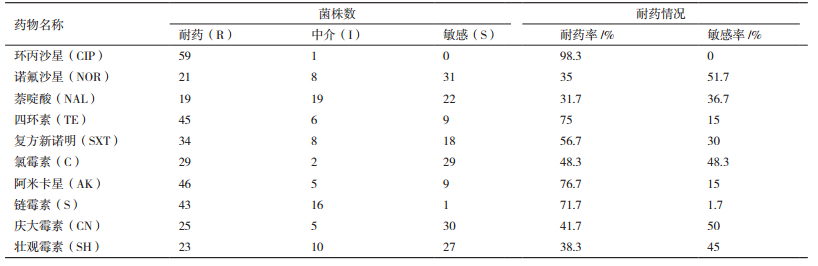
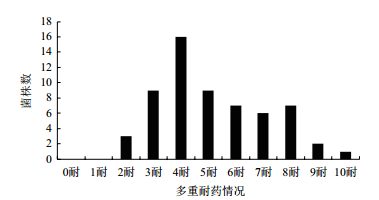
60株分离菌对临床常用的10种抗菌药物的耐药情况见表 4,可见分离菌对10种抗菌药物耐药性不同。其中环丙沙星耐药率比其他几种抗生素明显要高的多,所占总菌株的98.3%。其次,对四环素(75%)、阿米卡星(76.7%)、链霉素(71.7%)耐药率也较高,而对庆大霉素、氯霉素类药物比较敏感,敏感率在48%以上。本实验分离出的60株沙门氏菌多重耐药情况见图 1,60株分离菌不存在对所有药物都敏感的菌株,耐2种以上药物达100%。多重耐药中3耐、4耐、5耐、6耐、7耐和8耐的菌株较多,且有一株菌同时耐10种抗菌药物。
|
| 图 1 60株沙门氏菌对10种抗菌药物的多重耐药性 |
12种耐药基因,7种耐药基因被扩增出相应大小的目的片段。喹诺酮类耐药基因gyrA、parC占总菌株的66.7%、98.3%。四环素类耐药基因tetA占总菌株的53.3%。磺胺类耐药基因sul Ⅰ、sul Ⅱ,以sul Ⅰ检测率最高46.7%,而sul Ⅱ检测率为33.3%,氯霉素类耐药基因floR,占总菌株的48.3%,氨基糖苷类耐药基因aadA1检测率为63.3%,其中Aaca(3)-Ia、tetB、tetC、tetG及cat1未检测出。
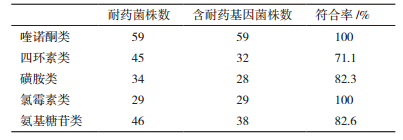
2.6 药敏实验与耐药基因检测结果比较60株分离菌对喹诺酮类、四环素类、磺胺类、氯霉素类和氨基糖苷类的药敏结果与耐药基因的检测结果的符合率为100%、71.1%、82.3%、100%和82.6%,通过表 5可知两者的一致性很高(≥87.2%)。而且同一菌株也存在同种类型的耐药基因,如有数株菌同时含有磺胺类耐药基因suiⅠ和suiⅡ,数株菌同时含有喹诺酮类耐药基因gyrA和parC。
沙门氏菌是公共卫生中最常见的人畜共患疾病之一,沙门氏菌引起的食源性疾病比其他食源性病原体更为显着。本研究中沙门氏菌的分离率为12%,且60株分离菌对喹诺酮类、四环素类及氨基糖苷类的耐药性很高,特别是喹诺酮与氨基糖苷类其耐药性高达98.3%、76.7%。现今,针对沙门氏菌引起的疾病治疗(动物)时,主要使用环丙沙星,导致目前分离出的猪源沙门氏菌对此抗生素的耐药性很高,可能会导致喹诺酮类药物的临床实用价值大幅度下降,这一发现与王晓泉等[20]的研究不符。因为近几年来养殖场为使幼小的牲口生长迅速、增强抗病毒的能力、提高经济效率,把喹诺酮类抗生素作为饲料添加剂并长期使用,由于大量使用这些抗生素,细菌耐药性也随之逐渐增加[21-23]。由表 4和图 1可知,本实验所分离的60株猪源沙门氏菌的耐药性居高不下,且大部分菌株同时耐3-8种药物。另有调查数据显示[24],沙门氏菌的多重耐药性从1990年的20%-30%增加到本世纪初的70%,并且耐药率将随时间的增加而增加,将很快成为沙门氏菌疾病的主要原因,因此研究沙门氏菌的耐药性及耐药基因的问题已成为全球关注的热点之一,值得人们更加深入的研究。
另外,从这60株分离菌中扩增得到喹诺酮类耐药基因parC、gyrA,磺胺类耐药基因sulⅠ、sulⅡ,氯霉素类耐药基因floR,四环素类耐药基因tetA,氨基糖苷类耐药基因aadA1,这一结果与El-Tayeb等[25]的研究报道一致。其中parC检测率最高为98.3%,gyrA、sulⅠ、sulⅡ、floR、tetA及aadA1检测率也很高,分别为66.7%、46.7%、33.3%、48.3%、53.3%及66.3%,与药敏实验的结果一致,在一定程度上表明,耐药基因可以决定细菌的耐药表型。其中tetB、tetC、tetG、cat1及Aaca(3)-Ia没有扩增出来,表明这些耐药基因在江苏省徐州市的猪源沙门氏菌中不存在或占极少数。这与马孟根等[16]的研究结果存在一定差异,本实验并未检测出tetC和cat1,这主要是因为地区流行菌株的血清型和耐药物的不同所致。将耐药基因测序结果与GenBank序列进行比对发现,两者同源性高达98.95%,与Ng等[26]报道一致。证明扩增到的PCR片段为相对应的耐药基因,且耐药基因的存在情况也符合药敏试验的结果(≥87.2%)这一现象可以表明,耐药基因可以决定细菌的耐药表型。耐药基因的检测为临床抗生素的合理选用作出一定的理论指导。
如今,具有多重耐药性的沙门氏菌成为主要的食源性疾病菌,这一现状大大地阻碍了医学的临床治疗[27-29]。为了明确沙门氏菌的耐药机制,本研究采集猪源沙门氏菌进行血清型鉴定、生化鉴定、药敏实验和耐药基因的检测,并对耐药基因进行克隆与序列分析,测序后发现耐药基因序列发生突变,与药物的亲和力下降,揭示了耐药基因可以决定细菌的耐药表型这一现象。将gyrA基因序列与GenBank已发表的序列进行对比,发现第408位A→C,即丝氨酸转变为丙氨酸。最近的研究发现gyrA基因的位点83与87是突变热点,若发生突变则表明沙门氏菌对喹诺酮类药物耐药强的原因之一。沙门氏菌对于喹诺酮类的耐药性与突变数目密切相关,单一突变可以导致较低水平的耐药,多个突变则导致高水平的耐药性,具体的耐药机制有待进一步研究。
4 结论本实验的药敏实验和耐药基因的检测结果表明,江苏省徐州市猪源沙门氏菌的耐药性已经相当严重,特别是对喹诺酮类的耐药性居高不下,耐药菌会通过食物的摄取传播给人,猪源沙门氏菌的耐药机制将直接影响人源沙门氏菌的耐药性。针对此现象,本研究明确了耐药性和耐药基因的关系,并对已成功分离的沙门氏菌中几种常见的耐药基因进行克隆及特性分析发现,耐药基因可以决定细菌的耐药表型。
| [1] |
Ahmed AM, Shimamoto T, Shimamoto T. Characterization of integrons and resistance genes in multidrug-resistant Salmonella enterica isolated from meat and dairy products in Egypt[J]. International Journal of Food Microbiology, 2014, 189: 39-44. DOI:10.1016/j.ijfoodmicro.2014.07.031 |
| [2] |
Zhao X, Ye C, Chang W, et al. Serotype distribution, antimicrobial resistance, and class 1 integrons profiles of Salmonella from animals in slaughterhouses in Shandong province, China[J]. Frontiers in Microbiology, 2017, 8: 1049. DOI:10.3389/fmicb.2017.01049 |
| [3] |
Bacci C, Boni E, Alpigiani I, et al. Phenotypic and genotypic features of antibiotic resistance in Salmonella enterica isolated from chicken meat and chicken and quail carcasses[J]. International Journal of Food Microbiology, 2012, 160(1): 16-23. DOI:10.1016/j.ijfoodmicro.2012.09.014 |
| [4] |
Ombui JN, Mitema ES, Kikuvi GM. Serotypes and antimicrobial resistance profiles of Salmonella isolates from pigs at slaughter in Kenya[J]. The Journal of Infection in Developing Countries, 2010, 4(4): 243-248. |
| [5] |
Voss-Rech D, Potter L, Vaz CSL, et al. Antimicrobial resistance in nontyphoidal Salmonella isolated from human and poultry-related samples in Brazil: 20-year meta-analysis[J]. Foodborne Pathogens and Disease, 2017, 14(2): 116-124. DOI:10.1089/fpd.2016.2228 |
| [6] |
Ren D, Chen P, Wang Y, et al. Phenotypes and antimicrobial resistance genes in Salmonella isolated from retail chicken and pork in Changchun, China[J]. Journal of Food Safety, 2017, 37(2): e12314. DOI:10.1111/jfs.2017.37.issue-2 |
| [7] |
Lebdah MA, Mohammed WM, Eid S, et al. Molecular Detection of some antimicrobial resistance genes in Salmonella species isolated from commercial layers in Egypt[J]. Zagazig Veterinary Journal, 2017, 45(1): 29-38. |
| [8] |
马婧嘉, 施春雷, 李可, 等. 沙门氏菌耐药谱及质粒耐药基因的筛查[J]. 中国食品学报, 2014(4): 184-190. |
| [9] |
张金宝, 马春芳, 余婷, 等. 宁夏地区奶牛乳房炎大肠杆菌毒力基因检测和耐药性分析[J]. 畜牧兽医学报, 2015, 47(1): 177-182. |
| [10] |
郭立明, 张雪, 厉华明. 沙门氏菌检测方法的研究进展[J]. 生物技术世界, 2014(9): 97. |
| [11] |
李爱华, 王胜启, 秦东春. 沙门菌属stn基因LAMP快速检测方法的建立及应用[J]. 中国卫生检验杂志, 2014(9): 1217-1220. |
| [12] |
郭万柱, 吴彤, 陈瑶先. 动物微生物学[M]. 成都: 四川科学技术出版杜, 1997, 234-235.
|
| [13] |
CLSL. M100-S19 performance standards for antimicrobial susceptibility testing[S]. Wayne: Clinical and Laboratory standards Institute, 2009. http://www.sciencedirect.com/science/article/pii/S0196439901880090
|
| [14] |
Zhu Y, Lai H, Zou L, et al. Antimicrobial resistance and resistance genes in Salmonella strains isolated from broiler chickens along the slaughtering process in China[J]. International Journal of Food Microbiology, 2017, 259: 43-51. DOI:10.1016/j.ijfoodmicro.2017.07.023 |
| [15] |
Liljebjelke KA, Hofacre CL, White DG, et al. Diversity of antimicrobial resistance phenotypes in Salmonella isolated from commercial poultry farms[J]. Frontiers in Veterinary Science, 2017, 4: 96. DOI:10.3389/fvets.2017.00096 |
| [16] |
马孟根, 王红宁, 余勇, 等. 猪源致病性沙门氏菌耐药基因的分析[J]. 畜牧兽医学报, 2006, 37(1): 65-70. |
| [17] |
Lindstedt BA, Heir E, Nygård I, et al. Characterization of class Ⅰ integrons in clinical strains of Salmonella enterica subsp. enterica serovars Typhimurium and Enteritidis from Norwegian hospitals[J]. Journal of Medical Microbiology, 2003, 52(2): 141-149. DOI:10.1099/jmm.0.04958-0 |
| [18] |
Chen S, Zhao S, White DG, et al. Characterization of multiple-antimicrobial-resistant Salmonella serovars isolated from retail meats[J]. Appl Environ Microbiol, 2004, 70(1): 1-7. DOI:10.1128/AEM.70.1.1-7.2004 |
| [19] |
Dawar R, Ganjoo A, Imdadi F, et al. Multidrug resistant invasive nontyphoidal Salmonella isolated from and masquerading healed tubercular constrictive pericarditis and study of virulence markers[J]. Cureus, 2017, 9(4): e1198. |
| [20] |
王晓泉, 焦新安, 刘晓文, 等. 江苏部分地区食源性和人源沙门氏菌的多重耐药性研究[J]. 微生物学报, 2007, 47(2): 221-227. |
| [21] |
黄马兰, 刘湘新. 饲料添加剂中数种抗菌药与喹诺酮类药物的作用[J]. 中兽医医药杂志, 1996, 4(35): 46. |
| [22] |
Donado-Godoy P, Clavijo V, Leoón M, et al. Counts, serovars, and antimicrobial resistance phenotypes of Salmonella on raw chicken meat at retail in Colombia[J]. J Food Prot, 2014, 77(2): 227-235. DOI:10.4315/0362-028X.JFP-13-276 |
| [23] |
Matayoshi M, Kitano T, Sasaki T, et al. Resistance phenotypes and genotypes among multiple-antimicrobial-resistant Salmonella enterica subspecies enterica serovar Choleraesuis strains isolated between 2008 and 2012 from slaughter pigs in Okinawa Prefecture, Japan[J]. Journal of Veterinary Medical Science, 2015, 77(6): 705-710. DOI:10.1292/jvms.14-0683 |
| [24] |
姜肖明. 我国细菌耐药性监测应注意的几个问题[J]. 中外健康文摘:医药月刊, 2007, 3(12): 126-127. |
| [25] |
El-Tayeb MA, Ibrahim A SS, Al-Salamah AA, et al. Prevalence, serotyping and antimicrobials resistance mechanism of Salmonella enterica isolated from clinical and environmental samples in Saudi Arabia[J]. Brazilian Journal of Microbiology, 2017, 48(3): 499-508. DOI:10.1016/j.bjm.2016.09.021 |
| [26] |
Ng LK, Mulvey MR, Martin I, et al. Genetic characterization of antimicrobial resistance in Canadian isolates of Salmonella serovar Typhimurium DT104[J]. Antimicrobial Agents and Chemotherapy, 1999, 43(12): 3018-3021. |
| [27] |
Peirano G, Agersø Y, Aarestrup F M, et al. Occurrence of integrons and antimicrobial resistance genes among Salmonella enterica from Brazil[J]. Journal of Antimicrobial Chemotherapy, 2006, 58(2): 305-309. DOI:10.1093/jac/dkl248 |
| [28] |
Crump JA, Sjölund-Karlsson M, Gordon MA, et al. Epidemiology, clinical presentation, laboratory diagnosis, antimicrobial resistance, and antimicrobial management of invasive Salmonella infections[J]. Clinical Microbiology Reviews, 2015, 28(4): 901-937. DOI:10.1128/CMR.00002-15 |
| [29] |
Ta YT, Nguyen TT, To PB, et al. Quantification, serovars, and antibiotic resistance of Salmonella isolated from retail raw chicken meat in Vietnam[J]. J Food Prot, 2014, 77(1): 57-66. DOI:10.4315/0362-028X.JFP-13-221 |