海带(Saccharina japonica)是具异型世代交替的重要经济海藻。在食品、医药、工业及能源等方面都有巨大的应用价值[1-3]。经过半个多世纪的发展,我国的海带养殖体系已经较为成熟,养殖规模日趋壮大。创立于20世纪50年代的夏苗培育法,曾经使我国的海带养殖业产生了一次巨大飞跃。但随着海藻生物技术的发展,传统的育苗方法存在以下几点不足:(1)培育时间长,成本较高,风险大。(2)难以实现稳定和高度一致的良种化养殖。种海带混合采孢子使得栽培苗种容易混杂,难于保持品系的纯度[4-6]。(3)种苗易发生病害并且能源消耗高[7]。近年来,利用海带配子体克隆,将育种和育苗相结合的新方法,利用海带杂种优势,克服了传统育种和育苗技术的缺点和不足,工艺简单、品种纯度高、育苗时间短、不受季节限制、成本低[8-11],既可延长优良品种的寿命,又可作为杂交海带育苗的保证。因此,配子体克隆成为海带种质资源保存和利用的主要手段,对海带育种、育苗、生理及遗传学研究越来越重要。
培育海带高产新品种,为养殖业提供优良苗种才能保障养殖业的可持续发展。近年来,作为海水养殖生物遗传改良和品种培育高新技术之一,分子标记技术发展迅速,目前主要用于海带群体遗传多样性和遗传结构分析、亲缘关系分析、种质鉴定等方面。如李言等[12-18]应用RAPD、ISSR、SSR、AFLP和ITS标记技术对海带及长海带进行了遗传多样性分析和系统进化分析。其中,微卫星标记(Simple sequence repeats,SSR)是一种常用的分子生物学标记,由于它的共显性特征,广泛应用于非模式生物的基因多样性及亲缘关系的分析[19-20]。已有报道表明,在海带中可以用多态性高的微卫星引物确定杂交品种的亲缘关系[21],以此了解种质材料的遗传信息。本研究采用不同物种微卫星标记(包括海带SSR、裙带菜SSR)以及海带与裙带菜的内转录间隔区(Internal transcribed spacer,ITS)通用引物,在获得可区分亲本♀与亲本♂配子体的数对微卫星引物的基础上,对不同试验组合子代样品和其亲本进行遗传构成的分析,为育种材料的选择提供准确的参考信息,通过对配子体克隆和孢子体幼苗的初代筛选,使得培育出纯度更高,性状更优良的海带与裙带菜品种。
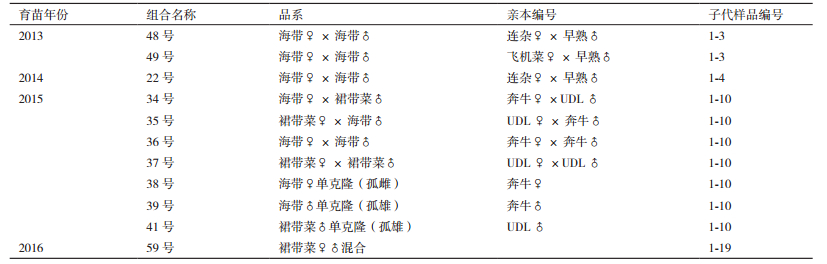
1 材料与方法 1.1 材料试验材料亲本均为山东东方海洋科技股份有限公司实验室保存的海带及裙带配子体克隆,子代样品均为山东东方海洋科技股份有限公司实验海区栽培的海带孢子体(表 1)。
采用天根DNA植物基因组提取试剂盒提取所有亲本和子代样品DNA,利用1%琼脂糖凝胶电泳和紫外分光光度计检测其质量和浓度,根据所测样品的质量浓度,将样品稀释至50 ng/μL,置于4℃暂存待用。
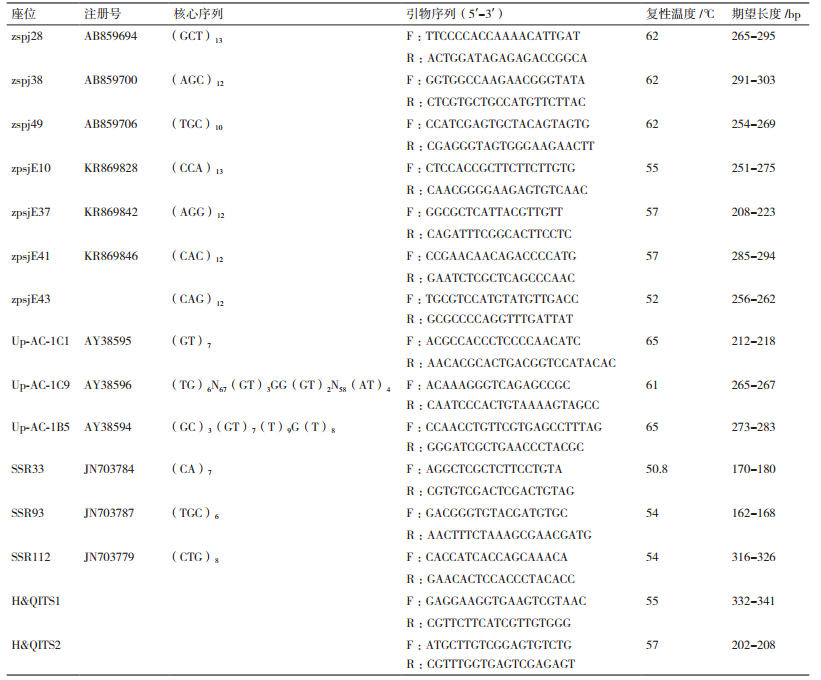
1.2.2 引物的筛选和优化首先选取数对海带SSR引物、裙带菜SSR引物、海带与裙带菜通用的SSR引物对不同组合的亲本进行初筛,从中得到能够区分不同组合父本与母本的SSR标记,利用这些有效的标记对不同组合子代样品进行检测,获得不同组合的遗传构成分析结果,共筛选出13对能区分亲本的微卫星标记,海带SSR引物为zspj28、zspj38、zspj49、zpsjE10、zpsjE37、zpsjE41和zpsjE43;裙带菜SSR引物为1C1、1C9和1B5;海带与裙带菜通用引物为SSR33、SSR93和SSR112。同时,使用海带与裙带菜通用的ITS引物进行物种间的鉴定。在某些引物扩增结果不理想的情况下,对其退火温度和Mg2+浓度进行摸索。
1.2.3 PCR的扩增共选用30对多态性高的微卫星引物,其中12对海带中的引物为国家海藻与海参工程技术研究中心开发所得,7对海带与裙带菜的通用引物来自徐静娟等[22]报道,9对裙带菜的引物参考Claire等[23]报道合成,还有2对海带与裙带菜通用的ITS引物是国家海藻与海参工程技术研究中心根据海带与裙带菜序列设计(筛选出的13对微卫星引物与2对海带与裙带菜通用的ITS引物相关信息见表 2)。PCR扩增体系为2×PCR supermix 10 μL、10 μmol/L上下游引物各1 μL、50 ng/μL DNA模板1 μL,ddH2O补至20 μL。PCR反应条件为95℃ 5 min;95℃ 30 s,58-63℃ 30 s,72℃ 60 s,循环30次;72℃ 5 min,4℃保温。利用1%琼脂糖凝胶电泳检测扩增效果,6%变性聚丙烯酰胺凝胶电泳分离,银染显色。
通过得到的琼脂糖凝胶与聚丙烯酰胺凝胶上的基因带型,比较所有组合的子代个体与其亲本的条带分布情况,若子代在所用微卫星标记中具有与亲本相同的等位基因位点,基本可以确定亲本和子代的亲缘关系。若子代与亲本位点不符,则认为无亲缘关系。
2 结果 2.1 2013年育苗的48号组合与49号组合 2.1.1 区分亲本引物的初筛共选取4对海带中的微卫星引物zspj19、zspj28、zspj38和zspj49对48号组合亲本连杂♀×早熟♂、以及49号组合飞机菜♀×早熟♂进行扩增,筛选得到zspj38和zspj49可以区分48号组合的父本与母本,zspj28、zspj38和zspj49可以区分49号组合的父本与母本。
2.1.2 48号组合与49号组合的子代与亲本的遗传构成经过以上3对微卫星标记鉴定(图 1)得到,48号组合3个子代个体全部与其亲本遗传构成一致;49号组合3个子代个体只有1个与亲本遗传构成一致,即48号组合中所有个体都是其亲本的后代,49号组合中只有1个个体是其亲本的后代。
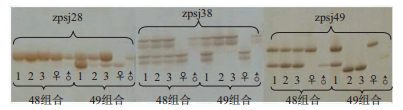
|
| 图 1 2013年育苗48组合、49组合的亲缘关系鉴定 |
共选取9对海带中的微卫星引物zspjE10、zspjE37、zspjE41、zspjE43、zspjE44、zspjE45、zspjE55、zpsj49和zpsj81对其亲本连杂♀×早熟♂进行扩增,筛选得到zspjE10、zspjE37、zspjE41、zspjE43和zpsj49可以区分其父本与母本。
2.2.2 22号组合的子代与亲本的遗传构成分析经过以上5对微卫星标记鉴定得到:22号组合4个子代个体全部与其亲本遗传构成一致,即22号组合中所有个体都是其亲本的后代(图 2)。

|
| 图 2 2014年育苗22号组合的亲缘关系鉴定结果 |
由于2015年的海带与裙带菜杂交试验中用到的裙带菜亲本只有UDL ♀×UDL ♂,因此选取这两个亲本作为模板,用引物1B2、1B5、1C1、1C9、1G2、1H4、1H5、2A2、2A8、2B2、2B4和2C1进行筛选。筛选得到1G2、2A2、2B2、1B5、1C1、1C9、1H4、2A8和2C1在2个亲本中都能得到有效条带,其中只有1C9引物可区分所用裙带的2个亲本。
2.3.1.2 海带与裙带菜通用SSR引物的筛选选取了此次杂交试验中的2个裙带菜亲本UDL ♀×UDL ♂、2个海带亲本奔牛♀×奔牛♂作为模板,用SSR23、SSR33、SSR93、SSR112、SSR134、SSR142、SSR147、SSR253和SSR259进行筛选。筛选得到(1)SSR23、SSR33、SSR93、SSR112、SSR142、SSR147、SSR253在海带中扩增结果较好,出现特异条带,其中只有SSR93能区分2个海带亲本。(2)SSR33、SSR93和SSR112在海带与裙带菜中条带较特异且差异较明显,可以用作海带与裙带菜的区分鉴定引物。
2.3.1.3 海带与裙带菜通用的ITS引物的鉴定选取此次杂交试验中的2个裙带亲本UDL ♀×UDL ♂、2个海带亲本奔牛♀×奔牛♂作为模板,运用海带与裙带菜的通用引物H&QITS1和H&QITS2进行检测,结果(表 3)得到2对ITS引物都能区分海带与裙带菜。
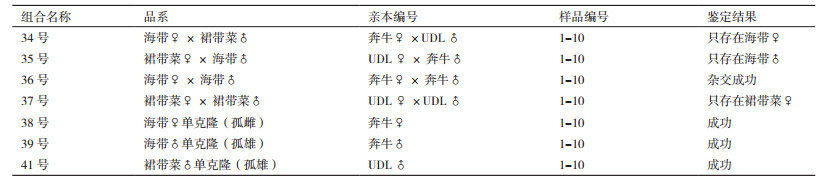
用筛选出的海带微卫星引物ssr93对杂交试验中的所有组合进行扩增,除37组合与41组合扩增不出条带,即检测不出海带成分外,其余均含有海带成分;且因为ssr93能够区分出海带♀×海带♂,因此,可以检测出34、35、36、38、39组合所有样品均与亲本所具有的等位基因吻合(图 3、图 4和表 4)。

|
| 图 3 SSR93引物在No.34、No.35、No.36组合中的鉴定 |

|
| 图 4 SSR93引物在No.38和No.39组合中的鉴定 |
用筛选出的裙带微卫星引物1c9对杂交试验中的所有组合进行扩增,只有37组合与41组合能扩增出条带,即可以检出裙带菜成分,说明其他组合均没有裙带菜杂交。其中1c9检测出37组合只有裙带菜♀的成分,41组合只有裙带菜♂的成分(图 5和表 4)。

|
| 图 5 1C9引物在No.37和No.41组合中的鉴定结果 |
由于H&QIts1与H&QIts2可以区别海带♀♂与裙带菜♀♂,所以试验中又采用这两对引物鉴定了34组合与35组合,结果(图 6、图 7和表 4)显示2个组合都只存在海带成分,无裙带菜成分。因此,34与35组合裙带菜杂交不成功。

|
| 图 6 H&QIts1检测No.34组合和No.35组合的子代与其海带亲本的遗传构成 |

|
| 图 7 H&QIts2检测34组合和35组合的子代与其海带亲本的遗传构成 |
由于59号组合的亲本为裙带菜雌雄混合克隆,因此选择3对常用的裙带菜引物进行检测,结果(图 8)显示,59号组合19个个体中,59-4、59-6、59-15、59-16、59-17、59-18、59-19与亲本(裙带菜雌雄混)带型不一致。
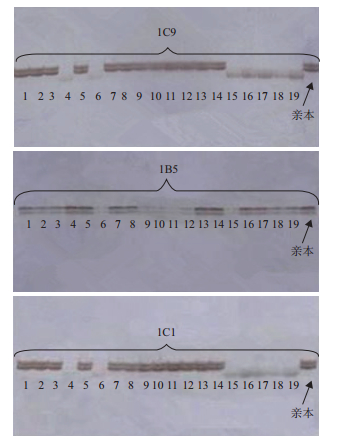
|
| 图 8 2016年育苗的No.59组合的遗传构成分析鉴定 |
将这些样品用褐藻的ITS引物扩增后进行测序,发现有polyG结构导致了其双峰的出现,测序正常的前420 bp与Undaria pinnatifida的“ITS1+5.8S+ITS2(部分)”的同源性达99%,可知这些个体均为裙带菜。但从SSR亲缘关系鉴定来看,本次取的20个个体有7个与当时育苗用的混克隆没有亲缘关系。
3 讨论海带分子标记辅助选择育种主要用于育种群体材料的遗传多样性和遗传结构分析、亲缘关系分析和种质鉴定上,这些可对育种亲本材料的选择提供参考信息[24]。其中,最常用的微卫星DNA标记是进行物种亲缘关系研究及遗传多样性分析的有效工具。之前的研究中,不同物种不同群体都是利用微卫星标记通过试验方法和后期数据统计以及指标(等位基因数、香农信息指数等)的分析来进行群体的遗传多样性检测、亲缘关系分析、杂种优势预测等生物学研究[25-27]。而本研究中所有的亲本均为配子体材料,配子体是单倍性的,无需通过后期各项指标的数据分析来得到组合中子代与亲本的关系,而是通过聚丙烯酰胺凝胶电泳得到的子代与亲本的基因型带型,直接确定其家系遗传构成组成,是海带与裙带菜育种试验中最简单快速有效的鉴定方法。
试验中的每个组合都要先进行区分亲本引物的筛选,因为不同的SSR标记在不同亲本中扩增位点不同,只有先筛选出能够区分亲本的SSR引物,才能准确地得到子代的分析结果。在亲本中扩增没有差别的引物,也无法区分出子代的差异。其次,对于2015年海带与裙带菜杂交试验的每个组合,都利用海带与裙带菜的引物进行了双重验证,因为必须保证该组合的子代中有并且只有该亲本的成分,尤其是孤雌、孤雄的组合,一旦混有其他的亲本,杂交必然失败,只有这样才能保证遗传构成分析结果准确有效。另外,59号组合分析的结果显示有几个子代与亲本不吻合,因此在此试验的基础上又用褐藻的引物进行了测序分析,但是这些非子代个体的真实信息有待深入研究。
将分子育种与常规育种的紧密结合,通过整合资源、优势互补,是未来藻类遗传育种的发展方向。分析家系遗传构成的前提是需要有足够多的多态性丰富的微卫星标记位点。国家海藻与海参工程技术研究中心已经开发出大量可用于鉴定海带的微卫星标记[28-30],而对于裙带菜等其他褐藻,可用标记并不多,因此在未来的研究中,开发特定品种的分子标记也很必要。
4 结论利用30对微卫星引物对11个海带与裙带菜的克隆育苗组合的子代与亲本进行了遗传结构分析,有11对引物能够区别出各组合的亲本♀与亲本♂,经过这11对引物的分析后,部分个体是其亲本的后代,部分个体遗传构成与亲本不一致。
| [1] |
黄晓林, 郑优, 单琰婷, 等. 海带化学成分和药理活性研究进展[J]. 浙江农业科学, 2015, 56(2): 246-250. |
| [2] |
Adams JM, Ross AB, et al. Seasonal variation in the chemical comp-osition of the bioenergy feedstock Laminaria digitata for thermoche-mical conversion[J]. Bioresour Technol, 2011, 102(1): 226-234. DOI:10.1016/j.biortech.2010.06.152 |
| [3] |
Adams JMM, Toop TA, Donnison IS, et al. Seasonal variation in Laminaria digitata and its impact on biochemical conversion routes to biofuels[J]. Bioresour Technol, 2011, 102(21): 9976-9984. DOI:10.1016/j.biortech.2011.08.032 |
| [4] |
李大鹏, 芦永红, 吴超元. 海带遗传育种及育苗生物技术历史及现状[J]. 生物学通报, 2002, 37(8): 1-3. |
| [5] |
张全胜, 丛义周, 曲善村, 等. 海带良种化生产的研究现状与展望[J]. 水产科学, 2006, 25(8): 424-427. |
| [6] |
李静, 逄少军, 单体锋, 等. 基于海带孤雌生殖研究海带夏苗制冷水工艺中的品种混杂问题[J]. 中国农业科技导报, 2016, 18(4): 163-167. |
| [7] |
冯蕾, 唐学玺, 张培玉. 海带育种育苗技术研究进展[J]. 科学技术与工程, 2005, 5(8): 30-39. |
| [8] |
Li DP, Zhou ZG, Liu HH, et al. A new method of Laminaria japonica strain selection and spore ling raising by the use of gametophyte clones[J]. Hydrobiologia, 1999, 398/399: 473-476. DOI:10.1023/A:1017090130586 |
| [9] |
李志凌, 张全胜, 杨迎霞, 等. 海带配子体克隆大规模培养技术的研究[J]. 齐鲁渔业, 2003(5): 1-3. |
| [10] |
李大鹏, 吴超元, 刘晚昌, 等. 海带单倍体无性繁殖系育苗技术的研究[J]. 海洋学报, 2003, 25(5): 141-145. |
| [11] |
张培玉, 张全胜, 等. 海带配子体克隆育苗生产中采苗技术的研究[J]. 应用于环境生物学报, 2005, 11(5): 545-548. |
| [12] |
李言, 刘延岭, 崔翠菊, 等. 海带种质资源遗传多样性的RAPD分析[J]. 生物技术通报, 2016, 32(4): 151-158. |
| [13] |
Li BJ, Shi YY, Yang GP, et al. Microsatellite DNA variation of the gametophyte clones isolated from introduced Laminaria japonica(Phaeophyta)and L. longissima of China and varieties derived from them[J]. J Integr Plant Biol, 2008, 50(3): 352-359. DOI:10.1111/jipb.2008.50.issue-3 |
| [14] |
石媛媛, 杨官品, 廖梅杰, 等. 海带和长海带配子体无性繁殖系微卫星[J]. DNA多态性比较分析.中国海洋大学学报, 2008, 38(1): 303-308. |
| [15] |
张全胜, 石媛媛, 丛义周, 等. 我国引种海带和栽培品种(系)来源配子体克隆的AFLP分析[J]. 中国海洋大学学报, 2008, 38(3): 429-435. |
| [16] |
Shan TF, Pang SJ, Zhang YR, et al. An AFLP-basedsurvey of genetic diversity and relationships of major farmed cultivars and geographically isolated wild populations of Saccharina japonica(Phaeophyta)along the northwest coasts of the pacific[J]. Journal of Applied Phycology, 2011, 23(1): 35-45. DOI:10.1007/s10811-010-9530-x |
| [17] |
Bi Y H, Hu Y J, Zhou Z G. Genetic variation of Laminaria japonica(Phaeophyta)populations in China as revealed by RAPD markers[J]. Acta Oceanologica Sinica, 2011, 30: 103-112. DOI:10.1007/s13131-011-0110-y |
| [18] |
崔翠菊, 李言, 张立楠, 等. 基于ITS序列海带目种质资源的物种鉴定研究[J]. 中国农学通报, 2014, 30(26): 27-32. DOI:10.11924/j.issn.1000-6850.2013-3055 |
| [19] |
Ayres-Ostrock LM, Mauger S, Plastino EM, et al. Development and characterization of microsatellite markers in two agarophyte species, Gracilaria birdiae and Gracilaria caudata(Gracilariaceae, Rhodophyta), using next-generation sequencing[J]. Journal of Applied Phycology, 2015. DOI:10.1007/S10811-015-0592-7 |
| [20] |
Jones AG, Small CM, et al. A practical guide to methods of parentage analysis[J]. Mol Ecol Resour, 2010, 10(1): 6-30. DOI:10.1111/men.2009.10.issue-1 |
| [21] |
石媛嫄. 海带连锁图谱构建及东方1号杂交海带亲缘关系分析[D]. 青岛: 中国海洋大学, 2008. http://cdmd.cnki.com.cn/Article/CDMD-10423-2008175061.htm
|
| [22] |
徐静娟, 赵翠, 等. 海带微卫星引物对裙带菜群体分析适用性的初步研究[J]. 中国海洋大学学报, 2012, 42(10): 41-46. |
| [23] |
Claire D, Marie V, Carolyn E, et al. Microsatellites isolation and polymorphism in introduced populations of the cultivated seaweed Undaria pinnatifida(Phaeophyceae, Laminariales)[J]. Conservation Genetics, 2005(6): 647-650. |
| [24] |
刘福利, 王飞久, 孙修涛, 等. 分子育种及其在海带育种中的研究进展[J]. 海洋科学, 2012, 36(9): 128-134. |
| [25] |
田萍萍, 李晓捷, 等. 烟威沿海大叶藻居群遗传多样性研究及对海草场修复的启示[J]. 水产科学, 2014, 33(2): 108-114. |
| [26] |
辜青青, 曾涛, 等. 基于SSR标记技术的南丰柑橘种质资源亲缘关系研究[J]. 果树学报, 2017, 34(6): 653-659. |
| [27] |
姚海芹, 刘福利, 等. 食用海带(Saccharina japonica)新品系遗传多样性研究[J]. 渔业科学进展, 2017, 38(3): 155-162. DOI:10.11758/yykxjz.20160302002 |
| [28] |
Zhang LN, Peng J, Li XJ, et al. Development of 27 trinucleotide microsatellite markers for Saccharina japonica using next generation sequencing technology[J]. Conservation Genetic Resources, 2014(6): 341-344. |
| [29] |
Peng J, Zhang LN, Li XJ, et al. Development of genic SSR markers from an assembled Saccharina japonica genome[J]. Journal of Applied Phycology, 2016(28): 2479-2484. |
| [30] |
Zhang LN, Peng J, Li XJ, et al. Characterization of genome-wide microsatellites of Saccharina japonica based on a preliminary assembly of illumina sequencing reads[J]. Journal of Ocean University China, 2016, 15(3): 523-532. DOI:10.1007/s11802-016-2945-5 |