植物病原细菌不能直接从寄主表面进入植物细胞,一般从自然孔口和伤口侵入寄主植物,在细胞间隙或维管束中繁殖[1]。不同细菌入侵的途径不同,在引起病害症状过程中,具有各自的侵染特点[2-5]。借助分子标记方法,可以示踪病原细菌在寄主中的入侵、定殖和繁殖。标记病原微生物的方法主要有抗生素抗性基因、lacZ基因、lux 基因、inaZ 基因、xylE基因、gus基因以及来源于海洋生物水母的绿色荧光蛋白基因(Green fluorescent protein,gfp)[6]。运用GFP标记无需反应底物,具有易检测、灵敏度高、稳定性好等优点,成为研究微生物与寄主植物相互作用的理想工具[7-10]。国内外多家实验室报道,携带gfp基因的重组载体成功标记与寄主植物互作的微生物,如pGFP4412、pGPE-GFP、pHC60等[11-13]。
青枯劳尔氏菌(Ralstonia solanacearum)引起的茄科作物青枯病是农业生产上的一个重要细菌病害,从根茎部伤口或次生根根冠进入维管束,最终引起萎焉[3]。柑橘黄单胞柑橘亚种(Xanthomonas citri subsp. citri)通过气孔和机械伤口进入植物组织,在叶片、果实和茎上都可以造成为害[4]。丁香假单胞菌番茄致病变种(Pesudomonas syringae pv. tomato)也是通过植株自然气孔或伤口侵入,产生圆形或近圆形暗褐色斑[5]。这3种植物病原细菌分别是劳尔氏菌属、黄单胞菌属和假单胞菌属的代表性植物病原细菌,是研究植物-病原微生物互作的模式材料。每种细菌的寄主范围和症状类型不同,柑橘黄单胞柑橘亚种仅在柑橘上引起溃疡症状,青枯劳尔氏菌和丁香假单胞菌番茄致病变种可在多种植物上引起病害,分别引起萎焉和斑点症状。基因组测序和分子生物学技术的发展,促进了3种植物病原细菌侵染机制的研究,但仍然需要一个稳定的标记手段来研究它们的入侵过程,揭示其在寄主植物体内的扩散、趋化性及生物膜等现象。
本研究拟以广宿主载体pBBR1MCS-5为骨架,克隆青枯劳尔氏菌RipAK基因的启动子和gfp基因,构建表达GFP蛋白的重组质粒。所得到的质粒载体pBB-GFP,可以对青枯劳尔氏菌R. solanacearum、番茄细菌性斑点病菌P. syringae pv. tomato和柑橘溃疡病菌X. citri subsp. citri进行有效标记,旨在为进一步研究植物病原细菌的入侵、定殖和繁殖提供了很好的标记载体。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒本研究中所用菌株和质粒见表 1。试验中所使用的大肠杆菌DH5α在LB培养基中37℃培养;青枯菌GMI1000、柑橘溃疡病菌29-1及丁香假单胞菌番茄致病变种DC3000在NB培养基中28℃培养[14]。基本培养基M63用于检测青枯菌RipAK基因启动子活性,配制固体培养基时,加入1.5%琼脂粉[15]。实验中需要加入的抗生素和使用浓度分别如下:氨苄青霉素Amp(100 μg/mL),庆大霉素Gm(5 μg/mL),壮观霉素Spe(50 μg/mL),利福平Rif(25 μg/mL),多粘菌素PB(50 μg/mL),四环素Tc(15 μg/mL)。
实验中所用番茄植物为感病品种红洋梨,叶片为5叶龄植物的展开叶片;柑橘植株用的是葡萄柚品种,由福建省福州市农科所提供。
1.2 方法 1.2.1 RipAK基因启动子活性检测根据茄科作物青枯菌R. solanacearum基因组信息,设计特异性引物P1F(5'-CCGGGTACCCTTCGATGCGGGTGATGT-3')和P1R(5'-CCGCTCGAGGCTGGCACCGTCGA-TC-3')。引物5'端分别引入KpnI和XhoI酶切位点。以青枯菌GMI1000的gDNA为模板扩增545 bp RipAK基因启动子,与pLacZ-Basic载体连接后,用Kpn I和Sal I将启动子和lacZ基因一起切下,连接进入pHM1载体,得到pHM1:PRipAKLacZ。从pLacZ-Basic载体上用同样的酶将lacZ基因切下,与pHM1载体连接,得到pHM1:LacZ。采用电转化方法将两个重组质粒分别导入青枯菌GMI1000,转化子用NB液体培养基震荡培养至OD600=1.5左右时,将菌液浓度调至OD600=1.0。在加有20 μg/mL X-gal的NA和M63固体平板上分别滴加2 μL上述菌液,28℃黑暗条件下培养2 d后观察菌落颜色。采用Miller[21]方法定量测定培养菌体的LacZ活性。
1.2.2 pBB-GFP质粒的构建用引物P2F(5'-CCGCTCGAGCTTCGATGCGGGTGATGT-3')和P2R(5'-CCGGAATTCGCTGGCACCGTCGATC-3')扩增青枯菌RipAK基因启动子,经Xho I和EcoR I双酶切后与广宿主质粒pBBR1MCS-5连接;随后,将gfp基因片段连接到EcoR I位点,得到pBB-GFP。gfp基因片段从pHC60质粒上用EcoR I酶切下来,大小为1 047 bp。pBB-GFP载体质粒送福州尚亚生物技术有限公司测序确定正确后保存备用。
1.2.3 pBB-GFP标记植物病原细菌制备R. solana-cearum GMI1000、X. citri subsp. citri 29-1和P. syrin-gae pv. tomato DC3000的电转化感受态细胞,将质粒pBB-GFP用电转化仪(Micropulser,Biorad,USA)电击转化(2 KV,3 ms)进入相应的感受态细胞中,在含有Gm的抗性NA平板上筛选标记菌株,使用gfp基因特异性引物gfpF(5'-ATGGCTAGCAAAGGAGAAGA-3')和gfpR(5'-TTAGCAGCCGGA-TCCTTTGTA-3')对长出的单菌落进行PCR验证,确定质粒成功转化到相应的病原细菌。
1.2.4 荧光显微镜镜检GFP标记的3种病原微生物在NB液体培养基中培养至OD600=1.5左右时,取20 μL菌液置于载玻片上,轻轻加上盖玻片。使用荧光显微镜(Olympus BX53,Tokyo,Japan)油镜,在1 000倍下采用激发波长为488 nm的蓝光观察,标记成功的融合菌株可以看到明亮的绿色荧光。
1.2.5 标记菌株在植物叶片上的定殖用针尖在番茄和柑橘叶片主脉两侧形成创伤。3种标记菌株在NB液体培养基中培养至OD600=1.5左右时,用无菌NB培养液将菌体浓度调至OD600=1.0,取20 μL菌液轻轻滴加在伤口处。R. solanacearum GMI1000和P. syringae pv. tomato DC3000滴加番茄叶片伤口,X. citri subsp. citri 29-1滴加柑橘叶片伤口。24 h后,用打孔器取下滴加菌液的叶片伤口处,置于载玻片上,滴加少许无菌水,加盖玻片,在荧光显微镜200倍下采用蓝光观察,定殖成功的标记菌株可在叶片伤口位置看到明显的绿色荧光聚集。
1.2.6 致病力测定采用注射接种方法测定菌株的致病力。将标记菌株和各自的野生型菌株在NB液体培养基中培养至OD600=1.5左右时,用无菌水将菌液浓度调至OD600=0.3。挑选刚伸展开的番茄和柑橘叶片,用针头在叶片背面轻轻针刺,造成伤口。用无针头注射器将菌悬液轻轻打入伤口,野生型菌株接种在叶片主脉左侧,GFP标记菌株接种于右侧。注射接种结束后2 d,记录R. solanacearum和P. syringae pv. tomato在番茄叶片上的致病力情况;5 d后记录X. citri subsp. citri在柑橘上的隆起病斑。
1.2.7 序列分析试验中的引物合成是根据青枯菌GMI1000基因组信息(GenBank Accession No. AL646053.1),用Primer Premier 5设计特异性引物。RipAK基因及其启动子序列分析在Neural Network Promoter Prediction(http://www.fruitfly.org/seq_tools/promoter.html)网站完成。
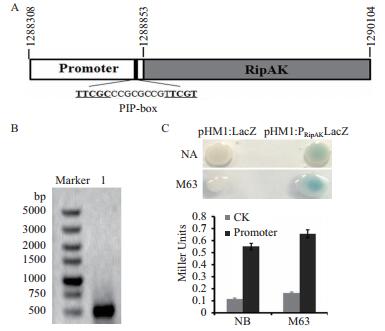
2 结果 2.1 RipAK启动子序列及活性检测青枯菌RipAK基因编码一个效应蛋白,在模式菌株GMI1000中位于质粒DNA的1288853-1290104(图 1-A)。将起始密码子前1 000 bp序列进行启动子序列预测,符合原核生物启动子序列特征的区域位于起始密码子上游366 bp处。在起始密码子上游94 bp位置,含有一个受HrpB调控的典型PIP-box序列(-TTCGCCCGCGCCGTTCGT-)(图 1-A)。为验证RipAK基因启动子的活性,用特异性引物扩增起始密码子上游545 bp的核苷酸序列(图 1-B),构建pHM1:PRipAKLacZ载体。转化GMI1000后,在涂有X-gal的NA和M63固体培养基上,可以观察到菌落变蓝,表明所克隆的RipAK基因启动子能够推动lacZ基因的表达(图 1-C)。在NB和M63液体培养基中培养青枯菌,对lacZ基因的活性进行定量测定,携带启动子的菌株用NB培养,活性为0.551;用M63培养基的菌株活性为0.656,活性增加19%(图 1-C)。
|
| 图 1 RipAK启动子及活性检测 A:RipAK基因及其启动子的序列;B:PCR扩增的545 bp启动子序列;C:RipAK启动子推动的LacZ基因活性检测。上图是在涂有X-gal的NA和M63平板上观察LacZ活性,下图是用液体培养后用Miller方法测定LacZ酶活力单位 |
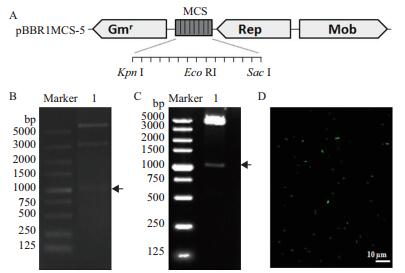
pBBR1MCS-5载体是一个4.7 kb大小的广宿主载体,在多克隆位点有13个常用酶切位点(图 2-A)。为获得RipAK基因启动子与gfp基因融合构建,用引物P2F和P2R扩增RipAK基因启动子区域,克隆到pBBR1MCS-5载体的Xho I和EcoR I位点(图 2-A)。gfp基因编码序列来源于pHC60载体,将该载体用EcoR I酶切后,含gfp基因的片段为1 047 bp(图 2-B),回收这个片段连接到pBBR1MCS-5载体的EcoR I位点后,经酶切和测序确定正确的连接方向,最终得到pBB-GFP重组载体(图 2-C)。携带pBB-GFP的大肠杆菌在荧光显微镜下可以看到荧光(图 2-D)。
|
| 图 2 pBB-GFP载体的构建 A:RipAK启动子克隆到pBBR1MCS-5载体的XhoI-EcoRI位点;B:pHC60载体质粒的EcoRI酶切。箭头所示为gfp基因的酶切片段;C:pBB-GFP载体质粒EcoRI酶切验证。箭头所示位置为gfp基因的酶切片段;D:携带pBB-GFP载体的大肠杆菌表达了GFP蛋白 |
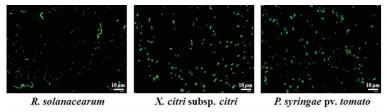
按照方法1.2.3将pBB-GFP质粒电转进入野生型R. solanacearum GMI1000、X. citri subsp. citri 29-1和P. syringae pv. tomato DC3000中。得到的转化子用gfpF和gfpR引物进行菌落PCR验证正确后(数据未显示),在NB液体培养基中培养2 d。将带有gfp标记的3种植物病原细菌菌液置于载玻片上,在1 000倍下用激发波长为488 nm的蓝光可观察到亮绿色的荧光(图 3),说明本研究中所构建的融合载体在3种病原细菌中表达了GFP蛋白,可以对病原菌进行标记。在荧光显微镜下,可清晰看到3种菌都是呈短杆状,大小约为1-2 μm。青枯菌GMI1000的液体培养菌经常可看到多个菌体串生,形成一个类似线状的结构。
|
| 图 3 荧光标记的3种植物病原细菌 |
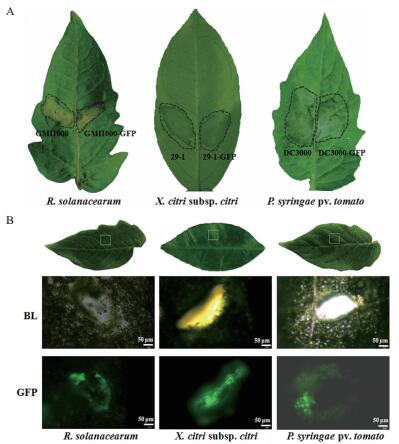
GFP标记菌株GMI1000-GFP、DC3000-GFP和29-1-GFP在寄主植物上的致病力没有变化(图 4-A)。注射接种2 d后,GMI1000-GFP和DC3000-GFP在番茄叶片上都可以形成病斑,与野生型菌株形成的病斑没有差异。接种后3 d,29-1-GFP与野生型29-1菌株一样,在柑橘叶片的注射区域开始出现水渍状症状,到第5天症状明显隆起(图 4-A)。为观察荧光标记菌株与寄主植物互作以后的情况,在番茄和柑橘叶片主脉两侧用针头形成创伤,滴加培养菌悬液1 d以后在荧光显微镜下观察。图 4-B结果显示,在伤口位置可以看到标记菌体的大量聚集,荧光亮度最强;伤口周围也有菌体分布,但亮度弱很多,基本呈随机弥散状态,这一现象符合这3种菌从伤口入侵,具有趋化性的特性。
|
| 图 4 荧光标记的3种植物病原细菌在寄主植物叶片上的致病力和定殖 A:标记菌株的致病力检测;B:标记菌株在叶片上的定殖 |
pBB-GFP载体可以标记多种植物病原细菌。利用GFP标记研究微生物与植物互作是示踪微生物的一种常用方法[22-23],但由于载体的兼容性问题,一种GFP表达载体能够标记的菌株种类有限。实验室曾尝试将pHC60质粒直接电转到青枯菌,但一直没有成功。本研究根据生物信息学分析和lacZ报道基因,确定了茄科作物青枯菌RipAK基因启动子。利用pBBR1MCS-5作为骨架,克隆了RipAK启动子和gfp基因,得到的质粒pBB-GFP载体在劳尔氏菌、黄单胞菌和假单胞菌中都可以表达绿色荧光蛋白,成功的标记了3个属的植物病原细菌。
在茄科作物青枯菌中,RipAK基因的表达受hrp调节基因hrpB的调控表达[24],在其启动子序列中也确实含有受hrpB调节的特性序列PIP-box。我们原初的设想是构建pBB-GFP对青枯菌进行标记,观察在维管束中的移动扩散、生物膜的形成。在实验过程中,发现携带pBB-GFP载体的大肠杆菌在荧光显微镜下可以看到荧光。因此,用构建的载体对丁香假单胞菌番茄致病变种和柑橘溃疡病菌进行标记。在丰富培养基中震荡培养和寄主植物接触过程中,荧光标记的信号都非常稳定,可能是载体上多克隆位点前的lac启动子和RipAK启动子都可以推动gfp基因的表达。构建的pBB-GFP载体还可以尝试应用于其它病原微生物的标记试验。
对病原微生物进行荧光标记后,可以对多种生物学现象进行观察。细胞的形态和大小可以在荧光显微镜下清楚的观察到,不必利用电镜技术,省去很多繁杂的材料准备。本研究将3种病原微生物标记后,观察到它们都是1-2 μm大小的短杆状细菌,在丰富培养基中还观察到青枯菌可以形成多个细胞串联的线状结构,在丁香假单胞菌番茄致病变种和柑橘溃疡病菌没有观察到。这样的特殊生长方式可能更有利于青枯菌从寄主根部侵染,特别是在没有伤口的情况下,有利于从次生根根冠入侵。生物膜和趋化性是植物病原细菌常用的侵染机制,将番茄和柑橘叶片创伤以后,荧光标记菌株在伤口部位大量聚集,表明这3种病原细菌都具有趋化性或形成生物膜的能力,这有助于病原菌在侵染过程中主动发现最适宜的入侵途径。
4 结论本研究构建了茄科作物青枯菌RipAK基因启动子推动的gfp基因重组载体pBB-GFP,成功标记了青枯劳尔氏菌、丁香假单胞菌和柑橘溃疡菌3种植物病原细菌。液体培养基中的标记菌株,在荧光显微镜下可以观察到细菌的形态,在番茄和柑橘叶片的创伤部位,可以看到标记菌体的大量聚集,可用于研究病原细菌侵染寄主植物的过程。
| [1] |
陈功友, 王金生. 植物病原细菌致病性决定因子[J]. 植物病理学报, 2002, 32(1): 1-7. |
| [2] |
方中达, 刘经芬, 等. 水稻白叶枯病(Xanthomonas oryzae)侵染循环的初步研究[J]. 植物病理学报, 1956, 2(2): 173-185. |
| [3] |
Genin S, et al. Pathogenomics of the Ralstonia solanacearum species complex[J]. Annu Rev Phytopathol, 2012, 50(1): 67-89. DOI:10.1146/annurev-phyto-081211-173000 |
| [4] |
Brunings AM, Gabriel DW. Xanthomonas citri: breaking the surface[J]. Molecular Plant Pathology, 2003, 4(3): 141-157. DOI:10.1046/j.1364-3703.2003.00163.x |
| [5] |
赵廷昌, 于莉, 孙福在, 等. 番茄细菌性斑点病及其防治[J]. 植物保护, 1999, 25(4): 56. |
| [6] |
王玲巧, 王继峰, 牛建昭. 报道基因技术及其应用[J]. 生命的化学, 2003, 23(3): 236-238. |
| [7] |
杜艳, 刘永锋, 常有宏, 等. 梨炭疽病菌原生质体遗传转化体系的建立及GFP标记菌株的获得[J]. 江苏农业学报, 2017, 33(2): 295-300. |
| [8] |
田涛, 王琦. 绿色荧光蛋白作为分子标记物在微生物学中的应用[J]. 微生物学杂志, 2005, 25(1): 68-73. |
| [9] |
王颖, 杨成德, 薛莉, 等. 生防菌株ZA1的GFP基因标记及其功能稳定性测定[J]. 植物保护学报, 2017, 44(4): 657-663. |
| [10] |
刘勇勤, 李赤, 徐秀德, 等. 利用绿色荧光蛋白(GFP)标记香蕉枯萎病菌及其稳定性检测[J]. 吉林农业大学学报, 2013, 35(1): 15-18. |
| [11] |
田涛, 亓雪晨, 王琦. 芽孢杆菌绿色荧光蛋白标记及其在小麦体表定殖的初探[J]. 植物病理学报, 2004, 34(4): 346-351. |
| [12] |
杨震元, 赵军, 郝蕾蕾, 等. 用gfp基因标记法研究金龟子绿僵菌在玉米根际的定殖动态[J]. 中国生物防治, 2009, 25(3): 215-219. |
| [13] |
Cheng HP, et al. Succinoglycan is required for the initiation and elongation of infection threads during nodulation of alfalfa by Rhizob-ium meliloti[J]. J Bacteriol, 1998, 180(19): 5183-5191. |
| [14] |
Sun DL, Zhuo T, Hu X, et al. Identification of a Pseudomonas putida as biocontrol agent for tomato bacterial wilt disease[J]. Biological Control, 2017, 114: 45-50. DOI:10.1016/j.biocontrol.2017.07.015 |
| [15] |
Guevara C, Zambrano MM. Sugarcane cellulose utilization by a defined microbial consortium[J]. FEMS Microbiology Letters, 2006, 255(1): 52-58. DOI:10.1111/fml.2006.255.issue-1 |
| [16] |
VandeBroek A, Vanderleyden J. The role of bacterial motility, chemotaxis, and attachment in bacteria-plant interactions[J]. Molecular Plant-Microbe Interaction, 1995, 8(6): 800-810. DOI:10.1094/MPMI-8-0800 |
| [17] |
Xin XF, et al. Pseudomonas syringae pv. tomato DC3000: A model pathogen for probing disease susceptibility and hormone signaling in plants[J]. Annu Rev Phytopathol, 2013, 51: 473-498. DOI:10.1146/annurev-phyto-082712-102321 |
| [18] |
Ye G, Hong N, Zou LF, et al. Tale-based genetic diversity of chinese isolates of the citrus canker pathogen Xanthomonas citri subsp. citri[J]. Plant Disease, 2013, 97(9): 1187-1194. DOI:10.1094/PDIS-12-12-1201-RE |
| [19] |
Innes RW, Hirose MA, Kuempel PL. Induction of nitrogen-fixing nodules on clover requires only 32 kilobase pairs of DNA from the Rhizobium trifolii symbiosis plasmid[J]. J Bacteriol, 1988, 170(9): 3793-3802. DOI:10.1128/jb.170.9.3793-3802.1988 |
| [20] |
Kovach ME, Phillips RW, Elzer PH, et al. pBBR1MCS: a broad-host-range cloning vector[J]. Gene, 1995, 166(1): 175-176. DOI:10.1016/0378-1119(95)00584-1 |
| [21] |
Miller JH. Experiments in Molecular Genetics[M]. New York: Cold Spring Harbor Laboratory, 1972.
|
| [22] |
薛松, 汪军, 王国芬, 等. 解淀粉芽胞杆菌的GFP标记及定殖能力[J]. 热带作物学报, 2017, 38(3): 500-507. |
| [23] |
王凯, 东保住, 张贵, 等. GFP标记的马铃薯大丽轮枝菌生物学特性研究[J]. 华北农学报, 2016, 31(6): 88-93. DOI:10.7668/hbnxb.2016.06.014 |
| [24] |
Mukaihara T, Tamura N, Murata Y, et al. Genetic screening of Hrp type Ⅲ-related pathogenicity genes controlled by the HrpB transcriptional activator in Ralstonia solanacearum[J]. Molecular Microbiology, 2004, 54(4): 863-875. DOI:10.1111/j.1365-2958.2004.04328.x |





