黄秋葵(Abelmoschus esculentus)为锦葵科秋葵属植物,是一种众所周知的天然的、健康的蔬菜品种,广泛分布于非洲、亚洲、美洲等地区,非洲和亚洲的种植面积超过了99%[1]。黄秋葵荚果中富含不饱和脂肪酸、膳食纤维、活性多糖、维生素等[2],经常食用帮助消化、增强体力、保护肝脏、健胃整肠,而且荚果中含有黄酮、类黄酮、叶黄素、生物碱和微量元素,能增强机体的抗病能力[3-5]。王君耀等[6]研究小鼠经黄秋葵水提取液灌胃15 d,然后进行游泳、耐缺氧等实验,结果显示黄秋葵水可以明显提高小鼠耐缺氧能力及耐寒耐热能力,降低小鼠剧烈运动后血乳酸水平,因而具有抗疲劳作用。由于其果实具有较高的营养价值,并且可以提高运动耐力,欧美等国把它列入21世纪最佳绿色食品名录之中,并被许多国家作为运动员首选蔬菜。黄秋葵被认为是次要的作物,直到现在都很少人关注其遗传改良工作。
转录组是某个物种或者特定细胞类型产生的所有转录本的集合。转录组研究能够从整体水平研究基因功能以及基因结构,揭示特定生物学过程以及疾病发生过程中的分子机理,已广泛应用于基础研究、临床诊断和药物研发等领域。在锦葵科植物中棉花的转录组研究最多,有针对棉花纤维早期发育的[7]、有针对根部水胁迫的[8]、有研究苗期棉花杂种优势的[9]等。而黄秋葵作为一个小物种,研究报道比较少,Roland等[10]对黄秋葵叶片和果实进行了转录组测序,获得了叶片和果实基因表达的基本信息;张少平等[11]对红秋葵叶片进行转录组测序,主要研究花青素的合成。而黄秋葵果实作为主要收获部位,果实的发育和营养成分变化应该是研究的主要方向。因此,针对黄秋葵果实转录组信息不全、次生代谢相关功能基因研究较少的情况,本研究利用RNA-seq技术研究黄秋葵果实发育过程中的转录组,可以全面了解黄秋葵果实基因表达相关信息,以及各代谢途径关键基因的表达情况,并且可以以此为基础开发SSR、SNP等多种分子标记,以期为品种鉴定和遗传分析提供依据。
1 材料与方法 1.1 材料黄秋葵种质GZ167(福建省农业科学院亚热带农业研究所资源编号)种植于福建省农业科学院亚热带农业研究所试验基地,7月下旬采集开花后1 d、5 d、12 d的黄秋葵果实(去除种子,可排除异花授粉造成的影响),每个处理采3株的果实,等量混合后提取RNA,即每个RNA样品含有3株黄秋葵果实的RNA,这样可以排除个体差异,3份样品分别标记为167-1、167-5、167-12。
1.2 方法 1.2.1 黄秋葵总RNA的提取黄秋葵总RNA的提取采用成都福际生物有限公司的多糖多酚植物总RNA提取试剂盒(产品编号:RE-05021),RNA浓度和纯度通过Agilent 2100检测。
1.2.2 文库构建检测合格的黄秋葵总RNA用带有Oligo(dT)的磁珠富集mRNA,向得到的mRNA中加入Fragmentation Buffer使其成为短片段,再以片段后的mRNA为模板,用六碱基随机引物(Random hexamers)合成cDNA第1链,并加入缓冲液、dNTPs、RNase H和DNA polymerase I合成cDNA第2链,经过QiaQuick PCR试剂盒纯化并加EB缓冲液洗脱经末端修复、加碱基A,加测序接头,再经琼脂糖凝胶电泳回收目的片段,并进行PCR扩增,从而完成整个文库制备工作,构建好的文库用Illumina HiSeq2000进行测序。
1.2.3 Unigene的获得与功能注释测序仪产生的原始图像数据经base calling转化为序列数据,称之为raw reads,再经过平台初步的过滤后称之为clean reads,然后使用短reads组装软件Trinity[10]做转录组从头组装。Trinity首先将具有一定长度overlap的reads连成更长的片段,这些通过reads overlap关系得到的不含N的组装片段作为组装出来的Unigene。3个转录组一起组装。
Unigene基本功能注释信息给出Unigene的蛋白功能注释、Pathway注释、COG/KOG功能注释、Gene Ontology(GO)功能注释等。首先,通过blastx将Unigene序列比对到蛋白数据库nr(非冗余蛋白数据库)、SwissProt(蛋白质序列数据库)、KEGG(京都基因与基因组百科全书)和COG/KOG(蛋白质原核/真核同源数据库)(evalue < 0.000 01),得到跟给定Unigene具有最高序列相似性的蛋白(如有并列,取第一条),从而得到该Unigene的蛋白功能注释信息。
1.2.4 转录组中SSR位点分析按照以下配置参数使用软件MISA(http://pgrc.ipk-gatersleben.de/misa/)对转录组的所有Unigene进行搜索,寻找Unigene中的SSR,搜索条件为2个碱基的重复单元,需要至少6个重复才会被认为是SSR,3个碱基的重复单元,需要至少5个重复才会被认为是SSR,4-6个碱基重复至少4次,在此基础上,如果两个SSR序列的距离短于100 bp,就会被合并当作一个SSR标记。
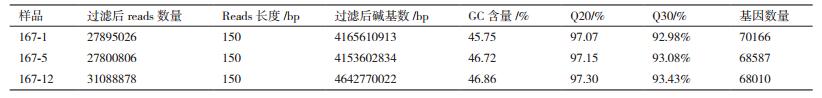
2 结果 2.1 转录组测序与组装分析采用Illumina HiSeqTM 2000高通量测序技术对黄秋葵不同发育时期果实转录组进行测序,从表 1可以看出,167-1、167-5、167-12三份样品转录组测序数据量和质量都较高,为后续的数据拼接组装提供了较好的数据源。
3个黄秋葵果实的转录组测序所获得的reads通过组装和拼接共获得77 476个Unigene,总长度达54 699 979 bp(约54.7 Mb),平均长度为706 bp,N50为1 033 bp,最大拼接长度为13 091 bp,最小为201 bp,表明组装效果良好。
2.2 黄秋葵果实Unigene功能注释通过blastx将Unigene序列比对到蛋白数据库nr、SwissProt、KEGG和COG/KOG,比对结果(图 1-A)显示,77 476个Unigene序列在Nr数据库中有61 559(占79.46%)个找到相似序列,在SwissProt数据库中有42 609(55.00%)个找到相似序列,在KEGG数据库中有34 972(45.14%)个找到相似序列,在COG/KOG数据库中有25 140(32.45%)个找到相似序列,4个数据库一共注释了61 891(79.88%)个Unigene序列,未能够得到注释的Unigene序列有15585(20.12%)个,有18 587(23.99%)个Unigene序列序列被4大数据库同时注释。如图 1-B所示,前3大物种的同源序列数量达到51 502个,占已注释基因总数的83.21%,它们分别是雷蒙德氏棉(Gossypium raimondii,28139个)、亚洲棉(Gossypium arboreum,13 116个)、可可(Theobroma cacao,10 247)。
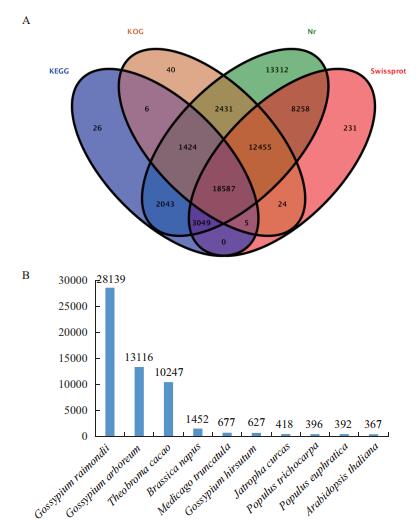
|
| 图 1 四大数据库注释维恩图和前10大物种分布统计图 |
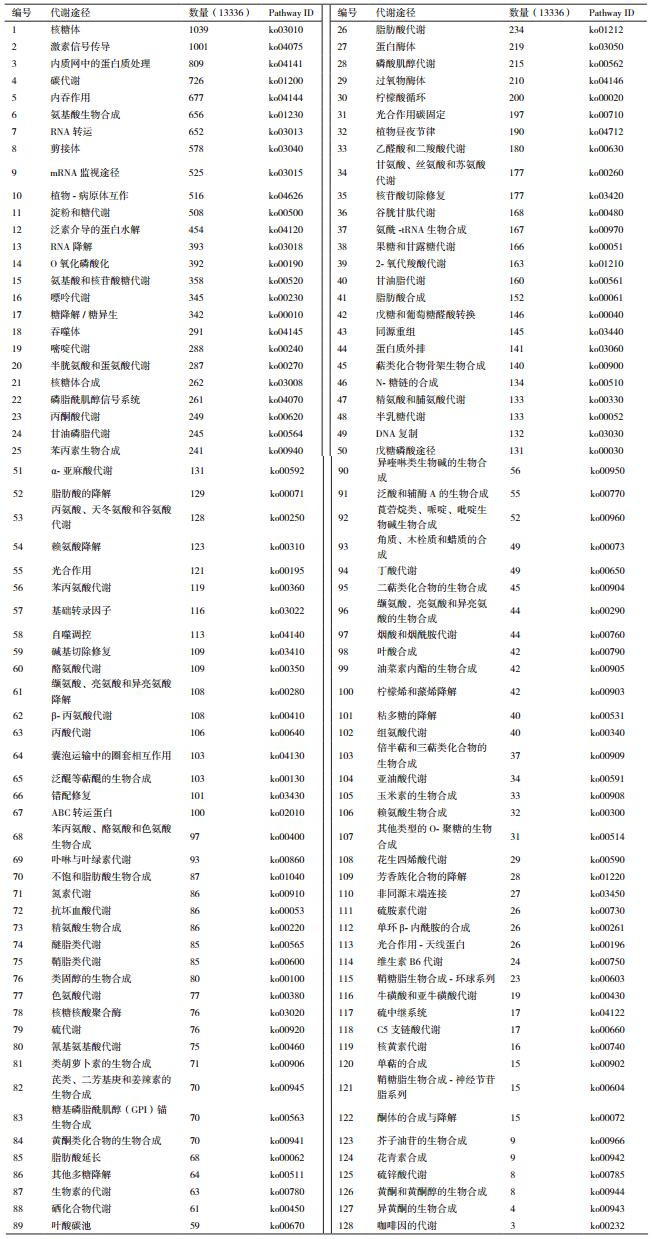
以KEGG代谢途径数据库为依据,可将13 336个黄秋葵果实的Unigene分成128个代谢途径(表 2),比张少平等[11]报道的黄秋葵叶片转录组多了9个代谢途径,体现出叶片与果实转录组的差异。其中涉及Unigene数量最多的前5个代谢途径分别是核糖体代谢、植物激素信号转导、内质网蛋白质代谢、碳代谢和内吞作用。
利用GO数据库,将黄秋葵果实的Unigene进行基因生物学特征功能分类。结果(图 2)显示,GO数据库注释到44 507个Unigene,可分为细胞组分、分子功能及生物学过程等3个本体共48个功能组,其中在参与的生物过程中,细胞进程(24 970)和代谢进程(25 434)含有Unigene最多;在细胞组分本体中,细胞进程(18 890)及其组织部分(18 884)含Unigene最多;在分子功能本体中,结合活性(23 656)和催化活性(23 117)含有的Unigene最多。
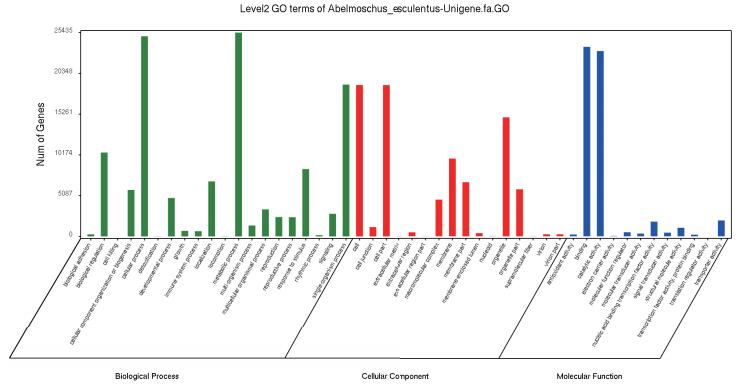
|
| 图 2 GO功能分类图 |
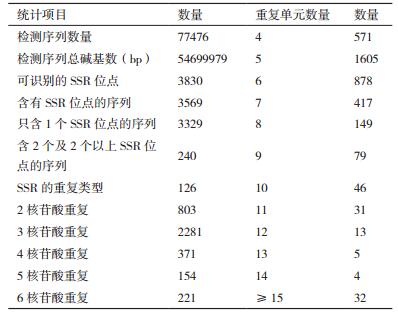
按照以下配置参数使用软件MISA对转录组的所有Unigenes进行搜索,寻找Unigenes中的SSR,在黄秋葵果实转录组77 476条Unigenes序列中发现3 830个SSR位点,分布在3 569条Unigenes中,发生频率(含有SSR的Unigenes数量与总Unigenes数量之比)为4.61%。其中有3 329条Unigenes序列中只含1个SSR位点,含2个及2个以上SSR位点的Unigenes序列有240条,SSR的分布频率(SSR的个数与总Unigenes的数量比)为4.94%,黄秋葵转录组序列中平均14.282 Kb就能发现一个SSR位点(表 3)。黄秋葵转录组中SSR的主要重复类型是三核苷酸重复,占SSR总数的59.56%;其次是二核苷酸重复,占SSR总数的20.97%;四核苷酸重复,占SSR总数的9.69%;五、六核苷酸重复类型的数量较少,总计9.79%(表 3)。SSR重复单元的重复次数分布在4-15次之间,其中5次重复的最多,有1605个SSR,占41.91%;其次为6和4次重复,分别有878、571个SSR,分别占22.92%和14.91%;15次以上重复的仅有32个SSR,占0.84%。从不同串联重复单元类型看(图 3),AAG/CTT重复类型最多,占19.4%,其次为AG/CT,占9.9%。
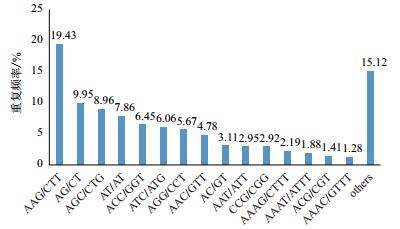
|
| 图 3 SSR不同重复单元 |
随着黄秋葵所含特殊成分及其营养保健功效的发掘,黄秋葵已经在非洲、欧美及东南亚等地进行了广泛种植,相关基础研究也受到极大的关注。前人对黄秋葵的研究主要集中在遗传育种[12-13]、栽培生理[14-15]、营养成分[16]和药用功效[5, 17]等方面,关于分子生物学方面特别是功能基因研究方面的研究较少。王旭等[18]采用同源序列克隆和RT-PCR技术,首次克隆了黄秋葵查尔酮合成酶基因(CHS)cDNA全长序列。张少平等[11]通过Illumina HiSeq 2500高通量测序获得了紫色黄秋葵叶片基因表达的基本信息,使研究者对叶片的代谢途径有了全面的了解。但是,对黄秋葵果实发育过程的代谢途径和转录组分析的研究至今还未见报道。本研究中利用RNA-seq技术分离了大量的多糖代谢、萜类化合物代谢、黄酮和异黄酮类代谢、脂肪酸代谢等次生代谢途径相关基因,为今后黄秋葵功能基因的开发利用等研究奠定了良好基础。
黄秋葵种质资源极其丰富,有些资源可以通过颜色、叶片形状、果形等形态学特征进行辨别,但形态标记数量有限、观测标准容易受到观测者的主观判断影响,因此,还必须结合其他的标记技术,进行更深层次的研究。SSR标记是近年来发展起来的一种以特异引物PCR为基础的分子标记技术,具有数量丰富、覆盖整个基因组、多等位基因等特性,目前该技术已广泛用于遗传图谱的构建、目标基因的标定、指纹图谱的绘制等研究中。Roland等[10]对黄秋葵叶片和果实进行了转录组测序,得到了935个SSR位点,而本研究中得到了3 830个SSR位点,远远超过其发现的个数,主要原因可能是选择的材料差异造成的。Roland等的测序结果得到最多的3类SSR位点是AT/TA(9.3%)、TTC/GAA(8.3%)和TCT/AGA(6.9%),而本研究中获得的最多的3类SSR位点是AAG/TCTT(19.43%)、AG/CT(9.95%)和AGC/CTG(8.96%),后期的工作将对Roland等获得的SSR位点与本研究结果进行对比,找出相同的SSR位点,丰富、可靠的SSR位点将为黄秋葵品种鉴定和资源分析提供有力依据。
4 结论采用RNA-Seq技术对3份黄秋葵果实进行测序分析,3份测序材料组装后共获得了77 476个Unigene序列,有61 891个Unigene在四大数据库中得到注释,占79.88%;以KEGG代谢途径数据库为依据,可将13 336个黄秋葵果实的Unigene分成128个代谢途径,使研究者全面了解了黄秋葵果实的代谢途径信息;在黄秋葵果实转录组中发现3 830个SSR位点,分布在3 569条Unigenes中,发生频率为4.61%,获得的最多的3类SSR位点是AAG/TCTT(19.43%)、AG/CT(9.95%)和AGC/CTG(8.96%)。
| [1] |
FAOSTAT, 2010. http://faostat.fao.org/site/567/default.aspx.
|
| [2] |
Kumar S, Dagnoko S, Haougui A, et al. Okra(Abelmoschus spp.)in West and Central Africa: potential and progress on its improvement[J]. African Journal of Agricultural Research, 2010, 5: 3590-3598. |
| [3] |
Islam M, Hossain N, Alamgir M, et al. Comparative effects of biogas plant residues, poultry manure and inorganic fertilizer on growth and yield of ladies finger[J]. IOSR Journal of Agriculture and Veterinary Science, 2014, 7(1): 2319-2372. |
| [4] |
黄阿根, 陈学好, 高云中, 等. 黄秋葵的成分测定和分析[J]. 食品科学, 2007, 28(10): 451-455. DOI:10.3321/j.issn:1002-6630.2007.10.112 |
| [5] |
Mairuae N, Connor JR, Lee SY, et al. The effects of okra(Abelmoschus esculentus Linn.)on the cellular events associated with Alzheimer's disease in a stably expressed HFE neuroblastoma SH-SY5Y cell line[J]. Neuroscience Letters, 2015, 603(10): 6-11. |
| [6] |
王君耀, 周俊, 汤谷平. 黄秋葵抗疲劳作用的研究[J]. 中国现代应用药学杂志, 2003, 20(4): 316-317. |
| [7] |
李锡花, 吴嫚, 于霁雯, 等. 棉花纤维发育早期RNA-Seq转录组分析[J]. 棉花学报, 2013, 25(3): 189-196. |
| [8] |
Megan JB, Wonkeun P, Phili JB, et al. RNA-seq transcriptome profiling of upland cotton(Gossypium hirsutum L.)root tissue under water-deficit stress[J]. PLoS ONE, 8(12): e82634. DOI:10.1371/journal.pone.0082634 |
| [9] |
丁元昊, 袁道军, 朱龙付, 等. 基于RNA-Seq技术的棉花苗期杂种优势的基因表达分析[C]. 作物杂种优势利用国际学术大会, 2012, 123-124. http://www.wanfangdata.com.cn/details/detail.do?_type=conference&id=7858503
|
| [10] |
Roland S, Sanjeet K, Lin CY, et al. The okra(Abelmoschus esculentus)transcriptome as a source for gene sequence information and molecular markers for diversity analysis[J]. Gene, 2013, 517: 27-36. DOI:10.1016/j.gene.2012.12.098 |
| [11] |
张少平, 邱珊莲, 郑云云, 等. 紫色黄秋葵转录组功能基因测序及分析[J]. 核农学报, 2017, 31(4): 643-653. DOI:10.11869/j.issn.100-8551.2017.04.0643 |
| [12] |
Mehta N, Asati BS, Mamidwar SR. Heterosis and gene action in okra[J]. Bangladesh Journal of Agricultural Research, 2007, 32(3): 421-432. |
| [13] |
Wammanda DT, Kadams AM, Jonah PM. Combining ability analysis and heterosis in a diallel cross of okra(Abelmoschus esculentus(L.)Moench)[J]. African Journal of Agricultural Research, 2010, 5(16): 2108-2115. |
| [14] |
Olayiwola MO, Ariyo OJ. Relative discriminatory ability of GGE Biplot and YSi in the analysis of genotype×environment interaction in okra(Abelmoschus esculentus)[J]. International Journal of Plant Breeding and Genetics, 2013, 7(3): 146-158. DOI:10.3923/ijpbg.2013.146.158 |
| [15] |
Islam M, Hossain N, Alamgir M, et al. Comparative effects of biogas plant residues, Poultry manure and inorganic fertilizer on growth and yield of ladies finger[J]. Iosrjournals Org, 2014, 7: 2319-2372. |
| [16] |
黄阿根, 陈学好, 高云中, 等. 黄秋葵的成分测定和分析[J]. 食品科学, 2007, 28(10): 451-455. DOI:10.3321/j.issn:1002-6630.2007.10.112 |
| [17] |
Monte LG, Santi-Gadelha T, Reis LB, et al. Lectin of Abelmoschus esculentus(okra)promotes selective antitumor effects in human breast cancer cells[J]. Biotechnology Letters, 2014, 36(3): 461-469. DOI:10.1007/s10529-013-1382-4 |
| [18] |
王旭, 韩春乐, 周亚楠, 等. 黄秋葵查尔酮合成酶基因AeCHS的克隆与表达分析[J]. 植物遗传资源学报, 2014, 15(3): 561-567. |