转录因子是植物应对各种逆境的重要调控手段,通过与目标基因启动子区的顺式作用元件结合,调控靶基因的转录水平[1]。NAC转录因子是植物特有的一类转录因子,其名称是矮牵牛(Petunia hybrida)的NAM基因、拟南芥(Arabidopsis thaliana)的ATAF1/2基因和CUC2基因的首字母缩写[2]。NAC转录因子典型的结构模型由两部分组成:存在于N端的高度保守的NAC结构域和存在于C端的高度多样的转录调控区[3]。N端的NAC结构域由150-160个氨基酸残基组成,分为A-E这5个亚结构域。其中,亚结构域C、D高度保守,含有核定位信号,与DNA结合[4]。C端的转录调控区由重复出现的简单氨基酸组成,并富含谷氨酸、脯氨酸、丝氨酸、苏氨酸等,具有激活或抑制转录的功能[5]。
NAC转录因子不仅参与植物生长发育的调控,还在植物应对环境胁迫时发挥了重要的作用[6]。拟南芥ORE1(ANAC092)与叶绿体发育关键基因GLK1/2互作,加速了叶绿体退化,促使叶片衰老[7]。酵母双杂实验证实香蕉(Musa acuminata)MaNAC1/2与乙烯信号组件的MaEIL5蛋白互作,推测MaNAC1/2基因通过乙烯信号通路参与香蕉果实的成熟调控[8]。拟南芥nac019突变体对高温敏感,而过表达该基因增强了植物对高温的抗性[9]。RhNAC3通过ABA依赖型途径参与了月季(Rosa hybrida)对干旱胁迫的响应[10]。小麦(Triticum aestivum)的基因表达谱显示TaNAC2响应干旱、盐害、冷害和ABA处理,在拟南芥中过表达TaNAC2提高了植株对干旱、盐和冷胁迫的耐受力[11]。
青杄(Picea wilsonii Mast.)为松科(Pinaceae)云杉属(Picea)常绿乔木,是我国特有的木本针叶树种,对干旱等恶劣环境具有非常强的适应能力,是进行抗逆基因研究的理想树种。本文从转录组数据中获得PwNAC42基因的cDNA全长序列,并对其进行生物信息学分析,预测理化性质及其蛋白结构等。采用实时荧光定量PCR检测基因在青杄各组织中的表达量,以及在干旱、盐害、冷胁迫和脱落酸(Abscisic Acid,ABA)处理下的表达模式。以期为研究木本植物中NAC转录因子的功能提供理论基础,为林木遗传育种提供分子依据。
1 材料与方法 1.1 材料青杄球果、种子、花粉采集于中国科学院植物研究所和北京植物园,三年生青杄的根、茎和针叶用于组织特异性表达实验。青杄种子在4℃春化后,放置于湿润滤纸上,待其萌发后移植到营养土:蛭石:珍珠岩为2:1:1(V/V/V)的培养基质中培养。温室光周期为16 h日照,温度为21℃,相对湿度55%-65%,生长到8周的青杄幼苗用于逆境响应实验。植物材料采集后用液氮处理,保存于-80℃备用。
1.2 方法 1.2.1 青杄PwNAC42基因编码区的克隆青杄cDNA文库由Invitrogen(上海)公司利用Gateway技术构建[12]。以实验室前期构建的均一化cDNA文库为模板,设计引物NAC42-F和NAC42-R(表 1)扩增PwNAC42的编码区序列,胶回收后连接到pEASY-T1载体上,获得PwNAC42单克隆。
将PwNAC42的cDNA序列导入DNAMAN软件中,通过翻译功能获得其ORF序列及氨基酸序列。ProtParam在线工具(http://web.expasy.org/protparam/)用于预测蛋白分子质量、等电点、分子式及不稳定指数。ProtScale工具(http://web.expasy.org/protscale/)用于分析蛋白亲/疏水性。NetPhos 3.1工具(http://www.cbs.dtu.dk/services/NetPhos/)用于预测蛋白可能存在的磷酸化位点。SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP/)用于预测蛋白是否存在信号肽结构域。TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM/)用于预测蛋白的跨膜区域。FoldUnfold工具(http://bioinfo.protres.ru/ogu/)用于预测蛋白无序化特征。GOR4在线工具(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)用于预测蛋白二级结构,SWISS-MODEL在线工具(https://www.swissmodel.expasy.org/)用于预测蛋白三级结构。
通过NCBI网站(https://www.ncbi.nlm.nih.gov/)的BLAST功能搜索PwNAC42氨基酸序列的同源序列,利用ClustalX软件进行多序列比对分析,并用MEGA5软件选择邻位相连法(Neighbour-joining)构建系统发育树(bootstap检测次数为1 000)。
1.2.3 组织特异性表达利用北京艾德莱(Aidlab)公司的EASYspin Plus植物RNA快速提取试剂盒提取青杄球果、种子、花粉、根、茎和针叶的RNA,通过天根生化科技(北京)有限公司的FastQuant cDNA第一链合成试剂盒反转录成单链cDNA。cDNA模版浓度调成一致之后通过天根公司的荧光定量PCR试剂盒(SYBR)在StepOnePlusTM Real-Time PCR System(ABI公司)上进行RT-qPCR实验,检测PwNAC42在不同组织中的相对表达量。用于荧光定量PCR的引物为NAC42-RT-F和NAC42-RT-R,以青杄EF1-α作为内参基因,序列见表 1[13]。试验设置3次重复。
1.2.4 逆境响应分析逆境响应试验的处理方法参考周燕妮等[14],选取长势相对一致的八周大的青杄幼苗。幼苗置于4℃处理0、3、6、12 h;再置于吸水纸上处理0、3、6、12 h;用200 mmol/L NaCl溶液处理0、3、6、12 h;用100 μmol/L脱落酸处理0、3、6、12 h。对照组为正常生长的植株。试验设置3次重复,处理后的青杄幼苗液氮速冻,于-80℃备用。
2 结果 2.1 青杄PwNAC42全长cDNA的获得通过转录组数据获得PwNAC42的cDNA序列全长,通过DNAMAN软件推导其编码的蛋白序列。PwNAC42基因cDNA序列全长共1 749 bp,其中编码区1 140 bp,共编码379个氨基酸。在91 bp处为起始密码子ATG,1 228 bp处为终止密码子TGA,1 741 bp处为Poly(A)9尾巴(图 1)。

|
| 图 1 PwNAC42全长cDNA的核苷酸序列及推导的氨基酸序列 |
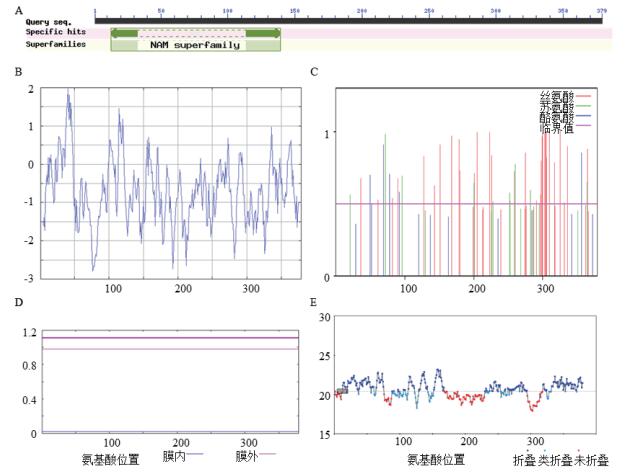
用NCBI网站上的BLAST功能对PwNAC42蛋白进行功能域分析,结果(图 2-A)显示PwNAC42蛋白有典型NAM(No apical meristem)结构域。
|
| 图 2 PwNAC42蛋白结构域及理化性质分析 A:蛋白结构域分析;B:蛋白疏水性分析;C:蛋白磷酸化位点分析;D:蛋白跨膜结构域分析;E:蛋白无序化分析 |
ProtParam预测结果显示,PwNAC42分子质量为42.92 kD,理论等电点为6.53,分子式为C1919H2934N522O582S9,其中丝氨酸(Ser)含量最高,为10.8%,其次为脯氨酸(Pro),含量为9.0%,并带有负电残基(Asp+Glu)48个,带有正电氨基酸残基(Arg+ Lys)46个,预测其半衰期约为30 h,不稳定指数46.78,预测蛋白不稳定。
PwNAC42蛋白疏水性分析结果显示,76位精氨酸(Arg)分值最小,为-2.789,亲水性最强,41位缬氨酸(Val)分值最大,为1.978,亲水性最弱,预测蛋白为亲水性蛋白(图 2-B)。磷酸化位点预测结果显示,PwNAC42有31个丝氨酸磷酸化位点,有11个苏氨酸磷酸化位点,有5个酪氨酸磷酸化位点(图 2-C)。SignalP 4.1预测发现该蛋白没有信号肽结构域(结果未显示)。蛋白跨膜结构域预测结果显示,PwNAC42整条多肽链都位于细胞膜外,不存在跨膜结构域(图 2-D)。蛋白无序化特征预测结果显示,PwNAC42在1-12、76-87、168-229、295-318处的氨基酸处于无序化区域(图 2-E)。
2.2.3 蛋白质二级结构和三级结构PwNAC42蛋白的二级结构预测结果(图 3-A)发现,α-螺旋含量为16.89%,延伸链为12.14%,无β-转角结构,无规则卷曲结构含量达到70.98%。PwNAC42蛋白三级结构预测结果(图 3-B)显示,蛋白无规则卷曲结构较多。

|
| 图 3 PwNAC42蛋白二级结构及三级结构分析 A:PwNAC42蛋白二级结构分析;B:蛋白三级结构分析 |
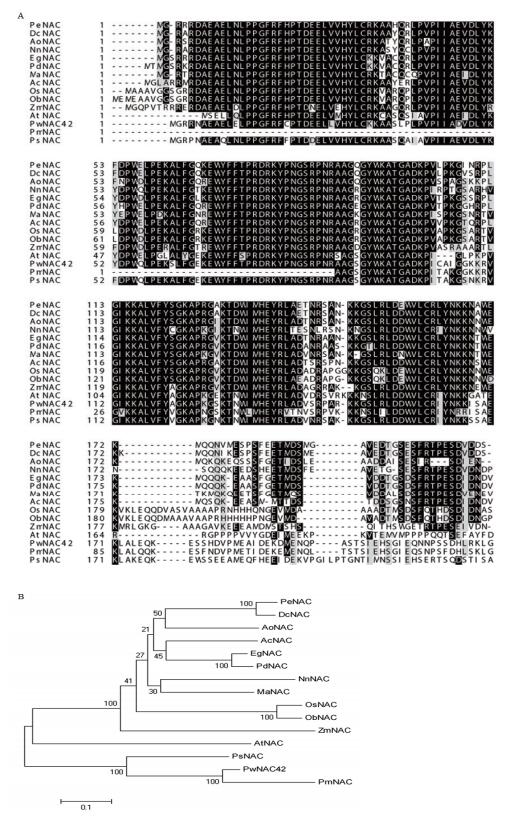
在NCBI网站上用BLAST功能对PwNAC42进行同源蛋白搜索,寻找到下列物种的同源蛋白:黑云杉(Picea mariana,PmNAC,AAC32123.1)、北美云杉(Picea sitchensis,PsNAC,ABK26029.1)、油棕(Elaeis guineensis,EgNAC,XP_010922868.1)、海枣(Phoenix dactylifera,PdNAC,XP_008792531.1)、凤梨(Ananas comosus,AcNAC,OAY70955.1)、石刁柏(Asparagus officinalis,AoNAC,XP_020254522.1)、莲(Nelumbo nucifera,NnNAC,XP_010257746.1)、水稻(Oryza sativa,OsNAC,BAA89798.1)、小果野蕉(Musa acuminata,MaNAC,XP_009394855.1)、小兰屿蝴蝶兰(Phalaenopsis equestris,PeNAC,XP_020578930.1)、铁皮石斛(Dendrobium catenatum,DcNAC,XP_020673301.1)、玉米(Zea mays,ZmNAC,ADK25055.1)、短花药野生稻(Oryza brachyantha,ObNAC,XP_015693359.1)、拟南芥(AtNAC,AT1G01720.1)。利用ClustalX工具进行多序列比对,发现PwNAC42与其它物种的NACs在N端相似性较高,在C端则仅与同为云杉属的黑云杉和北美云杉相似性较高(图 4-A)。用MEGA5邻接法构建系统进化树,结果(图 4-B)显示,PwNAC42与云杉属NACs聚为一类,拟南芥和其它植物则为另两类。
|
| 图 4 PwNAC42蛋白多序列比对(A)及系统进化树分析(B) |
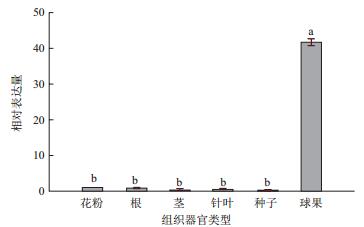
PwNAC42在各个组织器官中均有表达,但主要在球果中表达,球果中的表达量约为花粉中表达量的41倍(图 5)。PwNAC42对NaCl、干旱、4℃、ABA均有响应。200 mmol/L NaCl溶液处理下,PwNAC42表达量随着时间增加显著,在12 h时表达水平达到了对照组的88倍左右(图 6-A)。在干旱条件下,PwNAC42表达量变化显著,6 h前呈现上升的趋势,6 h和12 h的表达量则没有显著差异(图 6-B)。4℃处理下,PwNAC42表达量呈现上升的趋势,在处理12 h时基因的表达水平达到了对照组表达量的18倍(图 6-C)。ABA处理也显著提高了PwNAC42的表达量,表达量随时间持续上升,表达水平在12 h时约为对照组的14倍(图 6-D)。
|
| 图 5 PwNAC42在青杄各组织中的表达量 不同小写字母代表各个组织中PwNAC42表达水平差异显著(P < 0.05) |

|
| 图 6 PwNAC42在逆境处理下表达量的变化 不同小写字母代表各个处理时间之间PwNAC42表达水平差异显著(P < 0.05) |
NAC转录因子最早从矮牵牛中发现,被命名为NAM[15],之后越来越多的NAC转录因子被分离鉴定出来。全基因组分析发现,在拟南芥、水稻、烟草(Nicotiana tabacum)、大豆(Glycine max)、杨树(Populus trichocarpa)和中国白菜(Brassica campestris)等植物中分别有至少117、151、152、152、163和188个NAC转录因子家族成员[16-17]。本试验从青杄中克隆得到PwNAC42基因,通过BLAST工具显示,PwNAC42的N端存在典型的NAM结构域。多序列比对发现,PwNAC42与其它物种的NACs在N端相似性较高,而C端序列仅与云杉属NACs的相似性较高,与其它草本、木本植物相似性较低(图 4)。从而推测PwNAC42的C端序列在进化上较为保守。
前人的研究表明,NAC转录因子参与了植物果实成熟的调控过程。香蕉中的MaNAC1/2通过与乙烯信号组件相互作用,可能参与了香蕉果实的成熟调控[8]。番茄(Solanum lycopersicum)SlNAC3基因在果实的绿熟期和破色期中表达量较高,而干涉该基因表达后,类胡萝卜素的合成受到抑制,果实成熟的进程明显延迟[18]。本研究的组织特异性表达试验发现,青杄PwNAC42主要在球果中表达,推测其可能与球果成熟有关,但具体调控植物果实成熟的途径仍有待研究。
以往的研究发现,NAC转录因子在模式植物拟南芥和小麦、水稻、玉米、番茄等作物类植物响应逆境胁迫中发挥了重要功能[9、11、19-21]。本研究结果显示,盐胁迫、冷胁迫及干旱胁迫处理后PwNAC42表达量随时间变化而明显上升(图 6),推测其可能在植物响应这些逆境的过程中发挥了作用。ABA信号转导途径是植物响应逆境胁迫的重要途径之一,NAC转录因子既依赖于ABA途径发挥作用,也独立于ABA信号途径。Zhu等[21]研究了番茄中的SlNAC4基因,发现该基因的沉默能提高植物对盐和干旱的抗性,但基因的表达受到MeJA(Methyl Jasmonate)而不是ABA的诱导,从而推测SlNAC4可能通过不依赖ABA的途径参与植物对逆境的响应。前人对陆地棉(Gossypium hirsutum)中NACs进行了研究,发现GhNAC7响应乙烯、ABA、干旱、盐分、低温等多种处理,其启动子区存在ABREs(ABA response cis-element)元件,进而推断GhNAC7通过ABA信号通路参与了陆地棉对逆境的响应[22]。在本实验中,外源施加ABA时PwNAC42表达量显著上升,因此推测PwNAC42可能依赖于ABA信号通路响应环境胁迫,但具体的调控机制仍有待研究。
4 结论从青杄cDNA文库中克隆了一个NAC家族的转录因子PwNAC42。组织特异性表达结果表明PwNAC42主要在球果中表达。逆境响应实验结果发现,该基因响应多种非生物胁迫处理包括NaCl、干旱、低温及ABA,说明其参与了青杄对逆境胁迫的响应。
| [1] |
Singh KB, et al. Transcription factors in plant defense and stress responses[J]. Curr Opin Plant Biol, 2002, 5(5): 430-436. DOI:10.1016/S1369-5266(02)00289-3 |
| [2] |
Aida M, Ishida T, Fukaki H, et al. Genes involved in organ separation in Arabidopsis: an analysis of the cup-shaped cotyledon mutant[J]. The Plant Cell, 1997, 9(6): 841-857. DOI:10.1105/tpc.9.6.841 |
| [3] |
Shen H, Yin Y, Chen F, et al. A bioinformatic analysis of NAC genes for plant cell wall development in relation to lignocellulosic bioenergy production[J]. Bioenergy Research, 2009, 2(4): 217-232. DOI:10.1007/s12155-009-9047-9 |
| [4] |
Ooka H, Satoh K, Doi K, et al. Comprehensive analysis of NAC family genes in Oryza sativa and Arabidopsis thaliana[J]. DNA Research, 2003, 10(6): 239-247. DOI:10.1093/dnares/10.6.239 |
| [5] |
Wang Z, Dane F. NAC(NAM/ATAF/CUC)transcription factors in different stresses and their signaling pathway[J]. Acta Physiologiae Plantarum, 2013, 35(5): 1397-1408. DOI:10.1007/s11738-012-1195-4 |
| [6] |
Olsen AN, Ernst HA, Leggio LL, et al. NAC transcription factors: structurally distinct, functionally diverse[J]. Trends Plant Sci, 2005, 10(2): 79-87. DOI:10.1016/j.tplants.2004.12.010 |
| [7] |
Rauf M, Arif M, Dortay H, et al. ORE1 balances leaf senescence against maintenance by antagonizing G2-like-mediated transcription[J]. Embo Reports, 2013, 14(4): 382-388. DOI:10.1038/embor.2013.24 |
| [8] |
Shan W, Kuang J, Chen L, et al. Molecular characterization of banana NAC transcription factors and their interactions with ethylene signalling component EIL during fruit ripening[J]. J Exp Bot, 2012, 63(14): 5171-5187. DOI:10.1093/jxb/ers178 |
| [9] |
Guan Q, Yue X, Zeng H, et al. The protein phosphatase RCF2 and its interacting partner NAC019 are critical for heat stress-responsive gene regulation and thermotolerance in Arabidopsis[J]. Plant Cell, 2014, 26(1): 438-453. DOI:10.1105/tpc.113.118927 |
| [10] |
Jiang G, Jiang X, Lü P, et al. The rose(Rosa hybrida)NAC transcription factor 3 gene, RhNAC3, involved in ABA signaling path-way both in rose and Arabidopsis[J]. PLoS One, 2014, 9(10): e109415. DOI:10.1371/journal.pone.0109415 |
| [11] |
Mao X, Zhang H, et al. TaNAC2, a NAC-type wheat transcription factor conferring enhanced multiple abiotic stress tolerances in Arabidopsis[J]. J Exp Bot, 2012, 63(8): 2933-2946. DOI:10.1093/jxb/err462 |
| [12] |
张盾, 刘亚静, 李长江, 等. 青杄均一化cDNA文库构建及EST序列分析[J]. 生物技术通报, 2012(6): 71-76. |
| [13] |
Yu Y, Li Y, Huang G, et al. PwHAP5, a CCAAT-binding transcription factor, interacts with PwFKBP12 and plays a role in pollen tube growth orientation in Picea wilsonii[J]. J Exp Bot, 2011, 62(14): 4805-4817. DOI:10.1093/jxb/err120 |
| [14] |
周燕妮, 李艳芳, 张通, 等. 青杄PwUSP2基因的克隆和表达分析[J]. 植物生理学报, 2015, 51(8): 1307-1314. |
| [15] |
Souer E, Houwelingen AV, Kloos D, et al. The no apical meristem gene of petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J]. Cell, 1996, 85(2): 159-170. DOI:10.1016/S0092-8674(00)81093-4 |
| [16] |
Puranik S, Sahu PP, Srivastava PS, et al. NAC proteins: regulation and role in stress tolerance[J]. Trends Plant Sci, 2012, 17(6): 369-381. DOI:10.1016/j.tplants.2012.02.004 |
| [17] |
Ma J, Wang F, Li MY, et al. Genome wide analysis of the NAC transcription factor family in Chinese cabbage to elucidate responses to temperature stress[J]. Scientia Horticulturae, 2014, 165(3): 82-90. |
| [18] |
韩芹芹. 番茄SlNAC3基因通过延迟果实软化和改变类胡萝卜素的合成调控果实成熟[D]. 武汉: 华中农业大学, 2011. http://cdmd.cnki.com.cn/Article/CDMD-10504-1011405548.htm
|
| [19] |
Jeong JS, Kim YS, et al. Root-specific expression of OsNAC10 improves drought tolerance and grain yield in rice under field drought conditions[J]. Plant Physiol, 2010, 153(1): 185-197. DOI:10.1104/pp.110.154773 |
| [20] |
Lu M, Ying S, Zhang DF, et al. A maize stress-responsive NAC transcription factor, ZmSNAC1, confers enhanced tolerance to dehydration in transgenic Arabidopsis[J]. Plant Cell Reports, 2012, 31(9): 1701-1711. DOI:10.1007/s00299-012-1284-2 |
| [21] |
Zhu M, Chen G, Zhang J, et al. The abiotic stress-responsive NAC-type transcription factor SlNAC4 regulates salt and drought tolerance and stress-related genes in tomato(Solanum lycopersicum)[J]. Plant Cell Reports, 2014, 33(11): 1851-1863. DOI:10.1007/s00299-014-1662-z |
| [22] |
Shah ST. 陆地棉中NAC家族与叶片衰老和胁迫应答相关功能的遗传学分析[D]. 北京: 中国农业科学院, 2013. http://cdmd.cnki.com.cn/Article/CDMD-82101-1013357022.htm
|





